微卫星DNA,又称为简单重复序列(simple sequencerepeat,SSR),是一种广泛分布于原核和真核生物基因组中的遗传标记,由于其具有共显性遗传、多态性丰富、操作简单和成本低等优点,已在群体遗传学研究中得到大量应用[1, 2]。传统的微卫星标记开发方法主要是从构建的基因组文库中富集、回收、克隆微卫星序列,费时费力,且阳性克隆率较低。因此对于基因组背景信息较少的物种,传统方法在进行微卫星标记的大规模开发方面就受到一定的限制。随着高通量测序技术的成熟及成本的下降,基于转录组数据开发微卫星标记技术的优点正逐渐显露出来。首先,与基因组测序相比,转录组测序费用低、周期短、获得的信息量足够丰富可完全满足标记开发需要。其次,来源于转录组的微卫星标记与功能基因紧密连锁,可为后续基因的定位、性状关联分析等提供有用信息。利用转录组数据库开发微卫星标记的方法已在动植物中被大量报道[3, 4]。
诸氏鲻虾虎鱼(Mugilogobius chulae)是具有我国特色的一种小型海水鱼类,经过近10 年比较研究发现诸氏鲻虾虎鱼在各种生物学性状方面均较具实验动物的开发潜力[5, 6],如个体小、繁殖周期短、繁殖力强、便于实验室内饲养管理以及对污染物生物毒性敏感等。目前实验室封闭群已经繁殖到第14代,近交系已培育至第6 代,诸氏鲻虾虎鱼的实验动物化将会是对国际海洋模式鱼类空白的填补。目前公共数据库中关于诸氏鲻虾虎鱼基因组信息资源极其匮乏,且多为线粒体序列,远不能满足诸氏鲻虾虎鱼遗传质量控制和种质资源保护的需求。为更好地了解诸氏鲻虾虎鱼遗传背景知识,指导诸氏鲻虾虎鱼作为实验动物在封闭群建群过程中的遗传质量控制以及近交系近交过程中遗传纯合度检测,本研究利用Illumina 高通量测序平台对诸氏鲻虾虎鱼肝脏转录组进行测序,并对微卫星标记进行初步筛选和分析,以期为诸氏鲻虾虎鱼功能基因及遗传学研究提供基础数据。
1 材料与方法 1.1 材料转录组测序以及微卫星引物筛选所用的诸氏鲻虾虎鱼样本均取自广东省实验动物监测所海洋生物保种基地,共20 尾,平均体重(0.63±0.15)g,全长范围在2.6-4.1 cm 之间。挑选10 尾诸氏鲻虾虎鱼样本分别取新鲜肝脏组织并混合,液氮冷冻保存,用于总RNA 提取。另取10 尾样本固定于无水乙醇中,-20℃保存,用于DNA 提取。
1.2 方法 1.2.1 转录组测序参照 Trizol 试剂(Invitrogen 公司产品)说明书提取肝脏总RNA,利用华大基因科技服务有限公司Illumina HiSeq2000 高通量测序平台进行RNAseq 转录组测序。用SOAPdenove 软件[7]对转录组数据进行从头组装获得大量Unigenes。
1.2.2 微卫星序列的查找及引物设计使用MISA(MIcroSAtellite identification tool) 软件对组装的Unigenes 进行微卫星序列的查找和筛选,筛选标准为:单碱基重复次数在12 次或12 次以上,两碱基在6 次或者6 次以上,三和四碱基在5 次或者5 次以上,五和六碱基在4 次或者4 次以上。用Primer3.0 批量设计程序设计微卫星引物,设计好的引物送上海生工生物工程有限公司合成。
1.2.3 微卫星引物的筛选微卫星引物筛选所用诸氏鲻虾虎鱼样本共10 尾,按照广州欣研生物科技有限公司生产的组织DNA 提取试剂盒进行样品基因组DNA 的提取,并检测纯度和浓度。PCR 扩增总体系为25 μL,包括Premix TaqTM Version 2.0(TaKaRa)12.5 μL,上下游引物(10 pmol/μL)各1 μL,DNA模板200 ng,无菌去离子水补齐至25 μL。PCR 反应程序为:94℃预变性4 min ;94℃ 40 s,退火(温度依据引物而定)30 s,72℃ 30 s,30 个循环;72℃延伸10 min。PCR 产物在浓度为10% 的非变性聚丙烯酰胺凝胶中分离,硝酸银染色。用NR、Swiss-Port、KEGG 和COG 蛋白质数据库对含有多态性的序列进行基因注释。
2 结果 2.1 诸氏鲻虾虎鱼转录组序列中微卫星的数量和分布特点利用SOAPdenove 软件对转录组数据进行组装,共获得47 979 条Unigenes,总长度为43.7 Mbp,平均长度911 bp,N50 达到1 658 bp。对获得的Unigenes 进行微卫星标记的查找,共得到6 225 个微卫星位点(12.97%),分布在5 868 个Unigenes 上,其中有297 条Unigenes 含有两个及以上微卫星位点(5.1%),诸氏鲻虾虎鱼转录组序列中平均每7.02 kb就出现1 个微卫星位点。最短的微卫星序列为12bp,最长的为393 bp。重复单元类型中1-6 碱基重复类型均有出现,且分布数量不均。
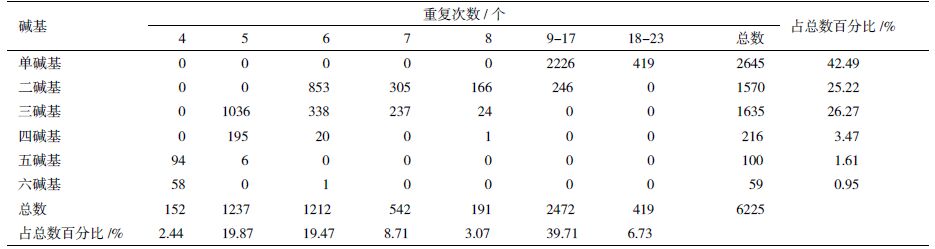
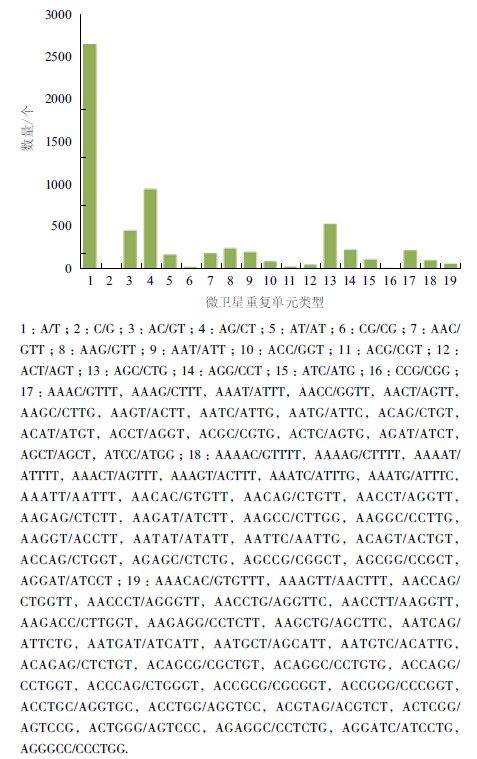
2.2 诸氏鲻虾虎鱼转录组序列中微卫星的重复类型及特征诸氏鲻虾虎鱼转录组序列中微卫星重复基序共226 种,1-6 碱基重复类型数量分别为3、11、59、61、53 和39 个。其中单碱基重复类型最多,有2 645 个,占42.49%,其次为三碱基和二碱基重复,分别为1 635 和1 570 个,占26.27% 和25.22%,四、五和六碱基重复类型数量较少,总共占6.03%。单碱基重复序列中除1 个为C/G 重复单元外,其余全为A/T 重复单元,二碱基重复序列中以AG/CT 和AC/GT 重复单元为主,分别占59.8% 和28.5%,三碱基重复序列中以AGC/TCG、AAG/CTT 和AGG/CCT 为优势类型,分别占32.4%、14.6% 和13.5%,其余重复单元各种重复类型分布较均匀,具体分布见图 1。在重复次数上,单碱基微卫星的核心重复次数主要集中在12-17 次,二、三碱基核心序列重复次数主要集中在5-8 次,四碱基以5 和6 次重复为主,五和六碱基以4 次重复为主,具体数据见表 1。
|
| 图 1 诸氏鲻虾虎鱼转录组中不同微卫星重复单元类型分布 |
利用Primer3 对获得的微卫星序列进行引物设计,共设计引物2 671 对,挑选部分二、三和四单元重复类型微卫星引物共76 对送出合成,其中二碱基重复次数≥ 9 次,三碱基重复次数≥ 7 次,四碱基重复次数≥ 6 次。以10 个诸氏鲻虾虎鱼野生群样本基因组DNA 为模板,对76 对引物进行了PCR 扩增。可稳定扩增出目的条带的有55 对,其中有32对具有多态性,23 对为单态,部分扩增结果见图 2。初步筛选获得的具有多态性的32 个微卫星位点及引物序列,见表 2,相关序列已上传至美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)。

|
| 图 2 引物2153-1、2368-4 和2489-2 在10 尾诸氏鲻虾虎鱼野生群体中的扩增结果 |
利用NR、Swiss-Port、KEGG 和COG 蛋白质数据库对32 对多态性微卫星序列进行基因功能注释,共有20 个微卫星位点获得注释,涉及到20 个已知的功能基因,包括到转录因子、受体、激酶和一些细胞组成成分等重要功能基因(表 2)。
3 讨论本研究在47 979 条,总长度为43.7 Mbp 的Unignges 中共发现微卫星位点6 225 个,平均每7.02kb 就出现1 个微卫星位点,发生频率为12.97%。与其他海洋鱼类相比,诸氏鲻虾虎鱼转录组中大于二碱基重复微卫星标记的出现频率介于中间状态(7.46%),比斑点叉尾鮰(Ictalurus punctatus)(11.2%)[8]和红鳍东方鲀(Fugu rubripes)(11.5%)[9]低,但又高于真鲷(Chrysophrys major)(4%)[10]和大黄鱼(Pseudosciaena crocea)(4.24%)[11],与牙鲆(Paralichthys olivaceus)(7.95%)[12] 相似。在出现密度上,与其他鱼类的差异较小,如鮸鱼(Miichthysmiiuy) 为1/6.04 kb[13]、鲫鱼(Carassius auratus)为1/6.39 kb[14],牙鲆(Paralichthys olivaceus) 为1/7.9 kb[12]。这种出现频率和密度上的差异可能与微卫星查找所选用数据量的大小、数据拼装的质量、组织来源以及物种间差异有关。
一般认为微卫星标记的多态性与重复单元的重复次数呈正相关[15]。对本研究获得的微卫星序列统计发现,除单碱基重复微卫星外,其余微卫星重复次数范围在5-8 次之间,比来源于基因组的微卫星重复次数低。推测可能是由转录组微卫星主要来自编码区导致,与基因组中非编码序列相比,编码区序列受到选择压力更大,相对不易发生变异[16]。
在微卫星重复类型上,除单碱基微卫星重复类型外,多数物种中是以二碱基重复类型为主[17],本研究发现诸氏鲻虾虎鱼转录组序列微卫星重复类型中以三碱基重复类型为最多,结果与之存在差异。这种现象在大黄鱼、鮸鱼和缢蛏(Sinonovacula constricta)中也见报道[11, 13, 18]。由于转录组序列主要来源于外显子,自然选择机制对转录区的三核苷酸基序表现出积极选择作用,导致在编码区由于受重大突变压力的影响而存在丰富的三核苷酸重复序列[19, 20]。在重复类型分布上,单碱基、二碱基和三碱基均表现出一定的偏倚性,单碱基重复类型中除发现1 个C/G 类型外,其余均为A/T 类型,在二碱基重复序列中,AG/CT 占主要优势,三碱基重复类型中最多的是AGC/TCG,这种二、三重复单元的重复类别在不同物种间差异较大[13, 14, 21],这种重复单元数量的偏倚以及类型的差异性可能与物种间差异性有关[22]。另外,在二碱基重复类型中还检测到18 个比较罕见的GC/GC 类型。
考虑到标记多态性问题,在进行微卫星引物筛选时,为避免盲目,尽量选取重复单元重复次数较高的序列进行筛选。本研究挑选部分二碱基重复次数≥ 9 次、三碱基重复次数≥ 7 次、四碱基重复次数≥ 6 次的序列,共设计76 对微卫星引物,以10个诸氏鲻虾虎鱼野生保种群体样本基因组DNA 为模板,对76 对引物进行了初步筛选。其中能稳定扩增出目的片段的引物有55 对,剩余21 对未扩增或者扩增非目的片段。55 对有效扩增引物中有32 对具有多态性,多态位点比例达到42.67%。分布上仅见于二和三碱基重复类型,四碱基重复类型序列没有发现多态位点,可能是选用的位点数量较少导致。通过对获得的32 个多态性微卫星位点进行蛋白库比对,共有20 个位点获得注释,这种位于外显子区域的微卫星位点更适合用于功能基因的定位和表达调控分析。初步开发的32 个具有多态性的微卫星位点,为诸氏鲻虾虎鱼种群遗传结构的分析、封闭群和近交系群体的遗传质量监测奠定了基础。
4 结论本研究利用高通量测序技术首次对诸氏鲻虾虎鱼肝脏转录组进行了测序,共获得了47 979 条Unigenes。在47 979 条Unigenes 中共查找到6 225个微卫星位点,主要以单碱基、二碱基和三碱基重复类型为主。挑选部分二、三和四单元重复类型微卫星序列,共设计76 对引物,可稳定扩增出目的条带的有55 对,其中32 对具有多态性。
| [1] | Dor L, Shirak A, Gorshkov S, et al. Construction of a microsatellitesbasedlinkage map for the white grouper(Epinephelus aeneus)[J]. Genomes, 2014, 45(5): 699-708. |
| [2] | Grattapaglia D, Amaral D, Santos G. Performance of microsatellitesfor parentage assignment following mass controlled pollination in aclonal seed orchard of loblolly pine(Pinus taeda L.)[J]. TreeGenetics & Genomes, 2014, 10(6): 1631-1643. |
| [3] | Norrell AE, Crawley D, Jones KL, et al. Development andcharacterization of eighty-four microsatellite markers for the redsnapper(Lutjanus campechanus)using Illumina paired-endsequencing[J]. Aquaculture, 2014, 430 : 128-132. |
| [4] | Vukosavljev M, Esselink GD, Westende WPC, et al. Efficientdevelopment of highly polymorphic microsatellite markers basedon polymorphic repeats in transcriptome sequences of multipleindividuals[J]. Molecular Ecology Resources, 2014, 15(1).DOI : 10.1111/1755-0998.1289. |
| [5] | 李建军, 陈小曲, 林忠婷, 等. 诸氏鲻虾虎鱼的形态与生长特性分析[J]. 实验动物与比较医学, 2012, 32(4): 334-340. |
| [6] | 李建军, 吴美慧, 叶慧欣, 等. 不同发育期诸氏鲻虾虎鱼对钻井液的敏感性比较[J]. 中国比较医学杂, 2013, 23 : 48-51. |
| [7] | Luo RB, Liu BH, Xie YL, et al. SOAPdenovo2 : an empiricallyimproved memory-efficient short-read de novo assembler[J].GigaScience, 2012, 1(18): 1-6. |
| [8] | Serapion J, Kucuktas H, Feng JN, et al. Bioinformation miningof type I microsatellites from expressed sequence tags of channelcatfish(Ictalurus punctatus)[J]. Marine Biotechnology, 2004, 6(4): 364-377. |
| [9] | Edwards YJ, Elgar G, Clark MS, et al. The identicication andcharacteriction of microsatellites in the compact genome of theJapanese puffer fish, Fugu rubripes : Perspectives in functional andcomparative genomic analysis[J]. Journal of Molecular Biology,1998, 278 : 843-854. |
| [10] | Chen SL, Liu YG, Xu MY, et al. Isolation and characterization ofpolymorphic microsatellite loci from an EST library of red seabream(Chrysophrys major)and cross-species amplification[J].Molecular Ecology Notes, 2005, 5 : 215-217. |
| [11] | 谢芳静, 张子平, 邹志华, 等. 大黄鱼EST 微卫星标记初步筛选[J]. 福建水产, 2011, 33(5): 9-14. |
| [12] | 陈松波, 龚丽, 刘海金. 牙鲆EST 资源的SSR 信息分析[J].东北农业大学学报, 2010, 41(10): 82-86. |
| [13] | 孙典巧, 孙悦娜, 王日昕, 等. 鮸鱼EST 序列中微卫星标记的初步筛选及特性分析[J]. 水生生物学报, 2011, 35(5): 753-760. |
| [14] | 杨曦. 鲫鱼(Carassius auratus)表达序列标签资源的SSR 构成与分布分析[J]. 生物技术通报, 2012(11): 139-143. |
| [15] | Weber JL. Informativeness of human(dC-dA)n·(dG-dT)npolymorphisms[J]. Genomics, 1990, 7(4): 524-530. |
| [16] | 懂迎辉, 吴国星, 姚韩韩, 等. 泥蚶34 个EST-SSR 标记的开发及在格粗饰蚶的通用性检测[J]. 水产学报, 2013, 37(1): 70-77. |
| [17] | 郭文久. 微卫星在基因组上的分布与功能及其计算方法初步研究[M]. 西安: 陕西科学技术出版社, 2009 : 5-6. |
| [18] | 刘博, 邵艳卿, 滕爽爽, 等. 缢蛏(Sinonovacula constricta)EST-SSR 分布特征及引物开发利用[J]. 海洋与湖沼, 2012,43(1): 133-137. |
| [19] | Blanca J, Cañizares J, Roig C, et al. Transcriptome characterizationand high throughput SSRs and SNPs discovery in Cucurbita pepo(Cucurbitaceae)[J]. BMC Genomics, 2011, 12 : 1-15. |
| [20] | Garg R, Patel RK, Tyagi AK, et al. De novo assembly of chickpeatranscriptome using short reads for gene discovery and markeridentification[J]. DNA Research, 2011, 18(1): 53-63. |
| [21] | Kantety RV, Rota ML, Matthews DE, et al. Data mining for simplesequence repeats in expressed sequence tags from barley, maize,rice, sorghum and wheat[J]. Plant Mol Biol, 2002, 48 : 501-510. |
| [22] | 许晓军, 张海琪, 张超, 等. 中华鳖表达序列标签源中的微卫星信息分析[J]. 经济动物学报, 2013, 17(1): 15-18. |