2. 广东省水产经济动物病原生物学及流行病学重点实验室,湛江524088 ;
3. 广东省教育厅水产 经济动物病害控制重点实验室,湛江 524088 ;
4. 仲恺农业工程学院,广州 510225
2. Guangdong Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals,Zhanjiang 524088 ;
3. Key Laboratory of Diseases Controlling for Aquatic Economic Animals of Guangdong Higher Education Institutions,Zhanjiang 524088 ;
4. Zhongkai University of Agriculture and Engineering,Guangzhou 510225
1989 年,IL-10 首次在鼠类T 细胞中作为细胞因子合成抑制因子被报道[1]。除T 细胞外,B 细胞、嗜酸性粒细胞、单核巨噬细胞、NK 细胞等,几乎所有淋巴细胞均能合成IL-10[2]。IL-10 主要调控炎症反应,它不仅能通过抑制单核巨噬细胞释放免疫介质、抗原呈递和细胞吞噬作用,抑制CD4+ T 细胞的增殖和细胞因子合成,抑制呼吸爆发等,从而抑制免疫作用;也能通过刺激NK 细胞、CD8+ T 细胞的细胞毒活性增强免疫作用[3, 4, 5, 6]。在哺乳动物研究中表明,IL-10 是以二聚体形式行使其生物学功能,通过与IL-10R1/R2 受体配体复合物结合,使JAKSTAT信号通路上JAK1 和Tyk2 的磷酸化,从而招募和激活转录因子STAT3 等,选择性激活或抑制基因转录,发挥其抗炎生物学功能[7, 8, 9, 10]。除JAK-STAT信号通路,IL-10 还通过其他信号通路介导免疫反应。
目前为止,IL-10 已在河豚、鲤、斑马鱼、黑鲈、鳕鱼、金鱼、虹鳟等硬骨鱼中被发现[11, 12, 13, 14, 15, 16, 17],其中虹鳟中发现两个同源性92% 的IL-10 剪切体。但对于斜带石斑鱼IL-10 相关的研究还未见相关报道。本实验对斜带石斑鱼IL-10 基因进行克隆及其生物信息学分析,以期为进一步研究该基因的结构特点和免疫调节作用奠定基础;同时构建IL-10 原核表达载体,表达IL-10 融合蛋白,旨在为进一步研究斜带石斑鱼IL-10 蛋白生物学功能提供参考。
1 材料与方法 1.1 材料 1.1.1 实验用鱼实验用斜带石斑鱼采集自湛江某水产品市场。
1.1.2 载体与菌株克隆载体pMD18-T 质粒和表达载体 pET-32a(+)质粒均购自TaKaRa 公司,大肠杆菌 DH5α 和BL21 菌株来自本实验室保存的菌株。
1.1.3 主要试剂UNIQ-10 柱式Trizol 总RNA 提取试剂盒购自上海生工生物技术有限公司;质粒提取试剂盒及DNA 胶回收试剂盒购自Thermo 公司;PrimeSTAR HS DNA 聚合酶、Ex Taq DNA 聚合酶、限制性核酸内切酶Xho I 和BamH I、T4 DNA 连接酶、末端转移酶TdT、dNTPs、DNA Marker、蛋白Marker等购自TaKaRa 公司;M-MLV 反转录酶购自NEB 公司;用PCR 的引物及序列测定均由上海生工生物技术有限公司完成。
1.2 方法 1.2.1 斜带石斑鱼总RNA 的提取及cDNA 第一链 合成斜带石斑鱼脾脏总RNA 提取按照UNIQ-10柱式Trizol 总RNA 提取试剂盒说明书进行;按照M-MLV 反转录酶说明书以3adapter 为引物合成第一链cDNA,进行同源克隆及3'-RACE ;以5-RECE-D1为引物合成cDNA,按照DNA 胶回收试剂盒说明书对cDNA 进行纯化,纯化后的cDNA 按照末端转移酶TdT 说明书对其加上ployA 尾,产物用于进行5'-RACE。
1.2.2 斜带石斑鱼IL-10 中间片段获得根据GenBank 上已登录的舌齿鲈Dicentrarchus labrax(DQ821114.1)和鲫Carassius auratus(HQ259106.1)设计简并引物IL-10-F1 和IL-10-D1,同源克隆获得IL-10 的基因片段。
1.2.3 斜带石斑鱼IL-10 基因3'-RACE根据获得的中间片段设计3'-RACE 特异引物(3-RACE-F1 ;3-RACE-F2)。3'-RACE 反应体系:第一轮PCR :cDNA 2.5 μL,3-RACE-F1(10 μmol/L)1 μL,long(0.4 μmol/L)/short(2 μmol/L)2 μL,PrimeSTARHS DNA 聚合酶0.25 μL,5×PrimeSTAR Buffer 5 μL,dNTP mix(2.5 mmol/L)2.5 μL,加水补足25 μL。PCR 反应条件:94℃ 30 s,66℃ 45 s,72℃ 2 min,5 个循环;94℃ 30 s,60℃ 45 s,72℃ 2 min,25 个循环,共计30 个循环。第二轮PCR :首轮PCR 产物稀释20 倍1 μL,3anchor 1 μL,3-RACE-F2(10μmol/L)1 μL,Ex Taq DNA 聚合酶0.25 μL,10×ExTaq Buffer 2.5 μL,dNTP mix(2.5 mmol/L)2.5 μL,加水补足25 μL。反应条件同第一轮PCR。
1.2.4 斜带石斑鱼IL-10 基因5'-RACE根据获得的中间片段设计5'-RACE 特异引物(5-RECE-D1 ;5-RACE-D2 ;5-RACE-D3)。5'-RACE 反应体系:第一轮PCR :加尾产物 2.5 μL,5-RACE-D2(10μmol/L)1 μL,5oligodT-anchor 1 μL,Ex Taq DNA 聚合酶0.25 μL,10×Ex Taq Buffer 2.5 μL,dNTP mix(2.5mmol/L)2.5 μL,加水补足25 μL。PCR 反应条件:94℃ 30 s,61℃ 45 s,72℃ 1 min,5 个循环;94℃30 s,58℃ 45 s,72℃ 1 min,5 个循环;94℃ 30 s,56℃ 45 s,72℃ 1 min,18 个循环,共计28 个循环。第二轮PCR :首轮PCR 产物稀释25 倍 1 μL,5anchor 1 μL,5-RACE-D3(10 μmol/L)1 μL,Ex TaqDNA 聚合酶0.25 μL,10×Ex Taq Buffer 2.5 μL,dNTPmix(2.5 mmol/L)2.5 μL,加水补足25 μL。反应条件同第一轮PCR。本实验所用引物序列,见表 1。
利用NCBI 在线软件ORF finder(http ://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)确定开放阅读框(ORF)及蛋白氨基酸序列;运用ExPASy ProtParam(http ://web.expasy.org/protparam/)预测蛋白分子量(Mw)和理论等电点(pI);采用SignalP 4.1 Server(http ://www.cbs.dtu.dk/services/SignalP/) 预测对信号肽进行预测;通过TMHMM Server v.2.0(http ://www.cbs.dtu.dk/services/TMHMM/)分析是否存在跨膜结构域;功能位点分析使用ExPASy PROSITE 在线软件(http://prosite.expasy.org/);蛋白结构预测采用PredictProtein(https ://www.predictprotein.org/)、Phyre 2 server(http ://www.sbg.bio.ic.ac.uk/phyre2/ html/page.cgi?id=index) 和SWISS Model(http ://swissmodel.expasy.org/);用DNAMAN6.0 软件对斜带石斑鱼IL-10氨基酸序列与其他物种IL-10 进行同源比对分析及序列拼接;利用MEGA6 软件进行系统进化树的构建。
1.2.6 斜带石斑鱼IL-10 原核表达载体pET-32a-IL10构建及其原核表达条件的优化根据已得到的IL-10基因cDNA 全长设计含酶切位点的引物扩增IL-10 去信号肽核心片段,引物为IL10F(BamH I)及IL10R(Xho I)。将用Ex Taq 酶PCR 扩增获得的含酶切位点的基因片段连接到pMD18-T 载体,对测序正确的阳性克隆和pET-32a(+)载体进行用BamH I 和XhoI 双酶切,1.5% 琼脂糖电泳后回收相应的目的条带,用T4 DNA 连接酶进行连接后转化至大肠杆菌BL21感受态细胞中,对阳性克隆进行测序。用终浓度为0.1 mmol/L IPTG 诱导4 h 进行原核表达。融合蛋白表达的优化按黄瑜等[18]的方法进行。
2 结果 2.1 斜带石斑鱼IL-10基因克隆结果(图 1)显示,获得全长1 104 bp 的IL-10基因,ORF 为564 bp,编码187 个氨基酸。GenBank登录号为KJ741852。蛋白理论分子量(Mw)为21.7kD,理论等电点(pI)为5.74,存在一个22 个氨基酸的信号肽,无跨膜区。ExPASy 预测斜带石斑鱼IL-10 蛋白存在4 个蛋白激酶C 磷酸化位点(24、73-75、135-137、177-179),3 个酪蛋白激酶II 磷酸化位点(69-72、85-88、150-153),1 个N 端酰基化位点(146-149)及1 个N 端糖基化位点(156-161)。

|
| 图 1 IL-10 cDNA 核苷酸及其编码的氨基酸序列 加粗为起始密码子ATG 和终止密码子TGA ;方框为信号肽;下划线为mRNA 不稳定信号基序 |
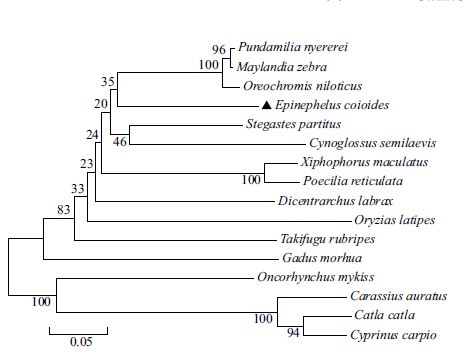
多序列比对结果(图 2)显示,斜带石斑鱼IL-10 的氨基酸序列与吉富罗非鱼的同源性较高,为72.25%,而与人和猴的仅有26.18% 和25.13%。在IL-10 多肽链的30、80、110 和116 位存在4 个非常保守的半胱氨酸。运用MEGA6 软件,以N-J 法构建IL-10 的系统进化树,结果(图 3)显示,斜带石斑鱼IL-10 与维多利亚湖慈鲷(P. nyererei)、斑马宫丽鱼(M. zebra)和吉富罗非鱼(O. niloticus)聚为一支。

|
| 图 2 IL-10 氨基酸序列的同源性比对 Fig. 2 Maylandia zebra:宫丽斑马鱼(XP_004545126.1);Epinephelus coioides:斜带石斑鱼(KJ741852);Oreochromis niloticus:尼罗罗非鱼(XP_003441414.1);Cynoglossus semilaevis :半滑舌鳎(XP_008318394.1);Oncorhynchus mykiss :虹鳟(NP_001232028.1);Homo sapiens :智人(BC104253.1);Gallus gallus :原鸡(AJ621254.1);Rattus norvegicus :褐家鼠(NM_012854.2);浅灰色背景为4 个构成二硫键的半胱氨酸,方框为IL-10 特征序列 |
|
| 图 3 IL-10 氨基酸的系统进化树 Fig. 3 Pundamilia nyererei :维多利亚湖慈鲷(XP_005739520.1);Maylandia zebra :宫丽斑马鱼(XP_004545126.1);Oreochromis niloticus :尼罗罗非鱼(XP_00-3441414.1);Epinephelus coioides :斜带石斑鱼(KJ741852);Stegastes partitus :深裂眶锯雀鲷(XP_008294254.1);Cynoglossus semilaevis :半滑舌鳎(XP_008318394.1);Xiphophorus maculatus :花斑剑尾鱼(XP_005804372.1);Poecilia reticulata :网纹鳉(XP_008407467);Dicentrarchus labrax :舌齿鲈(ABH09454.1);Oryzias latipes :青鳉(XP_004069312.1);Takifugu rubripes :红鳍东方鲀(XP_003973743.1);Gadus morhua :大西洋鳕鱼(ABV64720.1);Oncorhynchus mykiss : 虹鳟(NP_001232028.1);Carassius auratus : 金鱼(ADU34193.1) |
软件分析显示IL-10 蛋白二级结构中有117 处α-螺旋,占总二级结构的62.57% ;42 处无规则卷曲,占总二级结构的22.46% ;14 处β-转角和14 处共有延伸链,各占总二级结构的7.49%。PredictProtein和SWISS MODEL 的结果(图 4)显示,斜带石斑鱼IL-10 蛋白存在6 个α 螺旋,三维结构以二聚体形式存在,其空间结构与人IL-10 相似。

|
| 图 4 预测斜带石斑鱼IL-10(A)与人的IL-10(B)的空 间结构结构比较 |
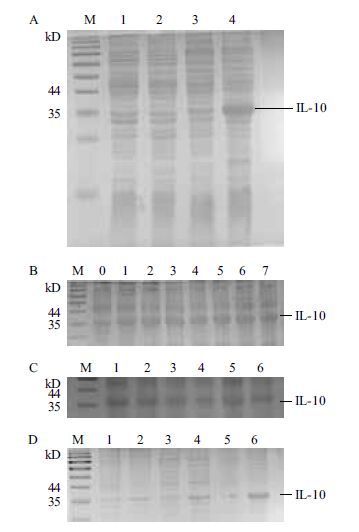
对测序正确的阳性菌与pET-32a(+)质粒的空载体菌分别在0.1 mmol/L IPTG 的浓度下诱导4 h 进行预诱导,结果(图 5-A)显示分子量为37.5 kD 的融合蛋白在大肠杆菌BL21 中表达。在IPTG 为0.1mmol/L 浓度时,融合蛋白在3 h 后表达量不再增加(图 5-B);在IPTG 浓度为0.02、0.06、0.1、0.4、0.6及1 mmol/L,诱导4 h 后,蛋白在0.02 mmol/L 和0.6mmol/L 时表达量最大(图 5-C);在IPTG 浓度为0.1mmol/L 时,温度为16℃、28℃和37℃,诱导4 h 后,37℃时蛋白在沉淀中表达量最大,而28℃时,上清中融合蛋白较多(图 5-D)。
|
| 图 5 IL-10 蛋白的原核表达 M :蛋白质Marker ;A :1 :pET-32a(+)未诱导;2 :pET-32a(+)诱导;3 :pET-32a-IL10 未诱导;4 :pET-32a-IL10 诱导。B :不同时间:0-7 :分别为0、1、2、3、4、5、6 和7 h。C:不同IPIG 浓度:1-6:分别为0.02、0.06、0.1、0.4、0.6 和1.0 mmol/L。D:不同温度:1,3,5:16℃、28℃、37℃诱导上清;2,4,6 :16℃、28℃、37℃诱导沉淀 |
本研究成功克隆了全长为1 104 bp 的斜带石斑鱼IL-10 基因,ORF 为564 bp,编码187 个氨基酸,在3' 端非编码区存在2 个mRNA 不稳定基序(ATTTA),表明该基因可能在炎症反应和免疫应答中较为活跃[19]。在斜带石斑鱼IL-10 氨基酸序列中存在LLDESVEESFKSPFGCHAMNSILEFYLNTVL 和GLFKAMGDLDVLFNYIE 两个属于IL-10 特征基序L-[FILMV]-X3-[ILV]-X3-[FILMV]-X5-C-X5-[ILMV]和G-X2-KA-X2-[ED]-X-D-[ILV]-[FLY]-[FILMV]-X2- [ILMV]- [EKQZ]的序列。4 个非常保守的半胱氨酸(Cys-30、Cys-80、Cys-110 和Cys-116)在斜带石斑鱼IL-10 多肽链中被发现,在人和哺乳动物IL-10研究表明,IL-10 存在对蛋白空间结构的形成非常重要的两个二硫键,对蛋白行使生物学功能不可或缺[8, 9]。不同于人和哺乳动物IL-10,斜带石斑鱼IL-10 成熟肽还存在一个额外的半胱氨酸(Cys-26),在其他已报道的鱼类IL-10,如斑马鱼[13]、鲤[12]、虹鳟[17]等,也存在类似的现象,IL-10 通常以二聚体行使生物学功能,这一可能的二硫键形成位点,是否对IL-10 的蛋白具有生物学意义还有待研究。哺乳动物IL-10 通过磷酸化行使其一些生物学功能[20],在鼠IL-10 发现一个潜在的糖基化位点,但不是蛋白功能所必需的[21]。斜带石斑鱼IL-10 蛋白分析显示其存在4 个蛋白激酶C 磷酸化位点、3 个酪蛋白激酶II 磷酸化位点以及1 个N 端酰基化位点和1 个N 端糖基化位点,这些修饰位点对蛋白功能是否有影响可以通过蛋白点突变来验证。
氨基酸同源性比对显示斜带石斑鱼IL-10 的氨基酸序列与吉富罗非鱼的同源性最高,为72.25%,而与人类和猴的同源性较低。斜带石斑鱼IL-0 蛋白结构预测显示斜带石斑鱼IL-10 含有6 个α 螺旋,以二聚体形式存在于生物体中,与对人和哺乳动物的研究一致。其三级结构与人IL-10 空间结构类似,蛋白的结构与蛋白功能之间的关系非常密切,斜带石斑鱼IL-10 可能和哺乳动物功能相同,以二聚体形式与其受体相结合,通过Jak-Sata 信号通路来实现其对细胞的免疫调控[7, 10, 20]。Grayfer 等[16, 22]基于对金鱼和斑马鱼IL-10 及其受体IL10R 特征和功能的研究,也认为IL-10 的功能在进化上可能高度保守。
本实验成功表达出分子量为37.5 kD 的斜带石斑鱼IL-10 融合蛋白。选择pET-32a(+)质粒构建原核表达载体是因为pET 系列拥有T7 强启动子的载体,能短时间内大量表达目的蛋白,而表达的融合蛋白带有6 个组氨酸标签有利于蛋白后续的分离与纯化,且多数情况下表达的融合蛋白不影响蛋白的免疫原性和免疫反应性。原核表达条件优化显示,在IPTG 浓度为0.02 mmol/L,37℃诱导3 h,蛋白包涵体的表达量最大。这些结果为后续进行IL-10蛋白的富集及抗体的制备奠定基础。而斜带石斑鱼IL-10 融合蛋白在低温诱导时,上清也有明显表达,可以省去包涵体蛋白复性等相关复杂操作,获取活性蛋白。本研究的后续实验将进行IL-10 的分离纯化,参照Grayfera 等[16]的实验操作,研究其对细胞呼吸爆发的影响,以及IL-10 对细胞内一些免疫相关基因表达的调控,探讨IL-10 在石斑鱼免疫反应中的作用与哺乳动物是否类似。
4 结论本研究从斜带石斑鱼成功克隆了IL-10 基因。该基因全长1 104 bp,编码187 个氨基酸,理论分子量为21.7 kD,理论等电点为5.74,同源分析显示与吉富罗非鱼亲缘关系较近。并对斜带石斑鱼IL-10的重组表达和表达条件进行了探索,其优化条件为:0.02 mmol/L 的IPTG,37℃诱导3 h。
| [1] | Fiorentino DF, Bond MW, Mosmann TR. Two types of mouse Telper cell. IV. Th2 clones secrete a factor that inhibits cytokineroduction by Th1 clones[J]. Exp Med, 1989, 170(6): 2081-095. |
| [2] | Saraia M, O' Garra A. The regulation of IL-10 production by immuneells[J]. Nat Rev Immunol, 2010, 10(3): 170-181. |
| [3] | Bogdan C, Vodovotz Y, Nathan C. Macrophage deactivation bynterleukin10[J]. Exp Med, 1991, 174(6): 1549-1555. |
| [4] | Oswald IP, Wynn TA, Sher A, et al. Interleukin 10 inhibitsacrophage microbicidal activity by blocking the endogenousroduction of tumor necrosis factor alpha required as a costimulatoryactor for interferon gamma-induced activation[J]. Proc Natl Acad Sci U S A, 1992, 89(18): 8676-8680. |
| [5] | Ding L, Shevach EM. IL-10 inhibits mitogen-induced T cellroliferation by selectively inhibiting macrophage costimulatoryunction[J]. Immunol, 1992, 148(10): 3133-3139. |
| [6] | Lang R, Patel D, Morris JJ, et al. Shaping gene expression inctivated and resting primary macrophages by IL-10[J]. Immunol,002, 169(5): 2253-2263. |
| [7] | Zdanov A, Schalk-Hihi C, Gustchina A, et al. Crystal structure ofnterleukin-10 reveals the functional dimer with an unexpectedopological similarity to interferon gamma[J]. Structure, 1995, 36): 591-601. |
| [8] | William TW, Rosalinda S, Anthony T, et al. Disulfide bondssignments and secondary structure analysis of human and marinenterleukin 10[J]. Biochemistry, 1993, 32(34): 8807-8815. |
| [9] | Syto R, Murgolo NJ, Braswell EH, et al. Structural and biologicaltability of the human interleukin 10 homodimer[J]. Biochemistry,998, 37(48): 16943-16951. |
| [10] | Weber-Nordt RM, Riley JK, Greenlund AC, et al. Stat3 recruitmenty two distinct ligand-induced, tyrosinephosphorylated dockingites in the interleukin-10 receptor intracellular domain[J]. Biolhem, 1996, 271(44): 27954-27961. |
| [11] | Zou J, Clark MS, Secombes CJ. Characterisation, expression andromoter analysis of an interleukin 10 homologue in the puffer fish,ugu rubripes[J]. Immunogenetics, 2003, 55(5): 325-335. |
| [12] | Savan R, Igawa D, Sakai M. Cloning, characterization andxpression analysis of interleukin-10 from the common carp,yprinus carpio L[. J]. Biochem, 2003, 270(23): 4647-4654. |
| [13] | Zhang DC, Shao YQ, Huang YQ, et al. Cloning, characterizationnd expression analysis of interleukin-10 from the zebrafish(Danioerion)[J]. Biochem Mol Bio, 2005, 38(5): 571-576. |
| [14] | Pinto RD, Nascimento DS, Reis MR, et al. Molecular characterization,D modelling and expression analysis of sea bass(Dicentrarchusabrax L.)interleukin-10[J]. Mol Immunol, 2007, 44(8): 056-2065. |
| [15] | Ingerslev HC, Ronneseth A, Pettersen EF, et al. Differentialxpression of immune genes in Atlantic salmon(Salmo salar L.)hallenged intraperitoneally or by cohabitation with IPNV. Scand.[J]. Immunol, 2009, 69(2): 90-98. |
| [16] | Grayfer L, Hodgkinson J, Hitchen SJ, et al. Characterization andunctional analysis of goldfish(Carassius auratus L.)interleukin-0[J]. Molecular Immunology, 2011, 48(4): 563-571. |
| [17] | Inoue Y, Kamota S, Ito K, et al. Molecular cloning and expressionnalysis of rainbow trout(Oncorhynchus mykiss)interleukin-10DNAs[J]. Fish Shellfish Immunol, 2005, 18(4): 335-344. |
| [18] | 黄瑜, 张雪利, 鲁义善, 等. 红笛鲷 tdt 基因融合蛋白原核表条件的优化及纯化[J]. 广东海洋大学学报, 2013, 33(1): 4-49. |
| [19] | Gay E, Babajko S. AUUUA sequences compromise human insulinlikerowth factor binding protein-1 mRNA stability[J].iochemical and Biophysical Research Communications, 2000, 2672): 509-515. |
| [20] | Sabat R, Grütz G, Warszawsk K, et al. Biology of interleukin-0[J]. Cytokine & Growth Factor Reviews, 2010, 21(5): 331-44. |
| [21] | Moore KW, O’Garra A, de Waal Malefyt R, et al. Interleukin-0[J]. Annu Rev Immunol, 1993, 11 : 165-190. |
| [22] | Grayfer L, Belosevic M. Identification and molecular characterizationf the interleukin-10 receptor 1 of the zebrafish(Danio rerio)nd the goldfish(Carassius auratus L.)[J]. Developmental andomparative Immunology, 2012, 36(2): 408-417. |





