2. 浙江大学生命科学学院,杭州 310058
2. College of Life Science,Zhejiang University,Hangzhou 310058
SPX结构域是由最早发现的3个蛋白名称的首个字母缩写来命名的,这3个蛋白分别是酵母的SYGI[1]和Pho81[2]以及人类的XPRI[3],它们的氨基端都含有一个相对保守共同结构域。目前在动物和植物以及很多高等真核生物中都发现了含SPX保守结构域的蛋白,根据所含结构域差异的分类,拟南芥和水稻基因组中所有编码含SPX结构域蛋白可以分成4个基因家族:SPX家族、SPX-RING家族、SPX-MFS家族及SPX-EXS家族[4, 5]。
酵母体内存在着一个被称为PHO体系的磷反应通路,该通路多个成员含有SPX结构域。在磷饥饿胁迫下的转录调控机制中,PHO通路研究最为详细,研究表明,植物缺磷时会产生类似于酵母PHO操纵子的多基因协同表达,组成一个高度协作的分子网络来适应磷胁迫[6, 7, 8]。植物中许多含有SPX结构域的基因参与对缺磷胁迫的反应,一些基因参与植物特异的磷高效吸收和利用机制,而其它基因则参与调控缺磷诱导的多基因表达[9, 10, 11, 12, 13]。植物中对磷饥饿响应的许多基因都已被克隆,如AtSPX1、AtSPX3,并对其表达调控做了深入研究[14, 15, 16]。在植物的根、叶、茎中存在着SPX蛋白家庭成员大量的表达[4]。MYB家族的转录因子PHR2是植物磷信号通路中心因子,研究表明在水稻中SPX4蛋白作为PHR2的负调控因子参与了植物磷信号转导和磷平衡[17]。在拟南芥和水稻中,SPX1蛋白具有同样的负调控PHR2的功能[10, 13, 16, 18],同时SPX1与SPX4蛋白均通过与PHR2蛋白结合抑制PHR2的功能[16, 17, 18]。
综上所述,SPX在植物磷信号通路中发挥了重要功能,但是SPX蛋白如何与PHR2结合并抑制后者的功能还未阐明清楚。对蛋白结构进行研究有助于阐明其中原因。本研究尝试通过克隆拟南芥AtSPX1基因,在原核细胞内过量表达AtSPX1蛋白,使用亲和层析和分子筛层析技术进行蛋白纯化,旨在为进一步解析SPX1的蛋白结构奠定基础。 1 材料与方法 1.1 材料
哥伦比亚野生型拟南芥由浙江农林大学亚热带森林培育国家重点实验室培育基地实验室栽培。E.coli DH5α感受态细胞,E.coli表达菌株BL21(DE3)以及表达质粒pE-SUMO为本实验室保存;RNA提取试剂盒购自北京鼎国昌盛有限公司;反转录试剂盒、DNA聚合酶、各种DNA限制性内切酶购自宝生物工程有限公司(大连);DNA连接酶购自New England Biolabs公司;DNA纯化试剂盒、胶回收试剂盒购自天根生化科技有限公司(北京);引物由生工生物工程股份有限公司(上海)合成;其余各试剂均为国产分析纯。 1.2 方法 1.2.1 AtSPX1基因克隆
用Trizol法提取拟南芥叶片组织RNA,反转录得到cDNA。根据拟南芥SPX1基因的cDNA保守序列以及该蛋白的二级结构预测进行引物设计进行AtSPX1基因全长克隆,上游引物A:5'-CGGGATCCATGAAGTTTGGTAAGAGTCTC AG-3'和下游引物B:5'-CCGCTCGAGCTATTTGGCT TCTTGCTCCAACA-3'(下划线为BamHⅠ和Xho Ⅰ酶切位点),由上海生工生物工程技术服务有限公司合成。聚合酶链式反应得到的产物连接到pMD-18T并转化到E.coli DH5α感受态细胞,经过菌检和双酶切实验后进行测序。将测序正确的菌株扩大提取质粒后,用BamHⅠ和XhoⅠ双酶切重组质粒和pESUMO、pET-HTT和pET-GTT载体,切胶回收目的片段。回收产物连接到含有同样黏性末端载体上,连接反应在16℃过夜。反应混合物转化到DH5α感受态细胞并涂布于抗性的LB平板上,挑菌培养后菌液PCR检测,并将检测正确的菌液进行测序,由此克隆得到了pE-SUMO-AtSPX1、pET-HTT-AtSPX1和pET-GTT-AtSPX1重组质粒。 1.2.2 AtSPX1理化性质分析
用DNASTAR5.0软件分析cDNA序列和开放阅读框;利用瑞士生物信息学研究所的蛋白分析专家系统(Expert Protein Analysis System,ExPASy)提供的生物信息学工具Protparam,分析预测蛋白质的氨基酸残基性质、分子量及理论等电点。采用Signal IP 3.0 Server分析信号肽序列。 1.2.3 AtSPX1的原核表达
重组表达质粒分别转化表达菌株RosettaTM (DE3)和BL21(DE3),从抗氨苄霉素和卡那霉素平板上挑取单克隆,接入2 mL LB液体培养基37℃培养,当菌体OD600达到0.6时,其中一部分加入0.3 mmol/L IPTG诱导,在37℃摇床上诱导5 h,收集菌体。另一部分加入0.3 mmol/L IPTG诱导,在20℃摇床上诱导14 h,收集菌体。超声(工作5 s间歇6 s,总时间1 min,功率20%)破碎,SDS-PAGE检测蛋白表达及可溶性情况。 1.2.4 重组AtSPX1蛋白的纯化与SUMO标签酶切
8 000×g离心收集诱导后的1 L菌液细胞,加入20 mL裂解液(1 mol/L NaCl,20 mmol/L Tirs-HCl pH 8.0,10% glycerol)冰水下功率为200 W,工作6 s间歇8 s超声20 min,静置15 min后,在4℃条件下转速17 000 r/min离心35 min。收集上清液,将上清液加入已经平衡好的Ni-NTA柱,放入冰浴摇床缓慢摇动结合30 min后,将纯化柱置于支架上,收集未特异性结合琼脂糖树脂上的杂蛋白液,用20个柱体积Wash Buffer (1 mol/L NaCl,20 mmol/L Tris-HCl pH 8.0,10% glycerol,100 mmol/L Imidazole)洗脱非特异性结合的杂蛋白,最后用15倍树脂体积的Elution Buffer (1 mol/L NaCl,20 mmol/L Tris-HCl pH8.0,10% glycerol,250 mmol/L Imidazole)收集目的蛋白。向蛋白洗脱液中加入ULP1蛋白酶,低温4℃酶切过夜。 1.2.5 硫酸铵沉淀纯化AtSPX1蛋白
将上述酶切反应液用超滤浓缩柱进行浓缩,在目的蛋白的浓度达到0.3 mg/mL时,测量浓缩液体积并查表确定硫酸铵的用量。将硫酸铵分4次加入浓缩液中,每次加入硫酸铵后都轻微摇晃离心管,最后将离心管冰上静置10 min,12 000 r/min离心5 min后弃上清,加入3 mL裂解液(1 mol/L NaCl,20 mmol/L Tirs-HCl pH8.0,10% glycerol)将离心管底部沉淀悬浮。 1.2.6 分子筛层析分析目的蛋白与质谱鉴定
将硫酸铵沉淀法所获的目的蛋白悬浮液用超滤浓缩柱进行浓缩,4 000×g离心浓缩至500 μL体积后上样,使用分子筛SuperdexTM 12 10/300 GL分离纯化目的蛋白,以缓冲液(1 mol/L NaCl,20 mmol/L Tirs-HCl pH 8.0)0.5 mL/min流速分离纯化目的蛋白,取10 μL fraction蛋白样品加入2 μL蛋白上样缓冲液,煮沸10 min,点样于12%聚丙烯酰胺凝胶(SDS-PAGE),完成电泳后观察目的蛋白的纯化情况。将切取的目的蛋白SDS-PAGE胶点转入离心管中,样品质谱鉴定由上海博苑生物技术有限公司进行。 2 结果 2.1 AtSPX1蛋白理化性质及信号肽预测
利用ProtParam工具预测表明AtSPX1蛋白的相对分子质量为30.07 kD,理论等电点(pI值)7.76,含酸性氨基酸(DE)16.4%,碱性氨基酸(KR)16.8%,亲水性氨基酸(AILFWV)、极性氨基酸(NCQSTY)和带电氨基酸(RKHYCDE)的比例分别为35.6%、22.6%和30.9%。根据在线网站Signal IP 3.0 Server分析AtSPX1蛋白,结果显示AtSPX1蛋白序列中预测到一个双向核定位信号,位于该蛋白序列的第30-46位氨基酸的位置。 2.2 pE-SUMO-AtSPX1原核表达载体的构建
测序验证表明成功构建重组AtSPX1质粒,转化成功的菌株提取质粒进行PCR鉴定(图 1-A)和双酶切(图 1-B)均得到目的片段,成功构建了pESUMO-AtSPX1原核表达载体。
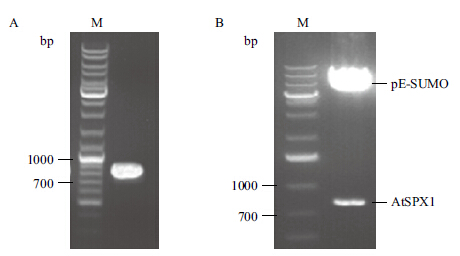 |
| 图 1 AtSPX1基因PCR扩增结果(A)和pE-SUMOAtSPX1限制性酶切分析(B) |
诱导温度、时间及载体对原核表达蛋白的可溶性有重要影响,高温条件下菌株快速表达目的蛋白容易使蛋白形成不正确的构象折叠最后以包涵体的形式存在,不利于获得可溶性蛋白。菌液浓度在OD600值0.4-0.6时,以37℃不加入IPTG培养5 h为对照组,以IPTG终浓度0.3 mmol/L的为诱导条件,分别在37℃,5 h和20℃,14 h的诱导条件下进行诱导培养。SDS电泳结果(图 2)显示,3种载体只有pE-SUMO-AtSPX1蛋白在20℃条件下上清有表达(图 2-C)。
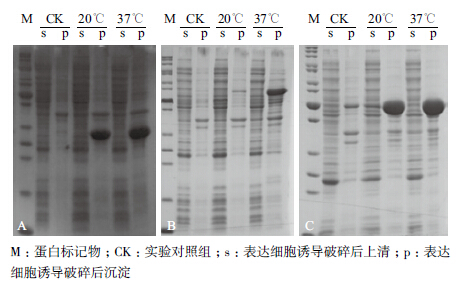 |
| 图 2 分别含pET-HTT (A)、pET-GTT (B)和pE-SUMO (C)三种载体的AtSPX1融合蛋白的表达检测 |
不同咪唑梯度洗脱蛋白后的SDS-PAGE检测结果(图 3)显示,从第7号孔道可以看出,在高浓度咪唑(1 mol/L NaCl,20 mmol/L Tris-HCl pH8.0,10% glycerol,250 mmol/L Imidazole)条件下,可洗脱出条带单一目的蛋白。
 |
| 图 3 SDS-PAGE检测AtSPX1 Ni-NTA亲和层析纯化 |
在15 mL洗脱下的蛋白液中(1 mol/L NaCl,20 mmol/L Tirs-HCl pH 8.0,250 mmol/L Imidazole,10% glycerol),加入0.1%(W/W) ULP1蛋白酶,低温4℃酶切过夜,用硫酸铵沉淀的方法能够将靶标蛋白和标签分开(图 4)。生物信息学分析表明,AtSPX1蛋白N端存在一个17个氨基酸的双向核定位信号,这个片段在纯化过程中很容易降解掉,所以图 4中酶切过后出现两条带。
 |
| 图 4 硫酸铵沉淀 |
将硫酸铵沉淀法获得的蛋白用10 kD超滤浓缩柱浓缩至体积500 μL,通过AKTA Purifier,上样至凝胶过滤层析柱SuperoseTM 12 10/300 GL作进一步纯化。作为标准的牛血清蛋白(BSA)、卵清蛋白(Ovalbumin)、木瓜凝乳蛋白酶(Chymopapain)大小分别为67、43和25 kD,对比作为标准的3种蛋白的洗脱体积和分子量关系可知[19],目的蛋白峰位置所对应的分子量大小在卵清蛋白(43 kD)位置之后和木瓜凝乳蛋白酶(25 kD)之前(图 5-A),初步确定其为单体(30 kD),并且重组蛋白只出现明显的单峰,说明蛋白的均一性较好。分子筛层析后,分管收集的蛋白变性后用聚丙烯酰胺凝胶变性电泳检测,结果(图 5-B)显示蛋白分子量为30 kD,蛋白纯度较高。
 |
| 图 5 AtSPX1的凝胶过滤层析图(A)及SDS检测(B) |
对SDS-PAGE考马氏亮蓝染色后的目的蛋白进行MALDI-TOT/TOF质谱。上海博苑生物技术有限公司提供的检索结果显示(检索程序是Mascot:http://www.matrixscience.com,数据库为NCBInr),目的蛋白为gi|15241275,检测结果中有21条肽段与目的蛋白AtSPX1序列相匹配。图 6-A红色部分表示与目的蛋白所匹配的多肽片段,覆盖率超过60%。图 6-B是Mascot Score直方图,右边红色表示匹配的结果,得分为938分且有显著性差异(P<0.05)。左边阴影区域为小片段相匹配的肽段,不具备显著性差异,为不可信任结果。
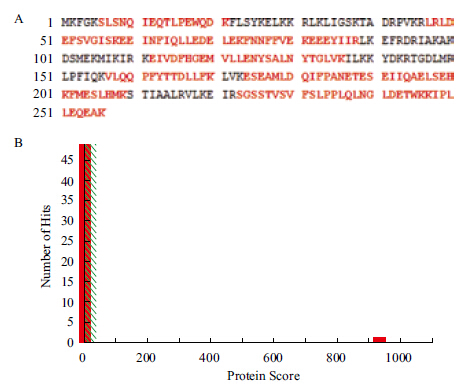 |
| 图 6 AtSEP3蛋白质肽质量指纹图谱鉴定(A)及Mascot Score直方图(B) |
近年来,关于植物响应磷营养胁迫的生理生化机制和信号传导途径的研究已经有了较大的进展,研究显示一部分SPX蛋白参与了磷的吸收代谢过程。但也有研究结果表明,OsSPX1基因表达水平下降引起植物对冷胁迫的敏感性增强[11, 12]。AtSPX1基因和OsSPX1具有50.5%的相似性,表明AtSPX1和植物低温耐受性也可能有关。同时SPX结构域在多个物种间有着较高的保守性,说明了SPX结构域对这些蛋白所行使的功能是不可缺少的。目前亚细胞定位的结果显示了AtSPX1定位于细胞核中,这与生物信息学预测AtSPX1蛋白在氨基端含有一个17个氨基酸的双向核定位信号相一致。这个片段在纯化过程中容易被降解掉,也解释了硫酸铵沉淀后出现两条带的结果,两条带经过质谱实验测定都是AtSPX1蛋白。在目前的原核表达过程中,20℃诱导发现有可溶性AtSPX1蛋白表达,而37℃诱导可溶性表达则明显减少。进一步改变诱导温度、时间以及IPTG浓度,发现可溶性没有明显增加,同时改变蛋白表达宿主细胞也没有明显效果。这个问题计划在后期的实验中通过重新构建不含核定位信号的表达载体来解决,这有可能提高蛋白的产量。
分子筛实验表明纯化的AtSPX1蛋白是以单体形式存在,同时这个蛋白只包含一个SPX结构域,表明SPX结构域自身并不倾向于形成同型二聚体。拟南芥的SPX2、SPX3、SPX4与SPX1相似性分别是66%、47%和44%,它们都只包含一个长度大约为150个氨基酸的SPX结构域。这个分析暗示拟南芥同源SPX家族成员也可能是单体。但是不同SPX蛋白的SPX结构域长度明显不同,例如,水稻的OsSPX4的SPX结构域包含170个氨基酸,拟南芥11个PHO1蛋白都含有300-360个氨基酸不等的SPX结构域,不能排除这些蛋白形成多聚体的可能性。有实验表明,拟南芥的AtSPX1蛋白的功能与铁和磷营养有关联[8],可能是这两种信号转导途径的交叉点之一,研究SPX结构域在其中的作用以及与它蛋白的互作是值得关注的方向。 4 结论
本研究成功构建了拟南芥SPX1基因原核表达载体pE-SUMO-AtSPX1,在大肠杆菌(E.coli)细胞中获得了该蛋白可溶性高表达,经过亲和层析、硫酸铵沉淀、分子筛层析方法得到高纯度的AtSPX1蛋白,分子筛层析分析表明AtSPX1以单体形式存在。
| [1] | Spain BH,Koo D,Ramakrishnan M,et al.Truncated forms of a novel yeast protein suppress the lethality of a G protein alpha subunitdeficiency by interacting with the beta subunit[J].J Biol Chem,1995,270(43):25435-25444. |
| [2] | Creasy CL,Madden SL,Bergman LW.Molecular analysis of the PHO81 gene of Saccharomyces cerevisiae[J].Nucleic Acids Res,1993,21(8):1975-1982. |
| [3] | Battini JL,Rasko JE,J Miller AD.A human cell-surface receptor for xenotropic and polytropic murine leukemia viruses:possible role in G protein-coupled signal transduction[J].Proc Natl Acad Sci USA,1999,96(4):1385-1390. |
| [4] | Duan K,Yi K,Dang L,et al.Characterization of a sub-family of Arabidopsis genes with the SPX domain reveals their diverse functions in plant tolerance to phosphorus starvation[J].Plant J,2008,54(6):965-975. |
| [5] | Wang C,Huang W,Ying Y,et al.Functional characterization of the rice SPX-MFS family reveals a key role of OsSPX-MFS1 in controlling phosphate homeostasis in leaves[J].New Phytologist,2012,196(1):139-148. |
| [6] | Bun-Ya M,Harashima S,Oshima Y.Putative GTP-binding protein,Gtr1,associated with the function of the Pho84 inorganic phosphate transporter in Saccharomyces cerevisiae[J].Mol Cell Biol,1992,12(7):2958-2966. |
| [7] | Ogawa N,DeRisi J,Brown PO.New components of a system for phosphate accumulation and polyphosphate metabolism in Saccharomyces cerevisiae revealed by genomic expression analysis[J].Mol Biol Cell,2000,11(12):4309-4321. |
| [8] | Ghillebert R,Swinnen E,De Snijder P,et al.Differential roles for the low-affinity phosphate transporters Pho87 and Pho90 in Saccharomyces cerevisiae[J].Biochem J,2011,434:243-251. |
| [9] | Wang Y,Ribot C,Rezzonico E,et al.Structure and expression profile of the Arabidopsis PHO1 gene family indicates a broad role in inorganic phosphate homeostasis[J].Plant Physiol,2004,135(1):400-411. |
| [10] | Wang C,Ying S,Huang H,et al.Involvement of OsSPX1 in phosphate homeostasis in rice[J].Plant J,2009,57(5):895-904. |
| [11] | Zhao L,Liu F,Xu W,et al.Increased expression of OsSPX1 enhances cold/subfreezing tolerance in tobacco and Arabidopsis thaliana[J].Plant Biotechnol J,2009,7(6):550-561. |
| [12] | Wang C,Wei Q,Zhang K,et al.Down-Regulation of OsSPX1 causes high sensitivity to cold and oxidative stresses in rice seedlings[J].PloS One,2013,8(12):e81849. |
| [13] | Liu F,Wang Z,Ren H,et al.OsSPX1 suppresses the function of OsPHR2 in the regulation of expression of OsPT2 and phosphate homeostasis in shoots of rice[J].Plant J,2010,62(3):508-517. |
| [14] | Yao ZF,Liang CY,Zhang Q,et al.SPX1 is an important component in the phosphorus signalling network of common bean regulating root growth and phosphorus homeostasis[J].Journal of Experimental Botany,2014,65:3299-3310. |
| [15] | Shi J,Hu H,Zhang K,et al.The paralogous SPX3 and SPX5 genes redundantly modulate Pi homeostasis in rice[J].Journal of Experimental Botany,2014,65(3):859-870. |
| [16] | Puga MI,Mateos I,Charukesi R,et al.SPX1 is a phosphatedependent inhibitor of PHOSPHATE STARVATION RESPONSE 1 in Arabidopsis[J].Proc Nat Acad Sci USA,2014,111(41):14947-14952. |
| [17] | Lv Q,Zhong Y,Wang Y,et al.SPX4 negatively regulates phosphate signaling and homeostasis through its interaction with PHR2 in rice[J].Plant Cell,2014,26(4):1586-1597. |
| [18] | Wang Z,Ruan W,Shi J,et al.Rice SPX1 and SPX2 inhibit phosphate starvation responses through interacting with PHR2 in a phosphate-dependent manner[J].Proc Natl Acad Sci USA,2014,111(41):14953-14958. |
| [19] | Zhang F,Xu Y,Zhang Z.Heterologous expression,purification,crystallization and preliminary X-ray diffraction analysis of KDO8P synthase from Arabidopsis thaliana[J].Protein Expression and Purification,2014,101:133-137. |




