MicroRNAs (miRNAs)是一类内源基因编码的单链小分子非编码RNA,长度为18-25个核苷酸,其主要通过两种机制调控其靶基因的表达:剪切靶基因mRNA或者抑制靶基因翻译[1, 2, 3]。miRNAs是植物应对生物和非生物逆境中的重要调控因子[4, 5]。miR169是受多种胁迫(如干旱、氮饥饿、高盐、冷害等)条件调控的一个保守miRNA家族[6, 7, 8]。核因子Y (nuclear factor Y,NF-Y)是普遍存在的一类转录因子,由NF-YA、NF-YB和NF-YC三个亚基组成。NF-YA家族含有miR169靶标位点,miR169可以抑制NF-YA基因转录[9, 10]。氮饥饿显著下调拟南芥中miR169的表达水平,上调靶基因NF-YA的表达,从而调控植物中氮素传感器NRT1.1的表达,因此调节了植物应对氮饥饿的反应[7]。高盐胁迫能够诱导拟南芥miR169的表达,其靶基因NF-YA作为CAAT结合因子,进一步参与基因表达调控和信号转导,从而抵抗高盐胁迫[11]。miR169还参与调控植物的花期,过量表达miR169d,抑制了靶基因AtNFYA2,从而减少FLC (Flowering Locus C,FLC)的表达,导致FLC的靶基因FT (FLOWERING LOCUS T)和LFY (LEAFY)表达量增加,从而促进拟南芥开花[12]。干旱胁迫下调拟南芥miR169a和miR169c的表达,共表达实验表明miR169a能更高效地抑制atNFYA5的mRNA水平。nfya5突变体和过表达ath-miR169a的转基因植株加快了叶片的失水速率,对干旱胁迫也更加敏感[9]。共表达实验和5'RACE实验都证明miR169可以剪切菜豆GmNFYA3,将GmNFYA3在拟南芥中过表达,则转基因植株叶片失水速率降低,抗旱性明显增强,而对高盐、ABA则更为敏感[10]。此外,miR169还参与了植物根发育的调控,通过miRNAs类似物和剪切位点突变的NF-YA基因的相关分析表明,miR169在根的生长和分支上发挥了特殊的调控作用。miR169defg通过抑制NF-YA2和 NF-YA10的转录和蛋白翻译影响了拟南芥主根的生长和侧根的起始[13]。干旱胁迫诱导了sly-miR169在番茄体内的积累水平,相应地下调了siNF-YA1/2/3的转录水平,过量表达sly-miR169c的转基因植株降低了气孔的开放、呼吸速率和叶片的失水率,从而增强了对干旱的耐受性[14]。这些结果表明,尽管miR169是一个非常保守的家族,但miR169在不同物种中的功能并不完全一致,参与干旱调节的主效靶基因也可能并不完全相同。
干旱胁迫是水稻生长发育的重要环境影响因子之一,能够导致水稻减产,甚至死亡。目前,水稻干旱胁迫反应研究已经深入到分子水平,涉及抗旱基因克隆、定位及功能分析等[15]。水稻miR169家族中对植物调控作用主要包括miR169g和miR169n/o两部分,其中miR169n和miR169o位于同一个miRNA基因簇中,二者相距3 707个碱基[16]。然而,在干旱胁迫条件下,水稻miR169o及其靶基因早期表达变化及其组织特异性尚不清楚。
为了明确缺水或干旱胁迫条件下,水稻miR169及其靶基因的表达调控模式,本研究利用qRTPCR法检测了胁迫初期水稻根、茎、叶中miR169o以及靶基因OsNF-YA1(Os03g48970)、OsNF-YA2 (Os12g42400)和OsNF-YA3(Os02g53620)的表达动态变化,以期明确缺水或干旱胁迫初期,miR169o及其靶基因的动态表达调控模式和组织特异性情况。 1 材料与方法 1.1 材料
水稻品种日本晴(Oryza sativa L.cv.Nipponbare)由本实验室保存;Trizol Regent购自Invitrogen公司; TaqMan® MicroRNA Reverse Transcription Kit购自ABI公司;RQ1 RNase-Free DNase、GoScriptTM Reverse Transcription System、GoTaq® qPCR Master Mix购自Promega公司;其他试剂均为国产分析纯;实验引物由北京华大基因科技有限公司合成。
水稻培养Hogland营养液[17]:10 mmol/L KH2PO4,2 mmol/L MgSO4,1 mmol/L CaCl2,0.1 mmol/L Fe-EDTA,50 μmol/L H3BO4,12 μmol/L MnSO4,1 μmol/L ZnCl2,1 μmol/L CuSO4,0.2 μmol/L Na2MoO4,pH5.5-6.0。 1.2 方法 1.2.1 水稻幼苗培养及其缺水胁迫处理
将水稻种子置于培养皿清水中,在37℃下催芽2 d。待幼芽长至1 cm长时,将其种植于泡沫板上,将泡沫板置于清水中,覆盖蛭石。4 d后将泡沫板移至含3 mmol/L KNO3的Hogland培养液中培养(28℃、16 h光照、8 h黑暗)。选取生长3周的水稻幼苗,将其从营养液中取出,用干燥吸水纸迅速吸除根部水分,进行脱水干旱胁迫处理0、0.5、1、2和4 h,然后采集根、茎和叶部样品,经液氮速冻后,置于-80℃保存备用。 1.2.2 水稻总RNA提取及cDNA合成
经缺水处理0、0.5、1、2和4 h的水稻根、茎和叶组织样品按照稍作改进的Trizol方法[18]进行水稻总RNA提取。经DNase处理后,用琼脂糖凝胶电泳检测RNA完整性及基因组DNA是否完全去除,用Nano Drop 2000分光光度计检测RNA浓度。miR169o反转录茎环引物为5'-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTAGGCA-3',按照ABI TaqMan® MicroRNA Reverse Transcription试剂盒方法进行反转录;靶基因OsNF-YAs反转录引物为Oligo dT,按照GoScriptTM Reverse Transcription System试剂盒方法进行反转录。 1.2.3 实时荧光定量PCR分析
利用Primer premier 5.0软件,设计miR169o、靶基因OsNF-YAs和内参基因U6的特异性引物(表 1)。以合成的cDNA为模板,按照GoTaq® qPCR Master Mix试剂盒方法,用ABI 7500实时定量PCR仪进行qRT-PCR检测。对照样品为未经胁迫处理的水稻样品。miR169o及其靶基因相对表达量按照2-△△ Ct法计算[19],其中△△Ct=(Ct样品-CtU6) TimeX-(Ct对照样品-CtU6) Time0,Time X表示任意时间点,Ct为荧光阈值。
以U6为内参基因、未处理样品为对照,进行缺水处理水稻根部样品qRT-PCR检测。实验中 miR169o、OsNFYAs和U6基因熔解曲线均为单一峰。分别计算各处理样品中基因的相对表达量。结果(图 1)表明,水稻根部miR169o表达量总体上呈升高趋势,在缺水处理0.5 h时出现极显著差异,在缺水处理2 h时达到最高峰,之后逐步下降,但仍明显高于对照。OsNF-YA2的表达动态变化与miR169o的完全相反,符合miRNA与靶基因的对应关系;OsNFYA1的表达动态与miR169o相似;OsNF-YA3在缺水处理1-4 h之间的表达动态与miR169o相反。
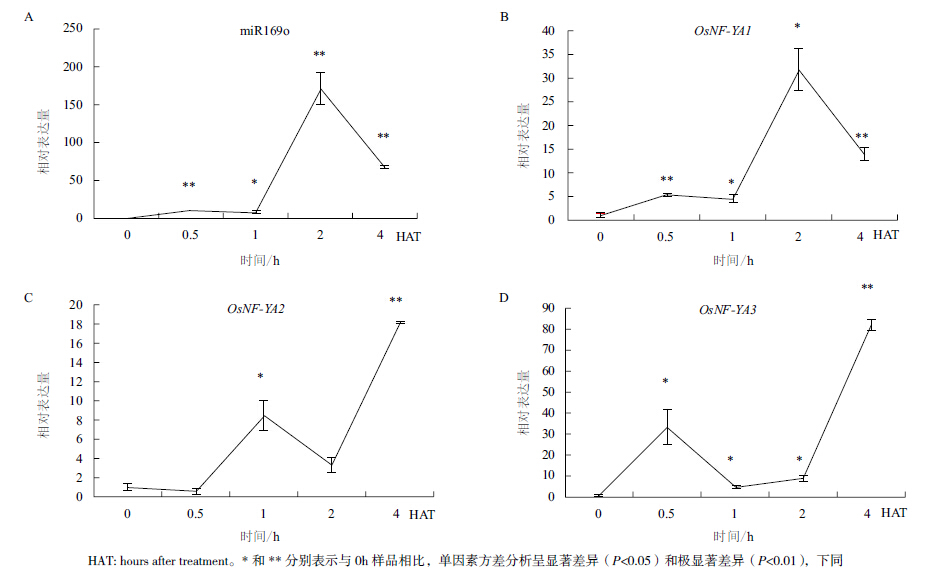 |
| 图 1 经缺水处理的水稻根部miR169o及OsNF-YAs表达 |
经缺水处理的水稻茎部miR169o表达动态与根部相似(图 2),但总体表达变化幅度不超过5倍。缺水处理1 h表达量迅速升高,2 h时出现一个2倍的峰值,随后下降,但4 h时但仍明显高于对照。3个OsNF-YAs的表达动态变化趋势基本一致,均与miR169o相反,符合miRNA与靶基因的对应关系。
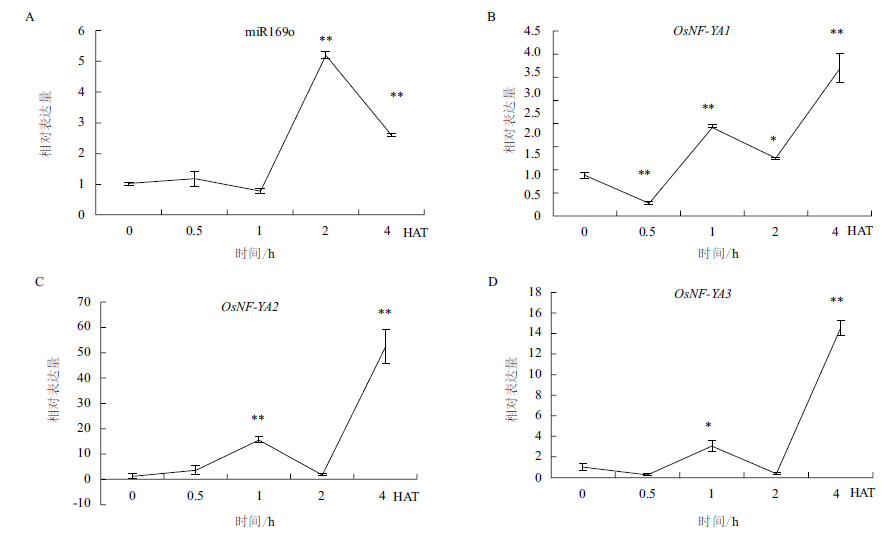 |
| 图 2 经缺水处理的水稻茎部miR169o及OsNF-YAs表达 |
经缺水处理的水稻叶部miR169o的表达量整体呈上升趋势(图 3)。缺水处理0-0.5 h迅速上升,在0.5 h时出现显著差异,0.5-1 h其表达基本维持此水平,随后迅速下降,在2 h时降至最底点,随后又上升至4倍峰值;OsNF-YA2和OsNF-YA3的表达在初始0.5 h显著下降,与miR169o呈相反趋势,但0.5-4 h的表达趋势同miR169o一致;OsNF-YA1的表达趋势与miR169o一致,同其在根部表达情况类似,不符合miRNA和靶基因的对应关系。
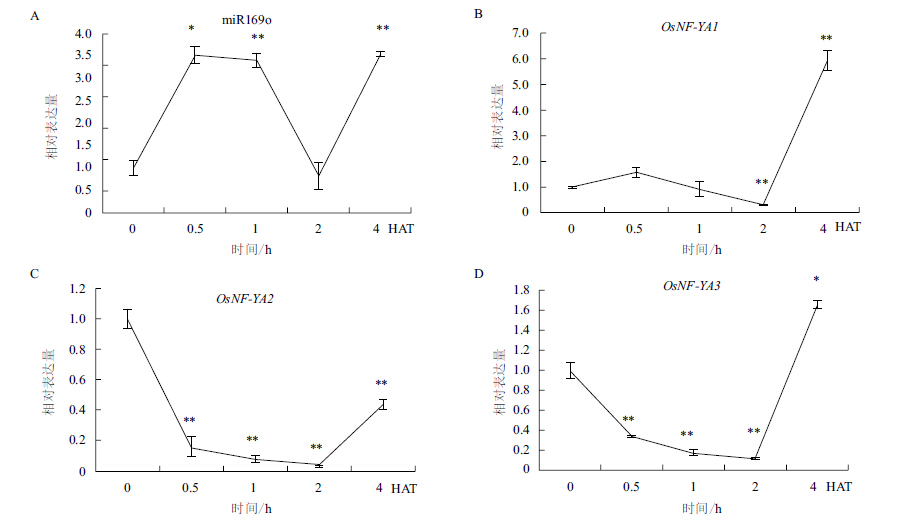 |
| 图 3 经缺水处理的水稻叶部miR169o及OsNF-YAs表达 |
miRNA在植物生长发育和应对逆境胁迫反应中发挥着重要的调控作用。近年来,针对miR169家族及靶基因NF-YAs的研究涉及到拟南芥[20]、水稻[21]、番茄[14]、大豆[10]、山杨[22]等植物,主要包括早花[12]、干旱[14]、高盐[16]、低氮[7]等性状的调控方面。水稻中miR169家族包含17个成员,代表 9个仅存在微小差别的不同的成熟序列。在高盐胁迫中,仅miR169g和miR169n/o受到诱导,其余miR169家族成员对盐胁迫无响应[16]。PEG 6000 (polyethylene glycol 6000)模拟干旱处理后,芯片分析表明水稻miR169家族中几个成员都能被诱导表达,然而Northern blot仅能验证miR169fg的诱导表达[23],这可能是由于不同技术方法的灵敏度和特异性所决定的。通常来说,盐胁迫和干旱胁迫之间存在着密切的联系,尽管已经报道miR169o在高盐胁迫下诱导表达,但对于干旱胁迫下水稻miR169o的动力学表达变化尚无报道。本研究对缺水干旱胁迫初期水稻miR169o及其靶基因的动态变化趋势进行了监测。对于胁迫条件设置,本实验室未采用PEG6000处理法,而是对植物进行缺水处理来模拟干旱胁迫初期的情况,这种处理方式较PEG6000处理更为剧烈,在4 h时植株已经出现明显萎蔫症状。在干旱胁迫初期0-4 h内,尽管水稻miR169表达水平在根、茎、叶中都有不同程度升降,但其表达总体上呈上升趋势。
本研究表明,干旱胁迫下miR169o在水稻不同组织中的表达具有明显的组织特异性。其在根、茎部的表达趋势基本一致,但根部变化幅度更大,可高达171倍。有报道在盐胁迫条件下玉米根部应答反应比叶部更快、更敏感[24]。本研究发现miR169o根部变化远高于茎部,可能是因为miR169o在干旱胁迫下根中的应答反应更为敏感。miR169o叶部表达趋势不同于根茎部,胁迫初始上调迅速,可能是干旱胁迫诱导了根部ABA积累、从根部运输到叶片,气孔开闭受抑制,从而减少水分流失[25, 26, 27]。因此,miR169o在叶部能迅速应答干旱胁迫,但其表达量变化范围远远小于根部。
本研究还发现,miR169与靶基因的表达变化趋势在茎部完全对应,但在根、叶部并不能完全对应。可能的原因是在干旱胁迫反应中,有其它miRNA、激素以及调控因子同时参与了调控过程[5]。有报道拟南芥NF-YAs家族分为3个亚组,不同亚组在根或地上部的积累量不同[7]。水稻OsNF-YAs家族也分为不同亚组,OsNF-YA1、OsNF-YA2和OsNFYA3处于不同亚组中,3个靶基因在根茎叶部表达趋势并不一致。其具体分布状况尚待研究。 4 结论
本研究定量分析了在干旱胁迫下水稻不同组织中miR169o及靶基因OsNF-YAs表达的动态变化,发现miR169o在干旱胁迫下表达量总体上升,其在根茎部动态变化趋势基本一致,根部在应对干旱胁迫应中更敏感,叶部反应更为迅速。靶基因OsNFYA1、OsNF-YA2、OsNF-YA3的表达在茎部与miR169o完全对应,而在根、叶部并不完全对应。
| [1] | Rogers K,Chen X.Biogenesis,turnover,and mode of action of plant microRNAs[J].Plant Cell,2013,25(7):2383-2399. |
| [2] | Bartel DP.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116(2):281-297. |
| [3] | Jones-Rhoades MW,Bartel DP,et al.MicroRNAs and their regulatory roles in plants[J].Annu Rev Plant Biol,2006,57:19-53. |
| [4] | Sunkar R,Chinnusamy V,Zhu J,et al.Small RNAs as big players in plant abiotic stress responses and nutrient deprivation[J].Trends in Plant Science,2007,12(7):301-309. |
| [5] | Khraiwesh B,Zhu JK,Zhu J.Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants[J].Biochim Biophys Acta, 2012,1819(2):137-48. |
| [6] | Covarrubias AA,Reyes JL.Post-transcriptional gene regulation of salinity and drought responses by plant microRNAs[J].Plant Cell Environ,2010,33(4):481-489. |
| [7] | Zhao M,Ding H,Zhu JK,et al.Involvement of miR169 in the nitrogen-starvation responses in Arabidopsis[J].New Phytol, 2011,190(4):906-915. |
| [8] | Lundmark M,Korner CJ,Nielsen TH.Global analysis of microRNA in Arabidopsis in response to phosphate starvation as studied by locked nucleic acid-based microarrays[J].Physiol Plant,2010, 140(1):57-68. |
| [9] | Li WX,Oono Y,et al.The Arabidopsis NFYA5 transcription factor is regulated transcriptionally and posttranscriptionally to promote drought resistance[J].Plant Cell,2008,20(8):2238-2251. |
| [10] | Ni Z,Hu Z,Jiang Q,et al.GmNFYA3,a target gene of miR169,is a positive regulator of plant tolerance to drought stress[J].Plant Mol Biol,2013,82(1-2):113-129. |
| [11] | Liu HH,Tian X,Li YJ,et al.Microarray-based analysis of stressregulated microRNAs in Arabidopsis thaliana[J].RNA,2008, 14(5):836-843. |
| [12] | Xu MY,Zhang L,Li WW,et al.Stress-induced early flowering is mediated by miR169 in Arabidopsis thaliana[J].J Exp Bot, 2014,65(1):89-101. |
| [13] | Sorin C,Declerck M,Christ A,et al.A miR169 isoform regulates specific NF-YA targets and root architecture in Arabidopsis[J]. New Phytol,2014,202(4):1197-1211. |
| [14] | Liu HJ,Chen LG,Zhu PP,et al.Effect of hyacinth mulching on rice (Oryza sativa L.) uptake and utilization of nitrogen[J]. Environmental Science,2011,32(5):1292-1298. |
| [15] | 潘雅姣,傅彬英,王迪,等.水稻干旱胁迫诱导DNA甲基化时空变化特征分析[J].中国农业科学,2009,42:3009-3018. |
| [16] | Zhao B,Ge L,Liang R,et al.Members of miR-169 family are induced by high salinity and transiently inhibit the NF-YA transcription factor[J].BMC Mol Biol,2009,10:29. |
| [17] | Hoagland DR,Arnon DI.The water-culture method for growing plants without soil[M].2nd ed.Circular.California Agricultural Experiment Station,1950:347. |
| [18] | Rio DC,Ares M,Hannon GJ,et al.Purification of RNA using TRIzol(TRI reagent)[J].Cold Spring Harbor Protocols,2010(6): pdb.prot5439. |
| [19] | Livak KJ,Schmittgen TD.Analysis of relative gene expression data using Real-Time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408. |
| [20] | Yang B,Ma HY,Wang XM,et al.Improvement of nitrogen accumulation and metabolism in rice (Oryza sativa L.) by the endophyte Phomopsis liquidambari[J].Plant Physiol Biochem, 2014,82:172-182. |
| [21] | Liu Q,Zhang YC,Wang CY,et al.Expression analysis of phytohormone-regulated microRNAs in rice,implying their regulation roles in plant hormone signaling[J].FEBS Letters, 2009,583(4):723-728. |
| [22] | Jia X,Wang WX,Ren L,et al.Differential and dynamic regulation of miR398 in response to ABA and salt stress in Populus tremula and Arabidopsis thaliana[J].Plant Molecular Biology,2009,71 (1-2):51-59. |
| [23] | Zhao B,Liang R,Ge L,et al.Identification of drought-induced microRNAs in rice[J].Biochem Biophys Res Commun,2007, 354(2):585-590. |
| [24] | Jia W,Wang Y,et al.Salt-stress-induced ABA accumulation is more sensitively triggered in roots than in shoots[J].Journal of Experimental Botany,2002,53(378):2201-2206. |
| [25] | Daszkowska-Golec A,Szarejko I.The molecular basis of ABA-mediated plant response to drought[J].Agricultwre and Biological Scieurs,2013:103-133. |
| [26] | Wilkinson S,Davies WJ.ABA-based chemical signalling:the co-ordination of responses to stress in plants[J].Plant,Cell&Environment,2002,25(2):195-210. |
| [27] | Chaves MM,Maroco JP,Pereira JS.Understanding plant responses to drought-from genes to the whole plant[J].Functional Plant Biology,2003,30(3):239-264. |





