RNA干扰(RNA interference,RNAi)技术作为分子生物学研究中发展迅速的一种新兴基因阻断技术,目前已成为生物领域的研究热点。它是一种进化上保守的作用机制,广泛存在于动物、植物和微生物中,包括真菌中的基因压制(Gene quelling)现象、植物中的共抑制(Co-suppression)现象和动物中的基因干扰现象[1]。这些现象的产生主要是通过反义RNA与正义RNA形成的dsRNA (Double-stranded RNA,dsRNA)分子,在细胞内被切割成21-23 bp大小的siRNA (Small interference RNA),特异性降解与之同源的单个内源靶基因的mRNA,从而阻断基因表达,产生基因沉默[2, 3]。近几年发展的RNAi技术能够高效特异的沉默功能基因,使生物体产生相应的功能缺陷表型,以此确定基因的功能。随着多种真菌如构巢曲霉(Aspergillus nidulans)、粗糙脉孢菌(Neurospora crassa)、大丽轮枝菌(Verticillium dahliae)和稻瘟病菌(Magnaporthe oryzae)等基因组测序计划的完成,真菌基因功能的鉴定迫在眉睫[4]。RNAi技术以其特异、高效、广泛性优势,被迅速应用到真菌基因功能的研究领域中,逐渐成为一种具有潜力的基因功能研究技术。 1 RNAi的发现
20世纪90年代初,Jorgensen研究组将一个强启动子控制的色素合成相关基因--查耳酮合成酶基因(chs)转入牵牛花中,试图用转基因技术手段使紫色矮牵牛花的花朵更加艳丽,结果并未获得预期的深紫色花朵,反而得到了斑驳状花朵及白色的花。深入的研究证明,被转入该植物中的chs基因与牵牛花中与该基因同源的色素基因的表达均受到了抑制,这种外源基因和自身同源基因均受到抑制的现象被命名为共抑制(co-suppression)现象[5, 6]。
该现象同样出现于真菌转基因研究中。1994年,当Cogoni等[7]把外源同源基因转入脉孢菌(Neurospora)时,该基因及Neurospora自身的同源基因的表达均显著性的下降,当时把这种在真菌中发现的类似于共抑制的现象称为"基因压制"(Gene quelling)现象,但当时并未明确其机制。
1995年,美国康乃尔大学的Guo和Kemphues博士等[8]发现由正义RNA和反义RNA形成的dsRNA均能有效并特异性地抑制秀丽新小杆线虫(Caenorhabditis elegans)中par-l基因的表达。这一发现有力地冲击了只有反义RNA形成的dsRNA才能有效地干扰具有同源序列的内源基因的表达这一传统观念。然而由于当时理论水平和实验依据的限制,这一现象并没有得到合理的解释。
1996年,Cogoni等[9]经过深入的研究证实真菌中的gene quelling与植物中的基因沉默机理基本相同。
1998年,华盛顿卡耐基研究院的科学家Fire[10]和麻省大学医学院的科学家Mello首次证实了Guo等发现的由正义RNA抑制同源基因表达的现象是由于在体外转录制备的RNA中污染了dsRNA而引发,并将这种现象命名为RNA干扰(RNAi)。由于RNAi的作用,功能基因无法表达而使其相对应的表型不能显现,导致基因功能的缺失或减弱,因此又将此作用机制称之为基因沉默(Gene silencing)。Fire和Mello的研究首次证明RNAi作用存在于生物界中,并因此获得2006年诺贝尔生理学和医学奖[11]。
目前研究发现,很早以前RNAi就普遍存在于小鼠、斑马鱼、果蝇、拟南芥和真菌等生物之中。在整个生物界进化过程中,它既是一种古老的生物学现象,又是一种保守的自我保护机制,因此又被称为"基因组的免疫系统"。这种免疫机制的存在不仅能避免生物体的基因组受到诸如病毒基因的外源基因分子的干扰和袭击,而且,也能通过实时定量地调控各阶段基因的表达来控制和调节细胞分化[12, 13]。 2 RNAi的分子机制
RNAi一经发现便引起了研究者们的高度重视,如今已成为一种高效的"基因沉默"技术,并在此技术基础之上衍生出了TALAN技术和CRISPR技术。该技术广泛应用于生物的基因功能鉴定和功能基因表达调控领域的研究,并且对dsRNA介导的基因沉默的分子机制的研究一直是生物学研究的热点。经研究证明,RNAi的分子作用机制在包括动植物的高等真核生物细胞中与真菌细胞中不尽相同。在动植物细胞中,依据dsRNA分子的来源,存在两种不同但又相关的RNAi途径:siRNA途径和miRNA (microRNA)途径。siRNA是由外源RNA或外源载体等导入细胞后产生的一系列小RNA,它能与靶mRNA完全互补并使其降解[14, 15];miRNA是一类由含颈环结构的前体RNA经过Dicer酶酶切而形成的非编码小RNA,它与靶mRNA的3'-非编码区结合,抑制靶mRNA降解和翻译表达,从而导致特定基因的沉默,而且大多数miRNA参与了生长与发育,生理代谢或压力应激过程[4, 16, 17]。在真菌细胞中,gene quelling属于进化上保守的siRNA途径,即典型的RNAi途径,而miRNA途径尚未被发现[1, 18]。另外,在粗糙脉胞菌中发现了第二种真菌基因沉默现象即减数分裂沉默(Meiotic silencing by unpaired DNA,MSUD),在第一次减数分裂前期,一段DNA在亲本一方正常,另一方不存在,或在亲本双方不同从而产生了未配对的DNA,使同源染色体不能正常配对,导致减数分裂异常,未配对的DNA就发生了沉默。这种现象发生在染色体配对到子囊孢子形成时期,MSUD参与了生长发育周期,因此,可能与miRNA起源于同一种古老的沉默机制[4, 15]。典型RNAi的分子机制作用途径大致可分为起始阶段、效应阶段和扩增阶段3个阶段。 2.1 起始阶段
dsRNA分为分子间双链和分子内双链。分子间双链是由分开的两个独立RNA分子互补形成,而分子内双链是由一个RNA分子回折,自身互补而形成。当细胞中的dsRNA扩增达到一定量时,外源或内源性的dsRNA被一种dsRNA特异的核酸酶RnaseIII家族中的Dicer酶特异识别,并将其逐步切割成长约21-23 nt的dsRNA片段(siRNA)。siRNA是RNAi作用赖以发生的重要中间效应分子,具有独特的特征性结构,其两条单链末端分别为5'-PO3和3'-OH,该siRNA与所作用的靶mRNA具有高度的核苷酸序列同源性。此外,由于每条siRNA单链的3'末端均有2-3个突出的非配对的碱基残基(图 1)[19, 20, 21],所以其化学性质相对稳定,无须像反义核苷酸那样通过大量的化学修饰来提高半衰期,这也是细胞赖以区分真正的siRNA和其他小dsRNA的结构基础。
2.2 效应阶段siRNA可与细胞质中特定的酶形成RNA诱导的沉默复合物(RNA-induced silencing complex,RISC),siRNA、核酸内切酶、核酸外切酶以及解旋酶是该复合体中的必备成分[22]。在RISC的作用下,siRNA解链成为单链,由其中的反义链特异识别目的mRNA并与之结合。一经结合,细胞内的核酸内切酶就在靶mRNA的近中点位置切割目的mRNA,被切割的mRNA片段被核酸外切酶降解后无法正常编码成相应的蛋白质,从而导致功能缺失[23, 24]。 2.3 扩增阶段
siRNA的反义链与靶mRNA的上游部分序列互补[25]。在依赖于RNA的RNA聚合酶(RNA-dependent RNA polymerase,RdRP)的作用下,经初级阶段产生的siRNA分子在细胞内能以自身反义链为扩增引物,以目标mRNA为模板,复制出新的dsRNA,新合成的dsRNA又可被降解成若干siRNA,这种新产生的siRNA (次级siRNA,secondary siRNA)又可形成更多的RISC复合物并作用于mRNA,使mRNA降解。这个过程使RNAi效应得到放大[26, 27]。 3 RNAi的特点 3.1 RNAi的高度特异性
siRNA与靶基因的mRNA在序列上严格遵循碱基互补配对原则,研究发现,即使发生一个碱基的错配,目的mRNA的沉默效率也会大大降低[28]。这种高精确度的序列特异性的识别能使dsRNA专一地降解与之同源的mRNA,从而精确沉默目的基因。 3.2 RNAi的高效性
细胞内的RNAi途径存在级联放大效应,极少量的dsRNA经起始阶段--效应阶段--扩增阶段的循环就能产生强烈的RNAi效应。靶mRNA被降解之后,细胞内的核糖核酸聚合酶继续催促dsRNA的合成并反复利用该dsRNA进行基因沉默,以维持高效、特异靶向的干扰作用。RNAi对基因表达的抑制率可达90%以上,这种高效的抑制效率是传统基因沉默实验技术所欠缺的[29, 30]。 3.3 RNAi的遗传性和传播性
目前已有大量的资料证实,在低等生物中RNAi可以传代,例如,在秀丽线虫(C.elegans)中通过生殖细胞可以传递好几代。另外,RNAi效应可以在细胞和组织间扩散,dsRNA可在生物体的不同组织间传递和维持[26, 31]。 3.4 RNAi的不完全性
RNAi对基因表达水平的抑制是不完全的,只是引起了部分下调,利用基因表达水平减弱这种不完全的沉默效应,可以对关键基因或致死基因的功能进行详细分析[32, 33]。同时,可根据基因沉默效率的不同程度来研究不同基因表达水平所引起的表型变化等更为详细的信息。且对于不同的基因其最低抑制率是不同的,例如根据抑制效率的多少,结合具体表型的变化来分析研究药物靶标[1, 34]。 3.5 RNAi的可调控性
RNAi是可以在不同组织和不同阶段进行实时调控的。已有研究报道称以可诱导型启动子或组织特异型启动子为骨架构建RNAi载体,可以实现时间特异性和组织特异性的基因沉默,从而调控RNAi作用的产生[35]。例如,Carneiro等[36]利用可诱导乙醇脱氢酶alcA启动子调控报告基因多聚半乳糖醛酸内切酶基因epg1的RNA沉默,由于选择的碳源的差异,EPG的表达能被不同程度的上调或下调。 3.6 RNAi的序列专一性
RNAi具有序列专一性的特点,而不具有基因座专一性特点[31, 37],该序列专一性可应用于基因家族的沉默表达。当靶基因所在的基因家族中存在一个或几个保守区域时,针对该保守区域设计的RNAi载体便可诱导整个基因家族的沉默。尤其是当存在基因冗余现象而掩盖了基因缺失突变体的表型时,采用RNAi技术可以避免基因互补[32]。 4 RNAi在真菌基因功能研究中的应用
在生物体生长发育过程中,抑制或沉默特定基因的表达技术通常用来研究该基因在该生理过程中的作用。目前,基因敲除和反义RNA技术已成为基因功能研究中最常用且很成熟的基因阻断技术。然而基因敲除技术,如通过同源重组进行敲除,操作复杂,一次只能完成一个基因的敲除,若靶基因是生长必需基因,则对该基因的敲除会导致个体的早期死亡而无法进行相关研究。同时,反义RNA技术抑制基因表达的效率较低,不利于目标基因的研究。与基因敲除和反义RNA技术相比,RNAi作为一种高效、特异的基因阻断技术,具有特异性、高效性、作用迅速性、高稳定性、操作相对简便、一次可研究多个基因等多方面的优势。目前RNAi正广泛地应用于功能基因组研究[38, 39],并显示出非常广阔的应用前景。
随着分子生物学技术的不断发展,镰刀菌(F.oxysporum)、稻瘟病菌及大丽轮枝菌等40多种真菌的基因组测序工作已完成,GenBank中积累了大量功能未知的真菌DNA序列[31, 34]。鉴定与明确这些基因的功能及生物学特性已成为当前研究中的热点问题和前进方向[40]。而功能基因的分析迫切需要一种高效、高通量技术来鉴定基因功能。RNAi技术可特异性地使特定基因沉默,其表现性状类似于基因功能缺失产生的表型[41],从而产生定向突变[42]。因此,RNAi技术以其快速、有效、能批量分析等优势,成为研究真菌基因功能的有效工具,在研究真菌的致病性及其营养生长方面尤为普遍[43],如对炭疽杆菌(Colletotrichum graminicola)[44]、柄锈菌(Puccinia triticina)[45]、绿僵菌(Metarhizium anisopliae)[46]和扩展青霉(Penicillium expansum)[47]等致病性的研究,以及对深绿木霉(Trichoderma atroviride)[48]菌丝生长的研究。 4.1 RNAi发夹结构表达载体在真菌基因功能研究中的应用
目前广泛应用于真菌基因功能研究的一种RNAi的方法是根据目的基因的反向重复序列结构构建可以诱发RNAi的发夹结构载体,即hpRNA (Hairpin RNA)表达载体。该表达载体导入真菌细胞后,在通用启动子Ptrpc作用下,反向重复序列会形成发夹结构的两臂,两臂之间由间隔序列相连,转录形成发夹结构的dsRNA,dsRNA被Dicer酶识别与加工后诱发目的基因沉默[15]。hpRNA表达载体于2002年首次应用于新型隐球菌(Cryptococcus neoformans)的RNAi研究中,以GFP作为间隔构建RNAi发夹结构表达载体,结果显示表现完全突变表型的转化子占总转化子的7%-10%[49]。
为了更为高效地利用hpRNA表达载体研究真菌基因功能,发展了两臂之间由真菌的内含子相连以载体pSilent-1(图 2)为基础的发夹结构表达载体,即ihpRNA表达载体。pSilent-1载体自身含有潮霉素抗性基因(Hygromycin resistant cassette)、多克隆位点和内含子间隔区,从而大大简化了载体构建程序,成为发夹结构表达载体构建的有效骨架。Nakayashiki等[34]用ihpRNA表达载体研究丝状真菌稻瘟病菌MPG1基因的功能,较普通的基因沉默技术,该沉默效率明显提高,高达70%-90%,完全突变表型占10%-50%。2007年,Krajaejun等[50]发展了以pFANTAi4载体为骨架的ihpRNA表达载体,载体构建利用Gateway技术,简便、快捷、成功率高、对原始载体和目的片段的限制少,避免了困难和繁琐的"酶切-连接"步骤,简化了克隆步骤,而且沉默效率同样很高。目前,ihpRNA表达载体在真菌基因功能研究中已经得到了广泛应用,并且技术已经成熟。如在荚膜组织胞浆菌(Histoplasma capsulatum)[51]、白腐真菌黄孢原毛平革菌(Phanerochaete chrysosporium)[32]、亚麻锈菌(Melampsora lini)[52]、产黄青霉(Penicillium chrysogenum)[53]和长孢黄萎病菌(Verticillium longisporum)[54]等许多真菌中都进行了基因功能的研究。
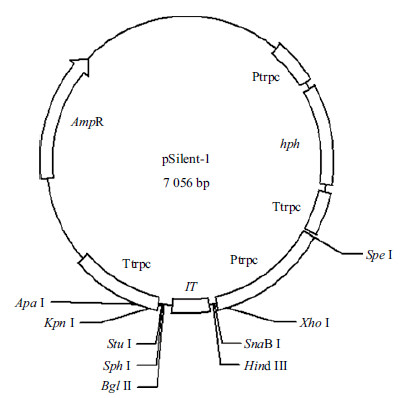 |
| AmpR :氨苄青霉素抗性基因;HPH :潮霉素抗性基因;IT :角质酶内含子2 图 2 沉默载体pSilent-1 图谱 |
针对大量单个基因或家族基因的基因功能的研究,若要构建hpRNA表达载体,需要确定目的基因的方向,工作量大又耗时,而反向双启动子系统RNAi载体的构建只需将目的基因非定性插入两个启动子之间,载体导入细胞后,靶基因就会在两个反向双启动子的作用下分别进行转录形成正义RNA链和反义RNA链,然后形成dsRNA,从而引起靶基因的沉默。这种载体非常适用于家族基因的沉默。
Nguyen等[55]利用含有TrpC启动子(The tryptophan synthetase)和gpdA启动子(Glyceraldehyde-3-phosphate dehydrogenase)两个方向相反的双启动子结构的pSD1(pSilent-Daul1)载体(图 3)研究了稻瘟病菌(M.oryzae)中所有涉及钙离子信号通路的37个相关基因,他们将编码增强型绿色荧光蛋白(eGFP)的基因与靶基因融合后插入两个启动子之间,使其形成转录嵌合体,并通过荧光信号的减弱程度来检测双干涉对突变菌株的沉默效率。另外,Nguyen等[56]采用"building blocks method"将人工合成的由GH10和GH11两个家族10个40 bp大小的内切木聚糖酶目的基因形成的400 bp序列插入到反向双启动子系统PSD1G (pSD1-eGFP)载体的EcoR V位点构成PSD1G-SDXYL沉默载体,从而使同源内切木聚糖酶家族基因同时发生沉默,研究结果表明内切木聚糖酶有利于稻瘟病菌在感病叶片中纵向穿透和水平侵染,而且在致病过程中是作为一个群体来发挥作用的。同时也证明了对于多基因同时沉默这种载体比hpRNA表达载体更有效。对单基因沉默,有研究报道其效率相对低些,可能是由于dsRNA形成率较低[15]。但是,Mu等[57]在对灵芝(Ganoderma lucidum)的URA3基因进行研究时,证明了反向双启动子系统沉默载体的沉默效率要比发夹结构沉默率高,可达81.9%。另外,他们通过对URA3和laccase以及一个报告基因的共沉默,说明了反向双启动子系统沉默筛选率可能更高。因此,反向双启动子系统RNAi载体更适用于高通量的真菌基因功能的研究。
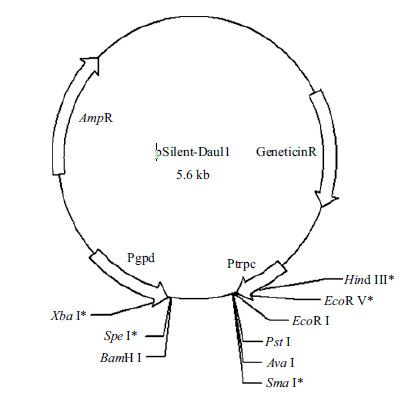 |
| 星号:酶切位点在载体中唯一;PtrpC:构巢曲霉trpC启动子;Pgpd:构巢曲霉gpd启动子;AmpR:氨苄青霉素抗性基因 图 3 沉默载体pSilent-Dual1 图谱 |
人工合成siRNA介导的RNAi在哺乳动物中的应用非常普遍,但是其在真菌中的应用报道却很少见。Khatri等[58]在研究构巢曲霉的鸟氨酸脱羧酶ODC基因时利用23 nt的siRNA duplex处理萌发的孢子引起孢子萌发和芽管生长明显减少,且培养18 h后沉默水平最高,并持续到培养48 h。而在哺乳动物中,人工合成siRNA介导的RNAi在数小时或1-2 d内明显激活并且可持续好多天[1]。但不可否认该研究为真菌细胞RNAi提供了一种简单、快捷的方法。Moazeni等[37]采用修改后的PEG/LiAc方法将不同浓度的19 nt siRNA导入白色念珠菌(Candida albicans)细胞,来抑制EFG1基因的表达,结果显示随着siRNA浓度的降低抑制作用也减弱,1 μmol/L的siRNA能够有效抑制EFG1基因的表达,其菌丝形成量明显减少,且降低了芽管的生长率,而采用100 nmol/L siRNA时得到了满意的结果。
Whisson等[59]通过脂质体介导原生质体转染的方法将150-300 bp大小的dsRNA直接导入致病疫霉菌(Phytophthora infestans)细胞,一种类似真菌的卵圆菌,促使inf1、edc14基因发生沉默,结果在转染12-15 d后,由于原来的dsRNA不再完整但在基因沉默过程中会发生级联放大效应,从而使mRNA表达水平明显降低。
上述3种研究真菌基因功能的方法,前两种方法最常用,而将人工合成的siRNA或dsRNA直接导入真菌细胞的RNAi方法虽然方便、快捷,但在多数真菌中不能起到有效的沉默作用[15]。 5 展望
RNA技术自1998年揭示以来,在植物、真菌、线虫等生物领域中取得了巨大成功,现已成为生命科学的研究热点之一。在显示出极大潜力的同时,在研究中也发现一些问题:(1) RNAi机制研究中:RISC组成成分如何,RISC与siRNA具体如何结合,靶向mRNA裂解过程中,除ATPase、解旋酶外,有没有其他大分子的参与,它们之间是如何相互作用的,与RNAi相关的基因有哪些、相关基因如何调控这一过程等问题尚需阐明。(2) siRNA表达载体的设计:由于一些mRNA序列在翻译之前可能掩藏在高度折叠的区域,或者与蛋白结合成牢固的复合体,从而干扰siRNA的特异性识别,导致siRNA并不能成功接近靶mRNA,完成降解过程。(3)如何保证导入的siRNA的稳定性,避免细胞内RNase的降解,仍是一个需解决的问题。(4) RNAi只需要靶基因的一小段序列,对于序列信息了解较少的真菌来说是一个优势,但可能会引起部分同源的非靶基因的沉默[1, 56]。沉默突变体无法通过重新导入靶基因形成恢复突变体。(5) RNAi载体在基因组中的随机插入可能会引起一些关键基因的插入突变从而影响表型,Nakayashiki等[34]在沉默稻瘟病菌PKSlike基因时,引起黑色素合成基因PKS的插入突变,导致菌落颜色发生改变。因此需要筛选大量的沉默突变子才能准确分析断定靶基因的功能。(6) RNAi技术常在细胞水平进行研究,其对整个生物体的作用效果尚不明确,并且作用于mRNA的不同靶位时,RNAi的效应机制存在差异。因此,应用RNAi技术需考虑其可行性,安全性和稳定性等问题。
尽管对RNAi的作用机制及应用还不是很清楚,但必须承认RNAi作为一种高效、特异性强的基因阻断技术,在功能基因组学上具有无穷的魅力,现已证明RNAi技术是一种无价的研究工具,在后基因组时代,可以更为快捷、有效地进行基因组功能研究,分析基因的功能。而且,这种技术将支持功能基因组学去发现和鉴定更多参与疾病和病害过程的新基因,为人类在基因研究领域开辟新路径。植物病原菌真菌是引起农作物病害的最大群体,是全球粮食安全的一个常见又主要的威胁,给全世界的农作物产量和市场化带来了严重损失[60],研究其致病基因,RNAi技术具有其他技术无可比拟的优势。对于GenBank中积累的大量未知的真菌基因,RNAi技术可作为鉴定其功能的一种重要方法。另外,许多真菌如粗糙脉胞菌、酿酒酵母(Saccharomyces cerevisiae)、粟酒裂殖酵母(Schizosaccharomyces pombe)和构巢曲霉等可作为模式生物应用于多种领域[4]。相信通过对真菌RNAi机制进行不断地深入研究,RNAi技术必将会更加完善和成熟,从而更好地探讨真菌与宿主间的相互作用,为疾病和病害防治提供新的策略[60],同时也会加快分子微生物学领域甚或整个生物学领域的研究步伐,显示出其广阔的应用前景。
| [1] | Nakayashiki H, Nguyen QB. RNA interference :roles in fungal biology[J]. Curr Opin Microbiol, 2008, 11(6):494-502. |
| [2] | Fire A. RNA-triggered gene silencing[J]. Trends Genet, 1999, 15 (9):358-263. |
| [3] | 张旭, 张树彪, 金梅. RNA 干扰在植物中的应用[J]. 安徽农 学通报, 2011(7):32-38. |
| [4] | Nakayashiki H. RNA silencing in fungi :mechanisms and applications[J]. FEBS Lett, 2005, 579(26):5950-5957. |
| [5] | Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric chalcone synthase gene into petunia results in reversible cosuppression of homologous genes in trans[J]. Plant Cell, 1990, 2 (4):279-289. |
| [6] | Jorgensen R, Cluster P, English J, et al. Chalcone synthase cosuppression phenotypes in petunia flowers :comparison of sense vs. antisense constructs and single-copy vs. complex T-DNA sequences[J]. Plant Mol Biol, 1996, 31(5):957-973. |
| [7] | Cogoni C, Romano N, Macino G. Suppression of gene expression by homologous transgenes[J]. Antonie Van Leeuwenhoek, Leeuw Int J G, 1994, 65(3):205-209. |
| [8] | Guo S, Kemphues KJ. Par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed[J]. Cell, 1995, 81(4):611-620. |
| [9] | Cogoni C, Irelan JT, Schumacher M, et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation[J]. Embo J, 1996, 15(12):3153-3163. |
| [10] | Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans[J]. Nature, 1998, 391(6669):806-811. |
| [11] | 魏敏. RNA 干扰及其应用[J]. 科技与企业, 2011(11): 129. |
| [12] | 胡大林, 庄志雄, 何云. RNA 干扰与基因沉默的分子机制研究 进展[J]. 中国职业医学, 2005, 32(4):46-48. |
| [13] | Hammond SM, Boettcher S, Caudy AA, et al. Argonaute 2, a link between genetic and biochemical analyses of RNAi[J]. Science, 2001, 293(5532):1146-1150. |
| [14] | Dugas DV, Bartel B. MicroRNA regulation of gene expression in plants[J]. Curr Opin Plant Biol, 2004, 7(5):512-520. |
| [15] | Dang YK, Yang QY, Xue ZH, et al. RNA interference in fungi : pathways, functions, and applications[J]. Eukaryot Cell, 2011, 10(9):1148-1155. |
| [16] | Cerutti H, Ma XR, Msanne J, et al. RNA-mediated silencing in algae :biological roles and tools for analysis of gene function[J]. Eukaryot Cell, 2011, 10(9):1164-1172. |
| [17] | He L, Hannon GJ. MicroRNAs :Small RNAs with a big role in gene regulation[J]. Nat Rev Genet, 2004, 5(7):522-531. |
| [18] | 樊荣辉, 武治印, 王永强, 等. RNAi 技术及其在真菌基因功能 研究中的应用[J]. 菌物学报, 2010, 29(3):315-320. |
| [19] | Zamore PD. RNA interference :listening to the sound of silence[J]. Nat Struct Biol, 2001, 8(9):746-750. |
| [20] | Harborth J, Elbashir SM, Bechert K, et al. Identification of essential genes in cultured mammalian cells using small interfering RNAs[J]. J Cell Sci, 2001, 114(24):4557-4565. |
| [21] | Nykänen A, Haley B, Zamore PD. ATP requirements and small interfering RNA structure in the RNA interference pathway[J]. Cell, 2001, 107(3):309-321. |
| [22] | Randall G, Grakoui A, Rice CM. Clearance of replicating hepatitis C virus replicon RNAs in cell culture by small interfering RNAs[J]. Proc Natl Acad Sci USA, 2003, 100(1):235-240. |
| [23] | Verma NK, Dey CS. RNA-mediated gene silencing :mechanisms and its therapeutic applications[J]. J Clin Pharm Ther, 2004, 29 (5):395-404. |
| [24] | Mahmood-Ur-Rahman, Ali I, Husnain T, et al. RNA interference : The story of gene silencing in plants and humans[J]. Biotechnology Advances, 2008, 26(3):202-209. |
| [25] | 侯义龙. RNA 干扰及其在植物上的应用研究进展与展望[J]. 信阳师范学院学报:自然科学版, 2010, 23(2):316-320. |
| [26] | Brantl S. Antisense-RNA regulation and RNA interference[J]. Biochimica et Biophysica Acta(BBA)- Gene Structure and Expression, 2002, 1575(1-3):15-25. |
| [27] | Lipardi C, Wei Q, Paterson BM. RNAi as random degradative PCR :siRNA primers convert mRNA into dsRNAs that are degraded to generate new siRNAs[J]. Cell, 2001, 107(3): 297-307. |
| [28] | McManus MT, Sharp PA. Gene silencing in mammals by small interfering RNAs[J]. Nat Rev Genet, 2002, 3(10):737-747. |
| [29] | Davenport RJ. A faster way to shut down genes[J]. Science, 2001, 292(5521):1469-1471. |
| [30] | 陈莉. 核酸干扰技术的应用前景[J]. 医学研究生学报, 2008, 21(10):1009-1010. |
| [31] | Singh N, Rajam M. RNA Interference and functional genomics in fungi[M]//Microorganisms in Sustainable Agriculture and Biotechnology. Springer Netherlands, 2012 :773-792. |
| [32] | Matityahu A, Hadar Y, Dosoretz CG, et al. Gene silencing by RNA interference in the white rot fungus Phanerochaete chrysosporium[J]. Applied and Environmental Microbiology, 2008, 74(17):5359-5365. |
| [33] | Liu H, Wang P, Gong G, et al. Morphology engineering of Penicillium chrysogenum by RNA silencing of chitin synthase gene[J]. Biotechnol Lett, 2013, 35(3):423-429. |
| [34] | Nakayashiki H, Hanada S, Quoc NB, et al. RNA silencing as a tool for exploring gene function in ascomycete fungi[J]. Fungal Genet Biol, 2005, 42(4):275-283. |
| [35] | Mouyna I, Henry C, Doering TL, et al. Gene silencing with RNA interference in the human pathogenic fungus Aspergillus fumigatus[J]. FEMS Microbiol Lett, 2004, 237(2):317-324. |
| [36] | Carneiro JS, de la Bastide PY, Hintz WE. Regulated gene silencing in the fungal pathogen Ophiostoma novo-ulmi[J]. Physiological and Molecular Plant Pathology, 2013, 82 :28-34. |
| [37] | Moazeni M, Khoramizadeh MR, Kordbacheh P, et al. RNAmediated gene silencing in Candida albicans :Inhibition of hyphae formation by use of RNAi technology[J]. Mycopathologia, 2012, 174(3):177-185. |
| [38] | Chi JT, Chang HY, Wang NN, et al. Genomewide view of gene silencing by small interfering RNAs[J]. Proc Natl Acad Sci USA, 2003, 100(11):6343-6346. |
| [39] | Thompson JD. Applications of antisense and siRNAs during preclinical drug development[J]. Drug Discov Today, 2002, 7 (17):912-917. |
| [40] | Caracuel-Rios Z, Talbot NJ. Silencing the crowd :high-throughput functional genomics in Magnaporthe oryzae[J]. Mol Microbiol, 2008, 68(6):1341-1344. |
| [41] | 粟挺, 刘爱玲, 陈信波. RNA 干扰载体构建方法的研究进 展[J]. 湖南农业科学, 2011(19):1-4. |
| [42] | Bachman J. Site-directed mutagenesis[M]// Lorsch J. Laboratory Methods in Enzymology :DNA. UD, USA :Academic Press, 2013, 529 :241-248. |
| [43] | Hofer U. Techniques and application RNAi‘ off-targets’ pathogen infection[J]. Nature Reviews Microbiology, 2014, 12(5): 314-315. |
| [44] | Oliveira-Garcia E, Deising HB. Infection structure-specific expression of beta-1, 3-glucan synthase is essential for pathogenicity of Colletotrichum graminicola and evasion of beta-glucan-triggered immunity in Maize[J]. Plant Cell, 2013, 25(6):2356-2378. |
| [45] | Panwar V, McCallum B, Bakkeren G. Host-induced gene silencing of wheat leaf rust fungus Puccinia triticina pathogenicity genes mediated by the Barley stripe mosaic virus[J]. Plant Molecular Biology, 2013, 81(6):595-608. |
| [46] | Wang YD, Yang PC, Cui F, et al. Altered immunity in crowded locust reduced fungal(Metarhizium anisopliae)pathogenesis[J]. PLoS Pathogens, 2013, 9(1):e1003102. |
| [47] | Barad S, Horowitz SB, Kobiler I, et al. Accumulation of the mycotoxin patulin in the presence of gluconic acid contributes to pathogenicity of Penicillium expansum[J]. Molecular Plant- Microbe Interactions, 2013, 27(1):66-77. |
| [48] | Carreras-Villasenor N, Esquivel-Naranjo EU, Manuel Villalobos- Escobedo J, et al. The RNAi machinery regulates growth and development in the filamentous fungus Trichoderma atroviride[J]. Molecular Microbiology, 2013, 89(1):96-112. |
| [49] | Liu H, Cottrell TR, Pierini LM, et al. RNA interference in the pathogenic fungus Cryptococcus neoformans[J]. Genetics, 2002, 160(2):463-470. |
| [50] | Krajaejun T, Gauthier GM, Rappleye CA, et al. Development and a application of a green fluorescent protein sentinel system for identification of RNA interference in Blastomyces dermatitidis illuminates the role of septin in morphogenesis and sporulation[J]. Eukaryot Cell, 2007, 6(8):1299-1309. |
| [51] | Rappleye CA, Engle JT, Goldman WE. RNA interference in Histoplasma capsulatum demonstrates a role for alpha(- 1, 3)-glucan in virulence[J]. Mol Microbiol, 2004, 53(1):153-165. |
| [52] | Lawrence GJ, Dodds PN, Ellis JG. Transformation of the flax rust fungus, Melampsora lini :selection via silencing of an avirulence gene[J]. Plant J, 2010, 61(2):364-369. |
| [53] | Liu H, Wang P, Hu YH, et al. Construction of an RNAi expression vector and transformation into Penicillium chrysogenum[J]. Annals of Microbiology, 2014, 64(1):113-120. |
| [54] | Singh S, Braus-Stromeyer S, Timpner C, et al. Silencing of Vlaro2 for chorismate synthase revealed that the phytopathogen Verticillium longisporum induces the cross-pathway control in the xylem[J]. Appl Microbiol Biotechnol, 2010, 85(6):1961-1976. |
| [55] | Nguyen QB, Kadotani N, Kasahara S, et al. Systematic functional analysis of calcium-signalling proteins in the genome of the riceblast fungus, Magnaporthe oryzae, using a high-throughput RNAsilencing system[J]. Mol Microbiol, 2008, 68(6):1348-1365. |
| [56] | Nguyen QB, Itoh K, Van Vu B, et al. Simultaneous silencing of endo-β-1, 4 xylanase genes reveals their roles in the virulence of Magnaporthe oryzae[J]. Mol Microbiol, 2011, 81(4):1008- 1019. |
| [57] | Mu DS, Shi L, Ren A, et al. The development and application of a multiple gene co-silencing system using endogenous URA3 as a reporter gene in Ganoderma lucidum[J]. PLoS One, 2012, 7(8): e43737. |
| [58] | Khatri M, Rajam MV. Targeting polyamines of Aspergillus nidulans by siRNA specific to fungal ornithine decarboxylase gene[J]. Medical Mycology, 2007, 45(3):211-220. |
| [59] | Whisson SC, Avrova AO, Van West P, et al. A method for doublestranded RNA-mediated transient gene silencing in Phytophthora infestans[J]. Mol Plant Pathol, 2005, 6(2):153-163. |
| [60] | Nunes CC, Dean RA. Host-induced gene silencing :a tool for understanding fungal host interaction and for developing novel disease control strategies[J]. Mol Plant Pathol, 2012, 13(5): 519-529. |





