天然吩嗪类衍生物是一类对环境安全、对植物病原真菌具有广谱抑菌活性的微生物源抗生物质[1]。吩嗪类衍生物种类繁多,主要包括吩嗪-1-羧酸(phenazine-1-carboxylic acid,PCA)、1-羟基-吩嗪(phenazine-1-hydroxy)、2-羟基-吩嗪(phenazine- 2-hydroxy,2-OH-PHZ)、吩嗪-1-甲酰胺(phenazine- 1-carboxamide,PCN)等[2]。合成吩嗪类衍生物的微生物主要是假单胞菌和链霉菌,绿针假单胞菌被公认为是一种安全、有效的植物根际促生菌,除了良好的抑菌作用,还能促进作物生长,目前仍然没有关于其致病性的相关报道,可以认为其是一种环境友好的促生菌[3, 4, 5]。据报道,许多绿针假单胞菌能够分泌多种具有抑菌活性的次级代谢产物,例如:吩嗪类衍生物、藤黄绿脓菌素、硝吡咯菌素、氢氰酸以及杀虫蛋白P.fluroscentinsect toxin D (FitD)等[6, 7]。其中,吩嗪类衍生物的分泌是其最为重要的抑菌机制之一。在前期的研究报道中,已阐明了绿针假单胞菌中吩嗪生物合成机理,发现了多个参与吩嗪合成的调控因子,初步构建了吩嗪的合成调控网络,这为利用微生物高效合成吩嗪类物质打下了良好的基础。但吩嗪类物质的产量仍然较低,影响了其作为生物农药的开发前景,因此,研发吩嗪类物质的高产菌株十分迫切。
Pseudomonas chlororaphis HT66是由本实验室从上海郊区水稻根际分离所得。其除了保留绿针假单胞菌的普遍性质外,更具有两大优点:一是其分泌的吩嗪类衍生物几乎全为PCN。据报道,在中性条件下,PCN的抑菌性远远高于PCA,为PCA的10倍[4],其他公开报道的绿针假单胞菌,如GP72、30-84以及O6等菌株,都产生多个吩嗪类衍生物,包括PCA、2-OH-PHZ[5, 8, 9, 10],这使得吩嗪类化合物的分离更为简便。二是在前期的实验中发现野生型HT66菌株,其PCN的产量高达400 mg/L,高于目前所报道的各假单胞菌野生型菌株的吩嗪产量,这提供了将其作为构建PCN高产菌株的研发优势。
在实验室前期对HT66进行化学诱变的实验中,通过多轮诱变最终得到P3株,其PCN产量达到了1 597 mg/L,较同一批次发酵的野生型而言产量提高了3倍[11]。经过全基因组测序,发现共有244个碱基发生了突变,其中有3个LuxR家族转录调控因子发生了突变。LuxR家族转录调控因子多与次级代谢产物调控相关[12]。在HT66野生株中,共有24个luxR家族调控因子,按照3个突变的luxR基因在基因组中出现的先后顺序排列,分别是luxR_13(M217_04406)、luxR_14(M217_04413)和luxR_19(M217_05833),其中luxR_19的第254个碱基由G突变为C,导致第85位氨基酸由Gly突变为Ala。通过氨基酸序列分析发现,在第85位氨基酸前后10-20个氨基酸位点均高度保守,与其活性密切相关。因此,luxR_19这个基因是否与吩嗪的合成相关,其第85位氨基酸的突变是否会对吩嗪合成调控产生影响,是我们首先关心的问题。同时,菌体的生长状况在一定程度上影响到次级代谢产物的合成,luxR_19是否会影响到菌株的生长状况,同样值得关注。
本研究通过分子操作手段,对luxR_19基因进行敲除,回补以及过表达,以研究luxR_19基因对HT66合成吩嗪能力的影响,进一步通过luxR_19(G85A)点突变菌株的构建,研究其对吩嗪合成的影响,旨在为进一步了解PCN高产的调控机制,构建高产PCN菌株打下良好的基础。 1 材料与方法 1.1 材料 1.1.1 菌株与质粒
本研究中所用到的菌株,质粒以及引物见表 1和表 2。野生型绿针假单胞菌HT66作为构建突变菌株的出发菌株。pK18mobsacB质粒用于得到不包含目的片段的基因片段。质粒pK18CL用于敲除luxR_19基因。质粒pK18CP用于在野生型HT66上获得点突变菌株。pME6032质粒用于构建回补以及过表达质粒。pME-luxR分别用于在野生型HT66中过表达luxR_19基因以及在HT66 △ luxR菌株中回补该基因。
Luria-Bertani (LB)培养基:10.0 g/L胰蛋白胨,5.0 g/L酵母提取物,10.0 g/L NaCl,固体培养基加入12.0 g/L琼脂。E.coli的培养温度为37℃,P.chlororaphis的培养温度为28℃。
King's Broth (KB)培养基:20.0 g/L胰蛋白胨,15.0 mL/L甘油,0.514 g/L K2HPO4,0.732 g/L MgSO4,固体培养基加入12.0 g/L琼脂,用于培养P.chlororaphis,培养温度为28℃。
抗生素浓度:对于E.coli:50 μg/mL kanamycin,15 μg/mL tetracycline;对于P.chlororaphis:50 μg/mL kanamycin,125 μg/mL tetracycline以及100 μg/mL Ampicillin。
泳动性(Swimming)实验培养基:10.0 g/L胰蛋白胨,5.0 g/L NaCl以及3.0 g/L琼脂糖。
从动性(Swarming)实验培养基:5.0 g/L葡萄糖,10.0 g/L胰蛋白胨,5.0 g/L酵母提取物和5.0 g/L琼脂。 1.2 方法 1.2.1 luxR_19无痕敲除菌株HT66CL的构建
设计两对引物LA (Xba I)-LB,LC-LD (Hind III)用于对luxR_19基因的敲除。利用LA-LB从HT66基因组上扩增M217_05833基因上游673 bp,利用LC-LD从HT66基因组上扩增luxR_19基因下游791 bp。利用LB与LC之间20 bp的同源片段,通过融合PCR将两段基因片段连接在一起。利用限制性内切酶Xba I和Hind III对获得的1.5 kb的融合片段进行酶切,将所获得的片段克隆至pK18mobsacB质粒中。所获得的质粒pK18CL首先被转入E.coli S17-1 (λpir),之后通过接合转移至P.chlororaphis HT66中,获得HT66C1菌株。将HT66C1菌株涂布于含有100 μg/mL Ampicillin (Amp)和50 μg/mL kanamycin (Kan)的LB平板上以筛选出单交换菌株,之后挑选单菌落涂布于含有15%蔗糖的LB平板上以获得双交换菌株。获得的菌株分别点种于含有50 μg/mL Kan的LB平板和无抗LB平板上,挑选在Kan 50平板上不生长,在无抗LB平板上生长的菌株,利用 PCR以及测序进行验证,最终得到无痕敲除luxR_19的菌株HT66CL。 1.2.2 luxR_19基因回补与过表达
利用引物LF (EcoR I)-LR (Xho I)从野生型HT66基因组中通过PCR扩增luxR_19基因,总长度为861 bp。利用限制性内切酶EcoR I和Xho I同时对扩增片段以及质粒pME6032进行酶切,连接得到pME-luxR质粒。将pME-luxR质粒分别电击转化至△ luxR以及HT66野生株中,得到回补以及过表达菌株。 1.2.3 点突变菌株HT66PM的构建
利用引物LF (EcoR I)-LR (Xho I)从诱变高产株P3基因组中利用PCR扩增出长达861 bp的基因片段,通过酶切连接获得pK18CP质粒。剩余操作同1.2.2,最后通过测序筛选得到luxR_19(G85A)点突变菌株HT66PM。 1.2.4 生长曲线的测定
挑取单菌落接种到5 mL LB液体培养基中,28℃,180 r/min过夜培养。利用分光光度计进行检测,检测波长为600 nm。将细菌悬浮液接种于60 mL KB培养基中,确保初始OD600=0.01,28℃,180 r/min培养。每株菌3个平行。 1.2.5 PCN产量的检测
用6 mol/L的HCl将发酵液调至pH2.0,加入9倍体积的乙酸乙酯,充分振荡混匀,6 000 r/min离心5 min分层,PCN转移至有机相中。取上层萃取液于通风橱中,使有机溶剂挥发干净。取适量乙腈溶解萃取物,HPLC检测发酵液中PCN产量。检测条件:流动相为乙腈-25 mmol/L乙酸铵,色谱柱为WondaSil C18-WR反相柱(5 μm; 4.6×250 mm,Shimadzu,Japan),检测波长254 nm,检测条件:0-2 min,8%乙腈-92% 25 mmol/L乙酸铵,2-20 min,乙腈浓度从8%升至60%,乙酸铵浓度从92%下降至40%,20-21 min,8%乙腈-92% 25 mmol/L乙酸铵。柱温30℃,流速1.0 mL/min。 1.2.6 泳动性(Swimming)和从动性(Swarming)的检测
挑取单菌落接种于LB液体培养基中28℃,180 r/min过夜培养。将细菌悬浮液稀释至OD600=0.1,取2 μL添加于置于平板中央的无菌滤纸片。28℃培养24 h。 1.2.7 qRT-PCR实验
由上海桑尼生物科技有限公司完成,所用引物如表格3所示。内参基因为 16S rRNA,以ΔΔCt值的方法分析基因的相对表达量。分别检测了HT66以及HT66CL两株菌的rpeA、phzR、phzI、phzE、rpoS、rpoD、psrA、gacA、gacS以及rsmE基因的相对表达量变化情况。
将luxR_19基因与NCBI数据库中的序列比对,结果(表 3和图 1)显示,luxR_19基因为转录调控因子,属于LuxR家族。比对还表明luxR_19基因与Pseudomonas protegens CHA0、Pseudomonas sp.UW4、Pseudomonas fluorescens Pf0-1三株菌的luxR序列相似度较高,在核苷酸水平达到了80%以上,而与Pseudomonas putida KT2440和Burkholderia cenocepacia J2315相比,相似度仅为69%。进一步的氨基酸序列分析结果显示,在第85位氨基酸前后的10-20个氨基酸序列均具有很高的同源性,高度保守,属于LuxR_19的活性区域。
 |
| 图 1 LuxR_19蛋白序列第74-107位氨基酸多序列比对图 |
成功构建了pK18CL与pK18PM质粒,利用热激转化导入S17菌株中,之后再通过接合转移导入HT66野生型中,经过两轮同源重组,最终提取基因组,PCR扩增后送上海华大基因公司测序,结果表明成功构建了重组菌株HT66CL和HT66PM。
构建pME-luxR质粒,利用电击转化分别导入HT66以及HT66CL菌株中,从含有四环素抗性的平板上挑取单克隆,提取质粒进行PCR,PCR扩增结果经测序表明HT66/pME-luxR和HT66CL/pME-luxR构建成功。 2.3 luxR_19基因敲除株HT66CL的生长影响
以HT66野生株为对照,同时测定luxR_19基因敲除株HT66CL与HT66WT生长曲线,在28℃,180 r/min的条件下检测48 h。结果(图 2)显示,HT66CL与HT66的生长情况十分相似,6 h以前处于适应期,约6 h进入对数期,到24 h以后,HT66CL与HT66均进入稳定期。由此可以推断,luxR_19基因的敲除对于HT66菌体生长情况而言,无较大影响。
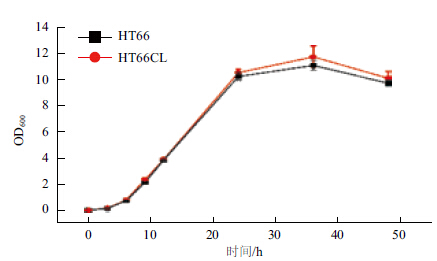 |
| 图 2 HT66与HT66CL生长曲线测定 |
同时,考察了HT66CL菌株的泳动性(Swimming)以及从动性(Swarming)变化。28℃条件下培养24 h,拍照记录菌落大小与形态。Swimming实验结果(图 3-A和图 3-B)显示,HT66与HT66CL菌株边缘均呈现锯齿状,菌落大小相近,无明显差异。Swarming实验结果(图 3-C和3-D)表明,HT66与HT66CL边缘不规则且界限模糊,HT66CL形成的生长圈大小明显大于HT66。luxR_19基因的敲除未对HT66的泳动能力产生较大影响,可能是由于其基因本身与控制泳动能力相关的基因无太多作用; luxR_19基因的敲除导致HT66从动能力的增加,说明其在一定程度上能够影响或者控制HT66的从动能力。
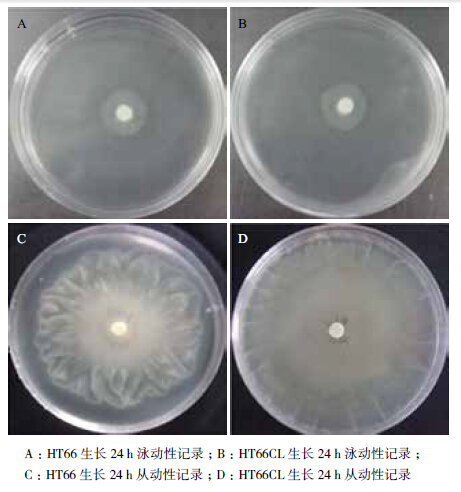 |
| 图 3 HT66与HT66CL的泳动性及从动性 |
将HT66、HT66CL以及HT66PM菌株发酵至36 h,萃取发酵液,利用HPLC检测PCN产量。结果(图 4)显示,野生型HT66菌株发酵至24 h,PCN产量为402.8 mg/L和473.5 mg/L。HT66CL发酵至24h,PCN产量为153.7 mg/L和198.9 mg/L。就PCN的产量而言,HT66CL比HT66下降了约50%。反观HT66PM,24 h的PCN产量为387.3 mg/L,整体而言虽然比HT66略有下降,但是下降幅度不明显,点突变后无明显变化。
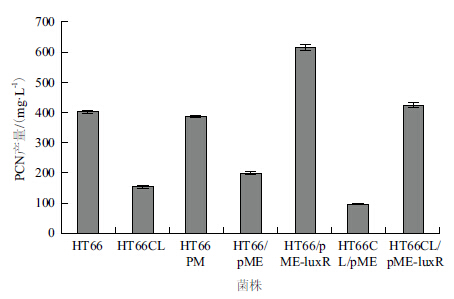 |
| 图 4 各菌株发酵24 h后的PCN产量 |
将HT66、HT66/pME、HT66/pME-luxR、HT66CL、HT66CL/pME以及HT66CL/pME-luxR菌株接种于大瓶KB培养基中发酵,培养至OD600=0.1,添加终浓度为1 mmol/L的IPTG,发酵至24 h,萃取发酵液,利用HPLC检测PCN产量,结果如图 4所示。首先是luxR_19的回补:HT66CL/pME-luxR在24 h PCN产量为475.3 mg/L,与HT66CL的153.4 mg/L相比有明显回升;较HT66的405.2 mg/L略高,说明通过luxR_19基因的回补,其产吩嗪能力回复到野生型;同时,HT66CL/pME-luxR的吩嗪产量明显高于HT66/pME的203.5 mg/L,是HT66/pME的2倍;一般来说,由于pME6032质粒较大,可能对菌株的生长带来一定影响,HT66CL/pME的PCN产量最低,仅有103.5 mg/L。然后是luxR_19基因的过表达,HT66/pME-luxR的PCN产量达到了623.7 mg/L,较HT66提高了约50%,较HT66/pME提高了2倍。 2.5 利用qRT-PCR检测相关基因表达水平
根据之前研究和文献报道,选取了参与吩嗪合成和调控吩嗪合成的重要基因,包括双元调控基因gacS/gacA,全局性调控基因rpeA、psrA、rsmE、rpoS,群体感应调控基因phzI/phzR,吩嗪合成必需基因phzE等进行qRT-PCR实验,以研究luxR_19基因的敲除对其产生的影响。以HT66野生型为参照对象,基因表达量的变化如表 4所示。
表 4显示,在HT66CL菌株中,直接参与吩嗪合成的基因phzI/phzR以及phzE基因,其表达水平都有一定的降低,其中phzI为野生型的0.822,phzR为野生型的0.739,phzE的表达量较phzI与phzR而言下降的较少,为野生型的0.901倍。这一结果与发酵结果,即PCN产量有一定下降所一致,从分子层面给上验证了luxR_19基因的敲除会导致吩嗪合成的降低。
从相关调控基因而言,HT66CL菌株的gacA的表达量显著降低,仅为野生型的0.593倍;而gacS的表达量基本维持在稳定的水平,为野生型的1.170倍。rpeA的表达量显著升高,为野生型的1.894倍,根据目前报道,rpeA能够在一定程度上抑制吩嗪类产物的合成[13],与本结果一致。对于两个sigma因子而言,rpoS的表达量保持稳定,rpoD有一定的下降,均与本研究的发酵结果一致。luxR_19基因的敲除导致rsmE与psrA基因表达量下降,分别为野生型的0.367与0.759,但根据相关文献,这两个基因均属于负调控基因。结果表明luxR_19能够影响多个基因的表达,应属于全局性调控基因。 3 讨论
根际促生菌株P.chlororaphis HT66是一株高产PCN的菌株,以HT66作为出发菌株,通过前期的菌种诱变,我们得到了PCN高产菌株P3,基于对突变株的全基因组比较分析,发现luxR_19发生了点突变。通过基因序列的比对,我们发现luxR_19在假单胞菌中具有较高同源性,进一步分析氨基酸序列,发现第85位氨基酸序列附近的氨基酸序列高度保守,处于luxR_19的活性区域。于是我们通过一系列的分子操作研究了luxR_19基因对其生长及吩嗪产量方面的影响。同时,利用点突变株HT66PM验证第85位氨基酸的改变对吩嗪产量的可能影响。
由于次级代谢产物的合成在很大程度上会受到菌株生长情况的影响,LuxR家族调控因子对于菌体生长的调控是多方面的,如通过控制群体感应,调节生物的从动能力以增加生物的环境适应性,如ppoR,通过调控相关代谢产物的合成以增加其环境竞争力等[14]。因此在将luxR_19基因敲除后,我们首先对其进行了生理生化能力的检测,包括测定生长曲线,菌体泳动性以及群体从动性3个指标。结果表明luxR_19基因的敲除不影响菌体的生长状况,但是会在一定程度上影响其群体从动性。在后期的工作中,可以尝试通过对该基因进行突变,从而达到增加群体从动性,确保菌株能够在自然环境中更有竞争力地定植于植物根部,从而促进合成PCN,更好地进行根际促生作用。
luxR_19基因敲除后,PCN产量有一定下降,在对其进行回补后,产量回到了正常水平,将该基因进行过表达后,产量较野生菌株有很大的提升。说明该基因确实对PCN的产量有一定影响,且对于 PCN的合成起正调控作用。通过qRT-PCR实验,我们检测了与PCN合成相关基因的表达水平,发现luxR_19基因的缺失直接导致gacA基因表达量明显降低,rpeA负调控因子表达量上升,并在一定程度上导致phz基因簇的表达情况。据此推测luxR_19对于PCN合成的调控作用可能是通过Gac系统来进行的,但具体的调控机制还有待进一步的研究。
通过点突变菌株的构建发现,该位点的点突变与P3菌株的高产无直接联系,也说明化学诱变株高产原因的复杂性。但无论是通过本实验或是相关报道,我们都能发现LuxR家族转录调控因子确实都参与到次级代谢产物的调控中。后期工作将继续分析鉴定P3菌株高产的影响位点,阐明P3菌株高产PCN的机制。
虽然HT66/pME-luxR较HT66而言,吩嗪产量仅仅提高了50%,但我们认为这是由于实验所选用的载体pME6032质粒较大,其本身会对菌体的生长带来一定影响。对比HT66/pME-luxR与HT66/pME两株菌的吩嗪产量,可以发现前者是后者的两倍,这足以展现luxR_19这一基因在提升吩嗪产量上的潜在能力。
在后续的实验中,我们可以通过以下两个方面来提升luxR_19在促进吩嗪生成方面的作用:选用拷贝数更加高的载体,减小对细菌生长的影响;对该基因作逐步的片段分析,发现其作用位点,从而直接在基因组上进行修饰,解除对载体的依赖,构建吩嗪高产菌株。同时,我们将对诱变株的高产原因作继续研究,揭示其高产机理。 4 结论
在Pseudomonas chlororaphisHT66菌株中:luxR_19对PCN的合成是正调控因子;luxR_19对细菌从动性有一定影响,而对菌体生长及泳动性无较大影响;诱变高产株P3的PCN产量提高不是由于luxR_19的碱基点突变造成。
| [1] | 张锋华,许煜泉,张雪洪.农用杀菌剂吩嗪-1-羧酸的生物合成与基因调控[J].农药研究与应用,2006(5):4-7. |
| [2] | 张云,胡洪波,彭华松,等.产PCA基因工程菌M18G反复补料分批培养研究[J].工业微生物,2007:16-20. |
| [3] | Han SH,Lee SJ,Moon JH,et al.GacS-dependent production of 2R,3R-butanediol by Pseudomonas chlororaphis O6 is a major determinant for eliciting systemic resistance against Erwinia carotovora but not against Pseudomonas syringae pv.tabaci in tobacco[J].Mol Plant-Microbe Interact,2006,19(8):924-930. |
| [4] | Chin-A-Woeng TF,Bloemberg GV,van der Bij AJ,et al.Biocontrol by phenazine-1-carboxamide-producing Pseudomonas chlororaphis PCL1391 of tomato root rot caused by Fusarium oxysporum f.sp. radicis-lycopersici[J].Mol Plant-Microbe Interact,1998,11(11): 1069-1077. |
| [5] | Liu H,He Y,Jiang H,et al.Characterization of a phenazineproducing strain Pseudomonas chlororaphis GP72 with broadspectrum antifungal activity from green pepper rhizosphere[J]. Curr Microbiol,2007,54(4):302-306. |
| [6] | Shen X,Hu H,Peng H,et al.Comparative genomic analysis of four representative plant growth-promoting rhizobacteria in Pseudomonas[J].BMC Genomics,2013,14(1):271. |
| [7] | Loper JE,Hassan KA,Mavrodi DV,et al.Comparative genomics of plant-associated Pseudomonas spp.:insights into diversity and inheritance of traits involved in multitrophic interactions[J].PLoS Genet,2012,8(7):e1002784. |
| [8] | Park J,Oh S,Anderson A,et al.Production of the antifungal compounds phenazine and pyrrolnitrin from Pseudomonas chlororaphis O6 is differentially regulated by glucose[J].Lett Appl Microbiol,2011,52(5):532-537. |
| [9] | Maddula V,Pierson E,Pierson L.Altering the ratio of phenazines in Pseudomonas chlororaphis (aureofaciens) strain 30-84:effects on biofilm formation and pathogen inhibition[J].J Bacteriol,2008,190(8):2759-2766. |
| [10] | Huang L,Chen MM,Wang W,et al.Enhanced production of 2-hydroxyphenazine in Pseudomonas chlororaphis GP72[J]. Appl Microbiol Biotechnol,2011,89(1):169-177. |
| [11] | 张平原,彭华松,张雪洪.高产吩嗪-1-甲酰胺的绿针假单胞菌的诱变与基因工程育种[J].上海交通大学学报:农业科学版,2015,33(2):90-94. |
| [12] | He W,Lei J,Jiu Y,et al.The LuxR family members gdmri and gdmrii are positive regulators of geldanamycin biosynthesis in Streptomyces hygroscopicus 17997[J].Arch Microbiol,2008, 189(5):501-510. |
| [13] | Whistler CA,Pierson III LS.Repression of phenazine antibiotic production in Pseudomonas aureofaciens strain 30-84 by RpeA[J].J Bacteriol,2003,185(13):3718-3725. |
| [14] | Fernández-piñar R,Cámara M,Soriano MI,et al.Ppor,an orphan luxr-family protein of Pseudomonas putida KT2440, modulates competitive fitness and surface motility independently of n-acylhomoserine lactones[J].Environmental Microbiology Reports,2011,3(1):79-85. |








