2.曲靖师范学院生物资源与环境科学学院,曲靖 655011
2. College of Biological Resource and Environmental Science,Qujing Normal University,Qujing 655011
小桐子(Jatropha curcas L.)属大戟科麻疯树属能源植物[1, 2],原产美洲,现广泛分布于全球的热带地区,在中国主要集中在四川、云南、广西、海南、福建等省区[3]。小桐子种子油含量高达40%-60%,流动性好,品质优良,是理想的柴油替代品[4, 5]。同时,小桐子还是干热河谷地区保水固土、防止沙化、增加有机质、构建防护林的优良树种[6],极具开发与利用价值。
在植物中,棉子糖系列寡糖(Raffinose family oligosaccharides,RFO)是由不同数量的半乳糖基通过α-1,6-糖苷键连接到蔗糖中葡萄糖基的6位羟基上形成的,根据连接半乳糖基的数量不同,主要包括:棉子糖(含1个半乳糖基)、水苏糖(含2个半乳糖基)和毛蕊花糖(含3个半乳糖基)。近年来的研究表明棉子糖系列寡糖的代谢在植物生长发育、逆境胁迫反应中发挥重要作用,尤其在种子成熟脱水过程中可以维持多种蛋白质的稳定性,更为重要的是寡糖可以促进种子细胞的玻璃化、避免脱水对细胞的伤害[7]。同时,棉子糖系列寡糖在植物冷驯化中也起到重要作用,很多研究已经证实低温导致植物叶片和种子中淀粉含量下降,而蔗糖和棉子糖系列寡糖含量升高。棉子糖系列寡糖的积累,特别是位于叶绿体外的寡糖,其通过储存碳源、渗透调节以及为植物提供低温保护剂等功能,对于植物抵抗低温环境有着重要帮助[8]。在棉子糖系列寡糖合成途径中,肌醇半乳糖苷(Galaetinol)是目前所知的唯一的半乳糖基供体,肌醇半乳糖苷合成酶(Galaetinol synthase,GS,EC 2.4.l.123)催化肌醇半乳糖苷的合成,被认为是棉子糖系列寡糖合成的关键酶,其催化反应是寡糖积累的限速步骤。我们先前通过RNA-Seq技术获得了小桐子低温锻炼下的转录组和数字基因表达谱数据,从中筛选到在低温锻炼过程中差异表达变化非常显著的小桐子肌醇半乳糖苷合成酶3基因[9, 10]。在此基础上,本研究进一步克隆其全长cDNA序列,并对其进行比较完整的生物信息学分析和转基因酵母的抗冷性功能初步验证,以期为研究可溶性糖在小桐子抗冷性形成中的作用机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 实验材料及处理供试小桐子种子取自云南省楚雄州元谋县。选取饱满的小桐子种子,用1.5% CuSO4消毒20 min,无菌水漂洗5次,于26℃的恒温培养箱中吸涨24 h[11]。将吸涨的种子在无菌水中漂洗3次,播于垫有5层用无菌水湿润滤纸的白磁盘中,于相对湿度(RH)75%、26/20℃、16/8 h光周期的恒温培养箱中萌发5 d。将发芽的种子播于消毒的培养土中,并于同样设置条件下的恒温培养箱中生长15 d至第2片真叶展开,每天用无菌水润湿培养土。将生长15 d的小桐子幼苗置于相对湿度(RH)75%、12℃、16/8 h光周期的低温培养箱中进行低温锻炼处理,分别取低温锻炼12、24和48 h与对照(正常培养)的第2片真叶,用铝箔纸包好,液氮速冻后保存于-80℃冰箱中用于RNA的提取。
1.1.2 菌株与主要试剂大肠杆菌Trans1-T1(DH-5α)、BL21(DE3)及酵母菌INVSc1菌株由本实验室保存;TransZol Up、DNase I、TransStart Taq DNA Polymerase、氨苄青霉素(Amp)、X-gal、IPTG、2×Easy Taq PCR SuperMix(+dye)、TransScript Two-Step RT-PCR SuperMix、EasyPure Quick Gel Extraction Kit、EasyPure Plasmid MiniPrep Kit、pEASY-T1 Cloning Kit、pEASY-E1 Expression Kit、Trans 2K Plus II DNA Marker购自北京全式金生物技术有限公司;BamH I、Xho I限制性内切酶购自大连宝生物公司;引物合成和测序由深圳华大基因有限公司完成。
1.2 方法 1.2.1 总RNA的提取及cDNA第一链的合成利用TransZol Up试剂提取小桐子12℃低温锻炼24 h叶片的总RNA,并利用DNase I消化RNA中的基因组DNA,得到纯化的总RNA。以Anchored Oligo(dT)18为逆转录引物,利用TransScript Two-Step RT-PCR SuperMix合成第一链cDNA。
1.2.2 小桐子JcGS3基因全长cDNA的克隆我们先前获得了小桐子低温锻炼转录组和数字基因表达谱数据,从中发现低温下差异表达变化非常显著的小桐子肌醇半乳糖苷合成酶3基因(CL2440.Contig2_JC-CK_1A)[9]。该基因序列长1 463 bp,包含完整ORF编码框(1 008 bp)。以此序列为基础设计全长扩增引物(上游F:5'-TTCTTCTTTCCAAACTCG-3';下游R:5'-TATAATCCACAACCACAT-3'),并送深圳华大基因公司合成。
以1.2.1反转录的cDNA为模板,使用热启动双封闭DNA聚合酶TransStart Taq DNA Polymerase进行PCR扩增,扩增条件为:94℃ 5 min;94℃ 30 s,50.7℃ 30 s,72℃ 1.5 min,35个循环;72℃ 10 min。扩增完成后用1%的琼脂糖凝胶电泳检测PCR产物,利用EasyPure Quick Gel Extraction Kit从琼脂糖凝胶回收目的基因条带(1 053 bp)。将目的基因片段与克隆载体pEASY-T1连接,转化大肠杆菌Trans1-T1感受态细胞,涂LB抗性平板(LB+Amp+IPTG+X-gal),过夜生长,进行蓝白斑筛选。挑取白斑克隆,进行菌落PCR验证,重组质粒命名为pEASY-T1-JcGS3,送深圳华大基因公司利用pEASY-T1质粒上的M13F与M13R通用引物进行双向测序。
1.2.3 小桐子JcGS3基因的生物信息学分析对于小桐子JcGS3基因的推导蛋白,利用在线工具ProtParam计算蛋白质的理论分子量、等电点等基本参数;利用WolfSport在线软件对蛋白质亚细胞定位进行预测;利用SignalP 3.0 Server分析蛋白质信号肽序列;利用在线工具TMHMM与Proscale检测其跨膜结构与亲水/疏水特性;利用NCBI CDD工具进行结构域与功能元件的鉴定;利用Swiss-Model与Phyre2进行蛋白质三维结构的同源建模,利用VMD软件显示其三维空间结构,并结合Ramachandram图验证其准确性。从NCBI下载其它物种的GS氨基酸序列,利用ClustalX进行序列相似性比对,然后用MEGA4.0软件通过邻接法构建系统进化树,并采用泊松法进行检验。利用Spidey软件将克隆的JcGS3 cDNA序列与其对应基因组序列(小桐子基因组数据库http://www.kazusa.or.jp/jatropha/)进行比对以确定基因内含子与外显子的结构,同时利用在线软件PlantCARE与PLACE进行启动子顺式作用元件的分析。
1.2.4 小桐子JcGS3基因酵母表达载体的构建、转化及功能验证利用BamH I与Xho I双酶切质粒pEASY-T1-JcGS3与酵母表达载体pYES2,切胶回收JcGS3片段与pYES2的酶切大片段,T4 DNA连接酶22℃连接5 h,转化大肠杆菌DH5α感受态细胞,涂LB+Amp平板,过夜生长,经菌落PCR验证的阳性克隆命名为pYES2-JcGS3。提取重组酵母表达质粒pYES2-JcGS3,转化酿酒酵母野生型菌株INVSc1,命名为INVSc1-pYES2-JcGS3;同时将对照空载体pYES2质粒也转化INVSc1,命名为INVSc1-pYES2;通过酵母菌落PCR验证以确定阳性克隆。
分别挑取重组酵母INVSc1-pYES2-JcGS3及对照INVSc1-pYES2单菌落,接种于15 mL YPD培养基中,30℃振荡过夜培养。用YPD培养基调至OD600=0.4-0.6,取20 mL菌液于8 000 r/min离心1 min,弃上清,用20 mL YPG诱导培养基重悬菌体,30℃诱导表达24-36 h至OD600>2.0。取诱导表达菌液2 mL至60 mL YPG诱导培养基中,调菌液OD600=0.2,在正常30℃条件下或18℃低温条件下继续振荡培养,每3 h间隔取样,测定样品的OD600值。以时间为横坐标,OD600为纵坐标,绘制酵母菌的生长曲线,比较重组酵母菌与对照酵母菌在正常和低温条件下的生长差异。
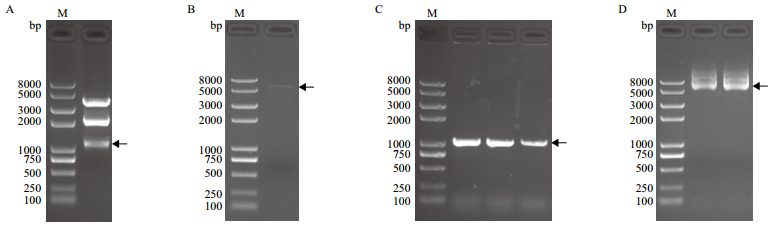
2 结果 2.1 小桐子JcGS3基因全长cDNA的克隆以小桐子12℃低温锻炼24 h的叶片材料提取总RNA,并反转录成cDNA为模板,利用JcGS3上下游引物扩增JcGS3基因的全长cDNA。结果表明,扩增产物约1.1 kb,与预期大小一致(图 1-A)。将目的条带切胶回收后与T/A克隆载体pEASY-T1连接、转化大肠杆菌,并经菌落PCR鉴定(图 1-B),阳性克隆命名为pEASY-T1-JcGS3,接菌提取其质粒DNA(图 1-C)。
 |
| A:PCR扩增;B:菌落PCR鉴定;C:重组质粒pEASY-T1-JcGS3 图 1 小桐子JcGS3基因的cDNA克隆 |
经过测序,克隆的JcGS3基因cDNA序列全长为1 053 bp,已提交至NCBI GenBank(登录号KJ670151.1)。利用NCBI ORFfinder分析表明其包含完整的开放阅读框(ORF)1 008 bp,编码一个由335个氨基酸(aa)组成的蛋白(图 2),其理论分子量推测为38 kD、等电点为5.44。SOPM服务器分析该蛋白的二级结构,α-螺旋109 aa,占32.54%;β-折叠76 aa,占22.69%;β-转角33 aa,占9.85%;无规则卷曲117 aa,占34.93%。预测分析该蛋白质没有信号肽、疏水区域及跨膜区的存在,说明该蛋白质属于可溶性胞质蛋白,这与PSORT预测该蛋白质主要定位在细胞质中相一致。
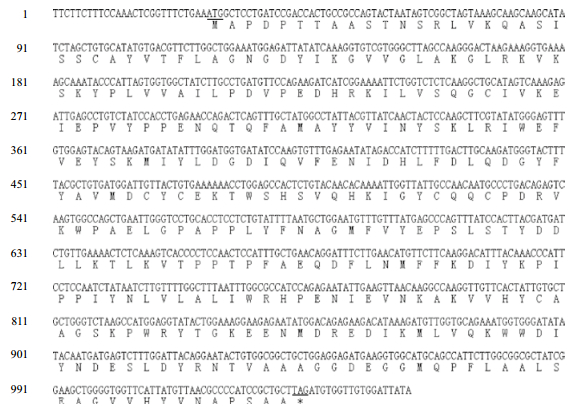 |
| 起始密码子(ATG)与终止密码子(TAG)用下划线标注 图 2 小桐子 JcGS3 cDNA 与推导的氨基酸序列 |
利用克隆得到的JcGS3 cDNA序列,对小桐子基因组数据库(http://www.kazusa.or.jp/jatropha/)进行在线Blast检索,得到其对应的基因组序列,发现JcGS3基因包含4个外显子与3个内含子。同时,也获取了该基因ORF上游1 000 bp的启动子序列,利用在线软件PlantCARE与PLACE对其进行了顺式作用元件分析发现,启动子区域转录因子结合位点较多,调控特性也很复杂(图 3)。具备多个TATA-box,TATA-box是RNA聚合酶II结合位点,保证转录的精确起始,并调控上游激活蛋白的作用;同时,其上游还包含多个CAAT-box,CAAT-box主要控制转录起始的频率并增强转录活性。以上两个顺式作用元件是启动子、增强子区域常见的作用元件,小桐子JcGS3基因均具有。另外,还鉴定到植物特有的CRT/DRE冷响应元件(CCGAC),以及脱落酸(ABA)应答元件,如ABRE(PyCGTGGC)、MYB(PyAACT/GC)和MYC(CANNTG)。除此以外,还鉴定出的元件,包括ASF、CCAAT-box、GATA-box、W-box等顺式作用元件。以上充分说明小桐子JcGS3基因是受多重信号系统的调节。
 |
| 图 3 小桐子 JcGS3 基因启动子的顺式作用元件 |
通过与NCBI中不同植物GS氨基酸序列进行比对分析(图 4-A)发现,GS氨基酸序列在N端和C端变化差异较大,而中间部位与C末端(PSAA)的氨基酸残基则较为保守。小桐子GS3中存在一个DxD基序(DGD,Asp121-Gly122-Asp123位),该基序是此类蛋白质的共同基序,也是Ca2+、Mn2+的结合位点(Asp121、Asp123、His258)。在N端还发现一个β-α-β超二级结构(图 4中用M标识),这也是该类蛋白催化所必须的。进一步通过CDD工具进行保守结构域预测分析(图 4-B)发现,JcGS3酶蛋白包含RfaJ结构域,该结构域具有脂多糖的合成活性及葡萄糖基转移酶活性,同时还鉴定到GAT结构域。小桐子JcGS3属于GT8糖基转移酶家族,该家族中除了转移葡萄糖残基外,还可以转移半乳糖残基,另外具有糖链分支的能力,负责形成脂多糖、寡聚糖等。通过CDD比对已经测定结构的GS3蛋白,推测小桐子GS3蛋白的催化活性中心由19个氨基酸残基组成(图 4中用*号标注),与文献报道的一致。
 |
| 双横线表示“ β-α-β”超二级结构(M1、M2、M3);组成活性中心的 19 个氨基酸残基用“*”表示;DxD 基序用单横线表示;Ca2+、Mn2+ 结合位点用“#”表示;涉及的物种:沙冬青(Ammopiptanthus mongolicus),拟南芥(Arabidopsis thaliana),牛耳草(Boea hygrometrica),甘蓝型油菜(Brassica napus),茶树(Camellia sinensis),辣椒(Capsicum annuum),欧洲板栗(Castanea sativa),阿拉伯咖啡(Coffea Arabica),大豆(Glycine max),小桐子(Jatropha curcas),木薯(Manihot esculenta),紫花苜蓿(Medicago sativa),毛果杨(Populus trichocarpa),丹参(Salvia miltiorrhiza),马铃薯(Solanum commersonii),番茄(Solanum lycopersicum),紫毛蕊花(Verbascum phoeniceum),复活草(Xerophyta viscose),玉米(Zea mays),下同 图 4 小桐子 JcGS3 推导氨基酸序列与其它植物 GS 的多重序列比对(A) 及其 CDD 预测(B) |
为了进一步阐明植物GS的进化关系,通过ClustalW软件对不同植物GS氨基酸序列进行了更广泛的多重比对分析,并使用MEGA软件以邻接法构建了其系统进化树(图 5)。生成的进化树分成两大主要分支:在分支I中,单子叶植物与双子叶植物都有;而在分支II中,全部是双子叶植物,小桐子JcGS3蛋白也归为II类。部分物种GS家族中的不同成员出现在不同大分支中,如:拟南芥在两种类型中基本数量一致,而与小桐子在进化上较近的毛果杨,有很大一部分分布于I类型中。这些结果说明植物GS在进化过程中不是平行渐进分化的,而是表现出不同程度的跳跃进化。
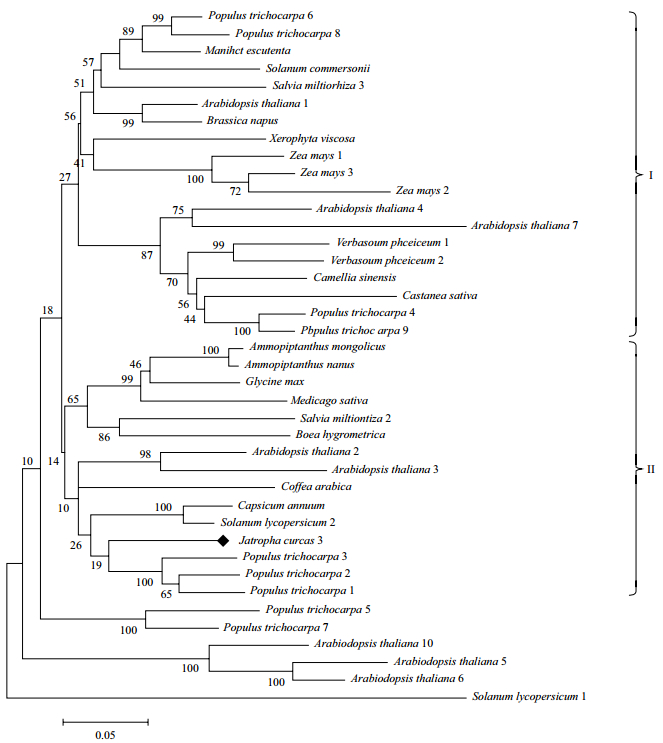 |
| 图 5 通过 ClustalW 序列比对和 MEGA4.0 邻接法构建的植物 GS3 进化树 |
根据已经测定的人类GS蛋白的晶体结构进行小桐子GS3蛋白质三维结构的同源建模,发现该蛋白质不仅在一级氨基酸序列非常保守,而且在空间结构上更加保守:核心7条基本平行的β-折叠构成一个保守结构域,周围则由5段进化上不太保守但不可或缺的α-螺旋围绕形成(图 6-A)。在β-折叠片层的中间凹陷部位被认为是该酶的催化中心部位,也是结合半乳糖与蔗糖的活性中心。
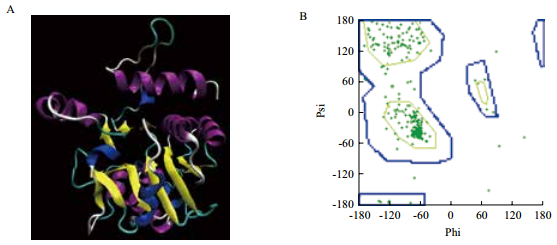 |
| A:三维结构卡通模型;B:Ramachandran能量图 图 6 同源建模得到的小桐子JcGS3蛋白三维空间结构 |
Ramachandran plots 是反映立体化学质量(Stereochemical quality)的参数。通过分析φ角和ψ 角的分布方式对模拟的三维结构是否与自然结构趋势相同进行评估,蓝线区域是最理想的φ 角和ψ角分布区域,而蓝线区域外部则为不合理区域。如果预测的蛋白质残基的二面角有90%以上位于蓝线区域,则表明其有稳定的空间结构。图 6-B显示,构建的小桐子GS3 蛋白三维结构的φ角和ψ角有95.8%位于Ramachandran 图中能量稳定区域,表明该结构是可靠的。
2.3 通过酵母表达初步验证小桐子JcGS3的功能将小桐子JcGS3基因通过BamH I与Xho I双酶切克隆到酵母表达质粒pYES2中,获得其重组酵母表达载体pYES2-JcGS3(图 7)。随后将其转化酵母菌INVSc1,利用JcGS3上下游引物进行酵母菌落PCR鉴定,结果(图 8)表明该重组酵母表达质粒已经成功转化酵母。
 |
| A:BamH Ⅰ与Xho Ⅰ双酶切目的基因片段;B:BamH Ⅰ与Xho Ⅰ双酶切pYES2;C:转化DH5α后菌落PCR验证;D:提取的重组质粒pYES2-JcGS3 图 7 pYES2-JcGS3重组质粒的构建 |
 |
| 图 8 转pYES2-JcGS3重组酵母INVSc1的菌落PCR验证 |
通过比较重组酵母菌INVSc1-pYES2-JcGS3与对照酵母菌INVSc1-pYES2的生长曲线(图 9)发现,在无胁迫条件下,两者的生长速率基本相似,且几乎不存在停滞期而直接进入对数生长期,在27 h时基本进入稳定平台期,这表明正常条件下JcGS3基因的表达对酵母菌生长几乎没有影响(图 9-A)。在18℃低温胁迫条件下,JcGS3重组酵母菌与对照酵母菌刚开始都出现了约3 h的生长停滞期,之后逐渐进入对数生长期;与对照相比,JcGS3重组酵母菌在生长的前6 h生长速率基本相同,之后到进入平台期的24 h内,JcGS3重组酵母菌的生长速率明显快于对照(图 9-B)。另外,JcGS3重组酵母菌在进入平台期后,对照酵母菌虽受低温胁迫,但仍保持生长的趋势,进入平台期的时间要稍晚3-6 h。该结果清楚表明JcGS3基因在酵母中过量表达能够显著增强酵母菌的低温抵抗能力,与抗冷性直接相关。
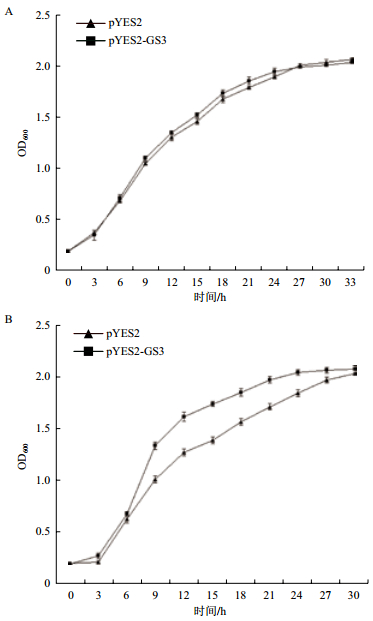 |
| 图 9 重组酵母菌INVSc1-pYES2-JcGS3与对照酵母菌INVSc1-pYES2在无胁迫(A)与18℃低温胁迫(B)条件下的生长曲线 |
植物在低温胁迫下,碳素同化总量下降,但可溶性糖增加,这样可以降低植物细胞的渗透势以抵抗逆境[12, 13]。此过程不仅在于可溶性糖参与细胞的渗透调节作用,更重要的原因可能在于许多可溶性糖是植物适应环境的信号物质[14]。淀粉分解与棉子糖系列寡糖合成代谢是诸多可溶性糖积累的枢纽。在拟南芥中,Kaplan等[15]通过代谢动力学研究表明,低温胁迫下第一个含量直接增加的碳水化合物是由淀粉经β-淀粉酶水解而来的麦芽糖和麦芽三糖,紧接着是6-磷酸葡萄糖和6-磷酸果糖,接下来是蔗糖、以及葡萄糖和果糖。另外,肌醇半乳糖、海藻糖、棉子糖以及水苏糖等低聚糖在之后的渗透调节中起更重要的作用[12]。而作为棉子糖系列寡糖合成中半乳糖基供体的肌醇半乳糖苷是此类低聚糖合成的关键底物,催化它生成的肌醇半乳糖苷合成酶(GS)已被认为是此代谢途径的限速关键酶。已报道的GS为分子量在36-38 kD的单体蛋白,活性最适pH为7-8,且活性可进一步被DDT(二硫苏糖醇)和低浓度的MnCl2增强。GS对其底物UDP-半乳糖和肌醇的米氏常数分别为0.16-1.8 mmol/L和4.0-6.5 mmol/L,对UDP-半乳糖和肌醇具有很强的专一性,而对ADP-Gal、GDP-Gal等糖类或肌醇的差向异构体均无作用[16]。
肌醇半乳糖苷合成酶属于多基因家族,已经报道在拟南芥[17]、紫花苜蓿[18]、番茄[19]、玉米[20]等植物中克隆到GS基因40多个。目前,在拟南芥中发现了10个编码GS的基因,其中有3个基因产物只存在于成熟后的种子中,AtGS1与AtGS2受干旱和高盐胁迫的诱导,但对低温没有响应,而AtGS3只在低温条件下被诱导表达[17]。玉米基因组分析表明它含有3个GS基因,主要在种子成熟组织中表达[20]。低温胁迫下,匍匐筋骨草的叶肉细胞和韧皮部细胞中GS1与GS2表达上调,很大程度上提高了其抗冷性[21]。Cunningham等[18]从紫花苜蓿(Medicago sativa)宿根中克隆得到一条长度为1 326 bp的GS全长cDNA,编码325个氨基酸。实验表明,未经低温处理的紫花苜蓿宿根中没有GS基因表达,而将植株转移至低温环境则开始转录,并且在2℃下持续2 d后达到最大表达量,转移至常温后,转录量再次减少直至消失。我们最近通过数字基因表达谱分析发现,小桐子肌醇半乳糖苷合成酶3基因(CL2440.Contig2_JC-CK_1A)在12℃低温锻炼12 h后表达上调近467.88倍[9],本研究中该基因JcGS3在酵母中过量表达能显著增强重组酵母菌的低温抵抗能力,推测该酶能在酵母中催化合成一系列起渗透调节作用的低聚糖类物质从而提高酵母的抗冷性。这些研究事实充分表明,GS与植物抗冷性密切相关。但是,植物GS作为一个大家族,只有部分GS基因的表达直接参与植物抗冷性形成,很多与之相关的抗逆性存在交叉性,即不同的GS基因可以被不同的逆境条件所诱导,同一逆境又可以同时诱导多种GS基因的表达,所以对于小桐子JcGS3基因进行交叉抗逆性及差异表达特性研究进而获得转基因小桐子株系是未来的研究方向。
4 结论本研究基于我们获得的小桐子低温锻炼下的转录组和数字基因表达谱数据,筛选到低温下差异表达变化较显著的小桐子肌醇半乳糖苷合成酶3基因JcGS3,并利用RT-PCR技术成功克隆到该基因。序列分析表明,其包含1 008 bp的开放阅读框,编码335个氨基酸,理论分子量为38 kD,等电点为5.44。生物信息学预测显示,该蛋白质没有信号肽、疏水区域及跨膜区的存在,主要定位在细胞质中;启动子序列中鉴定到了多个TATA框、CAAT框、CRT/DRE冷响应元件、脱落酸(ABA)应答元件;包含GT8糖基转移酶家族的典型RfaJ与GAT结构域。将JcGS3基因构建了酵母表达载体,发现该基因的表达能够显著提高其重组酵母菌的低温抵抗能力。
| [1] | 何璐, 虞泓, 范源洪, 等. 麻疯树(Jatropha curcas L. )植物学研究进展[J]. 长江流域资源与环境, 2010, 19:120-127. |
| [2] | 崔明昆, 林锦, 李红朝, 等. 云南的能源植物资源[J]. 云南师范大学学报:自然科学版, 2009, 29:39-46. |
| [3] | 刘杰, 李黔柱, 尹航, 等. 麻疯树植物资源的研究与开发利用进展[J]. 贵州大学学报:自然科学版, 2006, 23:105-109. |
| [4] | 邓志军, 程红焱, 宋松泉. 麻疯树种子的研究进展[J]. 云南植物研究, 2005, 27:605-612. |
| [5] | 王海燕, 文明富, 刘石生, 等. 麻疯树生物学研究进展及其开发利用[J]. 热带作物学报, 2010, 31:670-675. |
| [6] | 潘志贤, 纪中华, 方海东, 等. 金沙江干热河谷退化旱坡地小桐子造林关键技术[J]. 云南农业科技, 2008, 4:41-42. |
| [7] | Wang XF, Jing XM, Jian L, et al. Studies on membrane function and sugar components of ultradried seeds[J]. Acta Botanica Sinica, 2003, 45:23-31. |
| [8] | Imanishi HT, Suzuki T, Masuda K, et al. Accumulation of raffinose and stachyose in shoot apices of Lonicera caerulea L. during cold acclimation[J]. Scientia Horticulture, 1998, 72:255-263. |
| [9] | Wang HB, Zou ZR, Wang SS, et al. Global analysis of transcriptome responses and gene expression profiles to cold stress of Jatropha curcas L. [J]. PLoS ONE, 2013, 12:e82817. |
| [10] | Wang HB, Zou ZR, Wang SS, et al. Deep sequencing-based transcriptome analysis of the oil-bearing plant Physic Nut(Jatropha curcas L. )under cold stress[J]. Plant Omics Journal, 2014, 7:178-187. |
| [11] | 李忠光, 龚明. 不同化学消毒剂对小桐子种子萌发和幼苗生长的影响[J]. 种子, 2010, 30:4-7, 12. |
| [12] | Ruelland E, Vaultier MN, Zachowski A, et al. Cold signaling and cold acclimation in plants[J]. Advances in Botanical Research, 2009, 49:35-150. |
| [13] | Garcia AB, Engler JA, Iyer S. Effects of osmoprotectants on NaCl stress in rice[J]. Plant Physiol, 1997, 115:159-169. |
| [14] | Sheen J, Zhou L, Jiang JC. Sugar as signaling moleculaes[J]. Curr Opin Plant Biol, 1999, 2:410-418. |
| [15] | Kaplan F, Kopka J, Sung DY, et al. Transcript and metabolite profiling during cold acclimation of Arabidopsis reveals an intricate relationship of cold-regulated gene expression with modifications in metabolite content[J]. Plant Journal, 2007, 50:967-981. |
| [16] | Smith PT, Kuo TM, Crawford CG. Purification and characterization of galactinol synthase from mature Zucchini squash leaves[J]. Plant Physiol, 1991, 96:693-698. |
| [17] | Taji T, Ohsumi C, Iuchi S, et al. Important roles of drought- and cold-inducible gene fro galactinol synthase in stress tolerance in Arabidopsis thaliana[J]. The Plant Journal, 2002, 29:417-426. |
| [18] | Cuningham SM, Nadeau P, Castonguay Y, et al. Raffinose and stachyose accumulation, galactinol synthase expression, and winter injury of contracting alfalfa germplasms[J]. Crop Science, 2003, 43:562-570. |
| [19] | Downie B, Gurusinghe S. Expression of a galactinol synthase gene in tomato seeds is up-regulated before maturation desiccation and again after imbibition whenever radicle protrusion is prevented[J]. Plant Physiology, 2003, 131:1347-1359. |
| [20] | Zhao TY, Thacker R, Corum JW. Expression of the maize galactinol synthase gene family:Expression of two different genes during seed development and germination[J]. Physiologia Plantarum, 2004, 121:634-646. |
| [21] | Sprenger N, Keller F. Allocation of raffinose family oligosaccharides to transport and storage pools in Ajuga reptans:the roles of two distinct galactinol synthases[J]. The Plant Journal, 2000, 21:249-258. |




