鸡球虫病在世界范围内严重影响养鸡业健康发展,给养鸡业造成巨大的经济损失,该病主要是由几种艾美耳球虫寄生于鸡肠道引起。而在几种艾美耳球虫中,柔嫩艾美耳球虫(Eimeria tenella)是分布最广、致病力最强的球虫之一,常作为研究鸡球虫病的一种模式物种[1]。柔嫩艾美耳球虫寄生在绒毛上皮和盲肠的黏膜下,引起出血性肠炎,可导致鸡死亡,尤其对雏鸡危害最大。鸡球虫属于顶复器门原虫,顶复器原虫多数是专性细胞内寄生虫,都需要入侵宿主细胞,在宿主细胞内生长、发育、繁殖完成整个生活史,其共有的特征是具有由几种专门分泌蛋白的细胞器构成的顶端复合体,这些细胞器包括微线体、棒状体和致密颗粒,这些特殊细胞器分泌的蛋白参与了虫体入侵宿主细胞。在虫体入侵宿主细胞这一过程中,虫体黏附宿主细胞后,在虫体顶端和宿主细胞膜表面形成一个紧密的运动接触环(Moving Junction,MJ)[2]。对弓形虫、疟原虫等顶复器门原虫的研究表明,该结构对虫体成功入侵宿主细胞起至关重要的作用[3]。
研究发现由微线分泌的顶体膜抗原1(Apical membrance protein-1,AMA1)是组成MJ的关键分子,该分子与棒状体分泌的蛋白形成的复合体是MJ的主要成分[4]。由于MJ在顶复器原虫入侵宿主细胞过程中发挥重要作用,因此与顶膜抗原1相互作用的分子可能参与形成MJ,在虫体入侵宿主细胞过程中也是必不可少的。为了弄清楚柔嫩艾美耳球虫AMA1的相互作用分子以及在虫体入侵过程中发挥的作用,本实验室前期利用酵母双杂交技术,以柔嫩艾美耳球虫顶体膜抗原1(EtAMA1)为诱饵,筛选了柔嫩艾美耳球虫子孢子酵母双杂交cDNA文库,获得一些潜在的与EtAMA1相互作用蛋白的ESTs序列[5]。本研究选取其中编号为559的EST序列,运用RACE技术扩增获得该基因含完整开放阅读框(ORF)的全长cDNA序列。Blast分析发现该基因编码的蛋白是柔嫩艾美耳球虫的保守蛋白(conserved hypothetical protein),因此将该基因命名为EtCHP559。利用RT-PCR扩增其ORF区,构建该基因的真核表达重组质粒,并在真核细胞鸡胚成纤维细胞DF-1中进行表达,旨在为进一步研究该基因的功能奠定基础。
1 材料与方法 1.1 材料 1.1.1 虫株柔嫩艾美耳球虫纯种(CAAS211116-11),由中国农业科学院上海兽医研究所动物寄生虫病研究室保存提供。
1.1.2 菌种、细胞和载体大肠杆菌TOP10购自上海天根生物技术有限公司,真核细胞DF-1由本所丁铲老师馈赠,pGEM-T-easy vector购自美国Promega公司,真核表达载体pcDNA3.1-flag,由复旦大学上海医学院崔晓娴博士馈赠。
1.1.3 主要试剂DL2000(DNA Maker)、DNA胶回收试剂盒、质粒提取试剂盒、2*Taq PCR MasterMix,购自上海天根生物技术有限公司。Trizol、限制性内切酶BamH Ⅰ和Xho Ⅰ,购自大连宝生物工程有限公司。T4 DNA连接酶、购自美国Promega公司。GeneRacerTM Kit、Lipofectamine 2000,购自美国Invitrogen公司。胎牛血清、DMEM、opti-MEM,购自美国Gibco公司。近红外荧光羊抗兔二抗,购自LI-COR公司。RIPA裂解液购自上海碧云天生物技术有限公司。绿色荧光标记山羊抗兔二抗,购自Jackson Immuno Research公司。
1.2 方法 1.2.1 总RNA 的提取以纯化的柔嫩艾美耳球虫子孢子为材料按Trizol说明书操作,提取子孢子阶段虫体的Total RNA,经电泳和紫外分光光度计检测其浓度和纯度,符合要求后用于RACE模板的制备。
1.2.2 引物设计与合成 1.2.2.1 RACE 引物根据编号为559的EST序列,该序列含有3'端poly (A)尾,利用Primer Premier 5.00软件设计5'端的引物,RACE引物如下,5'Primer:5'-ACCTTCGACCGCTGCCGCTCGTGTTCCT-3';5'Nest Primer:5'-CCGCTGCCGCTCGTGTTCCTTTTCCC AT-3'。所有引物均由上海赛百盛生物有限公司合成。1.2.2.2全长cDNA扩增的引物将获得的5'-RACE扩增产物纯化回收后与pGEM-Teasy载体连接,转化TOP10感受态细胞,进行蓝白斑筛选,对PCR鉴定的阳性菌落进行测序,利用DNAstar软件查找5'-RACE PCR扩增产物的序列与原EST序列的重叠部分,将2段序列拼接为全长cDNA序列,根据拼接的全长cDNA序列,设计上下游引物进行RTPCR扩增含完整ORF的cDNA片段,在上下游引物引入酶切位点BamH Ⅰ和Xho Ⅰ,上游引物序列为5'-GGATCCATGCGATCCTCGCAGCGTCGGCGCC-3',下游引物序列为5'-CTCGAGTCGGCTTCATCTCCCGG CCCTTAC-3'。
1.2.3 EtCHP559 基因的克隆与测序 1.2.3.1 EtCHP559 全长基因5' 端的扩增与测序首先利用GeneRaceTMKit对提取的柔嫩艾美耳球虫子孢子总RNA依次进行去磷酸化反应、去mRNA帽结构、连接RNA Oligo、反转录,然后进行cDNA的5'端扩增。PCR扩增体系含模板1 μL,上游引物1.5 μL,下游引物1 μL,总体积为25 μL,反应条件:94℃ 2 min;94℃ 30 s,72℃ 1 min,5个循环;94℃ 30 s,70℃ 1 min,5个循环;94℃ 30 s,64℃ 30 s,72℃ 1 min,25个循环;72℃ 10 min。反应结束后,取10 μL进行1%琼脂糖凝胶电泳分析,若有特异性条带,则回收;若无特异性条带,则继续进行Nested PCR,反应条件:94℃ 2 min;94℃ 30 s,66℃ 30 s,72℃ 2 min,25个循环;72℃ 10 min。反应结束后取10 μL进行1%琼脂糖凝胶电泳分析,回收特异性条带与pGEM-T-easy vector连接,连接产物转化TOP10感受态细胞,进行蓝白斑筛选,挑选白斑菌落过夜培养后,进行PCR鉴定。经鉴定分子量大小与预期一致的阳性菌落,送上海桑尼生物技术有限公司进行测序。
1.2.3.2 EtCHP559 基因含完整ORF 的cDNA 克隆及测序利用DNAstar软件查找5'RACE PCR扩增产物的序列与原EST序列的重叠部分,将两段序列拼接为全长cDNA序列。根据拼接的全长cDNA序列,利用Primer Premier 5.0软件设计两端引物(引物引入酶切位点BamH Ⅰ、Xho Ⅰ),以mRNA反转录合成的cDNA第一链为模板,进行PCR扩增。PCR体系含模板2 μL,上下游引物各1 μL,总体积为20 μL。反应条件:94℃ 2 min;94℃ 45 s,57℃ 45 s,72℃ 2 min,35个循环;72℃ 10 min。反应结束后取10 μL进行1%琼脂糖凝胶电泳分析。回收的特异性PCR产物同5'RACE产物一样连接转化,经鉴定正确后测序。
1.2.4 EtCHP559 基因的生物信息学分析利用在线软件ORF finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析获得的新基因全长cDNA序列,找出编码框。利用ProtParam工具(http://cn.expasy.org/tools/protparam.html)计算编码蛋白质的理论分子量、等电点、氨基酸组成等。利用TMHMM服务器(http://www.cbs.dtu.dk/services/TMHMM-2.0/)分析编码蛋白有无跨膜结构。利用SignalP3.0在线分析软件(www.cbs.dtu.dk/services/SignalP)分析编码蛋白有无信号肽。利用ProtScale分析软件(www.expasy.org/cgi-bin/protscale.pl)分析编码蛋白的疏水性。采用motif_scan (http://myhits.isb-sib.ch/cgi-bin/motif_scan)寻找氨基酸序列的功能结构域,了解目的蛋白可能的功能。利用北京大学生物信息中心WebLab中提供的Antigenic程序(http://weblab.cbi.pku.edu.cn/program.inputForm.do?program=antigenic)分析其抗原为点。采用NCBI服务器中的CDD (http://www.ncbi.nlm.nih.gov/structure/cdd/cdd.shtml)程序对氨基酸序列进行功能保守功能域分析,判断该蛋白是否具有完整的保守结构域。
1.2.5 真核重组质粒pcDNA3.1-flag-EtCHP559 的构建按上海天根生物技术有限公司提供的质粒提取试剂盒提取重组克隆质粒pGEM-T-EtCHP559,并用BamH Ⅰ和Xho Ⅰ对载体pcDNA3.1-flag和重组克隆质粒pGEM-T-EtCHP559分别进行双酶切,酶切产物经1%琼脂糖凝胶电泳后,回收目的片段,4℃连接过夜,连接产物转入大肠杆菌TOP10感受态细胞中,涂板过夜培养,次日挑取单克隆菌落于5 mL LB培养基中培养12 h后,进行PCR鉴定;对鉴定为阳性的菌液提取质粒用BamH Ⅰ和Xho I限制性内切酶进行双酶切鉴定,阳性克隆送至上海桑尼生物有限公司进行测序。
1.2.6 DF-1 细胞的复苏培养及质粒转染质粒转染细胞前的一周复苏DF-1细胞,将细胞从液氮中取出,迅速放入37℃水浴中融化后,将细胞转入15 mL无菌离心管中,1 000 r/min离心5 min,用含10%胎牛血清的DMEM培养液悬浮细胞,转入细胞培养瓶中,于37℃,5% CO2浓度的培养箱中培养,每2 d更换一次培养液,传2-3代,至细胞生长状态良好。转染前,用0.25%胰蛋白酶-EDTA消化液37℃消化2 min,收集DF-1细胞进行计数,以每孔60万细胞铺被六孔板2块,37℃、5% CO2培养过夜,按照LipofectamineTM 2000脂质体转染操作步骤分别将空质粒pcDNA3.1-flag和重组质粒pcDNA3.1-flag-EtCHP559转染进DF-1细胞,置于37℃、5% CO2培养6 h,更换为含有2%胎牛血清的DMEM培养基。培养48 h后,收集一块六孔板的DF-1细胞,提取蛋白后进行Western blot检测,另一块六孔板的DF-1细胞进行间接免疫荧光检测。
1.2.7 间接免疫荧光(IFA)鉴定蛋白在真核细胞的表达为了检测真核表达重组质粒pcDNA3.1-flag-EtCHP559在DF-1细胞中的表达情况,取培养48 h的DF-1细胞,每孔用2%多聚甲醛2 mL固定15 min,PBS洗涤3次,加入1% Triton X-100 2 mL,室温下固定15 min,PBS洗涤3次,加入2% BSA/PBS 2 mL,4℃封闭过夜,PBS洗涤3次,加入抗rEtCHP559蛋白兔源血清(1:100倍稀释),37℃作用1 h;PBS洗涤3次,加入1:400稀释的绿色荧光标记的山羊抗兔二抗,37℃作用1 h,PBS洗涤3次后在荧光显微镜下观察结果。
1.2.8 Western blot 鉴定真核表达重组质粒pcDNA-3.1-flag-EtCHP559 在DF-1 细胞的表达情况收集培养48 h的DF-1细胞,加入RIPA裂解液,4℃,12 000 r/min离心2 min,取上清进行SDS-PAGE电泳。全湿法电转移至PVDF膜上,5%脱脂奶粉4℃封闭过夜;PBST洗涤3次,加入1:100稀释的抗rEtCHP559蛋白兔血清,37℃孵育2 h;PBST洗涤3次,加入1:15 000稀释的近红外荧光羊抗兔二抗,37℃孵育1 h,PBST洗涤3次,再经PBS洗涤5次后,用Odyssey双色红外激光成像系统进行拍照。
2 结果 2.1 CHP559基因的生物信息学分析根据Et CHP559基因的EST序列,利用5'RACE技术扩增获得了含一个完整开放阅读框(ORF:104-1 327 bp)的全长cDNA序列,该序列全长1 746 bp,包括5'端的UTR序列,长103 bp,含有CAAT序列,没有TATA序列;3'端UTR序列,长419 bp,含Ploy (A)尾,没有典型的AATAAA序列,ORF长1 224 bp,编码407个氨基酸(图 1);预测分子量为46.04 kD,等电点为9.12,在溶液中的不稳定指数为40.44,高于域值40,在溶液中性质不稳定,脂肪族指数81.45,总平均亲水性-0.223;信号肽、疏水性和跨膜结构的分析表明,该基因编码的蛋白有信号肽,信号肽的概率为0.998,其断裂位点位于第25个aa和第26个aa之间,该蛋白具有跨膜结构,其中第1-8个aa为蛋白的胞内区域,第9-31个aa为该蛋白的跨膜区域,第32-407个aa为蛋白的胞外区域。疏水性分析显示最大值为2.611,最小值为-3.511。抗原位点分析发现,该蛋白可能有15个抗原位点,推测具有良好的免疫原性。经结构功能域分析发现EtCHP559基因编码的蛋白可能含有2个N端糖基化位点,6个N端酰化位点,5个酪蛋白激酶Ⅱ磷酸化位点,6个蛋白激酶C磷酸化位点和1个酪氨酸激酶磷酸化位点。利用NCBI的CDD程序对该基因编码的氨基酸的保守结构域进行搜索,结果显示该基因编码的氨基酸没有保守结构域。与柔嫩艾美耳球虫基因组数据库(http://www.genedb.org/Homepage/Etenella)比较发现,该基因的ORF与保守的假象蛋白(hypothetical protein,conserved,ETH_00024035)100%同源,因此将该基因命名为Et CHP559。
 |
| 图 1 EtCHP559 cDNA 的核苷酸序列及其氨基酸序列 |
利用DNAstar软件查找5'RACE PCR扩增产物的序列与原EST序列的重叠部分,将两段序列拼接为全长cDNA序列。根据拼接的全长cDNA序列,利用合成的引物以柔嫩艾美耳球虫子孢子的cDNA第一链为模板进行PCR扩增含ORF的cDNA片段,扩增产物经1.0%琼脂糖凝胶电泳分析,在1 224 bp处有特异性的单一条带,与预期大小一致(图 2);其与pGEM-Teasy载体连接,转化感受态细胞TOP10,菌液PCR、双酶切、测序鉴定正确,表明成功扩增获得EtCHP559基因含完整ORF的cDNA片段。
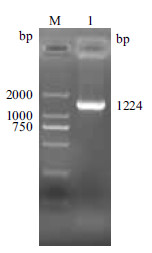 |
| M :DNA 分子量标准(DL2000);1 :EtCHP559 基因 图 2 EtCHP559 的PCR 扩增结果 |
分别用限制性内切酶BamH Ⅰ和Xho Ⅰ对鉴定正确的重组克隆质粒pGEM-T-EtCHP559和空载体pcDNA3.1-flag进行双酶切,纯化后进行连接,构建了真核重组表达质粒pcDNA3.1-flag-EtCHP559。对该重组质粒进行PCR和双酶切鉴定,结果(图 3)均为阳性,测序结果显示该基因正确插入,表明真核重组表达质粒pcDNA3.1-flag-EtCHP559构建成功。
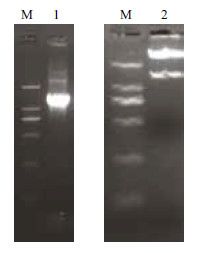 |
| M:DNA 分子量标准(DL2000);1:pcDNA3.1-flag-EtCHP559 菌液PCR 鉴定;2 :pcDNA3.1-flag-EtCHP559 双酶切鉴定 图 3 重组质粒pcDNA3.1-flag-EtCHP559 的PCR 与双酶切鉴定结果 |
利用抗rEtCHP559蛋白兔血清,对重组质粒转染48 h后的DF-1细胞进行了免疫荧光检测。结果(图 4)显示,在转染pcDNA3.1-flag-EtCHP559重组质粒的DF-1细胞中,绿色荧光通道可以看到特异的绿色荧光;而在转染pcDNA3.1-flag空载体和正常的DF-1细胞中,无任何可见荧光。结果表明pcDNA3.1-flag-EtCHP559重组质粒能在DF-1细胞中成功转染和表达。
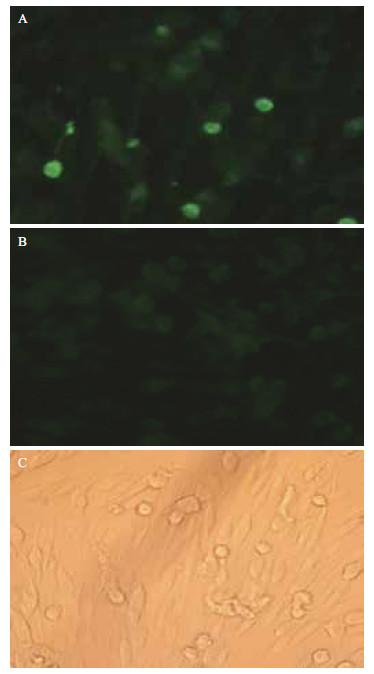 |
| A :pcDNA3.1-flag-EtCHP559 转染DF-1 细胞;B :pcDNA3.1-flag 质粒转染DF-1 细胞;C :正常DF-1 细胞 图 4 重组质粒pcDNA3.1-flag-EtCHP559 表达产物的间接免疫荧光检测 |
Western blot结果(图 5)显示,转染了pcDNA3.1-flag-EtCHP559重组质粒的DF-1细胞中可检测到大小约为47 kD的目的条带,而转染空载体的DF-1细胞中却没有相应的条带出现,表明pcDNA3.1-flag-EtCHP559重组质粒成功转染了DF-1细胞并在DF-1细胞中成功表达。
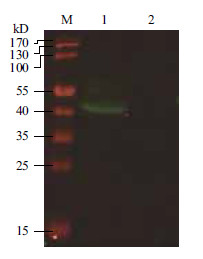 |
| M :预染的蛋白质标准分子量;1 :转染pcDNA3.1-flag-EtCHP559 的DF-1 细胞;2 :转染pcDNA3.1-flag 载体的DF-1 细胞 图 5 EtCHP559 重组蛋白Western blot 分析结果 |
虽然顶复器门原虫入侵的宿主细胞不同,包括红细胞、淋巴细胞、巨噬细胞和消化道细胞等,但它们都有一个共同的高度保守的入侵机制[6]。在这一过程中,运动接触环(MJ)的形成至关重要,此特殊结构作为支架推动虫体进入带虫空泡[7, 8],对顶复器门原虫弓形虫、疟原虫等研究表明,AMA1通过与棒状体颈部蛋白(RONs)相互作用形成的复合体是构成MJ结构的主要成分,参与入侵过程[3, 9, 10]。但到目前为止,对鸡球虫入侵宿主细胞时MJ的分子组成研究还未见报道,只是对微线分泌的顶膜抗原AMA1进行了一些研究。Jiang等[11]首次克隆到了柔嫩艾美耳球虫的AMA1基因全长,并在体外用多克隆抗体抑制EtAMA1蛋白后观察到子孢子细胞入侵率下降了70%,表明AMA1蛋白参与了子孢子入侵宿主细胞的过程。为了进一步研究柔嫩艾美耳球虫AMA1的功能,薛璞[5]利用酵母双杂交的方法从柔嫩艾美耳球虫子孢子酵母cDNA文库中筛选到了与EtAMA1相互作用的14个蛋白的ESTs序列。已有研究表明其中2个ESTs (EtZB1-A08与EtMIC2)参与了球虫入侵宿主细胞的过程,本研究扩增获得的EtCHP559基因,是根据其中的一个EST序列利用RACE技术扩增获得的,因此该蛋白可能与EtAMA1相互作用,参与了子孢子入侵宿主细胞的过程。
pcDNA3.1载体是一种能够在哺乳动物细胞或宿主内高水平稳定或瞬时表达的真核表达载体,其含有巨细胞病毒早期启动子CMV、BGH、polyA加尾信号和SV40早期启动子,全长5 400 bp,还带有Neo抗性基因便于转染细胞的筛选,常用来融合表达多种外源基因蛋白以及核酸疫苗的研究[12]。本研究选用的真核表达载体pcDNA3.1-Flag是由pcDNA3.1(+)改造而获得的,该载体在pcDNA3.1(+)多克隆位点的N端添加了flag标签,并且在标签序列之前插入了kozak序列。研究发现真核生物蛋白表达调控与蛋白起始密码子AUG周围的碱基种类有很大关系,当一个基因起始密码子AUG之前的碱基为kozak序列GCCACC时,该蛋白表达量会有相应提高[13]。Flag是序列为DYKDDDDK蛋白标签,由亲水性的8个氨基酸残基组成,序列简短,对目标蛋白的结构和生物活性影响小,容易构建成标签蛋白融合N端或C端,可用Anti-flag抗体检测[14]。Flag标签还可设计用于免疫亲和层析,在非变性条件下洗脱flag标签融合蛋白[15]。因此,本研究利用该载体成功构建了真核表达重组质粒pcDNA3.1-flag-EtCHP559,并在真核细胞DF-1中成功表达,表达的蛋白带有flag标签,在后续实验中可以用来纯化蛋白,也可以通过免疫沉淀来研究蛋白质之间的互作等。
近年来,为了对鸡球虫功能基因和核酸疫苗的研究,许多研究者将鸡球虫的功能基因连接到真核表达载体,成功构建了真核重组质粒,并在不同的真核表达细胞中获得表达。如王艳歌等[16]构建了柔嫩艾美耳球虫乳酸脱氢酶的真核表达质粒,并在293T细胞中成功表达,刘颖丽等[17]在Hela细胞中成功表达了柔嫩艾美耳球虫微线蛋白2(Et-MIC2),杜爱芳等[18]以减毒沙门氏菌为载体,在Vero细胞中表达了柔嫩艾美耳球虫微线蛋白4(Et-MIC4)。但这些细胞均为哺乳动物细胞,目前还未见利用球虫宿主鸡细胞来表达鸡球虫蛋白的报道。本研究尝试选择了鸡胚成纤维细胞(chicken embryo fibroblast cell line,DF-1)来表达EtCHP559,DF-1细胞来源于East Lansing Line (ELL-0),是一种可无限传代的细胞系,目前,在病毒研究领域应用广泛,如用于病毒疫苗的生产和重组病毒或蛋白的表达[19]。黄杰等[20]在DF-1细胞中成功表达了鸭瘟病毒的UL7蛋白,田志革等[21]将新城疫病毒V基因片段与pcDNA3.1载体连接,并在DF-1细胞中获得了表达。近来,DF-1细胞也被用于鸡球虫体外培养的研究,姜连连等[22]首次将DF-1细胞应用于鸡球虫研究领域,将柔嫩艾美耳球虫子孢子接入DF-1细胞后,子孢子能成功入侵DF-1细胞,并在细胞内发育至裂殖子,因此该细胞可用于鸡球虫体外研究。本研究在DF-1细胞中首次成功表达了柔嫩艾美耳球虫CHP559蛋白,由于DF-1细胞是柔嫩艾美耳球虫天然宿主鸡的本体细胞,因此以其为平台更有利于研究球虫基因的功能以及与宿主的相互作用关系。
4 结论本研究利用RACE技术成功获得了柔嫩艾美耳球虫保守蛋白EtCHP559基因,并构建了其真核重组表达质粒pcDNA3.1-Flag-EtCHP559,成功转染到DF-1细胞中。通过间接免疫荧光和免疫印迹的方法证实该基因在真核细胞DF-1中成功表达,这些结果将为进一步研究该蛋白的生物学功能和球虫核酸疫苗的研制奠定了基础。
| [1] | Williams RB. A compartmentalised model for the estimation of the cost of coccidiosis to the world’s chicken production industry[J]. International Journal for Parasitology, 1999, 29(8):1209-1229. |
| [2] | Aikawa M, Miller LH, Johnson J, et al. Erythrocyte entry by malarial parasites. A moving junction between erythrocyte and parasite[J]. J Cell Biol, 1978, 77(1):72-82. |
| [3] | Lamarque M, Besteiro S, Papoin J, et al. The RON2-AMA1 interaction is a critical step in moving junction-dependent invasion by apicomplexan parasites[J]. PLoS Pathogens, 2011, 7(2): e1001276. |
| [4] | Alexander D, Mital J, Ward G, et al. Identification of the moving junction complex of the apicomplexan parasite, Toxoplasma gondii : a collaboration between distinct secretory organelles[J]. American Journal of Tropical Medicine and Hygiene, 2005, 73(6, Suppl. S): 363. |
| [5] | 薛璞. 柔嫩艾美耳球虫子孢子酵母双杂交cDNA 文库的构建及 EtAMA1 相互作用蛋白的初步筛选[D]. 上海:上海师范大学, 2013. |
| [6] | Besteiro SSBU, Dubremetz J, Lebrun M. The moving junction of apicomplexan parasites :a key structure for invasion[J]. Cellular Microbiology, 2011, 13(6):797-805. |
| [7] | Proellocks NI, Coppel RL, Waller KL. Dissecting the apicomplexan rhoptry neck proteins[J]. Trends in Parasitology, 2010, 26(6): 297-304. |
| [8] | Opitz C, Soldati D‘. The glideosome’:A dynamic complex powering gliding motion and host cell invasion by Toxoplasma gondii[J]. Molecular Microbiology, 2002, 45(3):597-604. |
| [9] | Tyler JS, Treeck M, Boothroyd JCJB. Focus on the ringleader : the role of AMA1 in apicomplexan invasion and replication[J]. Trends in Parasitology, 2011, 27(9):410-420. |
| [10] | Besteiro S, Michelin A, Poncet J, et al. Export of a Toxoplasma gondii rhoptry neck protein complex at the host cell membrane to form the moving junction during invasion[J]. PLoS Pathogens, 2009, 5(2):e1000309. |
| [11] | Jiang L, Lin J, Han H, et al. Identification and characterization of Eimeria tenella apical membrane antigen-1(AMA1)[J]. PLoS One, 2012, 7(7):e41115. |
| [12] | 胡晓静, 刘棋, 付薇, 等. 鹅γ-干扰素基因真核表达载体的构 建及在幼仓鼠肾细胞中的表达[J]. 中国兽医科学, 2011(5): 526-529. |
| [13] | Petty AP, Dick CL, Lindsey JS. Translation of an atypical human cDNA requires fidelity of apurine-pyrimidine repeat region and recoding[J]. Gene, 2008, 414(1-2):49-59. |
| [14] | Hopp TP, Prickett KS, Price VL, et al. A short polypeptide marker sequence useful for recombinant protein identification and purification[J]. Biotechnology, 1988, 6(10):1204-1210. |
| [15] | Einhauer A, Jungbauer A. The FLAGTM peptide, a versatile fusion tag for the purification of recombinant proteins[J]. Journal of Biochemical and Biophysical Methods, 2001, 49(1-3):455-465. |
| [16] | 王艳歌, 董辉, 韩红玉, 等. 柔嫩艾美耳球虫乳酸脱氢酶真核 表达质粒的构建与鉴定[J]. 中国动物传染病学报, 2014(2):65-71. |
| [17] | 刘颖丽, 李建华, 郑君, 等. 鸡柔嫩艾美耳球虫EtMIC-2 基因 真核表达载体的构建及其在Hela 细胞中的表达[J]. 中国兽 医学报, 2011(8):1142-1146. |
| [18] | 杜爱芳, 王素华, 胡松华. 鸡柔嫩艾美耳球虫5401 基因真核 表达载体的构建及在Vero 细胞中的表达[C]. 桂林:中国 畜牧兽医学会家畜寄生虫学分会, 2004. |
| [19] | Himly M, Foster DN, Bottoli I, et al. The DF-1 chicken fibroblast cell line :Transformation induced by diverse oncogenes and cell death resulting from infection by avian leukosis viruses[J]. Virology, 1998, 248(2):295-304. |
| [20] | 黄杰, 程安春, 汪铭书, 等. 鸭瘟病毒UL7 基因真核表达质粒 的构建及其瞬时表达[J]. 中国兽医科学, 2013(8):861- 865. |
| [21] | 田志革, 柴洪亮, 李凤勇, 等. 野鸭源新城疫病毒V 蛋白基因 在DF-1 细胞中的表达及V 蛋白的亚细胞定位[J]. 中国兽 医科学, 2014(5):476-479. |
| [22] | 姜连连, 林矫矫, 韩红玉, 等. 柔嫩艾美耳球虫DF-1 细胞培养 体系的建立及应用[J]. 中国兽医科学, 2011(6):551-556. |




