哺乳动物生命的延续,需要生殖受精产生受精卵进而发育成个体,而精卵细胞的膜融合是细胞融合的中心环节。虽然这是一个十分重要的作用过程,但对于这个融合的分子机制还知之甚少。根据已有文献的报道,精卵膜融合所必须的蛋白分子有精子细胞膜上的IZUMO1[1, 2],卵细胞膜上的CD9[3, 4, 5]以及近期发现的IZUMO1配体JUNO[6, 7]。利用电子显微镜观察发现,精卵细胞膜融合最先发生在精子细胞膜的赤道段[8]。然而,参与精卵细胞膜融合的重要精子蛋白IZUMO1在精子顶体反应前并不存在于其膜表面,而是在顶体反应后才位于赤道和后顶体区域[1, 2, 9, 10, 11],因此,如果找到一个只存在于精子细胞膜赤道区域的蛋白,将能更清楚地研究精子赤道膜部分如何与卵细胞进行识别并引起下一步反应的。因此,精子赤道膜蛋白片段1(SPESP1)引起了本实验室注意,它不仅只位于精子膜赤道处[12],而且据报道抗SPESP1抗体可以抑制人类[13]和小鼠[14]的体外受精过程,敲除Spesp1基因的雄鼠生育能力明显下降。有实验结果表明,SPESP1与参与精卵膜融合并与一些精子膜蛋白重新定位有关,其中包括IZUMO1在顶体反应后的重新定位,同时还发现在精卵融合中与形成完整的精子膜有关[15]。本实验准备克隆绵羊SPESP1蛋白的cDNA,构建原核表达重组载体,并于大肠杆菌BL21中表达得到GST-SPESP1重组融合蛋白,旨在为之后研究其与IZUMO1等相关蛋白的相互作用奠定基础。
1 材料与方法 1.1 材料菌株E.coliDH5α和BL21、质粒pGEX-4T-1均为本实验室保存;绵羊睾丸购自西口子屠宰场;雌性小鼠购自于内蒙古大学动物中心。GST单克隆抗体、辣根过氧化物酶标记的羊抗小鼠IgG购自生工生物工程(上海)股份有限公司;质粒小提和琼脂糖凝胶DNA提取试剂盒购自天根生化科技(北京)有限公司;pMD19-T载体、RNAiso Reagent (D312)、PrimeScript RT reagent Kit、Premix Taq、T4 DNA Ligase、QuickCut限制酶BamH I、EcoR I与其它常规生化试剂均购自大连宝生物工程有限公司;华大基因有限公司进行DNA测序。小鼠抗羊SPESP1腹水多克隆抗体为本实验室制备。
1.2 方法 1.2.1 绵羊SPESP1 cDNA的克隆及重组表达载体pGEX-Spesp1的构建切取新鲜屠宰绵羊的睾丸组织50-100 mg,并根据TaKaRa RNAiso Reagent试剂盒操作步骤提取总RNA,利用反转录试剂盒制备绵羊睾丸组织总cDNA。根据GenBank中预测的绵羊Spesp1 cDNA序列(NC_019464.1),在其编码区的上下游各选取一段设计一对特异性引物。上游引物:5'-GACGCATGGCGGAGTGCTGT-3';下游引物:5'-CTAACATGCTTATTTCTCCC-3',引物由华大基因有限公司合成。预期扩增产物长度为1 095 bp。以反转录的cDNA第一链为模板,用设计好的特异性引物进行PCR扩增,程序为:94℃预变性3 min;94℃变性30 s,52℃退火30 s,72℃延伸1 min,共29个循环;最后72℃延伸10 min。PCR产物用1%琼脂糖凝胶电泳进行分离,切取1 000-2 000 bp之间的条带,利用琼脂糖凝胶DNA提取试剂盒提取纯化PCR产物,将纯化后的PCR产物与pMD19-T载体进行连接,转化进入E.coli感受态DH5α细胞中,选取5个阳性菌落进行测序。
根据测序结果设计含有BamH I和EcoR I酶切位点的表达引物,其中上游引物:5'-TCGGGATCCGCGTATCGAAGAGT-3'(下划线为BamH I酶切位点),下游引物:5'-GGAATTCGCATAGTTAGCTTGAATC-3'(下划线为EcoR I酶切位点,斜体为终止密码子),以已构建好的pMD19-T-Spesp1重组质粒为模板进行PCR扩增,预期扩增产物大小为1 054 bp,扩增程序为:94℃预变性3 min;94℃变性30 s,62℃退火30 s,72℃延伸1 min,共进行29个循环;最后72℃延伸10 min。将纯化的PCR产物以及质粒pGEX-4T-1同时用BamH I和EcoR I分别进行双酶切,回收纯化酶切后的产物,并将二者进行连接转化进入DH5α菌种中,选阳性菌落提取质粒用BamH I和EcoR I进行双酶切鉴定,之后测序确定片段正确。将构建成功的重组pGEX-Spesp1质粒转化到E.coli BL21(DE3)中进行表达。
1.2.2 重组融合蛋白表达将成功转化的菌株接种于10 mL的LB培养基中(Amp浓度50 μg/mL;葡萄糖浓度0.5%),37℃摇床培养,当菌液OD600=0.4-0.6之间时,加入IPTG (终浓度为0.01%)37℃诱导表达4 h。最后离心收集10 mL的诱导菌液,超声破碎后,加入含有β巯基乙醇的蛋白Loading Buffer,对其进行SDS-PAGE (12%分离胶)电泳鉴定目的蛋白表达情况。预期融合蛋白GST-SPESP1大小为64 kD。
1.2.3 最佳诱导温度细胞培养方法同上,当OD600=0.4-0.6时,加入终浓度为0.01%的IPTG诱导表达。再在25℃、28℃、31℃、34℃和37℃条件下诱导表达4 h后离心收集菌体,超声破碎,取部分液体离心收集上清,用SDS-PAGE分析目的蛋白的最佳表达温度条件
1.2.4 最佳诱导时间细胞培养方法同上,当OD600=0.4-0.6时,加入终浓度为0.01%的IPTG诱导表达。在上一步确定的最佳温度条件下,分别诱导表达2、3、4、5、6、7和8 h后,收集菌液超声破碎,用SDS-PAGE分析目的蛋白的表达量,确定最佳诱导时间。
1.2.5 最佳IPTG诱导浓度细胞培养方法同上,在已研究确定的诱导时间和温度条件下,加入终浓度为0.002%、0.005%、0.01%、0.015%、0.025%、0.03%、0.035%和0.04%的IPTG诱导表达。以SDS-PAGE分析目的蛋白的表达量,确定目的蛋白表达的最佳IPTG诱导浓度。
1.2.6 GST-SPESP1融合蛋白的Western blotting鉴定重组融合蛋白经SDS-PAGE电泳后转至PVDF膜,分别用GST单抗和小鼠抗SPESP1腹水多抗作为一抗,羊抗小鼠IgG-HRP作二抗,进行Western blot确认GST-SPESP1的正确性,具体操作同本实验室之前的报道[16]。
1.2.7 GST-SPESP1融合蛋白的纯化在E.coli BL21(DE3)中大量诱导表达目标蛋白,离心收集菌体,超声破碎;经SDS-PAGE电泳后,将PAGE胶放于低温的饱和KCl中浸泡5 min,切取对应片段大小位置的白色条带,并将其切碎;将碎胶末放入已经处理过的透析袋(透析袋用1 mmol/L EDTA煮5 min后再用ddH2O煮5 min处理)中,封口后放入水平电泳槽中60 V过夜;清除透析袋中胶块后,将袋子放在1×PBS中4℃进行蛋白复性12 h,每隔4 h换一次PBS;收集纯化后蛋白保存于-80℃备用。纯化蛋白要经过SDS-PAGE以及Western blotting鉴定。
2 结果 2.1 Spesp1 cDNA克隆及原核表达载体构建与鉴定以绵羊睾丸cDNA为模板,PCR扩增出大约1 000 bp的目的片段(图 1-A),与pMD19-T连接后,挑取3个阳性菌落双酶切鉴定后送往测序,结果显示,克隆的绵羊Spesp1 cDNA长度1 095 bp,与预期相同,其中不含信号肽的编码区(CDS)长度为1 032 bp,将序列信息与NCBI中预测的序列对比发现,有两个碱基差异,翻译为氨基酸后对比显示,除了NCBI预测的第88位的谷氨酸(Glu)变为了天冬氨酸(Asp),其它完全一致。序列已上传GenBank,登录号为KR003078。
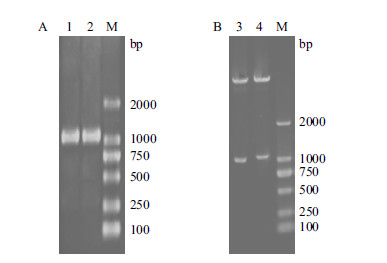 |
| M :DL2000 DNA Marker ;1,2 :Spesp1 cDNA 的PCR 产物;3,4 :pGEXSpesp1阳性克隆BamH I 和EcoR I 双酶切产物 图 1 Spesp1 cDNA 的PCR 结果(A)及pGEX-Spesp1 的BamH I 和EcoR I 双酶切鉴定(B) |
以重组pMD19-T-Spesp1为模板,用含有BamH I和EcoR I酶切序列的引物进行PCR,得到含有该酶切位点及Spesp1 cDNA序列的PCR产物。将纯化的PCR条带与pGEX-4T-1双酶切之后连接,转化后挑取3个阳性菌落,BamH I和EcoR I双酶切鉴定,在1 000 bp附近出现条带(图 1-B),测序后对比显示,所插入片段的序列没有发生变异。 2.2 绵羊SPESP1重组融合蛋白在E.coli BL21菌株中的表达以及条件优化 2.2.1 重组融合蛋白表达
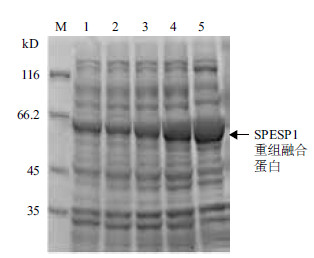
根据氨基酸分子量计算,不含信号肽的绵羊Spesp1蛋白的分子量为38 kD,GST标签是26 kD,即融合蛋白GST-SPESP1的分子量应为64 kD,经IPTG诱导后,用12% SDS-PAGE电泳后在66.2 kD与45 kD间出现特异性条带(图 2),而同样条件下的大肠杆菌BL21空菌以及含有pGEX-4T-1质粒菌体都不出现该条带。
 |
| M :蛋白分子量标准;1 :E.coli BL21 空菌诱导裂解菌液;2 :pGEX-4T-1诱导裂解菌液;3-5 :pGEX-Spesp1 诱导裂解菌液 图 2 pGEX-Spesp1 重组蛋白的表达分析 |
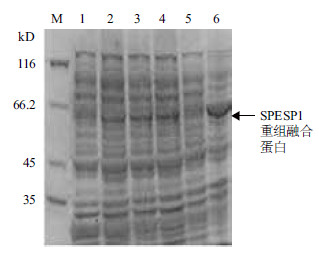
在0.01% IPTG诱导4 h后SDS-PAGE电泳发现,37℃时融合蛋白最多(图 3,泳道5),上清进行SDS-PAGE分析(图 4)后发现,上清几乎不含有该融合蛋白,说明该重组蛋白是以包涵体形式存在。
 |
| M :蛋白分子量标准;1-5 :分别为25℃、28℃、31℃、34℃和37℃超声裂解的菌液 图 3 不同温度诱导表达的SDS-PAGE 分析 |
 |
| M :蛋白分子量标准;1-5 :分别为25℃、28℃、31℃、34℃和37℃超声裂解的上清;6 :37℃超声裂解的菌液 图 4 不同温度诱导表达上清的SDS-PAGE 分析 |
在其他条件一致的情况下经不同时间诱导后,发现4 h时重组融合蛋白表达量达到最大(图 5),在4-8 h之间表达量不随时间延长而增加,故该重组融合蛋白最佳诱导时间为4 h。2.2.4最佳诱导IPTG浓度在其他条件一致的情况下,利用不同浓度的IPTG对其进行诱导,经过SDS-PAGE电泳(图 6)后发现,由于高浓度IPTG不利于细菌生长,所以该重组融合蛋白的IPTG最佳的诱导浓度为0.005%。
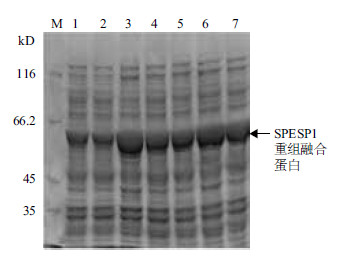 |
| M :蛋白分子量标准;1-7 :分别是诱导2、3、4、 5、6、7 和8 h 后超声裂解菌液 图 5 不同时间诱导表达的SDS-PAGE 分析 |
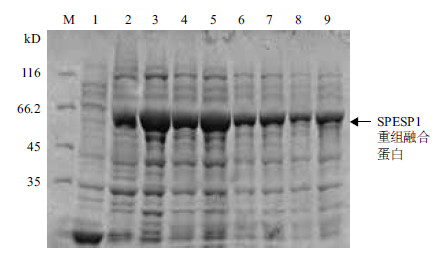 |
| M :蛋白分子量标准;1 :pGEX-4T-1 诱导裂解菌液;2-9 :IPTG 诱导终浓度分别为0.002%、0.005%、 0.01%、0.015%、0.025%、0.03%、0.035% 和0.04% 图 6 不同IPTG 浓度诱导表达的SDS-PAGE 分析 |
为了确定所表达的条带是带有GST标签的,用GST单克隆抗体进行Western blotting检验。用小鼠抗羊SPESP1腹水多克隆抗体作为一抗鉴定表达蛋白中含有融合蛋白GST-SPESP1蛋白(图 7)。其都在66.2 kD附近出现特异性条带,说明表达的特异性重组融合蛋白是带有GST的GST-SPESP1。
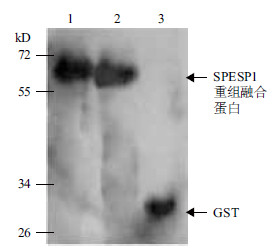 |
| 1,2 :pGEX-Spesp1 诱导破碎菌液;3 :pGEX-4T-1 诱导破碎菌液。其中,1是以鼠抗羊SPESP1 多抗作为一抗进行检测,2、3 是以鼠抗GST 单抗作为一抗 图 7 诱导表达融合蛋白SPESP1-GST 的Western blotting 检测 |
为了鉴定SDS-PAGE胶提取纯化的蛋白是所需的GST-SPESP1蛋白,将其进行SDS-PAGE电泳检测,发现其与pGEX-Spesp1转化菌诱导后大量表达的重组蛋白的位置相同,都出现在66.2 kD附近。分别用GST和小鼠抗羊SPESP1腹水多克隆抗体作为一抗进行检测,发现其都在66.2 kD附近处出现条带(图 8)。由此证明,所纯化的蛋白是GST-SPESP1重组融合性蛋白。
 |
| 1 :pGEX-Spesp1 诱导破碎菌液;2,3 :切胶纯化得到的融合蛋白SPESP1-GST。其中1、2 是以鼠抗GST 单抗作为一抗,3 是以鼠抗羊SPESP1 多抗作为一抗进行检测 图 8 切胶纯化的融合蛋白SPESP1-GST 的Western blotting 检测 |
精子赤道膜蛋白片段1(SPESP1)是已知的参与精卵细胞膜融合过程的一个重要蛋白,而对于绵羊中编码该蛋白的cDNA序列以及其在绵羊这一大型哺乳动物中发挥的生物学功能却未有报道。本实验利用RT-PCR得到绵羊睾丸组织总cDNA,之后参考GenBank中预测的绵羊Spesp1上下游附近非编码区序列设计引物,之后以总cDNA为模板通过PCR扩增得到大量含部分非编区的Spesp1的DNA片段,将其测序并与GenBank中预测的序列进行对比发现有两个碱基的不同,翻译成氨基酸序列后产生一个氨基酸的不同,其将预测的第88位谷氨酸(Glu)变异为天冬氨酸(Asp),这两氨基酸都是带负电荷的极性氨基酸,且差异点不位于蛋白预测的功能域中,猜测产生这种多态性的可能原因是由于个体差异。
构建重组表达载体pGEX-Spesp1,使得所表达的蛋白带有GST标签,以便后期对其进行检测与纯化。诱导实验结果显示,该蛋白的最佳表达条件是37℃、诱导4 h、终浓度0.005% IPTG,重组蛋白主要以包涵体形式存在。通过对上清与全液的SDSPAGE电泳图对比发现,可溶性重组蛋白量不随蛋白表达量增加而增加,说明表达蛋白的存在形态与其表达总量的相关性较弱。当IPTG终浓度大于0.015%时,蛋白表达量随着菌体生长受到抑制而开始减少,因此在表达量相差不大时,应该尽量选取浓度较低IPTG进行诱导。蛋白表达量在诱导时间到达4 h后,并不随时间增加而有所增长,但菌体培养时间过长会产生大量次级代谢产物影响后期的纯化,应尽量选取短的诱导时间。在Western blotting检测中,发现抗GST抗体与抗SPESP1抗体识别的条带位于同一水平上,说明诱导得到的表达蛋白是带有GST标签的SPESP1重组蛋白,为之后研究SPESP1体外与其他蛋白相互作用提供材料,为进一步阐明SPESP1在精卵融合中作用奠定基础。
4 结论本实验成功构建了pGEX-Spesp1重组载体,并在E.coli BL21成功表达了GST-SPESP1重组融合蛋白,并得到了最佳表达条件是37℃、4 h和IPTG终浓度0.005%。通过GST单克隆抗体和小鼠抗羊SPESP1腹水多克隆抗体作为一抗对其进行Western blotting检测,证实该蛋白是GST-SPESP1重组融合蛋白。在此基础上,本实验通过切胶纯化的方法得到了纯化的GST-SPESP1重组蛋白,并进一步证明纯化得到了目标重组蛋白。
| [1] | Yamashita M, Yamagata K, Tsumura K, et al. Acrosome reaction of mouse epididymal sperm on oocyte zona pellucid[J]. J Reprod Dev, 2007, 53(2):255-262.[2]Inoue N, Ikawa M, Isotani A, et al. The immunoglobulin superfamily protein Izumo is required for sperm to fuse with eggs[J]. Nature Publishing Group, 2005, 434(7030):234-238. |
| [3] | Miyado K, Yamada G, Yamada S, et al. Requirement of CD9 on the egg plasma membrane for fertilization[J]. Science, 2000, 287 (5451):321-324. |
| [4] | Le Naour F, Rubinstein E, Jasmin C, et al. Severely reduced female fertility in CD9-deficient mice[J]. Science, 2000, 287(5451): 319-321. |
| [5] | Kaji K, Oda S, Shikano T, et al. The gamete fusion process is defective in eggs of Cd9-deficient mice[J]. Nat Genet, 2000, 24(3): 279-282. |
| [6] | Chalbi M, Barraud-Lange V, Ravaux B, et al. Binding of sperm protein Izumo1 and its egg receptor Juno drives Cd9 accumulation in the intercellular contact area prior to fusion during mammalian fertilization[J]. Development, 2014, 141(19):3732-3739. |
| [7] | Bianchi E, Doe B, Goulding D, et al. Juno is the egg Izumo receptor and is essential for mammalian fertilization[J]. Nature, 2014, 508 (7497):483-487. |
| [8] | Yanagimachi R. Mammalian fertilization[M]. New York :Raven Press, 1994. |
| [9] | Sebkova N, Ded L, Vesela K, et al. Progress of sperm IZUMO1 relocation during spontaneous acrosome reaction[ J ] . Reproduction, 2014, 147(2):231-240. |
| [10] | Satouh Y, Inoue N, Ikawa M, et al. Visualization of the moment ofmouse sperm-egg fusion and dynamic localization of IZUMO1[J]. J Cell Sci, 2012, 125(21):4985-4990. |
| [11] | Kawai Y, Hama T, Mayumi T, et al. Flow cytometric analysis of mouse sperm using monoclonal anti-sperm antibody OBF13[J]. Journal of Reproductive Immunology, 1989, 16(1):71-82. |
| [12] | Wolkowicz MJ, Shetty J, Westbrook A, et al. Equatorial segment protein defines a discrete acrosomal subcompartment persisting throughout acrosomal biogenesis1[J]. Biology of Reproduction, 2003, 69(3):735-745. |
| [13] | Wolkowicz MJ, Digilio L, Klptz K, et al. Equatorial segment protein (ESP)is a human alloantigen involved in sperm-egg binding and fusion[J]. J Androl, 2008, 29(3):272-282. |
| [14] | Lv ZM, Wang M, Xu C. Antifertility characteristics of the N-terminal region of mouse equatorial segment protein[J]. Anat Rec (Hoboken), 2010, 293(1):171-181. |
| [15] | Fujihara Y, Murakami M, Inoue N, et al. Sperm equatorial segment protein 1, SPESP1, is required for fully fertile sperm in mouse[J]. Cell Science, 2010, 123(9):1531-1536. |
| [16] | Liu ZD, Xing WJ, Wang LQ, et al. Prokaryotic expression, ascitic polyclonal antibody preparation and identification of cashmere goat izumol[J]. Agriculture Science in China, 2010, 9(4):605- 613. |




