莱茵衣藻(Chlamydomonas reinhardtii)属于绿藻门、团藻目、衣藻科,是真核单细胞生物。进化上比较古老,介于高等植物和动物之间。衣藻的培养条件简单、生长周期短、光合效率高、遗传背景清楚,是重要的能源作物,素有"绿色酵母"之称[1]。其生物学特性与高等植物和动物密切相关,如纤毛病理学[2],是目前用于研究纤毛的模式生物中最为广泛使用的一个[3]。
近年来,人们关于纤毛的知识大多来自于对莱茵衣藻的研究,如我们对纤毛结构的了解,"鞭毛内运输"的机制的发现以及与纤毛相关疾病的确定等[3]。其中,巴德-毕氏综合症(Bardet-Biedl syndrome,BBS)就是一种由多种基因突变造成、与原生纤毛功能缺失相关的疾病。包含12种致病基因,分别为BBS1-12[4, 5]。BBS患者的症状包括视网膜衰退、肾囊形成、多指(趾)症、嗅觉失敏、高血压、肥胖症乃至精神发育迟滞症等[5]。最近的研究发现BBS蛋白位于纤毛或基体,它们参与了纤毛的组装过程和信号传导过程[6]。在莱茵衣藻中,抑制BBS5的表达,纤毛将不能形成[6];BBS1或BBS4基因突变的莱茵衣藻丧失趋光性[4],但是其失去趋光性的分子机制尚不清楚,有待人们进一步深入研究。
2007年莱茵衣藻基因组测序的完成为莱茵衣藻反向遗传学的研究提供了可能。RNA干扰(RNA interference,RNAi)是指在进化过程中高度保守、由双链RNA (double-stranded RNA,dsRNA)诱发、同源mRNA高效特异性降解的现象[7, 8]。它是一种高效的特异性强的基因阻断技术,近年来发展迅速,目前已成为功能基因组研究领域中一种简单、有效的代替基因敲除的遗传工具,在一定程度上起到了基因Knock-down的效果[9, 10]。因此若用RNAi技术对衣藻BBS1进行研究,将会促进人们深刻了解和认识莱茵衣藻趋光性的分子机制,同时有可能揭示出"纤毛相关疾病"发生的分子机理,为BBS1基因功能的进一步揭示及衣藻趋光性分子机制的阐明奠定基础。 1 材料与方法 1.1 材料
试材莱茵衣藻CC503,购自于美国Duke大学衣藻中心。藻株接种到固体TAP (Tris-Acetate-Phosphate)平板上进行继代培养。菌株大肠杆菌DH5α为本实验室保存。pEASY-T1 Cloning Kit购自于北京全式金生物技术有限公司,中间载体pChlamy_3载体质粒购自于Invitrogen公司。反转录试剂及各种内切酶购自于NEB公司,各种Taq聚合酶购于大连宝生物公司。 1.2 方法 1.2.1 衣藻RNAi骨架载体的搭建 1.2.1.1 pGH-intron中间载体的构建
利用CTAB法提取莱茵衣藻基因组DNA,以其为模板,根据已经发表的磷酸核酮糖激酶(Phosphoribulokinase)序列信息(GenBank登录号:AAA33090.1),设计一对引物PRF1:5'-GTGAGCAAGAGATGCGGC-3',PRR1:5'-CTGGTGGGGAGGAGGGAG-3',PCR扩增其第4段内含子,连接到pEASY-T1载体,并进行测序,获得磷酸核酮糖激酶的第4段内含子序列,即间隔序列。将设计好的两端带有合适多克隆位点的间隔序列委托捷瑞生物工程有限公司进行合成,并构建到由该公司提供的pGH载体上,形成中间载体pGH-intron。 1.2.1.2 pChlamy_RNAi骨架载体的构建
以pGHintron为载体的基本骨架,利用PCR技术设计4条引物分别扩增pChlamy_3载体上的启动子序列,引物为HSPF1:5'-AACTGCAG TCGCTGAGGCTTGACATG-3'(下划线部分为Pst Ⅰ酶切位点)与HSPR1:5'-GGGGTACC TTTAAGATGTTGAGTGACTTC-3'(下划线部分为Kpn Ⅰ酶切位点);扩增含有终止子(3'UTR)和潮霉素标记完整表达框序列,引物为UTRHYGF1:5'-GCTCTAGACCGCTCCGTGTAAATGGA-3'(下划线部分为Xba Ⅰ酶切位点)与UTRHYGR1:5'-AGAATGCGGCCGCAGTACCATCAACTGACG-3'(下划线部分为Not Ⅰ酶切位点);扩增产物酶切后相继插入到pGH-intron的多克隆位点之间,并进行测序验证,从而形成衣藻的RNAi骨架载体,命名为pChalmy-RNAi载体。 1.2.2 BBS1基因干涉片段的获得
为确认cDNA选取片段对BBS1基因干扰效果的影响,选取了BBS1基因上的两段序列进行干扰,克隆出来的片段分别命名为BBS1.1和BBS1.2。具体方法如下:TRIzoL提取莱茵衣藻总RNA,并用M-MuLV反转录酶进行cDNA第一条链的合成。然后再以合成的cDNA为PCR模板,用BBS1.1F与BBS1.1R引物对及BBS2.1F与BBS2.1R引物对进行PCR扩增,扩增产物分别连接到pEASY-T1载体上并测序,形成pEASY-T1-BBS1.1/2.1中间载体,获得BBS1基因两段干涉片段BBS1.1和BBS2.1。其中克隆BBS1基因上这两段序列所用的引物如下:
BBS1.1F:5'-GCTCTAGAGGGGTACCAGTCCAACCCAAACGATTAC-3'(下划线部分分别为Xba Ⅰ和Kpn Ⅰ酶切位点);BBS1.1R:5'-GATTCCATATGGACTAGTGTCGCCAAACAGATTACAAG-3'(下划线部分分别为Nde Ⅰ和Spe Ⅰ酶切位点);BBS2.1F:5'-GCTCTAGAGGGGTACCCGGGCGTATGTCAAGGTC-3('下划线部分分别为XbaⅠ和KpnⅠ酶切位点);BBS2.1R:5'-GATTCCATATGGACTAGTGCACCAGGAAGGGTATCG-3'(下划线部分分别为Nde Ⅰ和Spe Ⅰ酶切位点)。 1.2.3 BBS1基因RNAi载体的构建
酶切含有BBS1基因正向目的片段的pEASY-T1-BBS1.1/2.1中间载体质粒,与同样经Kpn Ⅰ和Spe Ⅰ酶切的pChlamy_RNAi骨架载体连接,获得含有BBS1基因正向目的片段的pChlamy_RNAi_BBS1.1/2.1_F中间载体。
酶切含有BBS1基因反向目的片段的pEASYT1-BBS1.1/2.1中间载体质粒,与同样经Nde Ⅰ和Xba Ⅰ酶切的含有BBS1基因正向目的片段的pChlamy_RNAi_BBS1.1/2.1_F中间载体连接,获得含有BBS1基因正向和反向目的片段的pChlamy_RNAi_BBS1.1/2.1_FR RNAi载体。 1.2.4 BBS1基因RNAi载体的转化与筛选
取对数期生长的无细胞壁的莱茵衣藻CC503,用基因枪方法转化,转化后在弱光下培养2 d后,将固体平板上的莱茵衣藻转移至含10 mg/L潮霉素的50 mL液体培养基中进行筛选,培养7-10 d直至液体培养基中绿色完全褪去,再取2 mL涂在固体平板上进行固体筛选(10 mg/L hygromycin),7 d左右长出单克隆,挑单克隆至液体培养。 1.2.5 Real-time PCR分析BBS1基因的沉默效果
分别提取CC503野生型及转pChlamy_RNAi_BBS1.1/2.1_FR质粒的莱茵衣藻总RNA。反转录合成cDNA第一链,然后以其为Real-time PCR的模板;S26 RNA作为内参[11],进行Real-time PCR扩增。检测野生型及转pChlamy_RNAi_BBS1.1/2.1_FR质粒的莱茵衣藻中目标基因mRNA的表达变化,以确定对目标基因的干扰效果。所用内参及靶基因引物对如下:S26F:5'-CGCCCTGCCCAAGATTTA-3';S26R:5'-CGCGGCTGTGGATAGCA-3';BBS1QF:5'-GGGCGTATGTCAAGGTC-3';BBS1QR5:5'-GCACCAGGAAGGGTATCG-3'。 1.2.6 莱茵衣藻趋光性分析
分别取对数生长期的野生型CC503及转基因莱茵衣藻于培养皿中,然后用锡箔纸包严,只留一定大小的空隙以便光源射入,2 h后观察两株系莱茵衣藻的趋光性变化。 2 结果 2.1 衣藻RNAi骨架载体的搭建
利用PCR技术,扩增了衣藻磷酸核酮糖激酶蛋白的第4段内含子,然后连接到pEASY-T1载体,并进行测序,大小为497 bp (图 1)。在该段序列两端设计合适的多克隆酶切位点,委托上海捷瑞生物工程有限公司进行合成,并构建到由该公司提供的pGH载体上,形成中间载体pGH-intron。
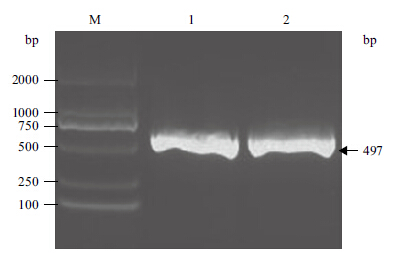 |
| M :D2000 Marker ;1,2 :莱茵衣藻磷酸核酮糖激酶第4 段内含子PCR 产物 图 1 莱茵衣藻磷酸核酮糖激酶第4 段内含子的PCR 产物 |
利用PCR技术,设计合适的含有相应酶切位点的引物,分别扩增Invitrogen公司pChlamy_3载体上的启动子序列及含有终止子和潮霉素标记完整表达框序列(图 2),PCR产物酶切后相继连接到中间载体pGH-intron上,并测序验证,形成衣藻RNAi载体的基本骨架,命名为pChalmy-RNAi载体(图 3)。
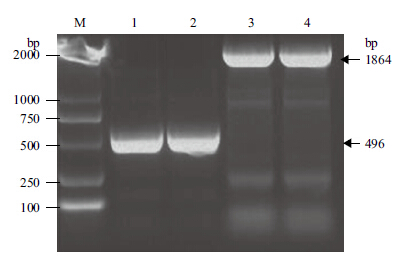 |
| M:D2000 Marker;1,2:莱茵衣藻嵌合组成型表达启动子HSP70A/RBC S2的PCR产物;3,4:莱茵衣藻3'UTR与潮霉素筛选标记表达框序列的PCR产物 图 2 莱茵衣藻启动子和3'UTR 与潮霉素表达框序列的 PCR 产物 |
 |
| HSP:嵌合的HSP70A/Rbc S2启动子;intron:衣藻磷酸核酮糖激酶蛋白的一段内含子;3'UTR:Rbc S2终止子;β2-T:β2-tubulin启动子;hpt:含有终止子的潮霉素选择标记基因序列;Forward:目的基因的正向片段;Reverse:目的基因的反向片段 图 3 衣藻RNAi 骨架载体构建示意图 |
根据phytozome数据库公布的BBS1基因(g17944.t1)报道的序列,设计了两对引物分别扩增BBS1基因上两段序列,然后连接到pEASY-T1载体,并进行测序形成pEASY-T1-BBS1.1/2.1中间克隆载体,扩增产物片段大小分别为188 bp和229 bp,结果与预期大小一致(图 4)。另外,由于我们设计的每条引物上均含有正向插入位点和反向插入位点合适的酶切位点,因此中间载体pEASY-T1-BBS1.1/2.1上相当于既含有靶基因的正向片段序列又含有反向片段序列,因此无需再进行反向片段的克隆,节省了构建时间和实验成本。
 |
| M:D2000 Marker;1,2:BBS1基因干涉片段BBS1.1的PCR扩增;3,4:BBS1基因干涉片段BBS2.1的PCR扩增 图 4 BBS1 基因干涉片段的PCR 扩增 |
用Kpn Ⅰ和Spe Ⅰ酶切含有BBS1基因正向目的片段的pEASY-T1-BBS1.1/2.1中间载体,然后与同样经这两个酶酶切的pChlamy_RNAi骨架载体连接,获得含有BBS1基因正向目的片段的pChlamy_RNAi_BBS1.1/2.1_F RNAi载体。由于靶基因片段较小,因此用Kpn Ⅰ和Nde Ⅰ进行酶切,多了中间的内含子序列,片段大小750 bp左右(图 5-A),结果与预期大小一致,表明获得了含有BBS1基因正向目的片段pChlamy_RNAi_BBS1.1/2.1_F中间载体。
用Nde Ⅰ和Xba Ⅰ酶切含有BBS1基因反向目的片段的pEASY-T1-BBS1.1/2.1中间载体质粒,与同样经这两个酶酶切的含有BBS1基因正向目的片段的pChlamy_RNAi_BBS1.1/2.1_F中间载体连接,获得含有BBS1基因正向和反向目的片段的pChlamy_RNAi_BBS1.1/2.1_FR RNAi载体,经Kpn I和Xba I酶切鉴定,结果(图 5-B)表明大小与预期大小一致。
 |
| M:D2000 Marker;1,2:Kpn Ⅰ和Nde Ⅰ双酶切鉴定含有BBS1基因正向片段pChlamy_RNAi_BBS1.1/2.1_F RNAi质粒;3,4:Kpn Ⅰ和Xba Ⅰ双酶切鉴定含有BBS1基因正向和反向片段的pChlamy_RNAi_BBS1.1/2.1_FR RNAi载体质粒 图 5 BBS1 基因RNAi 载体的酶切检测 |
采用基因枪法将pChlamy_RNAi_BBS1.1/2.1_FR RNAi载体转化莱茵衣藻CC503。转化后先在含有潮霉素的液体培养基上进行初步筛选,再涂在固体平板上进行筛选。然后随机挑取转干涉片段BBS1.1或BBS2.1 RNAi质粒的转基因衣藻单克隆(每个干涉片段选取10个单克隆)进行提取DNA,以扩增pChlamy_3载体上启动子序列的引物对HSPF1和HSPR1进行PCR检测。结果(图 6)显示,扩增条带大小约500 bp,与预期大小一致,其中只有一个单克隆未扩增出目标条带,阳性转化率高达95%,证明了转化和筛选方法的可行性。
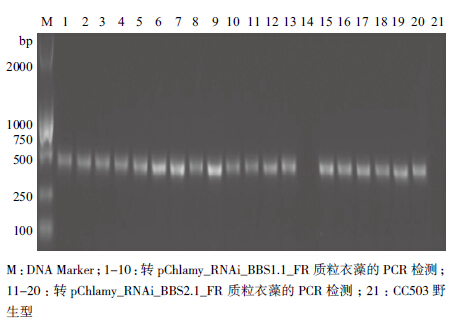 |
| M:DNA Marker;1-10:转pChlamy_RNAi_BBS1.1_FR质粒衣藻的PCR检测;11-20:转pChlamy_RNAi_BBS2.1_FR质粒衣藻的PCR检测;21:CC503野生型 图 6 转基因衣藻的PCR 检测 |
取野生型CC503及转pChlamy_RNAi_BBS1.1/2.1_FR质粒的莱茵衣藻,分别提取总RNA。其中转pChlamy_RNAi_BBS1.1_FR质粒获得的阳性转化子后代命名为BBS1.1_n (n代表哪个单克隆),转pChlamy_RNAi_BBS2.1_FR质粒获得的阳性转化子后代命名为BBS2.1_n (n代表哪个单克隆)。以其反转录合成的cDNA第一链为Real-time PCR模板,进行BBS1基因沉默效果分析。其中荧光定量PCR数据采用2-ΔΔCt法分析。结果(图 7)表明,针对BBS1基因设计的RNAi干扰片段,确实抑制了BBS1的表达。
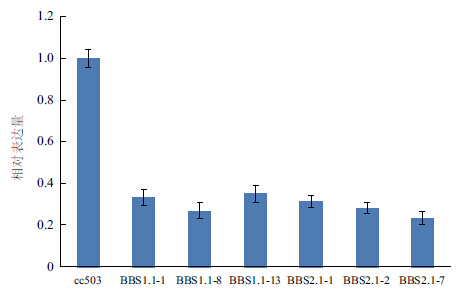 |
| 图 7 BBS1 基因的mRNA 表达水平 |
分别取对数生长期的野生型CC503及转基因莱茵衣藻,放入一定体积的培养皿中,然后用锡箔纸包严,只留一定大小的空隙以便光源射入,2 h后,观察两株系莱茵衣藻的趋光性变化。结果(图 8)显示,本研究所设计的针对BBS1基因上两段干扰片段的干扰效果均较好,野生型莱茵衣藻CC503绝大部分移动到靠近光源的一侧,而干扰BBS1基因后的转基因莱茵衣藻对光线不敏感。
 |
| 图 8 莱茵衣藻的趋光性分析 |
目前莱茵衣藻中已经有相应的RNAi载体,但其构建步骤较为繁琐,需要多次酶切和连接才能构建成功[12, 13],而且其转化后阳性克隆子的筛选效果也不理想。本研究搭建的RNAi载体只需在靶基因片段两段加上合适的酶切位点,克隆得到的cDNA片段再经两次酶切与连接即可得到目的基因的RNAi载体,试验方法简单易行,大大节约了构建时间与实验成本。
随后,针对BBS1基因设计的两个干涉片段被构建到我们所搭建的RNAi载体上,并转化莱茵衣藻CC503。转化后的藻株先在固体平板上长2 d后,转移到含有潮霉素的液体筛选培养基上再培养一周,绿色褪去后又涂在固体平板上进行筛选,长出的单克隆95%都是阳性,比未经液体培养基筛选,转化后直接涂在固体筛选培养基上获得的单克隆阳性率(4%)提高了几十倍。因此该筛选方案,不仅减少了工作量,还节约了实验成本,值得同行借鉴。
对转化BBS1基因后的藻株进行抑制效果分析结果表明,选取的两个干涉片段对BBS1基因均起到了明显的抑制作用,改变了莱茵衣藻的趋光性,说明BBS1基因在调控衣藻趋光性信号通路中起着重要作用。但其如何调控衣藻趋光性信号通路,具体由哪些基因参与,尚不清楚,是我们进一步要研究的重点。我们将应用本实验室的高通量测序平台,对转BBS1基因的藻株与对照进行转录组测序及分析,以找到其调控的信号网络及网络中的关键基因,为莱茵衣藻趋光性分子机制的阐明奠定基础。
另外,研究发现有些BBS患者具有不同程度的嗅觉丧失,在BBS1或BBS4基因敲除掉的老鼠中嗅觉纤毛数目降低[14]。BBS蛋白位于纤毛或基体,它们参与了纤毛的组装过程和信号传导过程。在线虫中,BBS蛋白是连接和协调"鞭毛内运输"蛋白颗粒中的蛋白质A和B复合体,是纤毛形成所必需的[15]。在斑马鱼中,BBS1基因突变后,没有影响纤毛的形成,而是影响其运动性。在衣藻中,抑制BBS5的表达,纤毛将不能形成。突变BBS1、BBS4或者BBS7基因,破坏了衣藻的趋光性[4],但并不影响鞭毛的组装,其趋光性的分子机制及是否影响鞭毛的运动尚未阐明,有待于进一步研究。
除此之外,本研究所构建的RNAi骨架载体直接可应用到衣藻中其它基因功能的研究,为后期进行BBS1基因所参与的信号通路中其它的功能基因的鉴定及同行进行衣藻功能基因的研究提供了宝贵的载体资源。因此,本研究具有较好的理论价值和应用价值。 4 结论
本研究成功构建了衣藻的RNAi骨架载体,并将衣藻中BBS1基因的干涉片段构建到该骨架载体上,转化衣藻CC503;抑制了BBS1基因表达,改变了衣藻的趋光性。
| [1] | Goodenough UW. Green yeast[J]. Cell, 1992, 70(4):533-538. |
| [2] | Merchant SS, Prochnik SE, Vallon O, et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions[J]. Science, 2007, 318(5848):245-250. |
| [3] | 柳林, 纪伟. 纤毛疾病和与之相关的基因[J]. 现代生物医学 进展, 2012, 12(2):373-375. |
| [4] | Lechtreck KF, Johnson EC, Sakai T, et al. The Chlamydomonas reinhardtii BBSome is an IFT cargo required for export of specific signaling proteins from flagella[J]. The Journal of Cell Biology, 2009, 187(7):1117-1132. |
| [5] | 潘俊敏. 衣藻、纤毛与“纤毛相关疾病”[J]. 中国科学C 辑: 生命科学, 2008, 38(5):399-409. |
| [6] | Oeffner F, Moch C, Neundorf A, et al. Novel interaction partners of Bardet-Biedl syndrome proteins[J]. Cell Motility and the Cytoskeleton, 2008, 65(2):143-155. |
| [7] | Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric chalcone synthase gene into petunia results in reversible cosuppression of homologous genes in trans[J]. The Plant Cell, 1990, 2(4):279-289. |
| [8] | B a s s B L . D o u b l e -s t r a n d e d R N A a s a t e m p l a t e f o r g e n e silencing[J]. Cell, 2000, 101(3):235-238. |
| [9] | Kusaba M. RNA interference in crop plants[J]. Current Opinion in Biotechnology, 2004, 15(2):139-143. |
| [10] | Wesley SV, Helliwell CA, Smith NA, et al. Construct design for efficient, effective and high-throughput gene silencing in plants[J]. The Plant Journal :For Cell and Molecular Biology, 2001, 27(6):581-590. |
| [11] | Simon DF, Descombes P, Zerges W, et al. Global expression profiling of Chlamydomonas reinhardtii exposed to trace levels of free cadmium[J]. Environmental Toxicology and Chemistry / SETAC, 2008, 27(8):1668-1675. |
| [12] | 李兴涵, 费小雯, 邓晓东. 莱茵衣藻磷酸果糖激酶RNAi 载体 构建及其对莱茵衣藻油脂积累的影响[J]. 广东农业科学, 2014, 10 :155-159. |
| [13] | 邓晓东, 蔡佳佳, 费小雯. 莱茵衣藻磷脂二脂酰甘油酰基转 移酶3 在三酰甘油合成中的功能研究[J]. 水生生物学报, 2014, 38(4):745-750. |
| [14] | Kulaga HM, Leitch CC, Eichers ER, et al. Loss of BBS proteins causes anosmia in humans and defects in olfactory cilia structure and function in the mouse[J]. Nature Genetics, 2004, 36(9): 994-998. |
| [15] | Ou G, Blacque OE, Snow JJ, et al. Functional coordination of intraflagellar transport motors[J]. Nature, 2005, 436(7050): 583-587. |
| [16] | Kim YH, Epting D, Slanchev K, et al. A complex of BBS1 and NPHP7 is required for cilia motility in zebrafish[J]. PLoS One, 2013, 8(9):e725. |




