2.中国人民解放军总后勤部卫生部药品仪器检验所,北京 100071;3.中国科学院北京基因组研究所,北京 100101
2. Institute for Drug and Instrument Control,Health Department,the General Logistics Department of People’s Liberation Army,Beijing 100071;3. Beijing Institute of Genomics,Chinese Academy of Sciences,Beijing 100101
随着测序技术的不断发展,我们获得越来越多的遗传信息。研究人员试图通过对特定靶向基因的破坏或敲除来研究它们的相关功能。与传统基因打靶技术相比,新型基因组编辑技术应用范围更广、作用效率更高、所需成本更低,可用于模式动物构建、基因功能研究、基因治疗等方面。
目前,常用的基因组编辑技术主要包括以下3种:锌指蛋白(Zinc finger proteins,ZFPs)技术、转录激活因子样效应因子(Transcription activator-like effectors,TALEs)技术、CRISPR(Clustered regula-rly interspaced short palindromic repeats)技术。它们的大致原理都是通过造成靶向DNA双链断裂(double-strand breaks,DSBs),引发细胞内固有的非同源末端连接(Nonhomologous end joining,NHEJ)[1]或同源重组(Homology-directed repair,HDR)[2]两种修复机制对DSBs处进行修复,从而实现基因的定点敲除、敲入或缺失。3种技术在具体操作方式上又有所不同。
锌指核酸酶(Zinc-finger nuclease,ZFN)与转录激活子样效应因子核酸酶(Transcription activator-like effector nulease,TALEN)这两种人工核酸内切酶,均由DNA识别域与非特异性核酸内切酶Fok I融合而成。其中,ZFN的锌指模块识别三联体碱基,在序列的识别上灵活性小,设计成本昂贵、技术被少数公司垄断,这些都限制了它的发展[3]。TALEN在结构上较之ZFN更加优化。其DNA结合域中的各单元可与靶向目标序列一一对应,组装更为简单,特异性更高。2013年,一种源自细菌适应性免疫系统的CRISPR/Cas9的出现,为基因靶向修饰领域带来了巨大变革。主要原理:向导RNA(guide RNA,gRNA)与Cas9核酸酶形成的复合物,在gRNA指导下与靶向目标序列结合,由靶向目标序列下游的原型间隔序列相邻基序(Protospacer adjacent motifs,PAM)激活Cas9核酸酶[4],使其对PAM上游3 nt处剪切造成DSBs。该系统较之前两种,应用范围更广、作用效率更高、使用成本更低,经过短短两年的发展,已被用于世界各地的实验室[5]。
线粒体是存在于大多数真核生物细胞中的细胞器,有“细胞中的能量工厂”之称。它主要通过氧化磷酸化以三羧酸循环腺苷(ATP)的形式为细胞活动提供能量。同时,还参与诸如铁硫簇的生物合成、钙稳态调节[6]、细胞分化、细胞信息传递和细胞凋亡[7]等过程,并且拥有调控细胞生长和细胞周期的能力。线粒体基因组,又称线粒体DNA(mtDNA)。长度一般为几万至数十万碱基对,人类mtDNA为16 569 bp,共编码2个rRNA、13个多肽、22个tRNA。基因排列紧凑,除与mtDNA复制及转录有关的一小段区域外,无内含子序列[8]。mtDNA表现为母系遗传,突变率高,是细胞核内DNA的10倍左右。且mtDNA突变是导致人类线粒体遗传病及衰老性疾病的重要原因之一。研究表明,它与衰老及神经退行性病变如阿尔茨海默症、帕金森氏病等老年化疾病有关,同时也与肿瘤的形成密切相关[9]。截至目前,已鉴定出超过500多个致病性突变来自mtDNA。而针对mtDNA致病性突变的精确改造能力则相对缺乏[10]。
随着Minczuk等[11, 12]将ZFN作用于mtDNA,Bacman等[13]首次将TALEN用于清除病人线粒体内的突变型mtDNA。这两种基于ZFPs技术、TALEs技术的线粒体核酸酶都为更好的进行线粒体功能研究、线粒体遗传病的治疗等提供了可能。此外,本实验室也基于人工CRISPR/Cas9系统独特地构建了作用于mtDNA的mtCRISPR/Cas9系统(结果未发表)。
任何一种基因组编辑技术,它的靶向剪切效率、切割特异性等问题不可回避。由于核基因组中的DNA损伤修复系统比较完整,当发生DSBs时,可通过NHEJ修复在靶向目标序列处引入插入或缺失突变。因此,改造核基因组的相关基因组编辑技术可通过错配酶法、靶向目标序列直接PCR后TA克隆测序或观察靶向目标序列测序峰图等方法鉴定。而相应的线粒体基因组编辑工具,由于线粒体缺乏完善的DNA损伤修复系统[14, 15, 16],修复能力较弱,无法通过上述方法检测。
因此,本研究利用转基因技术,将一段含有靶向目标序列的mtDNA序列随机整合到宿主基因组中,获得转基因细胞株。通过相应的核基因组编辑技术靶向剪切该细胞株的核基因组,对靶向目标序列处造成DSBs引发NHEJ修复。观察靶向目标序列测序峰图,鉴定mtDNA的靶向目标序列剪切活性。建立了一种检测线粒体核酸酶靶向剪切活性的新方法。
1 材料与方法 1.1 材料 1.1.1 实验用载体及细胞株所用载体为哺乳动物细胞表达载体pEGFP-N1,带EGFP标签,购自Clontech公司。所用细胞株为人肾上皮细胞HEK293,培养条件:DMEM+10%FBS。
1.1.2 主要药品及试剂DL2 000 DNA Marker、dN-TP、RNase A、T4 DNA Ligase、LA Taq、SYBR premix Ex Taq等均购自TaKaRa公司。胶回收试剂盒、质粒提取试剂盒、基因组提取试剂盒均购自天根生化科技(北京)有限公司。引物由生工生物工程(上海)股份有限公司合成,测序由北京六合华大基因科技股份有限公司完成。
1.2 方法 1.2.1 pEGFP-mtDNA转基因单克隆细胞株的获得 1.2.1.1 pEGFP-mtDNA重组克隆质粒的构建以pEGFP-N1为载体,将一段同时含有两个靶向目标序列(T1、T2)的mtDNA序列与EGFP的N端融合,除去EGFP前的起始密码子ATG,最终得到重组克隆质粒:pEGFP-mtDNA。
具体步骤如下:(1)以HEK293细胞基因组DNA为模板,用CL-F1/CL-R1引物对进行PCR扩增(335 bp),胶回收后得产物J;(2)以产物J为模板,用CL-F2/CL-R2(CL-F2带有flag标签,如表 1横线部分所示)引物对进行PCR扩增(167 bp),胶回收后得产物K;(3)以产物K为模板,用CL-F3/CL-R3(CL-F3带有Nhe I酶切位点和flag标签、CL-R3带有Age I酶切位点,如表 1相应下划线、下划线斜体部分所示)引物对进行PCR扩增(194 bp),得产物L;(4)对纯化后的产物L及pEGFP-N1质粒做Nhe I/Age I双酶切,分别以此为插入片段及载体进行连接、转化、挑单克隆鉴定、鉴定正确样品测序;(5)以上一步骤中的正确质粒为模板,用CL-F4/CL-R4引物对(CL-F4带有Age I酶切位点,如表 1横线斜体部分所示;删去EGFP起始密码子ATG,添加一个碱基C调整编码框,该碱基的选择应避免引入终止密码子)进行PCR扩增(902 bp),得产物M;(6)对纯化后的产物M及上一步骤所用质粒做Age I/Not I双酶切,分别以此为插入片段及载体进行连接、转化、挑单克隆鉴定、鉴定正确样品测序,即得最终质粒:pEGFP-mtDNA(图 1)。
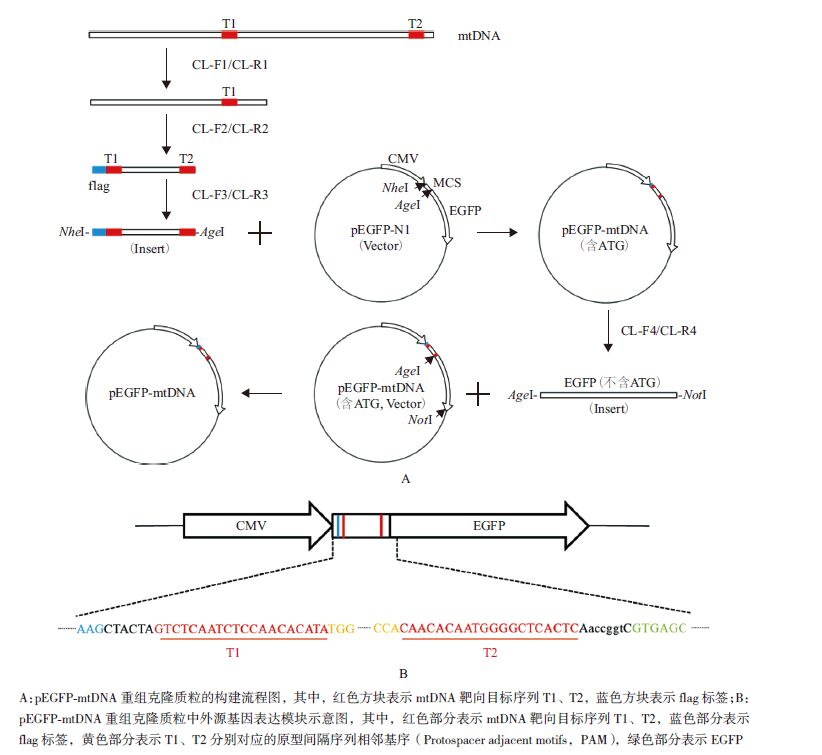
|
| 图 1 pEGFP-mtDNA 重组克隆质粒的构建 |
将pEGFP-mtDNA瞬时转染到培养于60 mm细胞培养皿的HEK293细胞中,24 h后,将细胞传代至10 cm培养皿中继续培养,加800 μg/mL的G418筛选,每隔3-4 d换液一次。培养至细胞不再大批量死亡,且形成单克隆细胞团后,挑单克隆于24孔板。对含有绿色荧光的单克隆细胞,收取细胞提基因组进行PCR鉴定。根据pEGFP-mtDNA序列,分别设计3对引物P1(P1-F/P1-R)、P2(P2-F/P2-R)、P3(P3-F/P3-R)(表 2),3对引物均可扩增出目的条带的细胞,则为阳性单克隆细胞株。
反应总体系20 μL(上/下游引物各0.5 μL、ddH2O 9 μL、2×SYBR premix Ex Taq 10 μL)。PCR反应程序:95℃ 3 min;95℃ 15 S、60℃ 30 S(40个循环),熔解曲线60-95℃,每个样品做3次重复。
1.2.2.2 标准曲线的建立将pEGFP-mtDNA质粒与HEK293细胞基因组DNA混合,设置分别含有1、2、4、8和16个外源基因拷贝数的标准品对照。具体方法如下:假设含有外源基因片段的质粒大小为a bp,HEK293细胞基因组DNA用量为b ng,大小为3×109 bp。外源基因片段完全随机的头尾相连的插入在一条染色体上,则b ng的基因组DNA中含有一个外源基因拷贝数的质量为:a×b×0.5/3×109 ng。设计特异性扩增外源基因序列的引物对RT-F/RT-R,以β-actin为内参的引物对Actin-F/Actin-R(表 3)。将外源基因片段扩增的CtpEGFP-mtDNA值减去内参扩增的CtActin值,得△Ct,再对样品拷贝数的对数值(以2为底)作图得绝对定量标准曲线。
构建含有相应mtDNA靶向目标序列的CRISPR/Cas9质粒。将该质粒瞬时转染到所选阳性单克隆细胞株中。48-72 h后,收取细胞提基因组DNA,用1.2.1.2所述P1引物对进行PCR扩增。扩增产物送样测序,测序引物为P1-R。如果靶向目标序列在核基因组中发生DSBs,则会引发细胞中的NHEJ修复机制,得到插入或缺失突变。而对样品全基因组DNA进行PCR扩增所得的扩增产物为混合产物,其中既含有野生型的原始靶向目标序列,也含有引入插入或缺失的突变型靶向目标序列。因此,使用Chromas软件显示测序峰图,查看对应靶向剪切位置处是否有套峰出现,如有套峰,则证明靶向目标序列有剪切活性,反之则无。此外,还可根据套峰强弱判断该系统对靶向目标序列的剪切效率。
2 结果 2.1 获得阳性单克隆细胞株 2.1.1 构建pEGFP-mtDNA重组克隆质粒pEGFP-N1为真核表达载体,含有EGFP基因和neo抗性基因。将一段含有两个靶向目标序列(T1、T2)的mtDNA序列,通过1.2.1.1所述方法与EGFP基因融合得到测序正确的重组克隆质粒pEGFP-mtDNA(图 1)。该质粒瞬时转染到HEK293细胞中,24 h后观察EGFP表达情况,转染效率达95%以上(图 2-A)。

|
| 图 2 阳性单克隆细胞株的筛选 |
将鉴定正确的pEGFP-mtDNA重组克隆质粒瞬时转染到HEK293细胞中(图 2-A),24 h后细胞传代,并用G418筛选。当有大批量未转入质粒的细胞死亡后,及时换液。药筛至细胞不再大量死亡,且形成单克隆细胞团时,挑单克隆于24孔板中。对含有绿色荧光的11个单克隆细胞,收细胞提基因组。分别用3个引物对P1(P1-F/P1-R)、P2(P2-F/P2-R)、P3(P3-F/P3-R)对单克隆细胞所提基因组进行PCR鉴定,3对引物均可扩增出目的条带的细胞,则为阳性单克隆细胞株,结果如图 2-B所示,单克隆细胞株2#、3#、5#、8#、10#为阳性单克隆细胞株。
2.2 检测外源基因拷贝数 2.2.1 建立标准曲线将pEGFP-mtDNA质粒与HEK293细胞基因组DNA混合,设置分别含有1、2、4、8和16个外源基因拷贝数的标准品对照,用RT-F/RT-R引物对扩增外源基因片段,Actin-F/Actin-R引物对扩增内参β-actin,每个反应重复3次,取平均Ct值。其中,特异性引物对RT-F/RT-R的扩增曲线、熔解曲线如图 3-A所示,内参引物对Actin-F/Actin-R的扩增曲线、熔解曲线如图 3-B所示,两个熔解曲线均在86℃附近有且只有一个单峰,证明两引物对均为特异性扩增。将RT-F/RT-R扩增的CtpEGFP-mtDNA减去Actin-F/Actin-R扩增的CtActin,得△Ct。△Ct与样品拷贝数的对数值(log2N)作图得到标准曲线(图 3-C),Log2N= -1.177 2△Ct+2.779 3,决定系数R2=0.997 1。
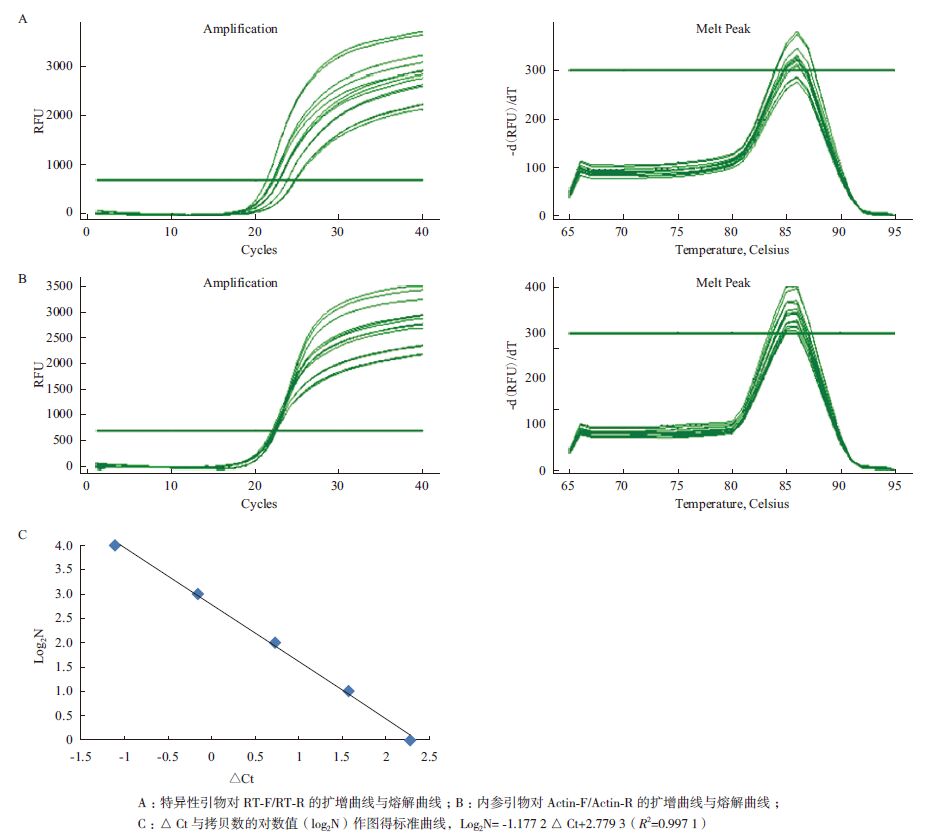
|
| 图 3 绝对定量PCR 检测外源基因拷贝数 |
提取阳性单克隆细胞株2#、3#、5#、8#、10#基因组,并以此为模板做实时荧光定量PCR。将这5个细胞株的△Ct分别带入计算公式:Log2N= -1.177 2△Ct+2.779 3,得其对应的拷贝数(N),标准差在0.935 1和0.138 7之间,结果可信。具体拷贝数检测结果如表 4所示。
构建分别含有mtDNA靶向目标序列T1、T2的两个CRISPR/Cas9质粒,命名为CRISPR/Cas9-T1、CRISPR/Cas9-T2。将CRISPR/Cas9-T1、CRISPR/Cas9-T2和相应不含T1、T2靶向目标序列的CRISPR/Cas9空骨架质粒(Control-T1、Control-T2)分别瞬时转染到pEGFP-mtDNA阳性单克隆细胞株10#中。48 h后,收取细胞提基因组DNA,用1.2.1.2所述P1引物对进行PCR扩增,扩增产物送样测序。测序结果表明,阴性对照Control-T1、Control-T2在靶向目标序列处均无套峰出现(图 4-B),而作用质粒CRISPR/Cas9-T1、CRISPR/Cas9-T2在靶向目标序列处均有套峰出现,且CRISPR/Cas9-T1处的套峰强于CRISPR/Cas9-T2处(图 4-A)。
3 讨论基因组编辑技术,是研究相关基因功能的重要手段。作为三大基因组编辑技术之一的CRISPR/Cas9系统,自2012年首次描述其对DNA的基本编辑能力后[17],经短短几年发展,已取得丰硕研究成果。它不仅可以编辑DNA用于构建癌症模型[18]、清除人类感染细胞中的HIV[19]、阻止杜氏营养不良小鼠模型中的肌肉退化[20]等,还可以编辑RNA[21],为更好的研究RNA功能提供了可能。目前的基因组编辑技术,大多只能改造核基因组,而很多人类遗传病是由于线粒体基因组突变所引起。mtDNA突变通常是异质性的,只有当mtDNA突变型占总mtDNA数量80%以上时,才会引起疾病症状[22]。在此之前,Minczuk和Bacman等[11, 12, 13]已分别将ZFN和TALEN两种人工核酸酶用于清除人类线粒体内的突变型mtDNA,本实验室也基于人工CRISPR/Cas9系统独特地构建了作用于mtDNA的mtCRISPR/Cas9系统(结果未发表)。这3种经改造后用于线粒体基因组编辑的线粒体核酸酶,同样面临着如何检测靶向剪切活性的问题。
对于核基因组编辑技术而言,由于核基因组中的DNA损伤修复系统比较完整,可在DSBs处通过NHEJ修复引入插入或缺失突变。因此,其检测方法主要有:(1)对靶向目标序列进行PCR扩增,扩增产物经变性、退火后形成错配,通过错配酶剪切不完全匹配的DNA序列,验证靶向目标序列剪切活性;(2)对靶向目标序列进行PCR扩增后,通过TA克隆测序与理论序列比对验证靶向目标序列剪切活性;(3)对靶向目标序列进行PCR扩增后,扩增产物直接测序,通过观察靶位点处的套峰情况验证靶向目标序列剪切活性。而线粒体对DNA的损伤修复能力较弱,无法通过上述方法检测。本研究构建了一个核基因组中含有单拷贝或低拷贝mtDNA靶向目标序列的转基因细胞系,利用相应核基因组编辑技术靶向剪切该细胞株的核基因组,通过上述第3种方法观察靶向目标序列测序峰图鉴定剪切活性,该法简洁明了。
首先,在构建重组克隆质粒时,可以如图 1-A所示,将一段含有一个或若干mtDNA靶向目标序列的串联序列连入克隆载体质粒中,同时检测多个靶向目标序列的剪切活性。在外源基因与EGFP基因融合时要注意是否需调整编码框、是否引入了起始密码子或终止密码子等问题。其次,在筛选阳性单克隆细胞株时,通过设计的3对扩增范围涵盖启动子区、编码区、终止区的特异性引物对的同时鉴定,既保证了筛选结果的准确性,又确保了外源基因能够完整表达。经过药物筛选得到的阳性单克隆细胞株,其外源基因往往随机整合在染色体上。因此,在获得阳性单克隆细胞株后,通过检测其外源基因拷贝数,筛选单拷贝或低拷贝的阳性单克隆细胞株,是有效鉴定靶向目标序列剪切活性的关键步骤。
目前,检测外源基因拷贝数的方法主要有Southern blot法和实时荧光定量PCR法。Southern blot法为传统外源基因拷贝数检测法,实时荧光定量PCR法为近年来新兴技术。通过比较,两种方法在检测外源基因拷贝数的准确性上结果十分接近[23, 24]。与Southern blot法相比,实时荧光定量PCR法起始模板量小、灵敏性高、简单高效、成本较低、不需要进行放射性实验[25, 26],该方法足以满足本研究的筛选需要。在筛选得到的单拷贝或低拷贝阳性单克隆细胞株中,瞬时转染定量的人工核酸酶后,可通过测序峰图鉴定靶向目标序列剪切活性更加清晰,减少背景干扰。
此外,利用实时荧光定量PCR法建立了检测转基因细胞株中外源基因拷贝数的实验体系。由于使用的SYBR Green I染料可广泛嵌合在DNA双链中,因此,引物的特异性尤为重要。设计引物时,尽量避免引物二聚体、引物发卡结构等对结果的干扰,并通过熔解曲线鉴定扩增产物是否特异。如图 3-A、3-B所示,本研究设计的内参引物对与外源基因检测引物对的扩增曲线、熔解曲线均在86℃附近有且只有一个单峰,两引物对均为特异性扩增。且两引物对的目的片段大小相近(分别为130 bp和127 bp),Tm值基本相等,扩增效率基本相同。由于实时荧光定量PCR所需起始模板量小、检测灵敏,在实验设计中每个样品均做3个重复,每个实验重复3次。以上措施更加保证了检测结果的真实可信。
当然,验证mtDNA靶向目标序列剪切活性的方法并不单一。如Bacman等[13]在酵母菌中利用单链复性(Single-strand annealing,SSA)[27],检测14459A-mitoTALEN的靶向剪切特异性。如发生特异性剪切,则促进同源重组恢复Lac-Z基因功能,得到蓝色菌落,反之为白色菌落。与本研究所构建的含有靶向目标序列的转基因细胞系相比,虽检测时间较短,但研究者可在构建重组克隆质粒时,将多个靶向目标序列或潜在发生非特异性剪切的序列串联整合到核基因组中,灵活多变。一个细胞株不仅可以检测多个靶向目标序列的剪切活性、比较不同靶向目标序列的剪切效率(图 4-A,T1、T2均具有靶向剪切活性,且T1剪切效率大于T2),还可以分析单碱基或多碱基不匹配下的潜在脱靶效应等问题,为更好的选择靶向目标序列、设计线粒体核酸酶提供了重要的参考依据和实验基础,一举多得。
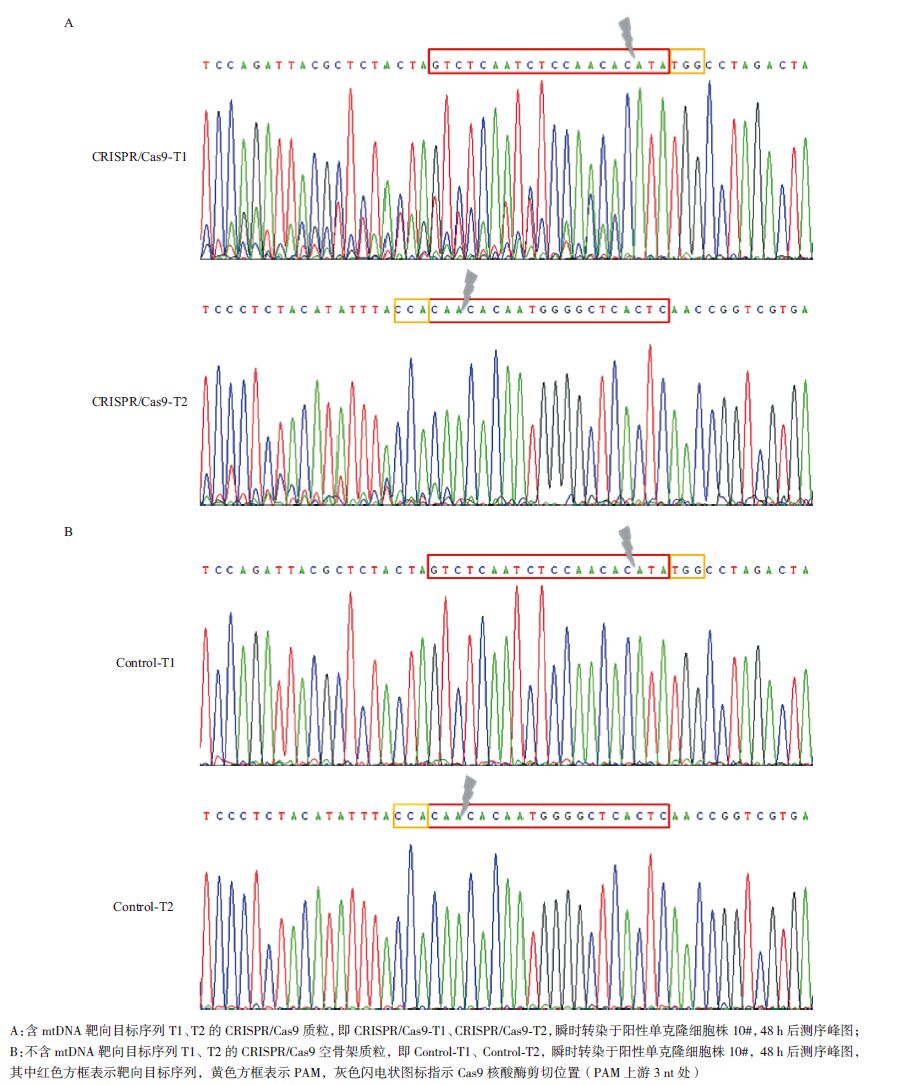
|
| 图 4 T1、T2 靶向目标序列剪切活性检测 |
本研究利用转基因技术,将一段同时含有两个靶向目标序列(T1、T2)的mtDNA序列整合到宿主基因组中。通过3对扩增不同位置的外源基因特异性引物对,筛选得到阳性单克隆细胞株后,利用实时荧光定量PCR法检测阳性单克隆细胞株中的外源基因拷贝数,挑选其中单拷贝或低拷贝的阳性单克隆细胞株。将含有相应靶向目标序列(T1、T2)的CRISPR/Cas9质粒瞬时转染到所选细胞株中,验证T1、T2的靶向剪切活性的同时证明了该方法的可行性。建立了一种高效检测线粒体核酸酶靶向剪切活性的新方法。
| [1] | Perez EE, Wang J, Miller JC, et al. Establishment of HIV-1 resistance in CD4+ T cells by genome editing using zinc-finger nucleases[J]. Nature Biotechnology, 2008, 26(7):808-816. |
| [2] | Urnov FD, Miller JC, Lee YL, et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases[J]. Nature, 2005, 435(7042):646-651. |
| [3] | Baker M. Gene-editing nucleases[J]. Nature Methods, 2012, 9(1):23-26. |
| [4] | Sternberg SH, Redding S, Jinek M, et al. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9[J]. Nature, 2014, 507(7490):62-67. |
| [5] | Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9[J]. Science, 2014, 346(6213):1258096. |
| [6] | Pozzan T, Magalhaes P, Rizzuto R. The comeback of mitochondria to calcium signalling[J]. Cell Calcium, 2000, 28(5-6):279-283. |
| [7] | Starkov AA. The role of mitochondria in reactive oxygen species metabolism and signaling[J]. Annals of the New York Academy of Sciences, 2008, 1147:37-1152. |
| [8] | Anderson S, Bankier AT, Barrell BG, et al. Sequence and organization of the human mitochondrial genome[J]. Nature, 1981, 290(5806):457-465. |
| [9] | Schapira AH. Mitochondrial disease[J]. Lancet, 2006, 368(9529):70-82. |
| [10] | Obara-Moszynska M, Maceluch J, Bobkowski W, et al. A novel mitochondrial DNA deletion in a patient with Kearns-Sayre syndrome:a late-onset of the fatal cardiac conduction deficit and cardiomyopathy accompanying long-term rGH treatment[J]. BMC Pediatrics, 2013, 13:27. |
| [11] | Minczuk M, Papworth MA, Kolasinska P, et al. Sequence-specific modification of mitochondrial DNA using a chimeric zinc finger methylase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(52):19689-19694. |
| [12] | Minczuk M, Papworth MA, Miller JC, et al. Development of a single-chain, quasi-dimeric zinc-finger nuclease for the selective degradation of mutated human mitochondrial DNA[J]. Nucleic Acids Research, 2008, 36(12):3926-3938. |
| [13] | Bacman SR, Williams SL, Pinto M, et al. Specific elimination of mutant mitochondrial genomes in patient-derived cells by mitoTALENs[J]. Nature Medicine, 2013, 19(9):1111-1113. |
| [14] | Croteau DL, Stierum RH, Bohr VA. Mitochondrial DNA repair pathways[J]. Mutation Research, 1999, 434(3):137-148. |
| [15] | Liu P, Demple B. DNA repair in mammalian mitochondria:Much more than we thought?[J]. Environmental and Molecular Mutagenesis, 2010, 51(5):417-426. |
| [16] | Boesch P, Weber-Lotfi F, Ibrahim N, et al. DNA repair in organelles:Pathways, organization, regulation, relevance in disease and aging[J]. Biochim Biophys Acta, 2011, 1813(1):186-200. |
| [17] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096):816-821. |
| [18] | Xue W, Chen S, Yin H, et al. CRISPR-mediated direct mutation of cancer genes in the mouse liver[J]. Nature, 2014, 514(7522):380-384. |
| [19] | Hu W, Kaminski R, Yang F, et al. RNA-directed gene editing specifically eradicates latent and prevents new HIV-1 infection[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(31):11461-11466. |
| [20] | Long C, McAnally JR, Shelton JM, et al. Prevention of muscular dystrophy in mice by CRISPR/Cas9-mediated editing of germline DNA[J]. Science, 2014, 345(6201):1184-1188. |
| [21] | O’Connell MR, Oakes BL, Sternberg SH, et al. Programmable RNA recognition and cleavage by CRISPR/Cas9[J]. Nature, 2014, 516(7530):263-266. |
| [22] | Schon EA, DiMauro S, Hirano M. Human mitochondrial DNA:roles of inherited and somatic mutations[J]. Nature Reviews Genetics, 2012, 13(12):878-890. |
| [23] | Yang L, Ding J, Zhang C, et al. Estimating the copy number of transgenes in transformed rice by real-time quantitative PCR[J]. Plant Cell Reports, 2005, 23(10-11):759-763. |
| [24] | Ingham DJ, Beer S, Money S, et al. Quantitative real-time PCR assay for determining transgene copy number in transformed plants[J]. Biotechniques, 2001, 31(1):132-134, 6-40. |
| [25] | Song P, Cai C, Skokut M, et al. Quantitative real-time PCR as a screening tool for estimating transgene copy number in WHISKERSTM-derived transgenic maize[J], Plant Cell Reports, 2002, 20(10):948-954. |
| [26] | 王晓建, 杨旭, 宋晓东, 等. 实时荧光定量PCR法检测转基因小鼠拷贝数[J]. 中国实验动物学报, 2007(3):170-174. |
| [27] | Liddell L, Manthey G, Pannunzio N, et al. Quantitation and analysis of the formation of HO-endonuclease stimulated chromosomal translocations by single-strand annealing in Saccharomyces cerevisiae[J]. Journal of Visualized Experiments, 2011(55):e3150. |







