植物生长通常会受到各种逆境因素的制约。逆境胁迫会引起植物体内一系列生理代谢的变化,抑制植物的生长发育,甚至会导致植物死亡。在长期进化过程中,植物逐渐形成了其自身所特有的形态特征和相应的逆境响应机制。如盐穗木和仙人掌的叶片或茎等器官高度肉质化,为防止水分过度蒸发,以适应荒漠高温和干旱的环境。植物形成这种自身保护机制是多方面的,除了特殊的自身形态结构特征外,在逆境胁迫下植物会改变信号通路,激活相应的转录因子,通过转录调控来提高植物的胁迫耐受性[1]。逆境胁迫通常会造成植物细胞内活性氧(Reactive oxygen species,ROS)物质的过度积累[2],最终导致氧化胁迫的产生。ROS能够直接与蛋白质,核酸和脂类等物质结合,从而导致DNA损伤,植物体内酶失活及细胞渗透压改变等一系列的过氧化链式反应,危害细胞膜系统正常生理功能,甚至直接导致植物死亡[3, 4]。ROS还能够促使膜脂的过氧化导致化学反应的产生,生成相应的醛类物质,造成醛类物质在生物体内积累。过量的醛会与蛋白质和核酸反应,破坏蛋白质和核酸的正常结构和功能。为了降低这些活性氧给细胞带来的毒害作用,植物体内会诱导醛脱氢酶(Aldehyde dehydrogenases,ALDHs)基因的表达水平,大量累积醛脱氢酶蛋白,促使对自身具有毒害作用的醛类物质氧化成相应的羧酸,并减少脂类物质的过氧化,从而提高植物的耐受性[5]。
醛脱氢酶(ALDHs)是一大类NAD(P)+依赖催化酶,它催化活性醛物质氧化成相应的羧酸类物质。在结构上,Perozich等[5]提出醛脱氢酶至少含有7-10个保守基序,这些序列中大部分β折叠区是高度保守的。这些区域一般含有3个特征序列:谷氨酸活性位点、Rossmann折叠辅酶结合位点和硫醇催化位点,这些位点可能是ALDH具备催化活性的基本特征。近来,Jimenez-Lopez等[6]对玉米的ALDH蛋白家族结构进行分析时发现,玉米ALDH2B2基因产物能调节其他配体与之结合;氨基酸序列分析发现,玉米ALDH2B2蛋白通道主要是由中性或疏水性残基组成,可结合大的亲脂性和疏水分子,能阻挡中长链脂肪分子对线粒体的损伤。目前,ALDHs在动物、植物及微生物中均有发现。研究表明,ALDH 在植物的不同组织中广泛表达,参与到植物对生物及非生物条件下的胁迫适应以及植物的发育调节[7],在植物中其功能主要包括氧化醛类物质、参与代谢以及提高植物的胁迫耐受能力。例如,念珠藻在响应干旱、盐、ABA和紫外(UV)胁迫时,ALDH21A1基因的转录水平升高[8];在厌氧条件下,过表达水稻ALDH2a蛋白可以氧化对植物产生毒害的乙醛,使得水稻在缺氧环境下耐受能力增强[9]。高盐诱导下的拟南芥 ALDH12基因编码合成 P5ADH酶,促使脯氨酸的合成[10],对植株体内渗透调节物质的积累起到关键作用。
本文将主要从醛脱氢酶的分类、功能及作用途径进行阐述。
1 植物中醛脱氢酶的分类醛脱氢酶家族最初是由Wood等[11]由藓类和藻类的研究中提出的。而后Jimenez-Lopez、Kirch和Gao等又分别对玉米、拟南芥和水稻的ALDH超家族进行了描述和总结[6, 12, 13]。Kotchoni等[14]进行了修正,详细地概述了水稻的ALDH超家族。目前在真核生物中发现了至少172个ALDH基因,根据醛脱氢酶基因命名委员会建立的分类标准进行分类,超过40% 序列与先前确定的蛋白序列相同则构成一族,超过60% 完全相同的则构成一个亚族,低于40% 完全相同的则为一个新的醛脱氢酶家族[15]。研究发现,ALDH超家族分属于20多个蛋白家族,根据同源性序列比对表明它们之间具有高度保守性[16]。真核生物中ALDHs根据序列保守程度可分为24个蛋白家族,在植物中发现13个ALDH超家族,包括:ALDH2、ALDH3、ALDH5、ALDH6、ALDH7、ALDH10、ALDH11、ALDH12、ALDH18、ALDH21、ALDH22、ALDH23和ALDH24。其中AL-DH10、ALDH12、ALDH21、ALDH22、ALDH23和ALDH24家族是植物所特有的。ALDH2、ALDH3、ALDH5、ALDH6、ALDH7和ALDH18家族与哺乳动物ALDH有较高的同源性[17, 18]。其中表 1列举植物各类醛脱氢酶及相关功能。
2 植物醛脱氢酶的生物学功能醛脱氢酶广泛分布于植物种属中,在功能方面的多样性充分体现了其在生物体中的重要性。生物体中的醛主要来源于膜脂过氧化、蛋白质糖基化和氨基酸氧化。它们具有较强的亲电子能力,易于核酸和蛋白等结合导致DNA损伤,破坏细胞稳态[3]。醛脱氢酶是一类广泛参与代谢内源性和外源性醛的酶类,参与植物的生长发育和应答植物遭受的生物及非生物胁迫等。醛脱氢酶可以通过结合NAD+或者NADP+催化内源性或外源性醛类物质氧化脱氢生成相应的羧酸类物质[20],从而减轻醛类物质在植物体内的积累,达到其解毒的目的,进而能够缓解在非生物胁迫条件下对植物产生的毒害,提高植物耐受能力。另一方面,醛脱氢酶还与生物因素产生的胁迫相关,在生物胁迫条件下同样能够诱使醛脱氢酶基因的上调表达。
2.1 植物醛脱氢酶参与植物的生长发育醛脱氢酶在许多植物的生长发育中发挥着重要作用。第一个被验证的植物ALDH2基因(RF2)是细胞质雄性不育(Cytoplasmic male sterility,CMS)的恢复基因,它存在于线粒体中,编码一个线粒体的ALDH2蛋白,是玉米雄蕊发育所必须的。当RF2基因发生突变时,花药绒毡层细胞功能就会受到严重损伤,造成花药败育。所以,RF2基因在玉米功能性雄配子体产生过程中发挥重要作用[21, 22]。ALDH18编码的Δ1-吡咯-5-羧酸合成酶(Δ1-pyrroli-ne-5-carboxylate,P5CS2),P5CS2定位在叶原基中,在胚胎发育中起到了重要作用,其突变会导致种子在发育晚期阶段死亡以及幼苗畸形[18, 23]。烟草线粒体的两个醛脱氢酶基因TobALDH2a和TobALDH2b主要在生殖组织中表达,TobALDH的活性能参与到花粉管的生长[24]。
2.2 植物醛脱氢酶对逆境胁迫的响应醛脱氢酶不仅参与调控植物的生长和发育,还参与到生物及非生物胁迫的调控。Wen等[25]利用葡萄白粉菌(Uncinula necator)和霜霉菌(Plasmopara viticola)处理中国野生华东葡萄,结果表明,VpALDH2B4基因分别在白粉菌和霜霉菌侵染一定时间后表达水平达到最高,且霜霉菌诱导表达水平较白粉菌的更为显著。将 VpALDH2B4基因转入拟南芥进行异源表达,能够增强拟南芥抗霜霉病能力。醛脱氢酶基因也参与到植物应对干旱、高盐、冷、水浸、氧化、紫外和重金属胁迫的应答[26, 27]。植株在受到离子胁迫时,促使植物体内活性氧的累积,也促使如丙二醛(Malondialdehyde,MDA)等醛类物质生成,丙二醛的总量决定着植物细胞中脂质的过氧化反应水平。在转基因拟南芥中MDA的含量显著低于野生型植株,这可能就是由于转基因植株体内高水平的ALDH氧化MDA所致。同时,高水平的ALDH还能激活抗氧化剂的生成,抑制活性氧物质在植株体内的累积,增强植物的抗氧化能力。Xu等[28]将葡萄线粒体中的ALDH2B8基因转入到拟南芥中过表达,能够增强拟南芥的盐胁迫耐受及抗氧化能力。
植物响应干旱胁迫的过程是复杂的。Kirch等[29]在研究复苏植物Craterostigma plantagineum的醛脱氢酶发现,Cp-ALDH与 ALDH3I1的氨基酸同源性为72%,属ALDH3家族。Cp-ALDH具有甘油醛-3-磷酸脱氢酶活性,免疫胶体金定位结果表明Cp-ALDH倾向于淀粉颗粒。作者推测Cp-ALDH除参与醛类的脱毒外,还可能参与糖和淀粉的代谢。而早期研究报道糖代谢在干旱胁迫中起关键作用,这为ALDH3在干旱胁迫中起到重要作用提供了依据。Sunkar等[30]研究也表明在干旱、重金属和过氧化氢(H2O2)处理拟南芥时,能够诱导Ath-ALDH3上调表达,而转有Ath-ALDH3基因的拟南芥植株在盐和干旱胁迫下膜脂过氧化衍生的醛类物质含量低于野生型。比较分析拟南芥的9个醛脱氢酶家族,属于两个不同家族的醛脱氢酶基因ALDH3I1、ALDH3H1和ALDH7B4在不同组织中,脱水和高盐胁迫和脱落酸(ABA)处理下被特异激活,从而提高植物对胁迫的抵抗能力[15]。无芒隐子草中的醛脱氢酶基因CsALDH12A1能够增强转基因拟南芥的干旱耐受能力[31]。ALDH7蛋白主要累积在植物的茎秆部位,可以提高植物对干旱、盐和氧化胁迫抗性能力,从而增强拟南芥和烟草交叉耐受多重胁迫的能力[21],水稻的OsALDH7基因能够受非生物胁迫诱导[32, 33]。ALDH10家族也是一类已被广泛证明可以提高植物对环境胁迫,如高盐、干旱和冷的抗性。由于ALDH10家族主要编码甜菜碱醛脱氢酶(BADH),能催化甜菜醛生成相应的甜菜碱,在植物抵抗渗透胁迫中发挥作用[11, 20]。胁迫条件下ALDH18基因表达也会上调,促使ALDH18编码的P5CS2合成脯氨酸,脯氨酸的作用累积类似于甜菜碱,是一种重要的渗透保护物质,在植物的抗性生理中发挥着重要作用[10]。
ALDH5F1编码一个拟南芥琥珀半醛脱氢酶,该基因发生突变会导致植物生长矮小,并伴随坏死病斑,同时会增加植物对紫外光和热激处理的敏感性[34]。水稻的OsALDH7与水稻的种子成熟及种子的活性有关,种子中OsALDH7缺失会造成丙二醛和黄色素在种子干燥和贮藏过程中积累,促进种子的老化,在种子的生存能力和解除醛类物质累积造成的毒害过程中起重要作用[32, 33]。对水稻的ALDH1a和ALDH2a基因研究较多,其中醛脱氢酶 ALDH1a定位在细胞质中,ALDH2a定位在线粒体中。Nakaz-ono等[9]将水稻幼苗从有氧光照转移到有氧黑暗条件下发现,水稻幼叶中ALDH2a mRNA的总量会有轻微的下降,推测ALDH2a并不是简单的通过光调节,而可能通过调节黄化或绿色幼苗中某些方面的代谢。
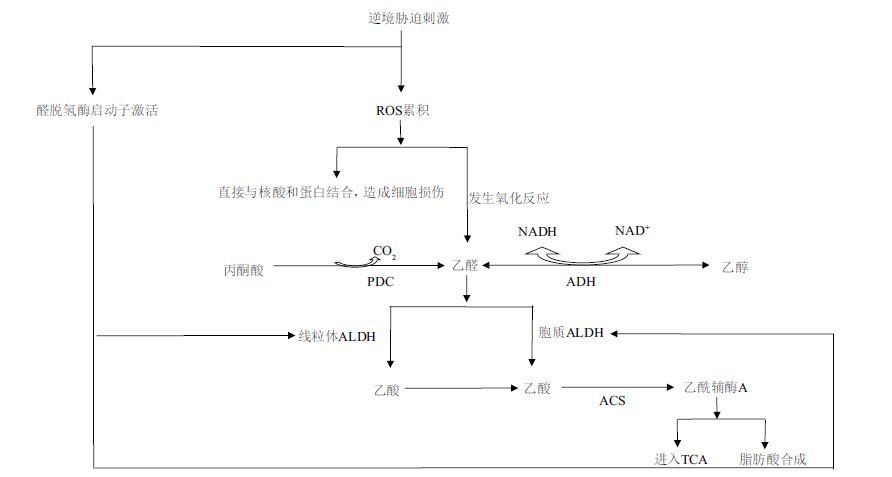
3 植物醛脱氢酶的作用途径植物受到逆境胁迫时会产生相应的机制来应对。例如,植物受到外界刺激会导致活性氧积累,促使膜脂过氧化导致醛类物质产生的同时,也会促使ALDH相关启动子的激活。对拟南芥ALDH7B4基因启动子的分析表明,在机械损伤应答中,虽然ALDH7B4基因启动子被激活的机制尚不清楚,但是机械损伤响应是由茉莉酸或者茉莉酸相关化合物植物二烯酸调控;当细胞受到损伤时,细胞壁的相关元件会释放糖醛酸从而激活ALDH7B4基因的启动子,促使ALDH7B4基因的表达。研究还发现ALDH启动子区具有如ACGT1、ACGT2和ACGT3的基序,这些临近翻译起始密码子的序列是盐和旱响应所必须的元件。如果剔除ACGT1或者ACGT2和ACGT3基序,会导致启动子活性降低甚至削弱盐、旱耐受能力[35]。
植物受到逆境胁迫时,其体内的糖酵解和酒精发酵对植物能量的产生起重要作用。酒精发酵作为植物应对逆境胁迫的一条途径,其反应过程一般包括两步。首先是丙酮酸脱羧酶(Pyruvate decarboxylase,PDC)催化丙酮转变成乙醛,而后在乙醇脱氢酶(Alcohol dehydrogenase,ADH)作用下催化乙醛转变成乙醇,最终完成整个发酵过程。这一反应中NAD+被重复利用,在这个代谢途径中维持在缺氧条件下糖酵解和ATP水平。在缺氧情况下,许多参与糖酵解和乙醇发酵过程的基因被强烈诱导表达。如水稻线粒体的ALDH2a在水淹条件下诱导表达,表明其可能参与水稻体内乙醛的氧化作用,从而解除乙醛对水稻在水淹情况下的毒性[9, 36]。
植物应对逆境胁迫的另一条途径可能是PDC-ALDH-ACS通路。在酵母中PDC-ALDH-ACS通路被称作丙酮酸脱氢酶(Pyruvate dehydrogenase,PDH)旁路。植物极有可能是通过此路径代谢其体内所产生的醛类物质。不同组织、细胞中乙酰辅酶A合成酶(Acetyl-coenzyme A synthease,ACS)所起到的作用是不同的[37]。线粒体和细胞质中ACS利用乙酸合成乙酰辅酶A,这一过程早在酵母及哺乳动物体内已经确立[37, 38, 39]。乙酰辅酶A是脂肪酸生物合成和参与三羧酸循环的重要原料。乙酰辅酶A可以看作是活化的乙酸,是辅酶A的乙酰化形式。因而,可通过研究乙酸的生物合成进而探讨ACS怎样促成质体乙酰辅酶A的生成提供一个有效途径。乙酸容易跨膜扩散,但是它在植物中的转运路径却不清楚,其中一个可能的通路与动物及酵母的路径是一致的,即利用ALDH通过PDC催化乙醇发酵生成的乙醛或者利用ADH催化乙醇脱氢获得。最终Kuhlemeier实验室[40, 41]已经确认在烟草花粉中乙醇高效发酵与PDC和ALDH的高表达相一致。他们用14C标记的乙醇供给花粉管生长所需,在脂肪酸中发现标记的原子表明,乙醇能够用于脂肪酸的合成,推测是通过PDC-ALDH-ACS通路完成这一过程[42],但仍需要进一步实验验证,这也为植物的PDH旁路提供了直接证据。
总而言之,植物受到逆境胁迫刺激会促使相应的醛脱氢酶启动子激活,调控胞质及线粒体中ALDH的合成。与此同时,也造成大量ROS物质的累积。ROS一方面直接与核酸或蛋白结合造成细胞损伤;另一方面促使氧化反应的发生,生成相应的乙醛类物质。乙醛进入植物体内,在线粒体和胞质中ALDH的作用下被氧化生成相应的乙酸,乙酸在ACS的作用下生成乙酰辅酶A,一部分乙酰辅酶A进入TCA循环氧化供能,另一部分用于脂肪酸合成。此外,通过酒精发酵代谢途径也促使部分乙醛类物质的生成。植物体内的丙酮酸在丙酮酸脱羧酶(PDC)作用下脱去一个CO2,生成乙醛,部分乙醛会被ADH催化还原生成乙醇;另一部分进入到细胞质基质以及线粒体,被氧化生成乙酸最终进入到三羧酸循环(TCA)以及脂肪酸的合成。其大致途径,如图 2。
4 展望植物响应逆境胁迫的机制较为复杂。不同种类的醛脱氢酶,在植物体内发挥着不同作用。对ALDH大部分研究集中在ALDH的作用、逆境胁迫下与ALDH的表达和活性关系以及异源表达ALDH提高植物的胁迫耐受性等方面。也有一些途径已逐渐被人们所认知,如PDC-ALDH-ACS旁路可能是ALDH缓解逆境胁迫的一条途径。同一植物对不同胁迫响应的信号途径也可能是不同的。到目前为止对这些复杂的调控网络的研究很多方面是不清楚的;其次,ALDH对植物生殖器官的发育也起到重要作用,但是相关研究大多处于一个表观状态,如某些基因缺失会引起败育,但其如何参与调节这一过程的机理还需进一步探索;植物的ALDH 相关功能已被认知,是否还有其他功能,不得而知。已知的ALDH会作用到哪些醛类物质也需要充分确认,为我们将来充分利用这类物质提供依据;第三,部分醛脱氢酶的生化功能及进化动力尚不明确。如ALDH23和ALDH24只存在于苔藓和藻类较低等植物中,而在高等植物中的相关报道极少。所以,对植物ALDH蛋白的体外研究,以及ALDH在缓解逆境胁迫和在植物的生长发育方面的具体机理探索将会成为ALDH研究的一个方向。最后,通过转基因技术将这些酶类基因转化到对干旱和盐碱耐受性较弱的作物体内,将会在一定程度上改良作物耐盐、旱的品质,提高这些作物对不良环境的适应能力,对干旱区盐碱地农业的发展具有重大的现实意义。
| [1] | Wang W, Vinocur B, Altman A. Plant responses to drought, salinity and extreme temperatures:towards genetic engineering for stress tolerance[J]. Planta, 2003, 218:1-14. |
| [2] | Singh S, Brocker C, Koppaka V, et al. Aldehyde dehydrogenases in cellular responses to oxidative /electrophilic stress[J]. Free Radical Bio Med, 2013, 56:89-101. |
| [3] | Jacobs AT, Marnett LJ. Systems analysis of protein modification and cellular response induced by electrophile stress[J]. Accounts Chem Res, 2010, 43:673-683. |
| [4] | Nadkarni DV, Sayre LM. Structural definition of early lysine and histidine adduction chemistry of 4-hydroxynonenal[J]. Chem Res Toxicol, 1995, 8:248-291. |
| [5] | Perozich J, Nicholas H, Wang BC, et al. Relationships within the aldehyde dehydrogenase extended family[J]. Protein Sci, 1999, 8:137-146. |
| [6] | Jimenez-Lopez JC, Gachomo EW, Seufferheld MJ, et al. The maize ALDH protein superfamily:linking structuralfeatures to functional specificities[J]. BMC Struc Bio, 2010, 10:43. |
| [7] | Missihoun TD, Schmitz J, Klug R, et al. Betaine aldehyde dehydrogenase genes from Arabidopsis with different sub-cellular localization affect stress responses[J]. Planta, 2011, 233:369-382. |
| [8] | Chen XB, Zeng Q, Andrew JW. The stress-responsive Tortula ruralis gene ALDH21A1 describes a novel eukaryotic aldehyde dehydrogenase prorein family[J]. Plant Physiol, 2002, 159(7):677-684. |
| [9] | Nakazono M, Tsuji H, Li Y, et al. Expression of a gene encoding mitochondrial aldehyde dehydrogenase in rice increases under submerged conditions[J]. Plant Physiol, 2000, 124:587-598. |
| [10] | Deuschle K, Funck D, Hellmann H, et al. A nuclear gene encoding mitochondrial Δ1-pyrroline-5-carboxylate dehydrogenase and its potential role in protection from proline toxicity[J]. Plant J, 2001, 27(4):345-356. |
| [11] | Wood AJ, Duff RJ. The aldehyde dehydrogenase(ALDH)gene superfamily of the moss Physcomitrella patens and the algae Chlamydomonas reinhardtii and Ostreococcus tauri[J]. Bryologist, 2009, 112:1-11. |
| [12] | Kirch HH, Bartels D, Wei Y, et al. The ALDH gene superfamily of Arabidopsis[J]. Trends Plant Sci, 2004, 9:371-377. |
| [13] | Gao C, Han B. Evolutionary and expression study of the aldehyde dehydrogenase(ALDH)gene superfamily in rice(Oryza sativa)[J]. Gene, 2009, 431(1-2):86-94. |
| [14] | Kotchoni SO, Jimenez-Lopez JC, Gao D, et al. Modeling-dependent protein characterization of the rice aldehyde dehydrogenase(ALDH)superfamily reveals distinct functional and structural features[J]. PLoS One, 2010, 5:e11516. |
| [15] | Kirch HH, Schlingensiepen S, Kotchoni S, et al. Detailed expression analysis of selected genes of the aldehyde dehydrogenase(ALDH)gene superfamily in Arabidopsis thaliana[J]. Plant Mol Bio, 2005, 57:315-332. |
| [16] | Sophos NA, Vasiliou V. Aldehyde dehydrogenase gene superfamily:the 2002 update[J]. Chem Biol Interact, 2003, 143-144:5-22. |
| [17] | Zhang Y, Mao L, Wang H, et al. Genome-wide identification and analysis of grape aldehyde dehydrogenase(ALDH)gene superfamily[J]. PLoS One, 2012, 7:e32153. |
| [18] | Brocker C, Vasiliou M, Carpenter S, et al. Aldehyde dehydrogenase(ALDH)superfamily in plants:gene nomenclature and comparative genomics[J]. Planta, 2013, 237:189-210. |
| [19] | Huang W, Ma X, Wang Q, et al. Significant improvement of stress tolerance in tobacco plants by overexpressing a stress-responsive aldehyde dehydrogenase gene from maize(Zea mays)[J]. Plant Mol Biol, 2008, 68:451-463 |
| [20] | Rodrigues SM, Andrade MO, Gomes AP, et al. Arabidopsis and tob-acco plants ectopically expressing the soybean antiquitin-like ALDH7 gene display enhanced tolerance to drought, salinity, and oxidative stress[J]. J Exp Bot, 2006, 57:1909-1918. |
| [21] | Cui X, Wise RP, Schnable PS. The rf2 nuclear restorer gene of male-sterile T-cytoplasm maize[J]. Science, 1996, 272:1334-1336. |
| [22] | Liu F, Schnable PS. Functional specialization of maize mitochond-rial aldehyde dehydrogenases[J]. Plant Physiol, 2002, 130:1657-1674. |
| [23] | Szekely G, Abraham E, Cseplo A, et al. Duplicated P5CS genes of Arabidopsis play distinct roles in stress regulation and developmental control of proline biosynthesis[J]. Plant J, 2008, 53:11-28. |
| [24] | Op Den Camp RGL, Kuhlemeier C. Aldehyde dehydrogenase in tobacco pollen[J]. Plant Mol Biol, 1997, 35:355-365. |
| [25] | Wen Y, Wang X, Xiao S, et al. Ectopic expression of VpALDH2B4, a novel aldehyde dehydrogenase gene from Chinese wild grapevine(Vitis pseudoreticulata), enhances resistance to mildew pathogens and salt stress in Arabidopsis[J]. Planta, 2012, 236:525-539. |
| [26] | Chugh V, Kaur N, Gupta AK. Role of antioxidant and anaerobic metabolism enzymes in providing tolerance to maize(Zea mays L.)seedlings against waterlogging[J]. Biochem Biol, 2011, 48:346-352. |
| [27] | Yang H, Zhang D, Wang J, et al. Molecular cloning of a stress responsive aldehyde dehydrogenase gene ScALDH21 from the desiccation tolerant moss Syntrichia caninervis and its responses to different stresses[J]. Mol Biol Rep, 2012, 39(3):2645-2652. |
| [28] | Xu X, Guo R, Cheng C, et al. Overexpression of ALDH2B8, an aldehyde dehydrogenase gene from grapevine, sustains Arabidopsis growth upon salt stress and protects plants against oxidative stress[J]. Plant Cell Tiss Org, 2013, 114:187-196. |
| [29] | Kirch HH, Nair A, Bartels D. Novel ABA- and dehydration-inducible aldehyde dehydrogenase genes isolated from the resurrection plant Craterostigma plantagineum and Arabidopsis thaliana[J]. Plant J, 2001, 28:555-567. |
| [30] | Sunkar R, Bartels D, Kirch HH. Overexpression of a stress inducible aldehyde dehydrogenase gene from Arabidopsis thaliana in transgenic plants improves stress tolerance[J]. Plant J, 2003, 35:452-464. |
| [31] | Bouche N, Fait A, Bouchez D, et al. Mitochondrial succinic-semialdehyde dehydrogenase of the gamma-aminobutyrate shunt is required to restrict levels of reactive oxygen intermediates in plants[J]. Proc Natl Acad Sci USA, 2003, 100:6843-6848. |
| [32] | Shin JH, Kim SR, An G. Rice aldehyde dehydrogenase7 is needed for seed maturation and viability[J]. Plant Physiol, 2009, 149:905-915. |
| [33] | Shen Y, Zhang Y, Yang C, et al. Mutation of OsALDH7 causes a yellow-colored endosperm associated with accumulation of oryzamutaic acid A in rice[J]. Planta, 2012, 235:433-441. |
| [34] | Zhang J, Duan Z, Jahufer Z, et al. Stress-inducible expression of a Cleistogenes songorica ALDH gene enhanced drought tolerance in transgenic Arabidopsis thaliana[J]. Plant Omics J, 2014, 7(6):438-444. |
| [35] | Missihoun TD, Hou Q, Dorothea Bartels DM. Sequence and functi-onal analyses of the aldehyde dehydrogenase 7B4 gene promoter in Arabidopsis thaliana and selected Brassicaceae:regulation patterns in response to wounding and osmotic stress[J]. Planta, 2014, 239:1281-1298. |
| [36] | Li Y, Nakazono M, Tsutsumi N, et al. Molecular and cellular characterizations of a cDNA clone encoding a novel isozyme of aldehyde dehydrogenase from rice[J]. Gene, 2000, 249:67-74. |
| [37] | Lin M, Behal R, Oliver DJ. Disruption of plE2, the gene for the E2 subunit of the plastid pyruvate dehydrogenase complex, in Arabidopsis causes an early embryo lethal phenotype[J]. Plant Mol Biol, 2003, 52(4):865-872. |
| [38] | Ke J, Behal RH, Back SL, et al. The role of pyruvate dehydrogenase and acetyl-coenzyme A synthetase in fatty acid synthesis in develo-ping Arabidopsis seeds[J]. Plant Physiol, 2000, 123(2):497-508. |
| [39] | Behal RH, Lin M, Back S, et al. Role of acetyl-coenzyme A synthe-tase in leaves of Arabidopsis thaliana[J]. Arch Biochem Bio-phys, 2002, 402(2):259-267. |
| [40] | Tadege M, Kuhlemeier C. Aerobic fermentation during tobacco pollen development[J]. Plant Mol Biol, 1997, 35(3):343-354. |
| [41] | Mellema S, Eichenberger W, Rawyler A, et al. The ethanolic fermentation pathway supports respiration and lipid biosynthesis in tobacco pollen[J]. Plant J, 2002, 30(3):329-336. |
| [42] | Wei Y, Lin M, Oliver DJ, et al. The roles of aldehyde dehydrogenases(ALDHs)in the PDH bypass of Arabidopsis[J]. BMC Biochem, 2009, 10:7. |