MicroRNAs(miRNAs)是一类长约22个碱基的非编码单链小分子RNA,普遍存在于各种生物中[1, 2, 3, 4],通过与靶基因的3'端非翻译区(3'-UTR)的互补结合[5, 6],进而诱导靶mRNA 的切割降解、翻译抑制等其他调节机制在转录后水平调控基因表达[7]。已有报道表明,多达30%的基因成为了miRNA的潜在靶基因[8, 9],因而miRNA靶标的检测逐渐成为研究miRNA功能的重要环节。全基因组研究已经证实了miRNA在哺乳动物性腺的发育中起着举足轻重的作用。我们先前已在牙鲆(Paralichthys olivaceus)性腺中鉴定了 141个成熟miRNA,有些miRNA如pol-miR-143、pol-let-7a等在精卵巢中均有较高表达,而一些miRNA如pol-miR-26a、pol-miR-26b等则存在明显的性别差异表达[10]。在猪的卵巢中,miR-26b被报道可通过直接下调ATM基因的表达而体外诱导颗粒细胞凋亡[11]。我们通过公共数据库 TargetScan、PicTar和miRBase等靶基因预测软件发现,牙鲆空通气孔同源框2基因(Empty spiracles homeobox 2,emx2)的3'UTR区可与pol-mir-26a,-26b完全互补结合从而可能成为其靶基因。
emx2基因是果蝇ems(Empty spiracles)的同源结构域基因,含同源转录因子,在果蝇头部的早期发育过程中发挥重要作用。脊椎动物emx2位于同源盒基因(Homeoticgenes,hox)家族的外侧,在哺乳动物发育中的大脑表达并对端脑背侧、大脑皮层和中枢神经系统发育起着关键作用[12, 13, 14]。近几年来有研究者发现,哺乳动物Emx2基因在子宫内膜上皮细胞中是一种必需的增殖抑制基因[15];而在发育中的泌尿生殖系统以及能形成排泄器官和生殖系统的原基细胞中也有表达,并呈生殖周期性变化[16, 17]。缺乏EMX2可使老鼠患特纳综合症即性腺发育不完全[18]。生殖周期中自然发生的EMX2表达下降对正常的增殖、蜕膜化等都是必需的。目前,对此基因在鱼类性腺发育中的功能及其与miRNA的关联研究甚少。因此,本研究以牙鲆emx2基因为例,克隆了emx2 3'UTR区,并对其潜在的pol-miR-26a-26b可能结合位点,即miRNA识别靶mRNA的“种子序列”进行定点诱变的基础上,构建野生型和突变型emx2基因 3'UTR双荧光素酶报告载体,旨在为后续研究miRNA通过调节emx2基因的表达而调控牙鲆性腺发育的作用机制奠定基础。
1 材料与方法 1.1 材料Trizol Reagent(Invitrogen 公司,美国);M-MLV、Rnasin inhibitor、Dpn I酶和psiCHECKTM-2靶标克隆载体(Promega,美国);DL2000、1 kb DNA Ladder Marker、PrimeSTAR HS DNA-Polymerse、T4 DNA Ligase、Not I、Xho I、TaKaRa TaqTM(TaKaRa公司,日本);DNA 凝胶回收试剂盒、质粒提取试剂盒(DONGSHENG BIOTECH,中国);E.coli DH5α菌株为本实验室保存。
1.2 方法 1.2.1 miRNA靶点预测根据先前我们已测序得到的牙鲆miRNA和emx2基因序列,通过靶基因预测软件 miRBase(http://www.mirbase.org/),PicTar WEB INTERFACE(http://pictar.mdc-berlin.de/cgi-bin/PicTar_vertebrate.cgi),TargetScanFish 6.2(http://www.targetscan.org/fish_62/)和computer-based RNAhybrid(http://bibiserv.techfak.uni-bielefeld.de/rnahybrid/submission.html)等预测miRNA与其靶基因3'UTR 区结合位点。
1.2.2 牙鲆总RNA提取、反转录及RT-PCR钓取基因根据我们已克隆的牙鲆emx2基因序列设计3' UTR区引物,上下游引物5'端分别携带Xho I、NotI 酶切位点。用于构建野生型靶标克隆载体的引物为3'-emx2XhoIF:5'-CCGCTCGAGGAGGAAGGCTCGGAGTCTCAG-3',3'-emx2NotIR:5'-ATAAGAATGCGGCCGCGCATTTCTGTACATGGAATACGC-3'。
取刚解剖完的牙鲆成鱼精卵巢混合组织1 g,研磨后采用Trizol法提取精卵巢混合组织总RNA,并将其反转录为cDNA。以cDNA为模板,3'-emx2XhoIF、3'-emx2NotIR为引物,进行PCR扩增。PCR反应在25 μL反应体系中进行,包含2.5 μL 2 mmol/L dNTP mixture、2.5 μL 10×KOD buffer、1.5 μL 25 mmol/L MgSO4、1.5 μL cDNA、0.3 μL KOD PLus Neo、17.1 μL ddH2O、上下游引物各0.3 μL。PCR扩增条件为:94℃预变性3 min;98℃变性15 s,58℃退火15 s,68℃ 延伸 1.5 min,共30个循环。扩增完成后取5 μL产物于1.5%琼脂糖凝胶电泳分析扩增产物的特异性。
采用DONGSHENG公司的DNA凝胶回收试剂盒,对上述目的片段进行回收。回收的目的片段与pMD19-T载体连接,蓝白斑筛选阳性克隆,提取质粒,送上海生工生物工程技术有限公司测序鉴定。
1.2.3 野生型重组报告载体的构建分别取野生型emx2 3'UTR PCR回收产物和psiCHECK-2载体各15 μL,用Xho I/Not I双酶切,在50 μL的酶切体系中加入1.5 μL Xho I、1.5 μL Not I、5 μL 10×buffer、27 μL ddH2O。37℃反应3 h左右。酶切产物采用DONGSHENG公司的DNA凝胶回收试剂盒进行回收。在10 μL目的片段和载体的连接体系中加入3 μL酶切回收的PCR产物、2 μL 酶切回收的载体psiCHECK-2、1 μL 10×Ligase Buffer、1 μL T4 DNA Ligase、3 μL ddH2O。16℃连接2 h,连接物转化到DH5α感受态细胞,将其涂布于含氨苄青霉素(100 μg/mL)的LB平板上,37℃生化培养箱培养过夜,随机挑取若干阳性克隆于3 mL LB管中摇床过夜培养后,采用G-SHUN高纯质粒小量提取试剂盒提取质粒,获得野生型psiCHECK-emx2-3'UTR重组质粒。利用双酶切和测序对阳性重组质粒进行鉴定。
1.2.4 突变型重组报告载体的构建根据miRBase、TargetScanFish 6.2、RNAhybrid预测出的miRNA与其靶基因3'UTR 区的结合位点的序列,对其进行定点突变,将emx2 3'UTR中第971位碱基GACTTGA突变为AGTCCAG。同时结合Primer 5.0 和Oligo 6来设计包含突变序列的引物,委托上海英骏公司合成。引物序列为:mutemx2F:5'-GCACGCAGCCTTTCTGTCAGAGTCCAGCCCTCCAGTGAAAGTTGCCAAG-3';mutemx2R:5'-CTTGGCAACTTTCACTGGAGGGCTGGACTCTGACAGAAAGGCTGCGTGC-3'。
以上述构建好的野生型重组报告载体psiCHEC-K-emx2-3'UTR的阳性质粒为模板,质粒稀释50倍后取1 μL做模板。采用KOD Plus neo DNA Polymerase进行体外定点诱变。在0.2 mL EP管中进行RT-PCR反应。在25 μL反应体系中加入2.5 L 2 mmol/L dNTP mixture、2.5 μL 10×KOD buffer、1.5 μL 25 mmol/L MgSO4、1 μL cDNA、0.3 µL KOD PLus Neo、16.6 μL ddH2O、上下游引物各0.3 μL。PCR 扩增反应:94℃预变性3 min;98℃变性15 s,58℃ 15 s,68℃ 4 min 30 s,共20 个循环。扩增完成后,取1 µL Dpn I酶 37℃ 4 h处理去除带甲基化的原始模板链,取1 μL Dpn I酶处理的DNA转化到DH5α感受态细胞,涂布于含氨苄青霉素(100 μg/mL)的LB平板上,37℃生化培养箱培养过夜,随机挑取若干阳性克隆于3 mL LB管中摇床过夜培养后,采用G-SHUN高纯质粒小量提取试剂盒提取质粒,获得突变型psiCHECK-mutated-emx2-3'UTR重组质粒。利用双酶切和测序对阳性重组质粒进行鉴定。
2 结果 2.1 miRNA靶点预测结果为确定emx2与miRNA之间的关系,通过利用4种生物信息学软件(TargetScan、PicTar、miRBase和RNAhybrid)预测出牙鲆emx2 是pol-miR-26a 和 pol-miR-26b 的潜在靶基因,pol-miR-26a和-26b与emx2 结合位点图,见图 1。
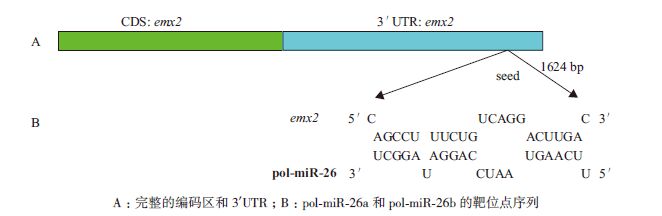
|
| 图 1 牙鲆信使RNA(mRNA)emx2 的序列鉴定 |
采用RT-PCR 法从牙鲆成鱼精卵巢混合组织中成功扩增出emx2 3'UTR长为1 027 bp 的特异性扩增条带(图 2)。与pMD19-T 克隆载体连接后,对阳性克隆进行酶切鉴定并进行DNA 测序,结果显示,测序所得序列与已知序列一致。

|
| 图 2 牙鲆emx2 3'UTR 的克隆 |
将构建的野生型psiCHECK-emx2-3'UTR和突变型psiCHECK-mutated-emx2-3'UTR重组报告载体,用 Xho I/Not I双酶切阳性克隆,得到线性质粒和emx2 3'UTR区基因片段,经电泳分析(图 3)显示,大小分别约为6 000 bp 和1 000 bp,与空质粒和牙鲆emx2 3'UTR 基因大小一致。而图 4的泳道1(大小位于6 000-7 000 bp之间)与图 3泳道1大小接近,表明野生型和突变型重组报告载体均构建成功。
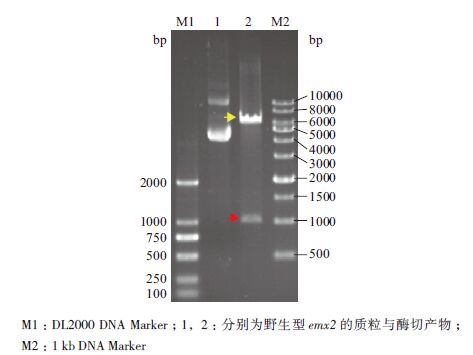
|
| 图 3 野生型重组质粒的双酶切验证 |

|
| 图 4 突变型重组质粒的PCR 验证 |
野生型和突变型质粒测序结果经BLAST分析表明定点突变成功,即emx2 3'UTR区miRNA识别“种子序列”GACTTGA突变为AGTCCAG。
3 讨论miRNA通过与其靶基因结合从而调控靶基因的表达。一些miRNAs通过下调它们作用的靶基因而在哺乳动物性腺发育过程中发挥着重要作用,例如已有报道称miR-26b通过直接下调其靶基因ATM可在体外诱导猪卵巢颗粒细胞凋亡;而且miR-26b的过表达可诱导猪卵巢颗粒细胞的凋亡,在miR-26b的mimic转染组中颗粒细胞的DNA断裂显著上升[11]。同样,在老鼠的卵巢中,mmu-miR-124与精巢发育重要基因SOX9直接结合。在原发性性腺细胞培养中mmu-miR-124的过表达可抑制SOX9基因的表达[19]。Lin等已详细阐述了pol-miR-26是性腺差异表达的miRNA[10, 11, 20],而Pellegrini等发现EMX2调控哺乳动物性腺的发育[17, 18, 21]。为了研究在牙鲆性腺发育中pol-miR-26a、-26b与 emx2之间的相互关系,本研究利用TargetScan、PicTar等软件成功预测了pol-miR-26a、-26b的靶基因并从中选取了性腺发育相关基因emx2,同时预测出pol-miR-26a、-26b与emx2的结合位点。在此基础上,本研究成功克隆了牙鲆emx2 3'UTR区,对其与pol-miR-26a、-26b的结合位点进行了定点突变,从而构建了其野生型和突变型的靶标报告载体。

|
| 图 5 野生型与突变型质粒的比对结果 |
根据罗师平等[22]的报道,盒式突变法、寡核苷酸引物介导的定点突变法及重叠延伸聚合酶链反应定点突变法等是比较常用的定点突变法。但与此同时这些方法操作复杂,非特异性突变较多等,使定点突变的实验受到一定阻力。本研究中采用了一种简便、快速、准确经济的基因定点突变的方法。它非常巧妙地抓住了甲基化的模板质粒对Dpn I酶十分敏感,而合成的突变质粒对Dpn I酶切不敏感这一特性,通过酶切去除模板质粒,只得到突变质粒,使之操作准确无误。此外,本研究所采用的KOD Plus neo DNA Polymerase是公认最好的高保真高效率高速的PCR酶之一(保真性约为Taq酶的80倍),能快速高效的避免延伸过程中不必要的错配。本研究采用低次数的循环延伸而非聚合酶链反应,有助于减少无意错配。整个突变只需20个循环、1次Dpn I酶切和1次转化就完成了定点突变,突变成功率较高,尤其适于DH5α来源的DNA。无需进行亚克隆,也无需对PCR产物进行末端处理等,大大减少了上述传统点突变的工作量,因而本研究通过该种定点突变,成功将emx2 3'UTR区miRNA识别“种子序列”GACTTGA突变为AGTCCAG。
实验采用的靶标克隆载体psiCHECK-2载体[23]是目前使用较多的靶标克隆载体,它可将RNAi与荧光素酶发光相偶联,如本研究中将RNAi的靶片段(emx2 3'UTR区)插入到海肾荧光素酶hRluc下游的多克隆位点中;当RNAi有效时,转录的荧光素酶RNA被降解,无发光反应;当RNAi无效时,转录的荧光素酶RNA被翻译成荧光素并参与发光反应,通过检测发光即可反应RNAi的效果。双报告基因用于实验系统,通常一个报告基因用于内对照,使另一个报告基因的检测均一化。此前使用萤火虫荧光素酶可结合β-半乳糖苷酶(β-Gal)、葡萄醛酸糖苷酶(GUS)或氯霉素乙酰转移酶(CAT)等作为双报告基因,但β-Gal、CAT等检测方式复杂、可检测范围和灵敏度均较差,不是理想的内参。本研究应用的psiCHECK-2载体,以海肾荧光素酶作为主要报告基因,还引入一个萤火虫荧光素酶表达归一化,二者反应都为发光反应,检测方式一致、检测范围和灵敏度相近,且二者发光反应的底物不同,不会相互影响,是目前理想的双报告基因系统。此外,psiCHECK-2载体含有单纯疱疹病毒胸苷激酶启动子(HSV-TK)、多聚腺苷酸加尾信号[SV40 Late poly(A)]和质粒所需的其他组成部分,可在哺乳动物细胞中稳定高表达外源插入片段,是目前研究miRNA与mRNA功能较为理想的表达载体。
4 结论本研究运用RT-PCR、基因重组和定点突变技术成功构建了牙鲆emx2基因的野生型(psiCHECK-emx2-3'UTR)和突变型(psiCHECK-mutated-emx2-3'UTR)双荧光素酶报告载体,为检测牙鲆emx2基因与miRNA间的作用关系奠定了基础。。
| [1] | Lagos-Quintana M, Rauhut R, Lendeckel W, et al. Identification of novel genes coding for small expressed RNAs[J]. Science, 2001, 294(5543):853-858. |
| [2] | Kim VN. MicroRNA biogenesis:coordinated cropping and dicing[J]. Nature Reviews Molecular Cell Biology, 2005, 6(5):376-385. |
| [3] | Pasquinelli AE, Reinhart BJ, Slack F, et al. Conservation of the sequ-ence and temporal expression of let-7 heterochronic regulatory RNA[J]. Nature, 2000, 408(6808):86-89. |
| [4] | Reinhart BJ, Slack FJ, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772):901-906. |
| [5] | Gregory RI, Chendrimada TP, Cooch N, et al. Human RISC couples microRNA biogenesis and posttranscriptional gene silencing[J]. Cell, 2005, 123(4):631-640. |
| [6] | Brennecke J, Stark A, Russell RB, et al. Principles of microRNA-target recognition[J]. PLoS Biology, 2005, 3(3):e85. |
| [7] | Bartel DP. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2):281-297. |
| [8] | Du T, Zamore PD. microPrimer:the biogenesis and function of microRNA[J]. Development, 2005, 132(21):4645-4652. |
| [9] | Lewis BP, Burge CB, Bartel DP. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120(1):15-20. |
| [10] | Gu Y, Zhang L, Chen X. Differential expression analysis of Paralichthys olivaceus microRNAs in adult ovary and testis by deep sequencing[J]. General and Comparative Endocrinology, 2014, 204:181-184. |
| [11] | Lin F, Li R, Pan ZX, et al. miR-26b promotes granulosa cell apopt-osis by targeting ATM during follicular atresia in porcine ovary[J]. PLoS One, 2012, 7(6):e38640. |
| [12] | Gulisano M, Broccoli V, Pardini C, et al. Emx1 and Emx2 show different patterns of expression during proliferation and differentiation of the developing cerebral cortex in the mouse[J]. European Journal of Neuroscience, 1996, 8(5):1037-1050. |
| [13] | Boncinelli E, Gulisano M, Broccoli V. Emx and Otx homeobox genes in the developing mouse brain[J]. Journal of Neurobiology, 1993, 24(10):1356-1366. |
| [14] | Pellegrini M, Mansouri A, Simeone A, et al. Dentate gyrus formation requires Emx2[J]. Development, 1996, 122(12):3893-3898. |
| [15] | Noonan FC, Goodfellow PJ, Staloch LJ, et al. Antisense transcripts at the EMX2 locus in human and mouse[J]. Genomics, 2003, 81(1):58-66. |
| [16] | Kusaka M, Katoh-Fukui Y, Ogawa H, et al. Abnormal epithelial cell polarity and ectopic epidermal growth factor receptor(EGFR)expression induced in Emx2 KO embryonic gonads[J]. Endocrinology, 2010, 151(12):5893-5904. |
| [17] | Pellegrini M, Pantano S, Lucchini F, et al. Emx2 developmental expression in the primordia of the reproductive and excretory systems[J]. Anatomy and eEmbryology, 1997, 196(6):427-433. |
| [18] | Miyamoto N, Yoshida M, Kuratani S, et al. Defects of urogenital development in mice lacking Emx2[J]. Development, 1997, 124(9):1653-1664. |
| [19] | Real FM, Sekido R, Lupiáñez DG, et al. A microRNA(mmu-miR-124)prevents Sox9 expression in developing mouse ovarian cells[J]. Biology of Reproduction, 2013, 89(4):78-97. |
| [20] | Reddy AM, Zheng Y, Jagadeeswaran G, et al. Cloning, characteriz-ation and expression analysis of porcine microRNAs[J]. BMC Genomics, 2009, 10(1):65-80. |
| [21] | Daftary GS, Taylor HS. EMX2 gene expression in the female reproductive tract and aberrant expression in the endometrium of patients with endometriosis[J]. The Journal of Clinical Endocrinology & Metabolism, 2004, 89(5):2390-2396. |
| [22] | 罗师平, 冷希岗. 基于 PCR 的体外诱变技术[J]. 国外医学:生物医学工程分册, 2005, 28(3):188-192. |
| [23] | 毕延震, 郑新民, 邵长伟, 等. 检测 miRNA 活性的双荧光素酶单载体[J]. 中国生物化学与分子生物学报, 2012, 8:775-780. |




