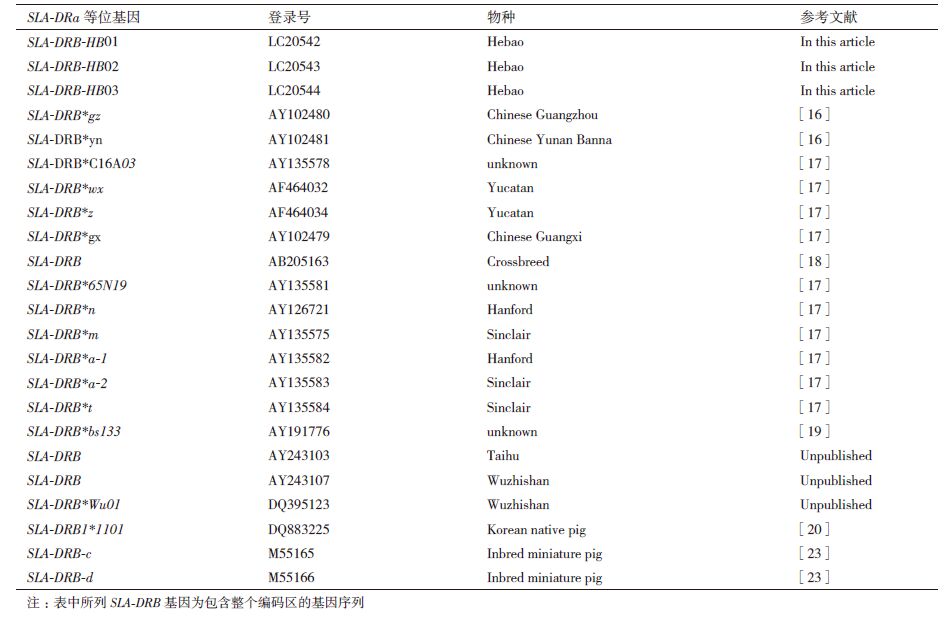
2.大连大学生命科学与技术学院,大连 116622
2. College of Life Science and Technology,Dalian University,Dalian 116622
动物识别自己、排除非己以维持机体正常生理活动和抵抗疾病的能力是免疫系统的核心职能。这种能力很大程度上是由各物种的主要组织相容性抗原复合体(Major histocompatibility complex,MHC)基因所决定[1]。猪的主要组织相容性复合体又称猪的白细胞抗原(Swine lymphocyte antigen,SLA),由Vaiman等[2]和Viza等[3]于1970年首次发现。Rabin等[4]发现其定位于猪的第7号染色体上;Geffrotin等[5, 6]用原位杂交、染色体标记和基因间常规连锁分析,进一步确定SLA基因位于第7号染色体的短臂上。目前SLA可分为3个主要类群:Ⅰ类区域基因、Ⅱ类区域基因和Ⅲ类区域基因[7]。其中,Ⅱ类基因位于SLA-D区,它控制着机体的细胞免疫和体液免疫应答,并在调节机体的抗病能力方面起到非常重要的作用[8]。
SLA Ⅱ类基因主要包括DRa、DRb、DQa、DQb、Dob、Dpa、TAP、LMP等。Ⅱ类基因有α和β两种基因,分别编码α肽链和β肽链。SLAⅡ类也包含多种基因座,其中只有SLA-DR和SLA-DQ在蛋白质水平上进行表达。SLA-DR分子是跨膜异二聚体,它由一条约34 kD的α链与一条约29 kD的β链以非共价键结合而成,每一条链分为3个区,N端的胞外区、中间的穿膜区和C端的胞质区[9]。其中穿膜区和胞质区与Ⅰ类分子的相应结构类似,而胞外区则不同,该区可分为两个结构域,在α链上为α1和α2,β链上为β1和β2(α和β链均包含约90个氨基酸残基),与免疫球蛋白的结构类似。α1和β1,特别是β1有较高的可变性,故呈现较高多态性,这样有利于机体适应不同外来抗原。α1和β1各形成4条β折叠股和1条α螺旋,8条β折叠股组成一个底层,支撑2个α螺旋,α链和β链2个α螺旋形成肽结合区(PBD)深槽的侧面。跨膜片段由25个疏水性氨基酸残基组成,可能形成α螺旋穿过脂质双分子层,并使分子固定在细胞膜上[10]。
荷包猪是辽宁省特有的珍贵地方品种,也是国家级的种质资源保护品种,已被列入《国家畜禽品种资源保护名录》[11]。荷包猪由于其地处辽西交通不便的山区一带——葫芦岛市建昌县和朝阳市凌源、喀左、朝阳三县与建昌接壤的地方,长期进行自繁自育,原始进化,因而推测其基因进化程度不高,稳定的基因特征非常适合作为科学研究的模式实验动物。目前为止,已经成功地克隆了荷包猪SLA-Ⅰ类基因全部序列,并分析了其分子进化特征[12]。为了更全面地研究荷包猪的SLA分子进化特征,有必要研究其他SLA功能基因。为此,本研究将选择SLA-DR基因为研究对象,克隆其β链序列,对其进行分子进化和氨基酸变异位点多态性分析,旨在为全面解析SLA-Ⅱ类分子的进化特征提供新的数据。
1 材料与方法 1.1 材料 1.1.1 材料荷包猪脾脏(3个个体)(辽宁省畜牧科学研究院荷包猪保种场)。
1.1.2 主要试剂pMD18-T Vector,E. coli JM109,总核酸提取试剂盒TRIZOL,反转录试剂盒Reverse Transcriptase M-MLV(RNase H-),DNA回收试剂盒Agarose Gel DNA Purification Kit Ver.2.0,EcoRⅠ及Hind Ⅲ限制性内切酶均为TaKaRa公司产品。
1.2 方法 1.2.1 引物设计扩增荷包猪SLA-DRB的引物的设计参考文献设计:上游引物:DRBF1:5'-GTTCTCCAGCATGTTGCATCTGTG-3';下游引物:DRBR1:5'-AGAGGATGCTTGCTTGGAGTCTC-3'。
1.2.2 总RNA提取及RT-PCR采用TRIZOL试剂盒并按照文献[13]方法从脾脏提取总RNA。然后以Oligo(dT)作为引物,按照反转录试剂盒说明将核酸反转录为cDNA。然后以引物DRBF1/DRBR1扩增SLA-DRB,PCR程序为:94℃ 3 min;94℃ 1 min,65℃ 1 min,72℃ 2 min,30个循环;72℃ 10 min。扩增PCR产物经纯化试剂盒回收。
1.2.3 基因克隆及重组子筛选将纯化后的SLA-DRB分别与pMD18-T载体连接,并转化宿主菌JM109。重组菌用含Amp的LB培养基进行抗性筛选,碱裂解法提质粒,EcoRⅠ和Hind Ⅲ双酶切鉴定,1%琼脂糖凝胶电泳分析[14]。
1.2.4 序列测定与分析通过将培养得到的3个体阳性克隆送至上海生工测序,将获得的3个个体的SLA-DRB基因cDNA分别命名为:SLA-DRB-HB01、SLA-DRB-HB02和SLA-DRB-HB03,应用GENTYX9.0(Software Development co.,Ltd)软件进行拼接,序列通过SAKURA系统登录入DDBJ/GenBank。
1.2.5 SLA-DRB同源性分析从DDBJ/EMBL/Gen-Bank中搜索所有SLA-DRB各等位基因cDNA全序列,见表 1。用DNAMAN Version 5.2.2(Lynnon Biosoft)对各等位基因之间氨基酸序列进行比较,按照文献[15]所述方法并略作改动进行关键位点氨基酸置换率的计算和统计。之后应用DNAStar 7.2分析各等位基因之间核酸序列同源性。
以人的HLA-DRB3(U66825)为参照,运用DNAMAN Version 5.2.2(Ly-nnon Biosoft)和Mega5.0软件(Mega software co.,Ltd)的Neighbor-Joining法绘制SLA-DRB各等位基因之间分子进化树。
1.2.7 SLA-DRB多态性分析首先通过获得荷包猪SLA-DRB基因序列推导其氨基酸序列,应用DNAMAN对获得的3个个体的氨基酸序列的主要氨基酸变异位点进行比较分析,发现其变异位点。然后与从GenBank中获得人的经典HLA-DRB基因序列推导获得的氨基酸序列,以HLA-DRB多态性位点作为参照,分析荷包猪SLA-DRB-HB的多态性。
2 结果 2.1 猪SLA-DRB基因座的克隆3个个体的RNA利用引物DRBF1/DRBR1经RT-PCR得到大约800 bp的条带,与理论计算值836 bp大小相符合,产物命名为SLA-DRB-HB01、SLA-DRB-HB02和SLA-DRB-HB03,(图 1-A)。分别将SLA-DRB-HB01-03克隆入pMD18-T载体,转化JM109,培养后用碱裂解法提质粒,EcoR Ⅰ和Hind Ⅲ双酶切筛选和鉴定,证实插入片段与目的基因大小相符(图 1-B)。

|
| 图 1 PCR 扩增SLA-DRB-HB 基因(A)及双酶切鉴定(B) |
经序列测定和GENTYX9.0(Software Developm-ent co.,Ltd)软件分析,SLA-DRB-HB长度为836 bp,其中1-801为ORF区,共编码266个氨基酸。3个个体SLA-DRB-HB01、SLA-DRB-HB02、SLA-DRB-HB03序列分别提交至GenBank,获得序列号分别为LC20542、LC20543和LC20544。
2.3 猪SLA-DRB基因座序列比较与分析经与DDBJ/EMBL/GenBank上所有SLA-DRB序列比较,发现SLA-DRB-HB推导的氨基酸序列与其它SLA-DRB等位基因的突变位点主要集中在信号肽区(1-29)、β1区(30-123)和β2区(124-217)。其中信号肽区超过4个等位基因发生氨基酸突变的位点包括2、10、11、17、18和19。在β1区超过4个以上等位基因发生氨基酸突变的位点有27个。β2区突变位点有11个,然而超过4个等位基因发生突变的位点只有201位。穿膜和胞浆功能区(218-226)变异位点共有6个,其中只有第232位超过4个等位基因发生氨基酸位点突变(图 2)。

|
| 图 2 SLA-DRB-HB 与DDBJ/EMBL/GenBank 上其他SLA-DRB 的多重序列比较 |
经计算各等位基因主要位点氨基酸置换率,发现变异主要集中在β1区,其中置换率超过0.5的有13个位点,置换率超过1的有5个位点。β2只有一个位点置换率超过0.5。其余的功能区氨基酸置换率均在0.5以下(图 3)。
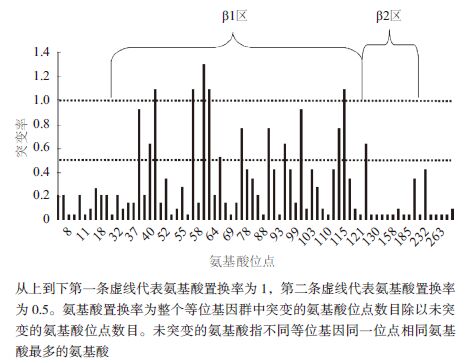
|
| 图 3 SLA-DRB 各等位基因氨基酸置换率 |
SLA-DRB-HB与其它SLA-DRB序列通过DNAStar 7.2进行同源比较和分析,同源性在90.3%-99.8%之间。其中与美国辛克莱Sinclair品系AY135575和中国Taihu品系AY243103同源性最高,均达到了99.8%,与AY135581同源性最低,仅为90.3%(图 4)。

|
| 图 4 SLA-DRB-HB 与其他SLA-DRB 同源性比对 |
根据猪SLA-DRB-HB各等位基因以及所有SLA-DRB等位基因,利用DNAMAN和Mega5.0软件绘制SLA-DRB-HB基因的分子进化树。结果(图 5)显示,荷包猪3个个体SLA-DRB-HB聚类成一个遗传关系最近的家族,其中SLA-DRB-HB01和SLA-DRB-HB02相对接近。荷包猪与我国贵州SLA-DRB-AY102480、美国的Sinclair品系SLA-DRB-AY24135575-Sinclair以及我国的太湖SLA-DRB-AY243103遗传关系相对接近,而与我国的五指山品系SLA-DRB-DQ395123以及韩国的SLA-DRB*1101-DQ883225遗传关系较远。
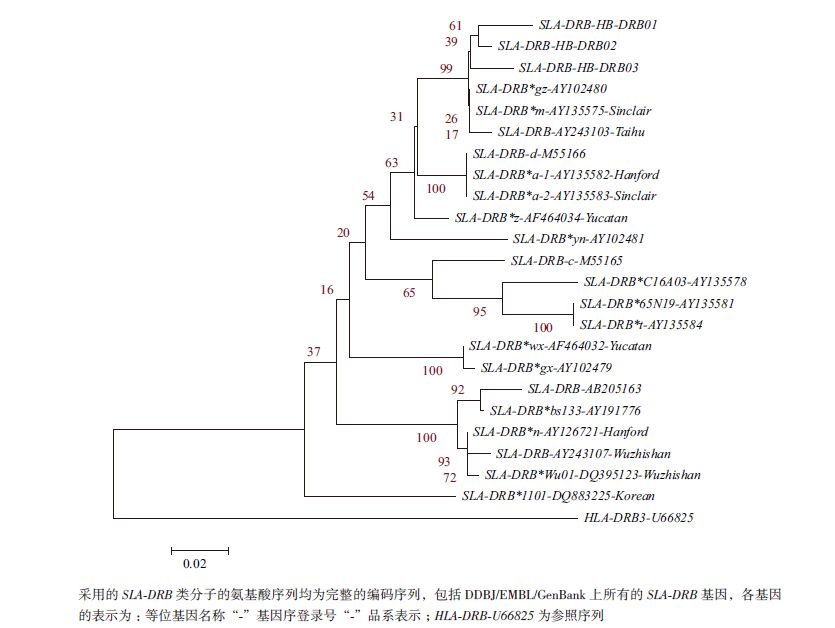
|
| 图 5 SLA-DRB-HB 各等位基因的分子内关系进化树 |
分子内可能的变异位点比较
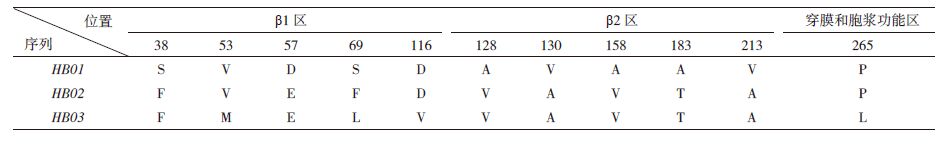
将SLA-DRB-HB 3个等位基因利用DNAMAN进行序列比较分析,结果(表 2)发现变异主要集中在β1和β2区,其中β1区存在5个变异位点,分别是38(S→F)、53(V→M)、57(D→E)、69(S→F→L)和116(D→V);β2区也有5个位点发生变异,分别在128(A→V)、130(V→A)、158(A→V)、174(A→T)、183(A→T)和213(V→A);而穿膜区则仅出现了1个变异位点,265位(P→L)。
与人HLA-DRB多态性位点比较分析 SLA-DRB-HB经过与人HLA-DRB相比较,发现在人DRB β1区17个典型的多态性氨基酸残基位点中,SLA-DRB-HB只有1个位点(β25)保留,而参照序列保留了4个位点(β25、β28、β30和β31)(图 6)。

|
| 图 6 SLA-DRB-HB 与人HLA-DRB β1 多态性位点比较 |
本研究克隆的荷包猪SLA-DRB-HB基因,与DDBJ/EMBL/GenBank上所有SLA-DRB序列进行比较,发现SLA-DRB-HB推导的氨基酸序列与其他SLA-DRB等位基因的突变位点主要集中在β1区(30-123)和β2区(124-217)。经过氨基酸置换率分析,进一步发现氨基酸置换率高的位点均分布在β1区。β1区的氨基酸突变主要集中在37-42以及52-60,该研究结果与之前的报道基本一致[21]。而β1区是承担SLA II类分子抗原多肽结合与递呈的功能区[16],从结果可以推测β1区某些关键的氨基酸位点或区域由于不断与不同抗原多肽结合,在进化上造成选择压力,从而导致氨基酸位点突变。因此,β1区的37-42以及52-60非常可能与抗原多肽结合及递呈有关。
SLA-DRB-HB与SLA-DRB其他等位基因同源性比较,同源率介于90.3%-99.8%之间。其中与美国辛克莱Sinclair品系 AY135575和中国的Taihu品系AY243103同源性最高,均达到了99.8%,而与AY135581的同源性最低,只有90.3%。结果说明荷包猪很可能与美国辛克莱Sinclair品或中国的Taihu猪在进化上或遗传上具有某种联系。
从分子进化树中发现,在人HLA-DRB作为参照的情况下,SLA-DRB-HB在遗传关系上位于SLA-DRB基因家族的较远分支且独立成一支。说明该基因与其他SAL-DRB具有相对远的遗传距离和亲缘关系,是SLA-DRB等位基因家族的一个远系成员,进化比较缓慢。其中,SLA-DRB-HB与我国贵州SLA-DRB-AY102480、美国的Sinclair品系SLA-DRB-AY24135575-Sinclair以及我国的太湖SLA-DRB-AY243103遗传关系相对接近,基本上与上述同源性分析的结果一致,荷包猪是否与这些同源性和进化关系密切的品种存在共同的祖先,有待于进一步调查。经分析,该荷包猪只分布于我国辽宁省偏僻的山区,这种原始低等进化的分子特征是由于地理生殖隔绝造成的[22]。今后,可以以荷包猪为选配品种,进行优化育种,而本研究为今后荷包猪的遗传育种提供基因方面的参考数据。
通过对3个个体SLA-DRB-HB的氨基酸序列进行比较发现,主要变异位点集中在β1区和β2区,而且这些位点比较分散,只有2个变异位点(38和57)位于β1区的37-42和52-60,另外还有一个位点位于胞质区。推测这些变异位点并不是由于在进化过程中不断接触不同的抗原肽形成选择压力等造成的,而可能是存在其他外来因素,如在该品系进化过程中某一阶段有过与其他品种发生杂交等造成。进一步分析荷包猪SLA-DRB-HB与人HLA-DRB主要多态性位点发现,荷包猪只保留了1个人HLA-DRB的典型多态性位点[23],而参照序列保留了4个。该结果进一步说明荷包猪与人的亲缘关系相对于其他品系更远,亦即其进化更原始,该结论与之前对荷包猪的研究所得出的结论基本一致[24]。该结果也揭示作为进化更原始的荷包猪,其基因资源非常宝贵,适合于作科学研究的模型材料。
4 结论本研究成功克隆了荷包猪SLA-DRB-HB三个等位基因,获得基因序列号,序列分析证明荷包猪的SLA-DRB相对于其他品系猪基因进化比较原始。
| [1] | Gao C, Jiang Q, Guo D, et al. Characterization of swine leukocyte antigen(SLA)polymorphism by sequence-based and PCR-SSP methods in Chinese Bama miniature pigs[J]. Dev Comp Immunol, 2014, 45(1):87-96. |
| [2] | Vaiman M, Renard C, LaFage P, et al. Evidence for a histocompatib-ility system in swine(SL-A)[J]. Transplantation, 1970, 10(2):155-164. |
| [3] | Viza D, Sugar JR, Binns RM. Lymphocyte stimulation in pigs:evidence for the existence of a single major histocompatibility locus, PL-A[J]. Nature, 1970, 227(5261):949-950. |
| [4] | Rabin M, Fries R, Singer D, et al. Assignment of the porcine major histocompatibility complex to chromosome 7 by in situ hybridization[J]. Cytogenet Cell Genet, 1985, 39(3):206-209. |
| [5] | Geffrotin C, Popescu CP, Cribiu EP, et al. Assignment of MHC in swine to chromosome 7 by in situ hybridization and serological typing[J]. Ann Genet, 1984, 27(4):213-219. |
| [6] | Smith TP, Rohrer GA, Alexander LJ, et al. Directed integration of the physical and genetic linkage maps of swine chromosome 7 reveals that the SLA spans the centromere[J]. Genome Res, 1995, 5(3):259-271. |
| [7] | Ando A, Imaeda N, Ohshima S, et al. Characterization of swine leukocyte antigen alleles and haplotypes on a novel miniature pig line, Microminipig[J]. Anim Genet, 2014, 45(6):791-798. |
| [8] | Shinkai H, Arakawa A, Tanaka-Matsuda M, et al. Genetic variability in swine leukocyte antigen class II and Toll-like receptors affects immune responses to vaccination for bacterial infections in pigs[J]. Comp Immunol Microbiol Infect Dis, 2012, 35(6):523-532. |
| [9] | Chardon P, Renard C, Vaiman M. The major histocompatibility complex in swine[J]. Immunol Rev, 1999, 167:179-192. |
| [10] | Lunney JK, Ho CS, Wysocki M, et al. Molecular genetics of the swine major histocompatibility complex, the SLA complex[J]. Dev Comp Immunol, 2009, 33(3):362-374. |
| [11] | 宋恒元. 辽宁地方良种—荷包猪[N]. 中国畜牧报, 2005, 01, 26. |
| [12] | Gao FS, Bai J, Gong XJ, et al. Analyzing the genetic characteristics and function of the swine leukocyte antigen 2 gene in a Chinese inb-reed of pigs[J]. Microbiol Immunol, 2012, 56(3):208-215. |
| [13] | 高凤山, 夏春, 张强, 等. 大肠杆菌表达的重组猪β2微球蛋白二级结构的圆二色谱分析[J]. 微生物学报, 2009, 49(12):1596-1600. |
| [14] | 高凤山, 姜平, 李新生, 等. 我国巴马小型猪SLA-2基因克隆及分子特征[J]. 微生物学通报, 2007, 34(4):686-690. |
| [15] | 张宏, 郑欣, 吴桂丹, 等. HBsAg-/HBV DNA+献血者生物学分布特征的研究[J]. 中国输血杂志, 2014, 27(8):838-842. |
| [16] | Chen F, Xie J, Zhou Y, et al. Novel SLA-DR alleles of three Chinese pig strains and the related function in human T cell response[J]. Cell Mol Immunol, 2004, 1(3):212-218. |
| [17] | Smith DM, Lunney JK, Martens GW, et al. Nomenclature for factors of the SLA class-I system, 2004[J]. Tissue Antigens, 2005, 65(2):136-149. |
| [18] | Gao FS, Wang L, Li XS, et al. Cloning α and β chains of SLA-DR loci and reconstruction of their complex in vitro[J]. Frontiers of Agriculture in China, 2008, 2(3):355-360. |
| [19] | Zeng R, Zeng YZ. Molecular cloning and characterization of SLA-DR genes in the 133-family of the Banna mini-pig inbred line[J]. Anim Genet, 2005, 36(3):267-269. |
| [20] | Lee YJ, Cho KH, Kim MJ, et al. Sequence-based characterization of the eight SLA loci in Korean native pigs[J]. Int J Immunogenet, 2008, 35(4-5):333-334. |
| [21] | 王燕, 邢小为, 薛立群, 等. 湖南大围子猪SLA-DR基因克隆及其生物信息学分析[J]. 细胞与分子免疫学杂志, 2009, 25(9):770-773. |
| [22] | 宋恒元, 郑旭, 王春艳. 荷包猪品种资源调查报告[J]. 猪业科学, 2014, 31(9):130-131. |
| [23] | Gustafsson K, Germana S, Hirsch F, et al. Structure of miniature swine class II DRB genes:conservation of hypervariable amino acid residues between distantly related mammalian species[J]. Proc Natl Acad Sci USA, 1990, 87(24):9798-9802. |
| [24] | Gao FS, Xu CB, Zhang XH, et al. Analysing molecular characteristics of the SLA-1 gene from Chinese Hebao pigs[J]. J Genet, 2012, 91(2):219-221. |