2.中国农业科学院植物保护研究所 植物病虫害生物学国家重点实验室,北京 100193
2. State Key Laboratory for Biology of Plant Diseases and Insect Pests,Institute of Plant Protection,Chinese Academy of Agricultural Sciences,Beijing 100193
全球转基因作物的商业化和大规模种植在一定程度上缓解了粮食紧缺问题,给农业生产带了巨大效益[1],转基因作物实际运用过程中还需兼顾其安全性,以保证转基因作物不会对生态环境及其他动植物产生非预期效应。水稻是我国重要的粮食作物,然而虫害的发生严重制约我国水稻经济发展,其中水稻螟虫危害占总虫害发生面积一半以上,是危害水稻并导致水稻减产的重要原因[2]。转cry1Ab/Ac基因水稻(TT51)是我国华中农业大学培育的具有自主知识产权的转基因水稻品种,该水稻对二化螟、三化螟、稻纵卷叶螟等害虫具有较强抗性[3],于2009年获得农业部颁发的转基因生物生产应用安全证书[4],并在2014年续期成功,更加有助于对其进一步商业化种植及推广。
转基因作物作为饲料对动物的影响是转基因作物安全性评价的重要内容。转基因作物经动物取食后,在动物肠道中的消化降解、残留情况可能会间接影响到动物体的发育,外源基因及蛋白也可能存在向重要器官和血液中转移的风险;如果动物体中存在外源基因和蛋白残留,食物链下一营养级也会暴露于低剂量Bt蛋白。为评价转基因水稻对啮齿类动物的影响,本研究以褐家鼠作为野生啮齿类动物的典型代表进行研究,褐家鼠个体大、数量多、分布广、繁殖力强,其取食活动容易造成粮食污染严重威胁到农业生产、食品工业及人类健康安全[5]。鼠类作为目前世界上种类和数量最多的哺乳动物是生态环境中食物链的中间环节,能为次级消费者提供食物来源,在生态系统的物质循环和能量流动中发挥着重要作用,一定条件下其取食活动也能够为种子的传播和萌发提供条件[6],而且鼠类本身代谢循环快,因此在生态系统养分循环中起到重要作用,对于维持生态系统的结构和功能的稳定性具有重要意义[7]。目前以实验用鼠作为转基因生物安全评价方面的研究主要从急慢性毒理、免疫、生殖系统方面进行评价,大部分研究结果显示,转基因作物并不会对动物体器官组织、生理指标、生长发育产生影响[8],虽然有实验显示大鼠在血生化、血常规指标中出现了一些差异,但所有指标均在该年龄段该品系大鼠的正常范围之内[9, 10]。无论是转基因作物中外源Bt蛋白[11]或是异源表达的Bt蛋白[12]都不会对鼠产生致敏性。迄今国内外尚无转cry1Ab/Ac基因水稻中外源基因及蛋白在野生褐家鼠体内残留研究的报道。褐家鼠其生存状况与常规实验用鼠相比更复杂,取食及消化规律也有所不同。为进一步丰富转基因水稻TT51饲用安全性评价,本研究以饲喂转Bt基因水稻50 d和110 d的褐家鼠为材料,采用小片段全覆盖PCR检测的策略,分析野生褐家鼠肠道内容物中cry1Ab/Ac基因的残留情况,同时采用ELISA和Western blot两种方法检测蛋白残留。
1 材料与方法 1.1 材料 1.1.1 实验材料供试水稻品种为华恢1号转cry1Ab/Ac基因水稻,亲本明恢63为对照组,由华中农业大学提供。
1.1.2 试剂QIAamp Fast DNA Stool Mini Kit购自QIGEN公司,QualiPlate试剂盒购自Envirologix公司;PVDF膜购自Promega公司;鼠源一抗由本实验室制备,二抗鼠抗羊lgG HRP购自北京天德悦生物科技公司;胃蛋白酶(3 000 U/mg)购自Sigma公司;PCR纯化试剂盒,购自Axygen公司。
1.2 方法 1.2.1 褐家鼠饲喂野生褐家鼠分成4组,其中2组为雄鼠,每组12只;雌鼠2组,每组14只。分别有1组雄鼠和1组雌鼠饲喂转基因水稻,另外1组雄鼠和1组雌鼠饲喂亲本水稻。分组方式采用同一窝后代同性两只鼠分成两组的方式,分别饲喂转基因水稻和亲本水稻。在50 d和110 d分别从每个处理中取出褐家鼠,解剖褐家鼠,分别取胃、空肠、回肠、盲肠部位置于离心管中液氮冷冻保存。
1.2.2 褐家鼠肠道中外源基因残留检测用QIAamp Fast DNA Stool Mini Kit试剂盒提取褐家鼠肠道内容物DNA,按照说明书中的方法提取,方法稍作改动,200 mg肠道内容物加入1 mL Inhibit Ex Buffer混匀,去除杂质和PCR干扰物,加入蛋白酶K去除体系中的蛋白,用无水乙醇沉淀DNA,过QIAamp spin柱洗脱后加入200 μL 超纯水。DNA提取后采用DNA定量仪(Thermo公司)测定DNA样品的浓度,并以OD260/280 nm检测DNA的纯度。DNA样品用超纯水稀释至40-50 ng/μL,-20℃储存备用。
分别以cry1Ab/Ac基因以及水稻内源蔗糖磷酸合酶基因SPS(sucrose phosphate synthase gene,U33175)[13]和淀粉分支酶基因RBE4(starch branching enzyme gene,EF055878)[14]、褐家鼠内源基因为GAPDH为对照设计引物25对,其中包括cry1Ab/Ac基因全长引物1对和100 bp小片段引物9对、扩增SPS基因100 bp小片段引物11对、扩增RBE4基因全长引物1对和100 bp小片段引物2对、扩增GAPDH基因引物1对,除鼠内源基因(表 1)以外其他引物序列及扩增长度参照文献[15],所有引物由生工生物工程公司合成。
扩增cry1Ab/Ac基因PCR反应条件:94℃预变性5 min;94℃ 30 s,58℃ 30 s,72℃ 1 min 45 s,30个循环;72℃ 10 min。扩增SPS基因PCR反应条件:94℃预变性5 min;94℃ 30 s,56℃30 s,72℃ 20 s,30个循环;72℃ 10 min。扩增RBE4基因PCR反应条件:94℃预变性5 min;94℃ 30 s,54℃ 30 s,72℃ 30 s,30个循环;72℃ 10 min。扩增GAPDH基因PCR反应条件:94℃预变性5 min;94℃ 30 s,59℃ 30 s,72℃ 20 s,30个循环;72℃ 10 min。PCR产物4℃保存,PCR反应体系为模板1 μL,上下游引物各0.4 μL,Taq Mix 10 μL,超纯水补至20 μL,设置水空白对照、阳性对照、待测样品,每个样品做3个重复,1%琼脂糖凝胶电泳检测。
1.2.3 体外消化cry1Ab/Ac基因模拟实验体外消化模拟实验采用添加肠道内容物和模拟胃液环境两种方法。
1.2.3.1 添加内容物方法cry1Ab/Ac基因50 ng和水稻基因组10 μg加褐家鼠肠道内容物5 mg以及对照组cry1Ab/Ac基因50 ng和水稻基因组10 μg分别在37℃温浴0、4、8和12 h,温浴后的产物用QIAamp DNA Stool Mini Kit试剂盒提取其中的DNA,DNA产物用根据cry1Ab/Ac基因所设计的10对引物检测cry1Ab/Ac基因片段。
1.2.3.2 模拟胃液方法同时将肠道内容物替换成模拟胃液0.5 μL[16](0.2 g NaCl,87.7 mg胃蛋白酶,盐酸调pH1.2,加水至100 mL),分别在37℃温浴0、4、8和12 h,混合反应后的产物用PCR纯化剂盒(Axygen公司)纯化,PCR反应条件同1.2.2,1%琼脂糖凝胶电泳检测。
1.2.4 褐家鼠肠道中的外源蛋白残留检测ELISA方法检测褐家鼠肠道内容物中的外源蛋白,检测前用BCA方法定量总蛋白,试剂盒购自北京艾德莱生物科技有限公司。Cry1Ab/Ac蛋白残留检测采用ELISA双抗夹心法,检测结果重复3次,QualiPlate试剂盒购自美国Envirologix公司,方法按照试剂盒说明书进行,其中放置温度均改为37℃,检测结果用Western blot验证。Western blot方法如下:待测样经过SDS-PAGE分离后转印至PVDF 膜上,将膜用TBS-T 溶液漂洗后用20 mL 5%脱脂奶粉封闭2 h,加入含一抗(0.5 μL)的TBS-T漂洗液15 mL,4℃孵育过夜或室温1 h,加入含鼠抗兔IgG二抗(1 μL)的TBS-T漂洗液15 mL,室温1 h,每步结束后漂洗膜3次,每次10 min,最后膜用显色液显色后拍照。
2 结果 2.1 褐家鼠肠道中外源基因残留检测提取饲喂转基因水稻及非转基因水稻雌雄褐家鼠肠道内容物总DNA,DNA定量仪检测DNA的纯度(OD260/280 nm)均在1.7-1.9,将总DNA样品用超纯水稀释至40-50 ng/μL,每个样品取1.0 μL用于检测。PCR检测的目标基因包括cry1Ab/Ac基因全长及100 bp小片段、水稻内源基因SPS及REB4和褐家鼠内源GAPDH基因,共25对引物,琼脂糖凝胶电泳结果(图 1-A和1-B)显示,仅能扩增出10个片段的相应正对照,在肠道内容物总DNA中,均未检测到cry1Ab/Ac基因全长和100 bp个小片段,其他扩增小片段的8对引物也未获得目的条带(电泳图未提供)。同样,在空肠、回肠、盲肠内容物提取的总DNA中,也检测不到水稻内源基因SPS及REB4(图 1-C、1-D和1-E);但所有的内容物总DNA中都可以检测到褐家鼠内源GAPDH基因(图 1-F)。
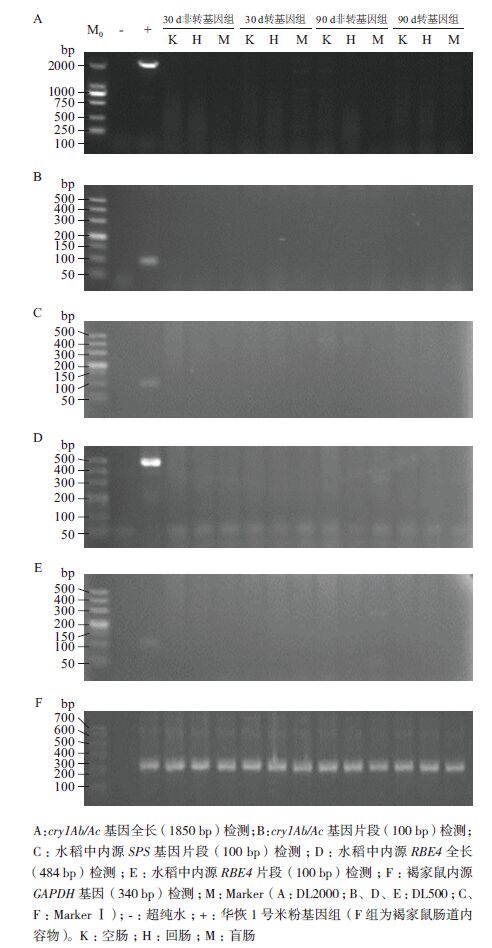
|
| 图 1 褐家鼠肠道内容物提取的总DNA 中cry1Ab/Ac 基因 的PCR 检测结果 |
通过将过量cry1Ab/Ac基因与褐家鼠肠道内容物孵育,分析外源基因体外降解规律。不加肠道内容物对照处理中在12 h仍能扩增出全长cry1Ab/Ac基因;然而,cry1Ab/Ac基因在加入了肠道内容物后,4 h开始逐渐降解,12 h后检测不到全长cry1Ab/Ac基因(图 2-A);但是在4 h和8 h时还能检测出cry1Ab/Ac基因9个100 bp小片段,而到达12 h时,9个小片段都无法检测到(图 2-B)。模拟胃液中,37℃ 4 h,cry1Ab/Ac基因已经完全降解,同样,cry1Ab/Ac基因9个100 bp小片段在4 h时完全降解(图 3)。
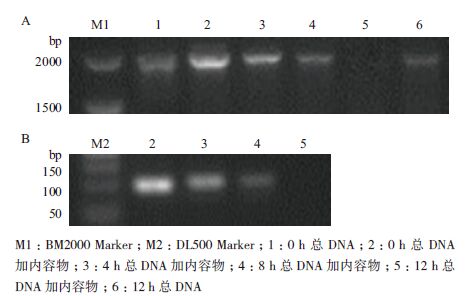
|
| 图 2 添加内容物体外消化cry1Ab/Ac 基因全长(A)及片 段(B) |

|
| 图 3 胃液体外消化cry1Ab/Ac 基因全长(A)及片段(B) |
先用BCA法对提取的蛋白进行定量,取20 μg可溶性总蛋白进行ELISA检测。用双抗夹心法对样品进行检测,检测结果(表 2)显示,50 d和110 d转基因和非转基因组中盲肠和直肠样品50TW-M、50TK-M、50TH-M、50TM-M、50TW-F、50TK-F、50TH-F、50TM-F、110CH-F、110CM-F、110TW-M、110TH-M、110TM-M、110TW-F、110TH-F、110TM-F出现比较明显的显色反应,检测值表明其中可能存在Cry1Ab/Ac蛋白残留。
对ELISA检测结果中产生颜色反应的样品进行SDS-PAGE和Western blot分析,SDS-PAGE结果(图 4-A和图 5-A)显示,在60 kD处50TM-M、110TM-F样品存在疑似条带,但Western blot分析结果表明这两个样品中并不存在Cry1Ab/Ac蛋白。
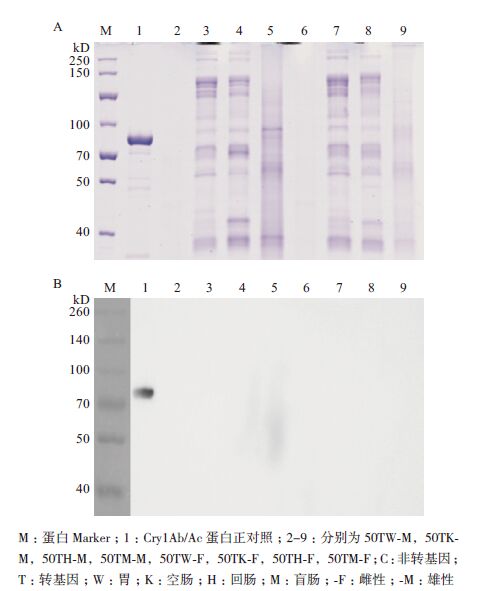
|
| 图 4 50 d 转基因和非转基因饲喂褐家鼠肠道内容物总蛋白 的SDS-PAGE 分析(A)及Western blot 分析(B) |
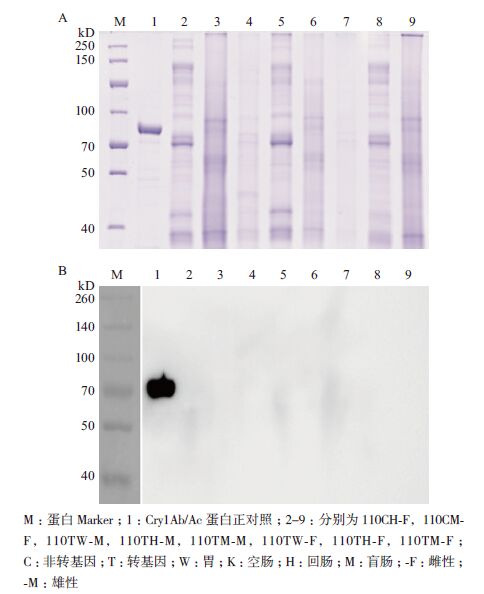
|
| 图 5 110 d 转基因和非转基因饲喂褐家鼠肠道内容物总蛋 白的SDS-PAGE 分析(A)及Western blot 分析(B) |
转基因作物中表达的外源蛋白在动物肠道中的残留是转基因作物安全性评价的重要环节,Bt蛋白不会破坏啮齿类动物的肠道是因为哺乳动物肠道中没有Bt蛋白的结合位点,但如果肠道中还存在蛋白残留可能会对宿主产生致敏性[17],外源基因残留也可能存在向动物体其他部位转移的风险,需要对转Bt基因水稻中外源成分的降解情况进行评估。
本研究采用多对引物同时对cry1Ab/Ac基因全长及基因碎片进行检测,尽可能确保能完整覆盖到全长外源基因片段,在褐家鼠肠道内容物中未检测到cry1Ab/Ac基因的残留,也有研究表明在饲喂转基因大豆的啮齿类动物的兔体内组织中检测不到外源基因[18]。为了进一步验证动物实验中外源基因在褐家鼠肠道中的降解情况,本研究进行了体外模拟实验,将过量cry1Ab/Ac基因与褐家鼠肠道内容物混合孵育,结果表明全长cry1Ab/Ac基因从4 h开始逐步降解,12 h时PCR扩增为阴性,而全长cry1Ab/Ac基因片段与肉鸡肠道内容物孵育时4 h后就检测不到基因片段[15]。用9对扩增100 bp小片段引物检测时发现,与全长基因有相同的降解规律,12 h完全降解。胃液中外源基因则在更短的时间内消化降解。
在ELISA检测结果中一些非转基因样品也出现了明显颜色反应,原因是褐家鼠肠道内容物中一些外源或内源的污染物可能会引起显色反应,对实验结果产生明显影响,Chowdhury[19]和Einspanier[20]的研究结果也出现这样的现象。进一步Western blot检测发现这些样品中并没有Bt蛋白残留,与Lutz等[21]的研究结果一致。很多研究表明Bt蛋白容易在鼠肠道中消化从而不会对肠道组织产生影响[22],更不会引起动物体过敏反应[12],在动物体吸收食物营养成分过程中,动物的血液[23]、内脏组织[24, 25]、肌肉组织[26]中也未检测到外源蛋白的残留。
上述研究充分说明转基因水稻中的外源基因和蛋白在褐家鼠肠道内容物中无残留,当然还可以从免疫毒理学、生长发育、繁殖性能等方面研究对转Bt基因对啮齿类动物的影响以全面评价转基因作物的安全性。
4 结论本研究以野生褐家鼠为模型,分析了饲喂转Bt水稻后雌雄褐家鼠肠道中的外源基因和蛋白的残留情况。DNA和蛋白水平的分析表明,褐家鼠肠道内容物中没有cry1Ab/Ac基因和蛋白残留,丰富了转Bt水稻饲喂安全性评价体系。
| [1] | James C. Global status of commercialized biotech/GM crops:2014[J]. ISAAA Brief 47, 2015. |
| [2] | 姜玉英, 曾娟, 陆明红. 2015年全国三大谷类作物重大病虫害发生趋势预报[J]. 植物保护, 2015(2):1-4. |
| [3] | Tu JM, Zhang GA, Datta K, et al. Field performance of transgenic elite commercial hybrid rice expressing Bacillus thuringiensis delta-endotoxin[J]. Nature Biotechnology, 2000, 18(10):1101-1104. |
| [4] | Lu C. The first approved transgenic rice in China[J]. GM Crops, 2010, 1(3):113-115. |
| [5] | 江佳富, 吴晓明, 左曙青. 北京市褐家鼠种群遗传多态性及与汉坦病毒分布关系的研究[J]. 中国媒介生物学及控制杂志, 2005(5):30-35. |
| [6] | Edwards GR, Crawley MJ. Rodent seed predation and seedling recruitment in mesic grassland[J]. Oecologia, 1999, 118(3):288-296. |
| [7] | Pearson DE, Hierro JL, Chiuffo M, et al. Rodent seed predation as a biotic filter influencing exotic plant abundance and distribution[J]. Biological Invasions, 2014, 16(5):1185-1196. |
| [8] | Wang ZH, Wang Y, Cui HR. Toxicological evaluation of transgenic rice flour with a synthetic crylAb gene from Bacillus thuringiensis[J]. Journal of the Science of Food and Agriculture, 2002, 82(7):738-744. |
| [9] | Schr∅der M, Poulsen M, Wilcks A, et al. A 90-day safety study of genetically modified rice expressing Cry1Ab protein(Bacillus thuringiensis toxin)in Wistar rats[J]. Food and Chemical Toxicology, 2007, 45(3):339-349. |
| [10] | Song H, He X, Zou S, et al. A 90-day subchronic feeding study of genetically modified rice expressing Cry1Ab protein in Sprague-Dawley rats[J]. Transgenic Research, 2014:1-14. |
| [11] | Kroghsbo S, Madsen C, Poulsen M, et al. Immunotoxicological studies of genetically modified rice expressing PHA-E lectin or Bt toxin in Wistar rats[J]. Toxicology, 2008, 245(1-2):24-34. |
| [12] | Cao SS, He XY, Xu WT, et al. Safety assessment of Cry1C protein from genetically modified rice according to the national standards of PR China for a new food resource[J]. Regulatory Toxicology and Pharmacology, 2010, 58(3):474-481. |
| [13] | Ding JY, Jia JW, Yang LT, et al. Validation of a rice specific gene, sucrose phosphate synthase, used as the endogenous reference gene for qualitative and real-time quantitative PCR detection of transgenes[J]. Journal of Agricultural and Food Chemistry, 2004, 52(11):3372-3377. |
| [14] | Jeong SC, Pack IS, Cho EY, et al. Molecular analysis and quantitative detection of a transgenic rice line expressing a bifunctional fusion TPSP[J]. Food Control, 2007, 18(11):1434-1442. |
| [15] | 徐灯, 耿丽丽, 张敏红. 转基因水稻cry1Ab/Ac基因及蛋白在肉鸡肠道中的残留检测[J]. 生物技术通报, 2015, 31(9):84-90. |
| [16] | Martin-Orue SM, O’Donnell AG, Arino J, et al. Degradation of transgenic DNA from genetically modified soya and maize in human intestinal simulations[J]. British Journal of Nutrition, 2002, 87(6):533-542. |
| [17] | Mills ENC, Madsen C, Shewry PR, et al. Food allergens of plant origin-their molecular and evolutionary relationships[J]. Trends in Food Science & Technology, 2003, 14(4):145-156. |
| [18] | Tudisco R, Lombardi P, Bovera F, et al. Genetically modified soya bean in rabbit feeding:detection of DNA fragments and evaluation of metabolic effects by enzymatic analysis[J]. Animal Science, 2006, 82:193-199. |
| [19] | Chowdhury EH, Shimada N, Murata H, et al. Detection of Cry1Ab protein in gastrointestinal contents but not visceral organs of genetically modified Bt11-fed calves[J]. Vet Hum Toxicol, 2003, 45(2):72-75. |
| [20] | Einspanier R, Lutz B, Rief S, et al. Tracing residual recombinant feed molecules during digestion and rumen bacterial diversity in cattle fed transgene maize[J]. European Food Research and Technology, 2004, 218(3):269-273. |
| [21] | Lutz B, Wiedemann S, Einspanier R, et al. Degradation of Cry1Ab protein from genetically modified maize in the bovine gastrointestinal tract[J]. J Agric Food Chem, 2005, 53(5):1453-1456. |
| [22] | Yuan Y, Xu W, He X, et al. Effects of genetically modified T2A-1 rice on the GI health of rats after 90-day supplement[J]. Scientific Reports, 2013, 3(1962):srep01962. |
| [23] | Swiatkiewicz M, Bednarek D, Markowski J, et al. Effect of feeding genetically modified maize and soybean meal to sows on their reproductive traits, haematological indices and offspring performance[J]. Bulletin of the Veterinary Institute in Pulawy, 2013, 57(3):413-418. |
| [24] | Walsh MC, Buzoianu SG, Gardiner GE, et al. Fate of transgenic DNA from orally administered Bt MON810 maize and effects on immune response and growth in pigs[J]. PLoS One, 2011, 6(11):12. |
| [25] | Furgal-Dierzuk I, Strzetelski J, Kwiatek K, et al. The effect of genetically modified maize(MON 810)and soyabean meal(Roundup Ready)on rearing performance and transfer of transgenic DNA to calf tissues[J]. Journal of Animal and Feed Sciences, 2014, 23(1):13-22. |
| [26] | Jennings JC, Kolwyck DC, Kays SB, et al. Determining whether transgenic and endogenous plant DNA and transgenic protein are detectable in muscle from swine fed Roundup Ready soybean meal[J]. Journal of Animal Science, 2003, 81(6):1447-1455. |





