2. 华中科技大学生命科学 与技术学院,武汉 430074 ;
3. 孟山都公司,美国;
4. 密歇根大学,美国;
5. 杰克森基因组医学实验室,美国
2. College of Life Science and Technology,Huazhong University of Science and Technology,Wuhan 430074 ;
3. Monsanto Company,USA ;
4. University of Michigan,USA ;
5. The Jackson Laboratory for Genomic Medicine, USA
基因表达调控在动植物生长发育过程中具有重要作用,是植物适应外界环境的分子基础,转录调控是基因表达调控的关键步骤。转录调控通过转录因子(Transcription factor)蛋白质序列中的DNA结合结构域和靶基因上游启动子区域特异DNA序列模体结合而实现。除DNA结合结构域(DNA binding domain,DBD)外,转录因子通常还包含转录调控结构域(Transcription regulation domain),主要用于调控靶基因转录活性,既可激活转录,也可抑制转录。转录因子中的核定位信号(Nuclear localization signal,NLS)可引导转录因子在胞浆内合成后通过核膜进入细胞核。此外,有些转录因子含寡聚化结构域可形成二聚体或多聚体复合物,具有更为复杂的调控机制。
转录因子种类繁多、功能复杂,它们通过与靶基因启动子结合,激活或抑制其转录活性,调控靶基因在不同组织、不同细胞、不同环境条件下特异表达,并通过转录因子级联调控网络,对许多生命过程进行调控。例如,果蝇体节发育由一类称为同源异型框(Homeobox)的基因调控,它们所编码的蛋白质为转录因子,含长度为60个氨基酸的DNA结合结构域。植物特异转录因子家族SQUAMOSA promotor binding protein(SBP)成员具有调控玉米果实发育和水稻分蘖等多种功能。
20世纪90年代开始的人类基因组计划,开创了生命科学研究的新时代。人类基因组计划指定的模式生物酿酒酵母、秀丽线虫和果蝇的基因组测序于2000年前先后完成。拟南芥基因组测序于2000年底完成。2000年12月15日,就在Nature杂志发表拟南芥基因组序列分析论文[1]的第2天,Science杂志发表了题为《拟南芥转录因子:从基因组水平上比较真核生物转录因子》的论文[2],首次系统预测了拟南芥基因组中的1 533个转录因子,将它们分为28个家族,并与酵母、线虫和果蝇等其它3个真核生物进行了系统比较,发现拟南芥中转录因子在整个基因组中所占比例远高于上述3个物种。
2004年,北京大学生命科学学院朱玉贤、邓兴旺主持的国家自然科学基金国际合作项目,对拟南芥中预测到的转录因子按家族逐个克隆,并对结果进行了初步分析[3]。为配合该课题的顺利进行,我们构建了拟南芥转录因子数据库[4](Database of Arabidopsis transcription factors,DATF)。DATF中预测到的转录因子数共1 922个,分为64个家族。此后不久,水稻和杨树基因组序列发布,我们又先后构建了水稻转录因子数据库[5](Database of rice transcription factors,DRTF)和杨树转录因子数据库[6](Database of poplar transcription factors,DPTF)。与此同时,苔藓类植物小立碗藓(Physcomitrella patens)和绿藻类植物莱茵衣藻(Chlamydomonas reinhardtii)基因组测序也先后完成,我们又构建了植物主要谱系中这两个代表性物种的转录因子数据库。
截止2007年,玉米、高粱、棉花、大豆、葡萄等重要经济作物的基因组测序尚未完成,但美国爱荷华州立大学植物基因组数据库PlantGDB收录了大量植物代表性转录本(Plant unique transcripts,PUT)序列数据[7]。这些PUT序列是由表达序列标签(Expressed sequence tag,EST)拼接而成,有些是全长mRNA序列,有些则是mRNA序列片段。我们从17个物种PUT序列中预测了转录因子,并和上述DATF等5个已完成基因组测序物种的转录因子数据库整合在一起,构建了植物转录因子数据库[8](Plant transcription factor database,PlantTFDB),为植物基因组学、遗传学和植物分子生物学研究提供宝贵的数据资源。2010年,玉米、高粱、大豆、葡萄等18个被子植物,代表性蕨类植物江南卷柏(Sela-ginella moellendorffii),以及9个绿藻基因组测序相继完成。此外,PlantGDB数据库也进行了更新,并增加了不少新物种。与此同时,许多转录因子家族、特别是植物特异转录因子家族的起源、演化、功能等研究成果相继发表,转录因子家族分类也得以更新。为此,我们对PlantTFDB进行了大规模更新,更新后的第2版包括从49个物种中预测到的53 315个转录因子,分为58个家族[9]。随着基因组测序技术不断改进,测序速度不断加快。2013年,已有67种植物的基因组测序完成,我们对PlantTFDB再次进行更新。更新后的第3版共包括129 288个转录因子,来自83个物种,其中67个已完成基因组测序,覆盖绿色植物各大门类[10]。
本文介绍植物转录因子分类规则和预测方法,以及植物转录因子数据库PlantTFDB的概况和注释信息。
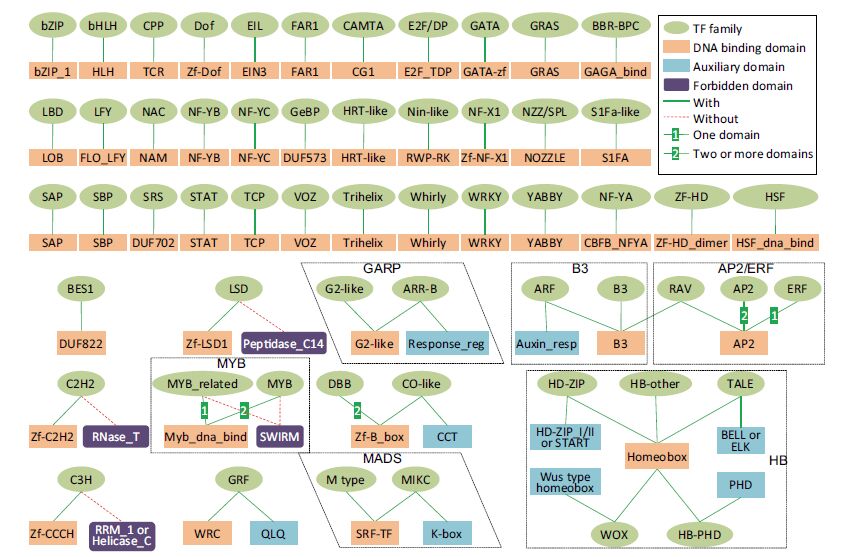
1 植物转录因子家族分类转录因子蛋白质序列中的DNA结合结构域DBD在很大程度上决定其与基因上游启动子区域DNA顺式元件结合的序列特异性[11]。DBD在演化上比较保守,通常用作区分不同转录因子家族的主要依据。2000年,Riechmann等[2]归纳整理了拟南芥中转录因子家族及其特征,将其分为28个家族。10多年来,我们先后检索和阅读了大量植物转录因子相关文献,文章总数累计达7 000余篇。在Riechmann等工作基础上,根据已有文献报道,总结了植物转录因子家族及其结构域序列特征,改进了植物转录因子家族分类规则,并不断加以修改和完善,用于植物转录因子家族划分和植物基因组中未知转录因子的预测(图 1)。
|
| 绿色椭圆表示转录因子家族,红色矩形表示DNA结合结构域,蓝色矩形表示辅助结构域,紫色矩形表示禁止结构域。绿色实线连接表示转录因子家族和DNA结合结构域、辅助结构域之间的对应关系,红色虚线连接转录因子家族和禁止结构域。数字“1”和“2”表示连接的DNA结合结构域数 图 1 植物转录因子家族分类规则 |
一般说来,根据转录因子蛋白质序列中所含DNA结合结构域种类,即可确定其属于某个特定家族。第3版PlantTFDB数据库58个转录因子家族中,36个家族(~62%)符合这种家族与DBD一一对应的简单规则,如调控植物生长发育的乙烯不敏感(Ethylene insensitive-like,EIL)转录因子家族均含EIN结构域,调控植物花、果实发育的SQUAMOSA基因启动子结合蛋白(SQUAMOSA-promoter binding protein,SBP)均含SBP结构域。
1.2 禁止结构域除上述具有简单对应关系的转录因子外,某些蛋白质家族情况比较复杂。例如,由两个半胱氨酸(Cys,C)和两个组氨酸(His,H)组成的C2H2锌指结构,是重要的蛋白质序列模体。这类蛋白质分子中,有些能与DNA结合,具有转录活性;有些则与RNA结合,具有核酸酶活性,除了能与RNA结合的C2H2锌指结构外,它们同时包含核酸酶相关RNase_T结构域。因此,我们将RNase_T结构域称为“禁止结构域”(Forbidden domain),用来降低转录因子预测中含C2H2锌指结构的蛋白质预测的假阳性率。又如,半胱氨酸型肽段内切酶MCP1B和AtMC2均具有DNA结合结构域Zf-LSD,但目前尚无证据表明它们具备转录调控功能。我们用禁止结构域“Peptidase_C14”用来滤除包含Zf-LSD结构域蛋白质中的非转录因子。除上述两个家族外,C3H和MYB家族也含禁止结构域。
1.3 辅助结构域有些转录因子中除了DBD外,还有其它一些特征结构域,称为“辅助结构域”(Auxiliary domain)。辅助结构域也可用作转录因子家族分类的依据。例如,生长调控因子(Growth regulation factor,GRF)转录因子家族中均含WRC结构域,该结构域中的特征序列为色氨酸(Trp,W)-精氨酸(Arg,R)-半胱氨酸(Cys,C)序列模体WRC。但并非所有含WRC序列模体的蛋白质都具有转录活性,只有既有WRC序列模体又有QLQ序列模体[谷氨酰胺(Gln,Q)-亮氨酸(Leu,L)- 谷氨酰胺(Gln,Q)]的蛋白质才是转录因子。
1.4 DBD结构域数有些转录因子中含两个或两个以上DBD,因此,DBD数目也常常用来区分不同转录因子家族。典型实例为AP2和ERF家族。这两个家族转录因子中均含AP2结构域,同属于AP2/ERF超家族,其中仅含一个AP2结构域的为ERF家族,含两个或两个以上的则为AP2家族。又如,MYB转录因子超家族均含Myb_dna_bind结构域,仅含一个的为MYB_related家族,而含两个或两个以上的为MYB家族。
1.5 超家族除上述基于DNA结合结构域、利用禁止结构域和辅助结构域对不同转录因子家族进行分类外,有些转录因子家族之间的关系比较复杂。例如,具有 DNA结合结构域G2-like的转录因子均属于GARP超家族,其中同时还含Response_reg结构域,而有的则仅有G2-like结构域。我们将仅含G2-like结构域的转录因子归为G2-like家族,而把兼有G2-like和Response_reg结构域的转录因子归为ARR-B家族。
更为复杂的是,AP2/ERF超家族中的另外一个家族RAV同时含有两个DNA结合结构域,一个为AP2,另一个为B3。而B3结构域又是另外一个超家族B3中两个家族的DNA结合结构域。该超家族中仅含B3结构域的为B3家族,同时含B3结构域和Auxin_resp辅助结构域的为ARF家族。
具有同源异型结构域(Homeodomain)的转录因子是一个具有多个家族的超家族,根据是否具有辅助结构域及辅助结构域类别,可细分为HD-ZIP、TALE、WOX等家族。
2 植物转录因子预测 2.1 预测方法利用上述家族分类规则,可以将文献中已经报道的植物转录因子分为若干家族,并以此为依据预测已经完成基因组测序的绿色植物基因组中未知转录因子。早期的预测主要采用BLAST序列相似性搜索,即以不同家族的已知转录因子DBD序列为检测序列,设置恰当的参数,用安装到本地的BLAST软件包,逐个搜索不同物种基因组中蛋白质编码序列,并对搜索结果进行计算机和人工筛选,剔除假阳性结果。
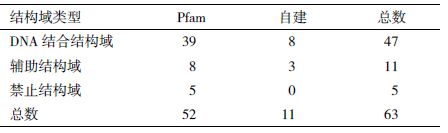
基于隐马氏模型(Hidden markov model,HMM)的序列分析软件包HMMER在蛋白结构域识别方面具有灵敏度高、特异性好的优势,多用于预测同一家族的远缘同源序列[12]。其主要原理为适当选取若干已知种子序列并进行多序列比对,基于隐马氏模型对序列比对结果进行分析并构建隐马氏模型,给出模型参数。因此,我们采用HMMER软件包为主要转录因子预测工具。欧洲生物信息学研究所(European bioinformatics institute,EBI)Bateman领导的研究组,利用HMMER软件包构建了蛋白质结构域数据库Pfam[13]。该数据库还无偿提供他们构建的用于预测蛋白质结构域的隐马氏模型。上述转录因子分类规则中共用到63个隐马氏模型,其中52个取自Pfam数据库,另外11个当时发布的第27版(Pfam V27.0)尚未公布。为此,基于文献和收集到的转录因子序列,利用HMMER软件包,我们构建了这11个结构域的隐马氏模型,用于预测植物基因组中的转录因子(表 1)。为提高预测的准确性,我们基于GO注释[14]、拟南芥信息资源数据库[15](The Arabidopsis information resource,TAIR)和国际蛋白质序列和功能知识库UniProtKB[16]等相关信息,人工检查序列比对结果,并参考Pfam确定阈值的方法,为每个结构域模型确定了一个阈值。
基于上述方法和隐马氏模型,我们构建了植物转录因子预测流程,用于预测植物基因组中未知转录因子[17]。
2.2 预测平台上述用于转录因子预测的隐马氏模型可免费提供国内外用户,便于用户自行构建本地转录因子预测系统,从基因组水平系统预测新测定的基因组中未知转录因子。为方便不具备自行构建本地转录因子预测系统的广大用户,我们在PlantTFDB数据库网站中构建了在线转录因子预测平台,用户可以上载序列,预测未知蛋白序列中的转录因子。目前,模式植物拟南芥的转录因子调控机制研究最为清楚,在PlantTFDB中注释信息也最为详尽。用户若在提交页面勾选“Best hit in Arabidopsis thaliana”,预测结果中则包括相似拟南芥转录因子的超链接,供用户参考。
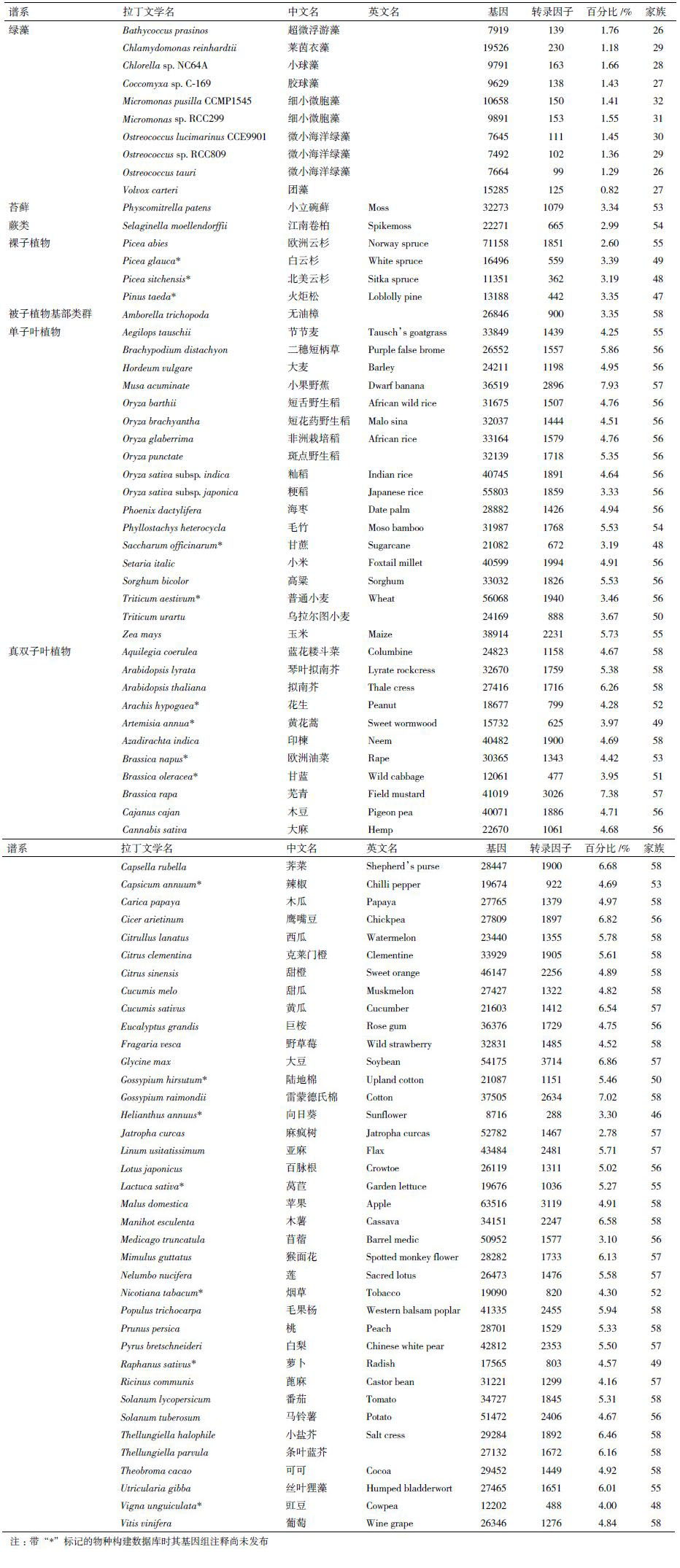
3 植物转录因子数据库构建 3.1 数据库概况2013年更新的第3版植物转录因子数据库PlantTFDB收录了从83个物种预测到的129 288个转录因子,分属58个家族(表 2)。这83个物种覆盖了绿色植物各大谱系,包括10个绿藻、1个苔藓植物、1个蕨类植物、4个裸子植物、1个被子植物基部类群、17个单子叶植物和49个双子叶植物。裸子植物中欧洲云杉(Picea abies)的基因组测序已经完成,填补了旧版PlantTFDB中没有裸子植物全基因组预测所得转录因子的空白。显然,这83个物种中,被子植物占绝大多数(~81%),包括单子叶植物水稻、玉米、高粱、小麦、大麦等主要粮食作物,双子叶植物中棉花、烟草、大豆、番茄、马铃薯、黄瓜、西瓜等重要经济作物,以及葡萄、苹果、梨、橙、橘等水果,为作物分子育种研究提供了宝贵资源。而与模式植物拟南芥同一属的琴叶拟南芥(Arabidopsis lyrata)、同为十字花科的小盐芥(Thellungiella halophila)和条叶蓝芥(Thellungiella parvula)的转录因子数据,则为转录因子家族的起源、演化和功能研究提供了基础。
植物从水生到陆生的演变是生命演化史上的重要事件。横跨绿色植物各大分支的转录因子全谱的发布,使我们可以从转录调控水平研究这一重要历史进程。与绿藻相比,陆生植物无论在转录因子家族数目、转录因子数目及转录因子在基因组中所占比例等方面都明显高于绿藻,与陆生植物更加复杂的多细胞形态发育相关[18]。
3.2 数据库注释高质量的注释信息是植物转录因子数据库PlantTFDB的重要特色。通过查看注释信息,从事植物转录调控研究的生物学工作者可获取该转录因子序列、功能、表达、调控等相关信息,并通过文献信息了解其研究现状。PlantTFDB中的注释信息可以分为两个层次,第一个层次为单个转录因子的注释,第二个层次为家族水平的注释。
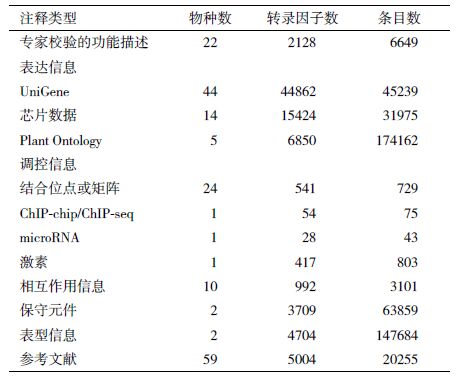
单个转录因子的注释,除名称、序列、结构域等基本信息外,也包括与其它重要数据库的链接。此外,我们从TAIR、UniProtKB和AthMap[19]等公共数据库中全面收集专家校验的功能描述、结合位点/矩阵、microRNA调控、激素调控、相互作用、突变和表型等信息。同时,还通过整合Entrez Gene[20]、GeneRIF[20]以及通过文本挖掘和人工校验获得的文献信息[18],为收录的转录因子提供了相关的参考文献列表。此外,我们还收录了分别基于9个十字花科物种的基因组比对和20个被子植物基因组比对所得到的转录因子结合位点保守元件序列[21, 22](表 3)。
家族水平的注释除了该家族简介和相关文献信息外,还包括该家族成员的演化信息,包括所有物种每个家族成员和每个物种内每个家族成员两类比对信息,以序列图标(Sequence logo)(图 2-A)和系统发生树方式(图 2-B)展示。
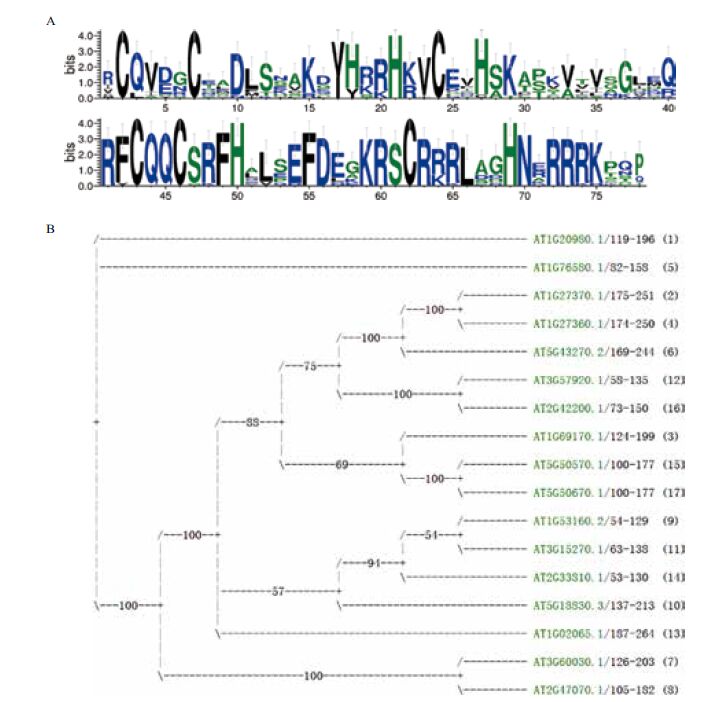
|
| A:拟南芥SBP转录因子DNA结合结构域序列图标;B:拟南芥SBP转录因子DNA结合结构域系统发生树 图 2 转录因子家族水平注释 |
自2005年首次发表拟南芥转录因子数据库DATF[4]至今已有10年,10年来,我们不断扩充和多次更新植物转录因子数据库PlantTFDB。在此期间,德国波茨坦大学、丹麦奥胡斯大学、美国俄亥俄州立大学、日本理化学研究所等单位也构建了相应的植物转录因子数据库(表 4)。与这些数据库相比,PlantTFDB包括的物种最多、注释信息最丰富、更新最及时。目前,该数据库年访问量逾千万次,已成为植物转录因子功能和演化研究的权威数据库和重要数据资源,我们构建的植物转录因子家族分类规则也被国内外同行用于新测序物种转录因子预测。
利用上述数据库资源,我们与其他课题组合作,对AP2/EREBP、MYB、SBP等植物转录因子家族进行了演化和功能分析[32, 33, 34]。同时,对拟南芥转录调控网络进行了深入分析,揭示了植物转录调控网络在结构和演化上的新特征[18]。
不言而喻,随着测序技术的飞速发展,更多植物基因组测序将完成,大量基因组、转录组数据不断发布。随着转录调控研究不断深入,转录因子分类规则有待改进。此外,SELEX等高通量DNA结合特异性测定技术的发展,为深入研究植物转录调控提供了新的契机。结合表达数据、启动子区域和保守元件等信息,预测转录因子下游靶基因,进而构建高质量转录调控网络,探索转录调控的分子机制,必将成为新的研究热点。开发转录调控分析平台,将植物转录因子数据库与数据分析整合起来,则是下一步研究目标。
| [1] | Arabidopsis Genome Initiative. Analysis of the genome sequence of the flowering plant Arabidopsis thaliana[J]. Nature, 2000, 408:796-815. |
| [2] | Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290:2105. |
| [3] | Gong W, Shen YP, Ma LG, et al. Genome-wide ORFeome cloning and analysis of Arabidopsis transcription factor genes[J]. Plant Physiol, 2004, 135:773-782. |
| [4] | Guo A, He K, Liu D, et al. DATF:a database of Arabidopsis transcription factors[J]. Bioinformatics, 2005, 21:2568. |
| [5] | Gao G, Zhong Y, Guo A, et al. DRTF:a database of rice transcription factors[J]. Bioinformatics, 2006, 22:1286. |
| [6] | Zhu QH, Guo AY, Gao G, et al. DPTF:a database of poplar transcription factors[J]. Bioinformatics, 2007, 23:1307. |
| [7] | Duvick J, Fu A, Muppirala U, et al. PlantGDB:a resource for comparative plant genomics[J]. Nucleic Acids Res, 2008, 36:D959-965. |
| [8] | Guo AY, Chen X, Gao G, et al. PlantTFDB:a comprehensive plant transcription factor database[J]. Nucleic Acids Res, 2008, 36:D966-969. |
| [9] | Zhang H, Jin J, Tang L, et al. PlantTFDB 2. 0:update and improvement of the comprehensive plant transcription factor database[J]. Nucleic Acids Res, 2011, 39:D1114-1117. |
| [10] | Jin J, Zhang H, Kong L, et al. PlantTFDB 3. 0:a portal for the functional and evolutionary study of plant transcription factors[J]. Nucleic Acids Research, 2014, 42:D1182-D1187. |
| [11] | Weirauch MT, Yang A, Albu M, et al. Determination and inference of eukaryotic transcription factor sequence specificity[J]. Cell, 2014, 158:1431-1443. |
| [12] | Eddy S. HMMER User’s Guide:Biological sequence analysis using profile hidden Markov models[W]. 2010, http://hmmer.janelia.org/. |
| [13] | Punta M, Coggill PC, Eberhardt RY, et al. The Pfam protein families database[J]. Nucleic Acids Research, 2012, 40:D290-D301. |
| [14] | Ashburner M, Ball CA, Blake JA, et al. Gene ontology:tool for the unification of biology. The Gene Ontology Consortium[J]. Nat Genet, 2000, 25(1):25-29. |
| [15] | Lamesch P, Berardini TZ, Li D, et al. The Arabidopsis Information Resource(TAIR):improved gene annotation and new tools[J]. Nucleic Acids Res, 2012, 40:D1202-210. |
| [16] | UniProt Consortium. Activities at the universal protein resource(UniProt)[J]. Nucleic Acids Research, 2014, 42:D191-D198. |
| [17] | He K, Guo AY, Gao G, et al. Computational identification of plant transcription factors and the construction of the PlantTFDB database[M]//Computational Biology of Transcription Factor Binding. Humana Press, 2010:351-368. |
| [18] | Jin J, He K, Tang X, et al. An Arabidopsis transcriptional regulatory map reveals distinct functional and evolutionary features of novel transcription factors[J]. Molecular Biology and Evolution, 2015, 32:1767-1773. |
| [19] | Bulow L, Engelmann S, Schindler M, et al. AthaMap, integrating transcriptional and post-transcriptional data[J]. Nucleic Acids Res, 2009, 37:D983-D986. |
| [20] | Maglott D, Ostell J, Pruitt KD, et al. Entrez Gene:gene-centered information at NCBI[J]. Nucleic Acids Research, 2011, 39:D52-D57. |
| [21] | Haudry A, Platts AE, Vello E, et al. An atlas of over 90, 000 conserved noncoding sequences provides insight into crucifer regulatory regions[J]. Nature Genetics, 2013, 45:891-898. |
| [22] | Baxter L, Jironkin A, Hickman R, et al. Conserved noncoding sequences highlight shared components of regulatory networks in dicotyledonous plants[J]. The Plant Cell Online, 2012, 24:3949-3965. |
| [23] | Pérez-Rodríguez P, Riaño-Pachón DM, Corrêa LGG, et al. PlnTFDB:updated content and new features of the plant transcription factor database[J]. Nucleic Acids Research, 2010, 38:D822-827. |
| [24] | Fredslund J. DATFAP:a database of primers and homology alignments for transcription factors from 13 plant species[J]. BMC Genomics, 2008, 9:140. |
| [25] | Mochida K, Yoshida T, Sakurai T, et al. TreeTFDB:An integrative database of the transcription factors from six economically important tree crops for functional predictions and comparative and functional genomics[J]. DNA Research, 2013, 20:151-162. |
| [26] | Yilmaz A, Nishiyama Jr MY, Fuentes BG, et al. GRASSIUS:a platform for comparative regulatory genomics across the grasses[J]. Plant Physiology, 2009, 149:171. |
| [27] | Mochida K, Yoshida T, Sakurai T, et al. LegumeTFDB:an integrative database of Glycine max, Lotus japonicus and Medicago truncatula transcription factors[J]. Bioinformatics, 2010, 26:290-291. |
| [28] | Iida K, Seki M, Sakurai T, et al. RARTF:database and tools for complete sets of Arabidopsis transcription factors[J]. DNA Res, 2005, 12:247-256. |
| [29] | Yilmaz A, Mejia-Guerra MK, Kurz K, et al. AGRIS:the Arabidopsis gene regulatory information server, an update[J]. Nucleic Acids Res, 2011, 39:D1118-1122. |
| [30] | Rushton PJ, Bokowiec MT, Laudeman TW, et al. TOBFAC:the database of tobacco transcription factors[J]. BMC Bioinformatics, 2008, 9:53. |
| [31] | Romeuf I, Tessier D, Dardevet M, et al. wDBTF:an integrated database resource for studying wheat transcription factor families[J]. BMC Genomics, 2010, 11:185. |
| [32] | Feng JX, Liu D, Pan Y, et al. An annotation update via cDNA sequence analysis and comprehensive profiling of developmental, hormonal or environmental responsiveness of the Arabidopsis AP2/EREBP transcription factor gene family[J]. Plant Mol Biol, 2005, 59:853-68. |
| [33] | Chen YH, Yang XY, He K, et al. The MYB transcription factor superfamily of Arabidopsis:expression analysis and phylogenetic comparison with the rice MYB family[J]. Plant Mol Biol, 2006, 60:107-124. |
| [34] | Guo AY, Zhu QH, Gu X, et al. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family[J]. Gene, 2008, 418:1-8. |