微生物氧化还原态无机硫化物和元素硫生成硫酸是自然界硫循环过程中的一个重要的反应。此反应能为微生物的生长提供能量[1]。已知在微生物中,主要存在两种硫氧化系统:第一种硫氧化系统——Sox系统,以化能自养型细菌Paracoccus pantotrophus为主要代表菌,该系统位于周质,由4种蛋白质SoxYZ、SoxAX、SoxB和Sox(CD)2组成,负责催化硫化物、元素硫、硫代硫酸盐及亚硫酸盐的氧化生成硫酸,此过程中伴随着电子转移到细胞色素c[2]。第二种硫氧化系统依赖于硫加氧还原酶(Sulfur oxygenase reductase,SOR),可催化元素硫的氧化和歧化反应,产生亚硫酸盐、硫代硫酸盐及硫化物[1, 2, 3, 4],反应过程中无需提供额外的辅酶和电子载体,反应如下:
加氧酶:S0+O2+H2O→HSO3-+H+
歧化反应:3S0+3H2O→2H2S+ HSO3-+H+
总反应式:4S0+ O2+4H2O→2H2S+2HSO3-+2H+
此反应体系中,亚硫酸盐和过量的元素硫会经非酶促反应迅速形成硫代硫酸盐:
S0+ HSO3- → S2O32-+H+
目前的研究表明,SOR或sor基因在自然界中并非广泛存在,主要存在于一些嗜热嗜酸古菌和嗜超高温细菌中。另外,一些嗜中温细菌中也存在sor基因,如嗜酸硫杆菌。嗜酸喜温硫杆菌(Acidithiobacillus caldus,A. caldus)是一种专性化能自养型嗜中温嗜酸性革兰氏阴性菌。它与嗜酸氧化亚铁硫杆菌、嗜酸氧化硫硫杆菌共同存在于浸矿的生态龛位,并广泛应用于生物冶金。嗜酸喜温硫杆菌能够氧化元素硫和各种还原态无机硫化物(Reduced inorganic sulfur compounds,RISCs)生成硫酸而释放出H+,从而使矿物溶解浸出。考虑到嗜酸喜温硫杆菌中RISCs的代谢与最终矿物的浸出有着直接的相关性,因此,研究此菌氧化RISCs的机理具有重大理论意义和实践价值。已知,嗜酸喜温硫杆菌的硫氧化系统分为3个亚类——缩短型Sox硫氧化亚系统、非Sox硫氧化亚系统和SOR亚系统[2]。其中,SOR亚系统在细胞质的硫氧化过程中起着重要的作用,其关键酶——硫加氧还原酶能够氧化细胞质中无论是胞外转运进入的还是由其他途径代谢生成的单质硫,由此与另两种硫氧化亚系统关联到一起,共同完成RISCs的氧化。研究该菌中的硫加氧还原酶的生化特性对于理解该菌如何代谢RISCs至关重要,但是目前关于这方面的报道很少。本研究克隆嗜酸喜温硫杆菌的硫加氧还原酶基因sor,在大肠杆菌中大量表达,并以Ni-NTA亲和层析柱纯化出重组蛋白SOR,研究其酶活性,旨在为研究嗜酸喜温硫杆菌SOR亚系统中的硫加氧还原酶的生化特性提供参考。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒嗜酸喜温硫杆菌TST3为本实验室从湖北大冶铜山口矿区酸性排污口附近的土壤中分离纯化、鉴定且保藏; 大肠杆菌表达载体pET-28a由安徽大学张部昌教授惠赠。
1.1.2 培养基Waksman培养基用于培养嗜酸喜温硫杆菌[5]; LB培养基用于培养大肠杆菌,筛选抗性菌株时,加入终浓度为100 μg/mL的卡那霉素[6]。
1.1.3 酶和化学试剂DNA Marker、loading buffer、dNTP mixture、pfu DNA聚合酶、溶菌酶溶液、蛋白酶K溶液、RNaseA溶液、大肠杆菌DH5α和BL21(DE3)感受态细胞及质粒小提中量试剂盒为天根生化科技(北京)有限公司产品; 限制性核酸内切酶Nde I、高保真内切酶EcoR I-HF及Protein Marker为New England Biolabs(NEB)公司产品; Micro BCATM Protein Assay Kit由Thermo Pierce公司提供; QIAquick Gel Extraction Kit为Qiagen公司产品; T4 DNA连接酶、IPTG为TaKaRa公司产品; 引物合成和测序工作由北京奥科鼎盛生物科技有限公司完成。
1.2 方法 1.2.1 引物的设计及硫加氧还原酶基因的克隆根据GenBank中登录号为NZ_ACVD010000-78的A.caldus ATCC 51756鸟枪法测序全基因组序列片段02162006.asm.C78中37 049-37 984 bp的sor基因,设计合成引物。为便于克隆,在正向引物和反向引物前分别加上NdeⅠ和EcoRⅠ酶切位点,设计的上下游扩增引物分别为:引物sor-1:5'-GCCATAT-GGACAAAAATCCTATCGTC-3'(正向引物,下划线为NdeⅠ酶切位点,前面GC为保护性碱基); 引物sor-2:5'-GGAATTCTCAGAGCACAAGTTTGCGCC-3'(反向引物,下划线为EcoRⅠ酶切位点,前面G为保护性碱基)。从嗜酸喜温硫杆菌TST3中提取基因组DNA[7],并以其为模板,利用上述的引物进行PCR扩增。采用的PCR扩增体系如下:嗜酸喜温硫杆菌TST3的基因组DNA 2 μL,dNTP(2.5 mmol/L)5 μL,10×pfu DNA聚合酶缓冲液5 μL,引物sor-1(2.5 μmmol/L)10 μL,引物sor-2(2.5 μmmol/L)10 μL,pfu DNA聚合酶0.5 μL,最后补无菌水至50 μL。PCR扩增条件为:95℃预变性5 min; 95℃变性45 s,63℃退火1 min,72℃延伸90 s,共30个循环; 72℃延伸10 min。
1.2.2 重组表达质粒的构建及目标序列分析PCR扩增产物及载体pET-28a分别采用NdeⅠ和EcoRⅠ双酶切,回收目的片段并连接,构建重组质粒pET-sor,并采用氯化钙转化法将质粒转化到E.coli DH5α。提取质粒进行双酶切来验证阳性克隆并测序,通过NCBI数据库提供的BlastN和BlastP功能分别对测序结果进行核苷酸序列及氨基酸序列对比分析。针对嗜酸喜温硫杆菌SOR氨基酸序列和NCBI数据库中查询得到的各种微生物的SOR同源类似物序列,利用Clustal Omega(http://www.ebi.ac.uk/Tools/msa/clustalo/,由EMBL数据库提供)和Mega 5.05软件进行各序列间的同源性和进化距离的分析与比较,并构建系统发育树[8]。
1.2.3 SOR酶在大肠杆菌BL21(DE3)中的诱导表达将测序正确的pET-sor重组质粒转化到大肠杆菌BL21(DE3)中。挑取若干单菌落,接种至含100 μg/mL卡那霉素的10 mL LB培养基中,37℃培养过夜后,按1%的接种量接入到100 mL含100 μg/mL卡那霉素的LB培养基(250 mL锥形瓶)中,37℃培养大约3 h(OD600约为0.8)后,加入终浓度为1 mmol/L的IPTG诱导酶的表达,继续培养4 h[9],离心(8 000 r/min,4℃,10 min)收集菌体,超声波破碎后,离心取上清进行SDS-PAGE电泳,分析蛋白质的分子量和存在性质,采用的分离胶浓度为10%,使用考马斯亮蓝R-250进行染色。利用BandScan 5.0软件分析目标蛋白的SDS-PAGE扫描图片,以了解重组菌株中目标蛋白质的表达量占全菌可溶性蛋白质的百分比。
1.2.4 SOR蛋白质的纯化方法离心(8 000 r/min,4℃,10 min)收集经IPTG诱导的E. coli BL21(pET-sor2)菌体,用1×PBS缓冲液(pH 7.4)洗涤3次后,再重悬,在冰水浴中进行超声波破碎(400 W下工作2 s,间歇3 s,共破碎30 min),离心(13 000 r/min,4℃,10 min),收集上清,即得SOR粗酶液。取粗酶液上样于经1×PBS缓冲液预平衡的Ni+-NTA柱,先用1×PBS缓冲液清洗,再用含20 mmol/L咪唑的1×PBS缓冲液洗脱,最后用含80 mmol/L咪唑的1×PBS缓冲液洗脱,并收集各步骤液体。
1.2.5 SOR酶活力的测定硫粉(2%)加入到含有0.005% 吐温20的70 mmol/L Tris-HCl缓冲液(pH 7.2)中,用最大功率的超声波处理10 min,粉碎硫粉。取1 mL处理后的硫粉液,加入100 µL的SOR粗酶液或纯化后的酶液(阴性对照加入的是1 mL的pET-28a裂解液上清)。在80℃条件下反应10 min后[1],分别测定反应液上清中的亚硫酸盐、硫代硫酸盐[10]及S2-浓度[11]。酶活单位定义为每分钟形成亚硫酸盐和硫代硫酸盐(SOR催化的氧化反应)或硫化氢(SOR催化的还原反应)的微摩尔数[4]。而比酶活为每毫克蛋白形成的亚硫酸盐和硫代硫酸盐的量或每毫克蛋白质形成的硫化氢量[10]。蛋白质含量的测定采用BCA法,以牛血清白蛋白为标准品。
SOR酶的Km和Vmax值的测定方法:取2-5 µg 纯化的酶,分别温育于1 mL的含有0.4%、0.8%、1.2%、1.6%及2% 硫处理液(含有0.1%吐温20)的70 mmol/L Tris-HCl(pH 7.2)中。在65℃条件下反应0、2、4、6、8及10 min后,测亚硫酸盐,硫代硫酸盐和硫化物的量(假设亚硫酸盐和硫化物是反应的唯一产物,而硫代硫酸盐来自硫和亚硫酸盐的非酶促反应)。反应速率V从产物浓度的线性增长中获得,再根据Linewaver-Burk作图法,以1/V对1/[S]作图,得到一条直线,直线在横轴上的截距为-1/Km,纵截距为1/Vmax,从而求出Km与Vmax。
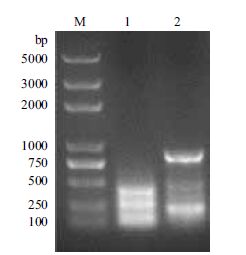
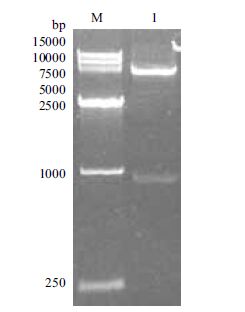
2 结果 2.1 pET-sor重组质粒的构建及鉴定以嗜酸喜温硫杆菌TST3的基因组DNA为模板,PCR扩增出嗜酸喜温硫杆菌硫加氧还原酶的基因片段sor,PCR产物进行1.0%的琼脂糖凝胶电泳,结果(图 1)显示,PCR产物位于750 bp和1 000 bp之间,与目标基因的序列长度936 bp吻合。将该片段克隆到pET-28a载体上,构建了重组质粒pET-sor。对该质粒进行双酶切鉴定(图 2),证实了质粒pET-sor已克隆了目标片段的重组质粒。
|
| M:DL5 000 DNA ladder; 1:阴性对照; 2:sor基因的PCR产物 图 1 硫加氧还原酶基因PCR产物琼脂糖凝胶电泳图 |
|
| M:DL15000 DNA ladder; 1:pET-sor质粒的双酶切产物 图 2 pET-sor质粒的双酶切产物琼脂糖凝胶电泳图 |
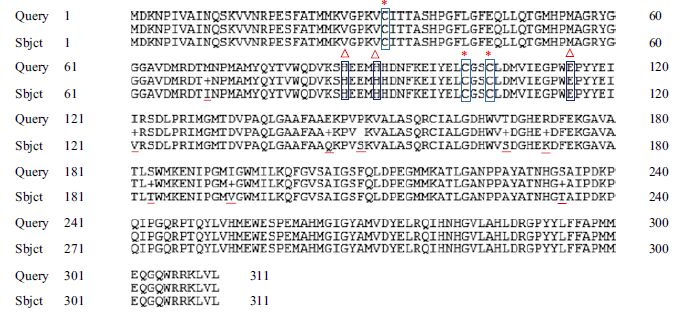
将重组质粒pET-sor送交测序,获得了一段长1 135 bp的核苷酸序列,利用NCBI数据库中的Vecscreen功能将其中pET-28a载体中的序列片段去除后,得到了长度为936 bp的嗜酸喜温硫杆菌TST3的sor基因序列,将此序列提交到GenBank,登录号为KJ958902。利用NCBI上提供的核苷酸比对功能(BlastN),对重组质粒的测序结果进行序列分析,得出重组质粒克隆的基因片段与登录号为NZ_ACVD01000078的A. caldus ATCC 51756鸟枪法测序全基因组的序列片段02162006.asm.C78中的37 049 bp至37 984 bp的sor基因序列之间的相似度为97%,两者的碱基序列有28处不同,而相应的嗜酸喜温硫杆菌TST3的SOR(简称AcSOR)氨基酸序列与NCBI数据库中登录号AIA55075.1的嗜酸喜温硫杆菌ATCC 51756硫加氧还原酶蛋白质序列的相似度达到97%,两者之间有9处不同(图 3中下标线标出),其中除了S→P的突变外,其余的突变都是理化性质基本相同的氨基酸残基,如S→T、T→S、I→M、V→I、K→R及Q→E。已知,硫加氧还原酶的活性中心由3个保守半胱氨酸(V-G-P-K-V-C和C-X-X-C ,其中X代表各种不同的氨基酸,图 3中*号标出)和一个Fe结合位点(H-X3-H-X23-E,其中X代表各种不同的氨基酸,图 3中Δ号标出)组成[1]。而AcSOR中的氨基酸突变均未发生在活性中心中。另外,对比各种微生物中的SOR氨基酸序列发现,这些突变位点并不保守,通常变化较大,且其他微生物的氨基酸序列中也出现过这些突变的氨基酸[3],推测这些氨基酸突变对酶的催化活性不会产生较大的影响。
|
| Query:嗜酸喜温硫杆菌TST3的SOR; Sbjct:嗜酸喜温硫杆菌ATCC 51756的SOR 图 3 pET-sor重组质粒测序结果的氨基酸序列比对分析结果 |
根据AcSOR的氨基酸序列比对(BlastP)结果,在GenBank数据库中找出与AcSOR序列一致性高于40%的蛋白质序列,进行多重序列比对,再利用Mega 5.05软件构建进化树(图 4),嗜酸喜温硫杆菌TST3与A.caldus ATCC 51756处于系统发育树中的同一分支,一致性可达97%。
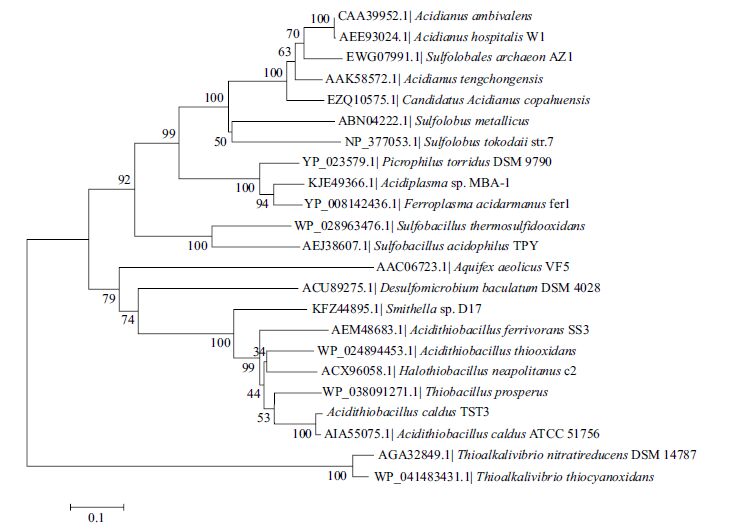
|
| 图 4 根据SOR氨基酸序列同源性构建的系统发育树 |
表达载体pET-sor转化到E.coli BL21(DE3)后,获得了8个E.coli BL21(pET-sor)重组菌株。以E. coli BL21(pET-28a)菌株为阴性对照,经IPTG诱导后,进行SDS-PAGE分析。结果(图 5)显示,与阴性对照相比,8个E.coli BL21(pET-sor)重组菌,均在相对分子量40 kD和35 kD之间,有明显的SOR蛋白表达条带,与目标蛋白SOR的相对分子量约为36 kD一致(包括了pET-28a载体表达的His-tag片段)。而且,利用BandScan 5.0软件分析目标蛋白的SDS-PAGE扫描图片,可知,在8个E.coli BL21(pET-sor)重组菌中,E.coli BL21(pET-sor2)的表达量最大(图 5中的孔道2),其目标蛋白质的表达量占全菌可溶性蛋白质的69.8%。另外,SDS-PAGE分析经IPTG诱导后的E.coli BL21(pET-sor2)重组菌株中的上清液、周质蛋白以及超声波破碎获得的胞内蛋白,结果显示目标蛋白分布于胞内,而且主要以包涵体形式存在。
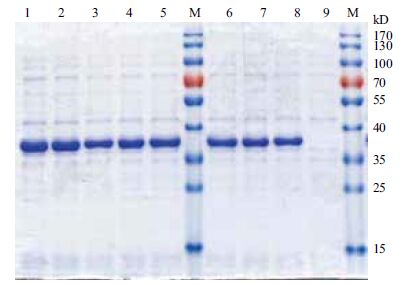
|
| M: 标准蛋白Marker ; 1-8: IPTG诱导的E.coli BL21 (pET-sor)蛋白, 从左到右的重组菌分别为:E.coli BL21 (pET-sor1)、E.coli BL21 (pET-sor2)、E.coli BL21 (pET-sor3)、E.coli BL21 (pET-sor4)、E.coli BL21 (pET-sor5)、E.coliBL21 (pET-sor6)、E.coli BL21 (pET-sor7)、E.coli BL21 (pET-sor8); 9: IPTG诱导的E.coli BL21 (pET-28a)蛋白(阴性对照) 图 5 E.coli BL21(pET-sor)重组菌株的SDS-PAGE图(全菌可溶性蛋白) |
对比不同的诱导温度、诱导时间及IPTG浓度的条件下的硫加氧还原酶的比酶活,获得E.coli BL21(pET-sor2)重组菌的最佳诱导条件。
2.3.1 诱导温度的优化在接种后3 h的菌液中加入1 mmol/L的IPTG后,分别置于16、20、25、30和37℃下进行诱导,诱导时长为4 h。结果(图 6)显示,比起较低的诱导温度,在较高温度下(如30和37℃)诱导表达的SOR催化氧化反应的比酶活相对较高,而催化还原反应的比酶活则相对较低。另外,由于低温下诱导酶的表达有助于酶的正确折叠[12],于是观察到16-25℃下诱导表达生成包涵体的量相对于30-37℃较低。根据两种反应的最适诱导温度和包涵体的生成量,选择25℃作为最适的诱导温度。
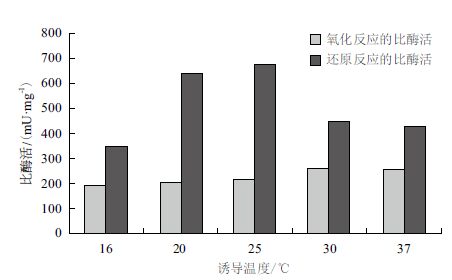
|
| 图 6 诱导温度的优化 |
接种3 h后,加入1 mmol/L 的IPTG,置于25℃分别诱导2、3、4、5、6、7及8 h(图 7),诱导6 h表达的SOR催化氧化反应的比酶活最优,而诱导4 h表达的酶催化还原反应的比酶活最优。结合两者,确定较好的诱导时间为4 h。
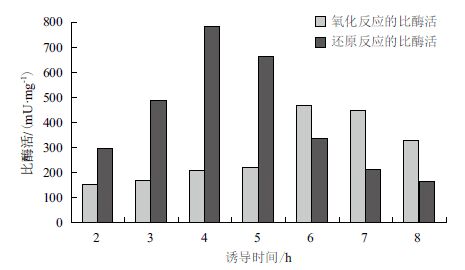
|
| 图 7 诱导时间的优化 |
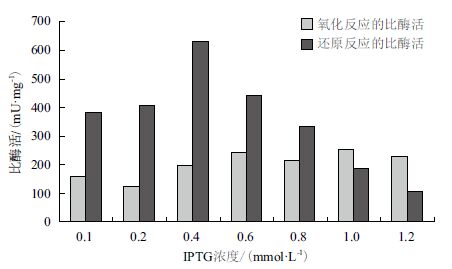
接种3 h后,分别加入0.1、0.2、0.4、0.6、0.8、1.0及1.2 mmol/L 的IPTG,置于25℃诱导4 h。图 8显示,在加入0.6 mmol/L 的IPTG的条件下,SOR催化氧化反应的比酶活相对最好,而加入0.4 mmol/L 的IPTG时,SOR催化还原反应的比酶活相对最好。由此确定最适的IPTG浓度为0.4 mmol/L。
|
| 图 8 IPTG诱导浓度的优化 |
综上所述,由以上的单因子实验可以得出,重组Sor酶优化的诱导条件为:加入0.4 mmol/L的IPTG后,在25℃的温度下诱导4 h。
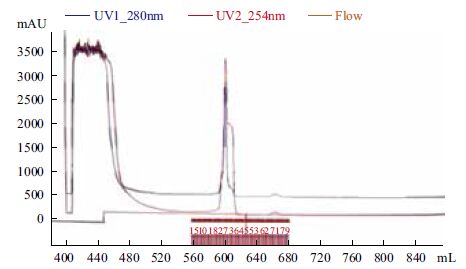
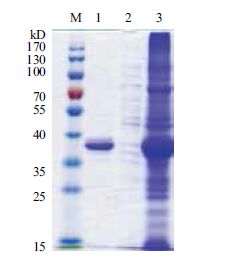
2.4 硫加氧还原酶的分离纯化及酶活研究在优化的条件下诱导E.coli BL21(pET-sor2)中硫加氧还原酶的表达。取经超声波破碎此重组菌获得的粗酶液,与1×PBS缓冲液预平衡的镍柱结合,目的蛋白吸附到镍柱上。之后分别用0、20 和80 mmol/L咪唑的1×PBS缓冲液进行洗脱,层析过程如图 9所示,经1×PBS缓冲液洗下来的峰为杂蛋白,而目标蛋白SOR是经20 mmol/L的1×PBS缓冲液洗脱下来。洗脱下来的纯化酶液和纯化前的粗酶液分别进行SDS-PAGE检测,结果(图 10)表明,纯化前的条带很多,而纯化后几乎所有的杂蛋白都消失了,说明纯化的效果较好。
|
| 图 9 SOR酶的Ni-NTA纯化层析图谱 |
|
| M:标准蛋白Marker; 1: 纯化后的SOR蛋白; 2: IPTG诱导的E.coli BL21 (pET-28a)的蛋白(阴性对照); 3: 纯化前的E.coli BL21 (pET-sor2)粗酶液 图 10 SOR纯化前后的SDS-PAGE图谱 |
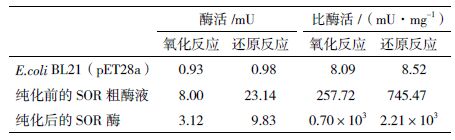
根据纯化前后的SOR与硫粉反应后的溶液中S2-离子、SO32-离子和S2O32-离子的浓度以及蛋白浓度,算出纯化前粗酶液及纯化后的SOR蛋白进行氧化反应、还原反应的酶活及比酶活,结果见表 1,纯化前的SOR粗酶液(100 μL)催化氧化反应的酶活为8.00 mU,催化还原反应的酶活为23.14 mU,而作为阴性对照的E.coli BL21(pET28a),经超声波破碎后获得的粗酶液(1 mL),催化氧化反应和还原反应的酶活仅为0.93 mU和0.98 mU。含3.10 mg蛋白质总量的经超声破碎后的重组菌上清液,经镍柱纯化后,获得含667.35 µg蛋白质总量的SOR酶。纯化后的SOR酶,催化氧化反应的酶活为3.12 mU,而催化还原反应的酶活为9.83 mU。另外,纯化前后的SOR酶催化氧化反应的比酶活分别为257.72 mU/mg和0.70 U/mg,而催化还原反应的比酶活分别为745.47 mU/mg和2.21 U/mg。
另外,研究SOR酶的反应动力学常数Km和Vmax时,考虑到硫代硫酸盐是非酶促反应的产物,所以以亚硫酸盐的量随着反应时间的变化绘制氧化反应曲线,以硫化物的量随着反应时间的变化绘制还原反应曲线。再根据氧化反应曲线和还原反应曲线中线性部分的斜率,分别计算在不同的底物浓度下氧化反应和还原反应的初始反应速率V,以1/V对1/[S]作图,得到两条直线。其中,氧化反应的1/V与初始底物浓度的倒数1/[S]的关系为y=1.228 7x+0.078 4(R2=0.976 5),而还原反应的1/V与1/[S]的关系为y=1.039 7x+2.050 9(R2=0.971 6)。根据这些直线的横截距和纵截距,可以计算得到氧化反应的Km和Vmax分别为15.672×10-2 g/mL和12.755×10-5 mol/(L·min),而还原反应的Km和Vmax分别为0.507×10-2 g/mL和4.876×10-5 mol/(L·min)。
3 讨论硫加氧还原酶依赖于氧气、催化元素硫的氧化和歧化反应,生成亚硫酸盐、硫代硫酸盐和硫化氢。普遍认为,SOR或sor基因主要存在于嗜热嗜酸古菌和嗜超高温(或高温)型细菌。天然SOR酶最早从化能自养型嗜热嗜酸型古菌Acidianus brierleyi(其SOR酶简称AbSOR)、Acidianus ambivalens(其SOR酶简称AaSOR)和Acidianus tengchongensis(其SOR酶简称AtSOR)中被分离纯化出来 [10, 13, 14, 15, 16]。例如,2000年,He等[14]报道Acidianus tengchongensis S5细胞裂解液中的AtSOR催化氧化反应和还原反应的比酶活分别为28.6 U/mg和3.1 U/mg。2004年,Urich等[15]发现直接从Acidianus ambivalens纯化获得的AaSOR酶催化氧化反应和还原反应的比酶活分别为2.96 U/mg和0.6 U/mg。后来,陆续有关于异源表达AaSOR和AtSOR的报道[3, 14, 15, 17]。2004年,Urich等[15]发现异源表达AaSOR的大肠杆菌粗酶液催化氧化反应和还原反应的比酶活分别为464 mU/mg和145 mU/mg,而经纯化后的重组AaSOR酶的比酶活可以分别达到2.82 U/mg和0.66 U/mg。2011年,Veith等[3]发现从异源表达AaSOR的大肠杆菌中分离纯化获得的重组AaSOR催化氧化反应和还原反应的比酶活分别为3.03 U/mg和1.69 U/mg。此外,在其他的一些古菌,如泉古菌门的Sulfolobus tokodaii和广古菌门的Ferroplasma acidarmanus、Picrophilus torridus,以及嗜超高温细菌Aquifex aeolicus中也都检测到sor基因的存在[18],并且相继有些报道完成了这些古菌中sor基因的异源表达。如2008年,Pelletier等[19]发现从嗜超高温菌Aquifex aeolicus中分离纯化得到的SOR催化还原反应的比酶活为5 U/mg,而该SOR酶在大肠杆菌中异源表达的重组酶催化氧化反应和歧化反应的比酶活分别为78.8 U/mg和3.05 U/mg。另外,发现sor基因存在于许多嗜中温细菌和中等嗜热菌中,如Halothiobacillus neapolitanus、Sulfobacillus acidophilus及Acidithiobacillus species等,显示SOR并非局限存在于极端嗜超高温细菌中[4, 20]。例如,在重组大肠杆菌中异源表达的嗜中温菌Halothiobacillus neapolitanus的SOR(简称HnSOR)催化氧化反应和还原反应的比酶活分别为42.4 U/mg和4.1 U/mg[4]。
虽然,国内外不断出现关于硫加氧还原酶的活性研究及异源表达的报道,但是有关嗜酸硫杆菌属中的SOR酶活及异源表达的研究却鲜有报道。2007年,Chen等[18]发现从异源表达嗜中温细菌Acidithiobacillus sp. SM-1(现更名为A.caldus SM-1)的SOR的大肠杆菌中获得的粗酶液催化氧化反应的比酶活为3.76 U/mg,但此研究并未报道该酶液催化还原反应的活性,也未尝试研究纯化后的酶反应动力学。2012年,You等[21]构建了克隆Acidithiobacillus sp. SM-1中的sor基因(sorSB)的重组质粒pBV220/sorSB,实现了SOR酶(SORSB)在大肠杆菌HB101中的异源表达。另外,此研究还比较了来自Acidianus ambivalens和Sulfolobus tokodaii及Acidithiobacillus sp. SM-1的SOR酶的温度稳定性,得出经Superdex 200凝胶过滤系统纯化后的重组SORSB酶在70℃的条件下催化氧化反应的比酶活大约可以达到0.3 U/mg,但该研究并未分析该酶催化还原反应的活性。本研究中首次采用具有His-tag标签的pET-28a表达载体来构建重组质粒pET-sor,并选择金属螯合亲和层析从重组菌中纯化SOR酶,这样简化了蛋白质纯化过程,且纯化后的SOR具有较高的催化活性,催化氧化反应和还原反应的比酶活可以分别达到0.70 U/mg和2.21 U/mg,纯化的回收率约为40%,纯化倍数可约达3倍。本研究还分析了该重组酶的反应动力学特性,获得了此酶催化氧化反应和还原反应的Km和Vmax值。
另外,本研究中所使用的菌种Acidihthiobacillus caldus TST3为本实验室从湖北大冶铜山口矿区酸性排污口附近的土壤中筛选鉴定获得的一株浸磷菌,从中克隆出的sor基因所编码的蛋白质序列(AcSOR)与GenBank中所报道的模式菌株A.caldus ATCC 51756的SOR氨基酸序列的相似性为97%,其中有9处突变位点。对比AcSOR与各种微生物中的SOR氨基酸序列,可知这些突变位点并不保守,在不同微生物中变化较大,且不存在于酶活性中心。另外,酶活测定结果显示这种突变的AcSOR仍可以催化单质硫的氧化和歧化反应,产生亚硫酸盐、硫代硫酸盐及硫化物,具有一定的酶活。以上结果均说明这些突变不会或只会微弱地影响酶的活性。为了进一步确定上述突变位点对SOR酶的活性产生的影响及相应的机理,目前本实验室致力于构建回复突变型重组SOR酶。通过对比突变前后的两种酶催化反应的差异,以了解AcSOR的酶促反应机制,为下一步进行分子改造奠定基础。
4 结论以嗜酸喜温硫杆菌TST3的硫加氧还原酶基因为模板,PCR扩增获得目的片段,选用pET-28a质粒为表达载体,构建了异源表达嗜酸喜温硫杆菌硫加氧还原酶的大肠杆菌。优化了重组菌株的诱导条件,并经Ni+-NTA层析柱分离纯化获得重组AcSOR,其催化氧化反应和还原反应的比酶活分别达到0.70 U/mg和2.21 U/mg。且该重组AcSOR催化氧化反应的Km和Vmax分别为15.672×10-2 g/mL和12.755×10-5 mol/(L·min),催化还原反应的Km和Vmax分别为0.507×10-2 g/mL和4.876×10-5 mol/(L·min)。
| [1] | Urich T, Gomes CM, Kletzin A, et al. X-ray structure of a self-compartmentalizing sulfur cycle metalloenzyme[J]. Science, 2006, 311(5763):996-1000. |
| [2] | Chen LX, Ren YL, Lin JQ, et al. Acidithiobacillus caldus sulfur oxidation model based on transcriptome analysis between the wild type and sulfur oxygenase reductase defective mutant[J]. PLoS One, 2012, 7(9):e39470. |
| [3] | Veith A, Urich T, Seyfarth K, et al. Substrate pathways and mechanisms of inhibition in the sulfur oxygenase reductase of Acidianus ambivalens[J]. Frontiers in Microbiology, 2011, 2:37. |
| [4] | Veith A, Botelho HM, Kindinger F, et al. The sulfur oxygenase reductase from the mesophilic bacterium Halothiobacillus neapolitanus is a highly active thermozyme[J]. Journal of Bacteriology, 2012, 194(3):677-685. |
| [5] | 吴为荣, 郑志宏, 刘金辉, 等. 氧化硫硫杆菌的耐氟驯化试验研究[J]. 有色金属:冶炼部分, 2007(3):38-40. |
| [6] | 沈萍, 范秀容, 李广武. 微生物学实验[M]. 第3版. 北京:高等教育出版社, 1999. |
| [7] | 吕早生, 关海燕, 李凌凌, 等. 一株高浸磷嗜酸氧化硫硫杆菌的分离与鉴定[J]. 应用与环境生物学报, 2011, 17:326-329. |
| [8] | 李凌凌, 吕早生, 吴敏, 等. 二苯并噻吩降解菌的筛选鉴定及煤脱硫的研究[J]. 环境工程学报, 2010, 4(10):2391-2396. |
| [9] | 李凌凌, 吕早生, 吴敏, 等. 重组的葡萄糖脱氢酶催化辅酶的再生性质[J]. 华中科技大学学报:自然科学版, 2010, 38(3):112-115. |
| [10] | Kletzin A. Coupled enzymatic production of sulfite, thiosulfate and hydrogen sulfide from sulfur:purification and properties of a sulfur oxygenase reductase from the facultatively anaerobic archaebacterium Desulfurolobus ambivalens[J]. Journal of Bacteriology, 1989, 171(3):1638-1643. |
| [11] | Rabinowitz JC. Analysis of acid-labile sulfide and sulfhydryl groups[J]. Methods Enzymol, 1978, 53:275-277. |
| [12] | 管启旺, 马江锋, 贺爱永, 等. 敏捷嗜酸菌腈水解酶基因在大肠杆菌中的表达[J]. 生物技术通报, 2015, 31(1):181-185. |
| [13] | Emmel T, Sand W, K?nig WA, et al. Evidence for the existence of a sulphur oxygenase in Sulfolobus brierleyi[J]. Journal of General Microbiology, 1986, 132(12):3415-3420. |
| [14] | He ZG, Li YQ, Zhou PJ, et al. Cloning and heterologous expression of a sulfur oxygenase/reductase gene from the thermoacidophilic archaeon Acidianus sp. S5 in Escherichia coli[J]. FEMS Microbiology Letters, 2000, 193(2):217-221. |
| [15] | Urich T, Bandeiras TM, Leal SS, et al. The sulphur oxygenase reductase from Acidianus ambivalens is a multimeric protein containing a low-potential mononuclear non-haem iron centre[J]. Biochem J, 2004, 381(Pt1):137-146. |
| [16] | Chen ZW, Jiang CY, She QX, et al. Key role of cysteine residues in catalysis and subcellular localization of sulfur oxygenase-reductase of Acidianus tengchongensis[J]. Applied and Environmental Microbiology, 2005, 71(2):621-628. |
| [17] | Sun CW, Chen ZW, He ZG, et al. Purification and properties of the sulfur oxygenase/reductase from the acidothermophilic archaeon, Acidianus strain S5[J]. Extremophiles, 2003, 7(2):131-134. |
| [18] | Chen ZW, Liu YY, Wu JF, et al. Novel bacterial sulfur oxygenase reductases from bioreactors treating gold-bearing concentrates[J]. Appl Microbiol Biotechnol, 2007, 74(3):688-698. |
| [19] | Pelletier N, Leroy G, Guiral M, et al. First characterisation of the active oligomer form of sulfur oxygenase reductase from the bacterium Aquifex aeolicus[J]. Extremophiles, 2008, 12(2):205-215. |
| [20] | Zhang HJ, Guo WB, Xu CA, et al. Site-specific mutagenesis and functional analysis of active sites of sulfur oxygenase reductase from Gram-positive moderate thermophile Sulfobacillus acidophilus TPY[J]. Microbiological Research, 2013, 168(10):654-660. |
| [21] | You XY, Meng Z, Chen DW, et al. Structural basis for the thermostability of sulfur oxygenase reductase[J]. Chinese Journal of Chemical Engineering, 2012, 20(1):52-61. |