2. 哈尔滨师范大学生命科学与技术学院,哈尔滨150025
2. College of Life Sciences and Technology,Harbin Normal University,Harbin 150025
不利的环境条件会影响植物的生长发育,导致作物的产量降低,大于50%的作物减产是由非生物胁迫引起的[1],其中日益严重的土壤盐渍化是主要的影响因素之一。植物耐盐基因工程是提高植物耐盐性的重要途径之一,但其前提基础是充分了解植物的耐盐分子机制,获得重要的耐盐工具基因。尽管目前植物耐盐基因工程取得了很多进展[2, 3, 4, 5, 6],分离到一些耐盐相关基因,获得一些耐盐性得到提高的转基因植物,但是,这些转基因植物的耐盐性仍然很有限。植物耐盐机制研究的不足,有效耐盐工具基因的缺乏已成为影响植物耐盐基因工程发展的主要障碍[7]。盐生植物星星草(Puccinellia tenuiflora)具有较强的耐盐能力,拥有丰富的耐盐基因资源,是研究植物耐盐分子机制的良好材料。星星草是禾本科碱矛属单子叶盐生草本植物,耐受NaCl的最高浓度可以达到600 mmol/L,耐受Na2CO3的浓度可以达到200 mmol /L[8],能在pH9-10以上的盐碱地上生长发育[9]。
类光诱导蛋白1(Light regulated protein 1,LIR1)基因LIR1与植物的抗逆性密切相关,LIR1在生长于盐碱地的星星草根中表达[10],在400 mmol/L NaHCO3胁迫6 h后于星星草叶中表达[11]。梁涵等[12]以100 mmol/L NaCl处理虎尾草(Chloris virgata)后,类光诱导蛋白1基因LIR1上调表达。在条锈菌CY31侵染早期,类光诱导蛋白1基因LIR1在小麦(Triticum aestivum)品种水源11中表达[13]。这些研究结果表明,LIR1基因参与植物对多种逆境胁迫的应答,可能在植物的耐盐性中发挥重要作用。类光诱导蛋白1基因LIR1可能协助光合作用的进行或光合作用产物的加工[14],而可溶性糖的存在是该过程的负调控因子[15]。在拟南芥(Arabidopsis thaliana)中可溶性糖会抑制类光诱导蛋白1基因的表达[16]。寒冷胁迫下黑麦草(Lolium perenne)类光诱导蛋白1基因LpLIR1先上调表达后下降到本底水平,表达水平的下降可能与可溶性糖的积累有关[15]。此外,类光诱导蛋白1基因还可能与蔗糖水平的调节有关[15]。
在前期的研究中,我们从盐碱地星星草根的消减杂交cDNA文库中分离到PtLIR1基因的表达序列标签(expression sequence tag,EST)[10],表明该基因可能在帮助星星草根抵御盐碱地逆境的过程中发挥重要作用。为了进一步了解该基因在星星草根中响应盐胁迫的分子机制,本研究从星星草中克隆PtLIR1基因,分析NaCl胁迫下PtLIR1基因的表达特性,并对PtLIR1基因表达与蔗糖水平、可溶性糖水平的相关性进行分析。
1 材料与方法 1.1 材料 1.1.1 实验材料以采自肇东盐碱地的星星草根和温室培养的星星草幼苗为实验材料。
1.1.2 主要试剂pMD19-T Simple载体(大连宝生物工程有限公司),大肠杆菌感受态细胞JM109(大连宝生物工程有限公司),PrimeScriptTM RT reagent Kit (Perfect Real Time)及SYBR® Premix Ex TaqTMⅡ(Tli RNaseH Plus) (大连宝生物工程有限公司),RNA清洁纯化试剂盒(天津原平皓生物技术有限公司),胶回收试剂盒(上海生工生物工程公司)。
1.2 方法 1.2.1 星星草的培养与处理以采自肇东盐碱地的星星草根为材料克隆PtLIR1基因。以温室培养的星星草幼苗为材料进行基因表达分析。将星星草种子萌发后种植在盛有草炭土和细沙(21)花盆中,温室培养8周,以300 mmol/L NaCl分别胁迫星星草幼苗0、6、12、24和48 h,将根和地上部分开,液氮速冻后于-70℃贮存,用于荧光半定量RT-PCR分析。
1.2.2 星星草PtLIR1基因的克隆以前期研究中分离到的星星草PtLIR1基因的EST序列为信息探针,在NCBI GenBank EST数据库中检索到星星草PtLIR1基因的EST序列EB104311.1,经生物信息学分析发现该EST序列含有完整的PtLIR1基因的开放读码框(ORF),根据该序列设计一对引物(PtLIR1-F1:5'- GTCGACATGCAGGCTGCCACTGGTTTG-3',划线部分为Sal I酶切位点; PtLIR1-R1:5'-CCATGGTTACTCCTTGGAGACTCCCGTCTG-3',划线部分为Nco I酶切位点)。采用SDS-苯酚法从盐碱地星星草根中提取总RNA,经DNaseⅠ去除基因组DNA污染以及RNA清洁纯化试剂盒纯化后,将总RNA反转录成cDNA,以此为模板以引物PtLIR1-F和PtLIR1-R1对星星草PtLIR1基因的ORF进行PCR扩增。PtLIR1基因的PCR反应体系为:2.5 μL 10×PCR缓冲液,2 μL dNTP(2.5 mmol/L),PtLIR1-F(10 µmol/L)和PtLIR1-R1(10 µmol/L)各1 μL,2 μL cDNA,0.25 μL rTaq(5 U/μL),加灭菌的去离子水至 25 μL。PCR反应条件为:94℃ 预变性4 min; 94℃ 30 s,60℃ 30 s,72℃ 30 s,共35个循环; 最后72℃ 延伸10 min。经琼脂糖凝胶电泳分析后将扩增片段用胶回收试剂盒回收,将回收产物与到pMD19-T Simple载体连接后转化大肠杆菌感受态细胞JM109。采用Amp抗性和蓝白斑反应筛选阳性重组子,送上海生工生物工程公司测序。插入片段的序列与预期相符,阳性重组质粒命名为pT-PtLIR1。
1.2.3 NaCl胁迫下星星草PtLIR1基因的表达特性分析分别提取NaCl处理后的星星草根和叶的总RNA,经DNA酶消化以及纯化后,用琼脂糖凝胶电泳检测总RNA的完整性,用核酸蛋白检测仪定量。采用荧光定量RT-PCR分析NaCl胁迫下PtLIR1基因的表达变化。取400 ng总RNA用PrimeScriptTM RT reagent Kit(Perfect Real Time)将其反转录成cDNA。反转录体系为:2 μL 5×PrimeSctipt Buffer(for Real Time),0.5 μL PrimeScript RT Enzyme MixⅠ,0.5 μL Oligo dT Primer(50 μmol/L),0.5 μL Random 6 mers(50 μmol/L),加灭菌的去离子水至10 μL。反转录条件为:37℃ 15 min,85℃ 15 s,4℃保存。以cDNA为模板,用SYBR® Premix Ex Taq TMⅡ(Tli RNaseH Plus)进行荧光定量RT-PCR分析。引物Pttubulin-F:5'-CGACTTGGCAAAGGTTCAGC-3'和Pttubulin-R:5'-TCATCGCCCTCATCATCTGC-3'扩增Pttubulin基因,用引物PtLIR1-F1和PtLIR1-R1 扩增PtLIR1基因。PCR反应体系为:10 μL 2×的SYBR® Premix Ex Taq TMⅡ(Tli RNaseH Plus),Forward Primer(10 µmol/L)和Reverse Primer(10 µmol/L)各0.8 μL,2 μL cDNA,加灭菌的去离子水至20 μL。PCR反应条件为:95℃ 5 min; 95℃ 30 s,55℃ 30 s,72℃ 30 s,共44个循环。目的基因的相对含量=2-∆∆CT,其中∆∆CT=∆ CT样本-∆ CT对照,∆ CT样本= CT样本-CT内参,∆ CT对照= CT对照-CT内参。PtLIR1基因表达的器官特异性分析采用半定量RT-PCR方法。
1.2.4 生物信息学分析用DNAman软件进行氨基酸序列分析,采用ClustalX1.8软件进行序列比对,用Mega4.0.2软件生成进化树。
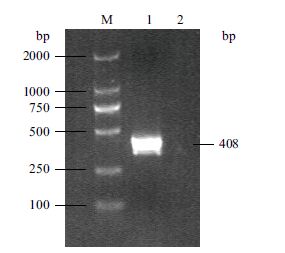
2 结果 2.1 星星草PtLIR1基因的克隆以前期研究中分离到的星星草PtLIR1基因的EST序列为信息探针,从NCBI GenBank EST 数据库中检索到星星草PtLIR1的EST序列EB104311.1,经ORF Finder 软件分析后发现该EST中含有PtLIR1基因完整的ORF。根据ORF序列设计一对引物(PtLIR1-F1和PtLIR1-R1),提取并纯化盐碱地星星草根的总RNA,将其反转录成cDNA后作为模板,扩增PtLIR1基因的ORF(图 1)。将扩增产物克隆后,经测序分析证实该扩增片段与预期的PtLIR1基因的ORF一致(GenBank登录号为JN871224)。
|
| M:DL2000 分子量标准; 1:PtLIR1基因ORF的扩增产物; 2:阴性对照 图 1 图1 星星草PtLIR1基因ORF扩增产物的琼脂糖凝胶电泳检测 |
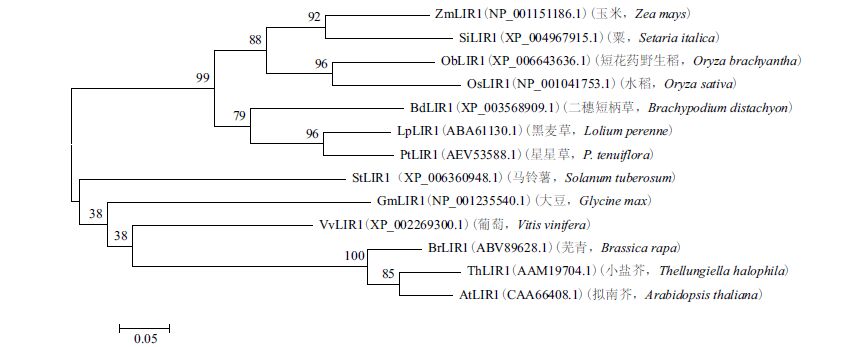
星星草PtLIR1基因的开放读码框长为408 bp,编码135个氨基酸。BlastX分析表明,PtLIR1基因编码的氨基酸序列与类光诱导蛋白1的匹配最佳。经过ProtParam计算,PtLIR1的分子量为14.1 kD、理论上的等电点值为4.45。在NCBI GenBank蛋白质数据库中选取黑麦草、二穗短柄草、玉米、短花药野生稻、粟、马铃薯、葡萄、芜青、拟南芥、小盐芥、水稻、大豆等植物LIR1氨基酸序列,与星星草PtLIR1进行了序列一致性比对,结果(图 2)表明,PtLIR1的氨基酸序列与黑麦草的类光诱导蛋白1的一致性最高,为80%。PtLIR1与水稻、玉米、芜青、小盐芥、拟南芥、短花药野生稻、粟、马铃薯、葡萄、二穗短柄草和大豆等植物的LIR1的一致性分别为57%、72%、42%、42%、54%、57%、71%、61%、67%、64%和47%。在相似性比较结果的基础上构建了LIR1的分子进化树(图 3),在比较的物种中,PtLIR1与黑麦草类光诱导蛋白1的亲缘关系最近。PtLIR1具有光诱导蛋白结构域。与其他光诱导蛋白相似,在PtLIR1的56-70和110-124位氨基酸序列中存在两个近似重复序列,形成α-螺旋结构,在重复序列内半胱氨酸残基之间的距离高度保守(8个氨基酸残基)。PtLIR1的重复序列与其他植物相似性较高,而重复序列外的相似性则较低,表明该结构域具有重要功能。

|
| 图 2 星星草和其他12 种植物LIR1 的氨基酸序列比对 |
|
| 图 3 植物LIR1 氨基酸序列的分子进化树 |
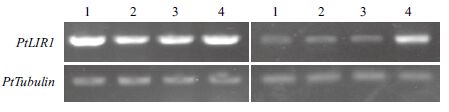
类光诱导蛋白1基因LIR1与植物的抗逆性密切相关,参与植物对盐胁迫[13, 14, 15]、寒冷胁迫[11]和病害[16]的应答反应。为了了解盐胁迫下星星草PtLIR1基因表达的器官特异性,利用半定量RT-PCR技术检测了PtLIR1基因在星星草根与叶中的表达(图 4)。无论在非胁迫条件下还是在盐胁迫条件下,PtLIR1基因在星星草的根与叶中均表达,PtLIR1基因在盐碱地星星草根中的表达量明显高于PtLIR1在其它条件下星星草根中的表达量,表明PtLIR1基因可能对星星草根适应盐碱地生境具有特别重要的意义。
|
| 1:非胁迫; 2:150 mmol/L NaCl 6 h; 3:300 mmol/L NaCl 6 h; 4:盐碱地 图 4 星星草PtLIR1基因表达的器官特异性分析 |
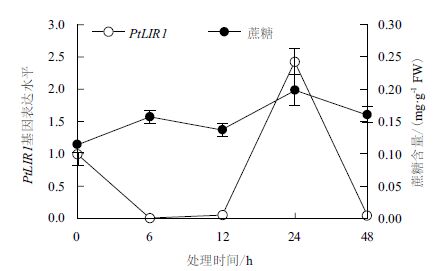
为了了解星星草根中PtLIR1基因对NaCl胁迫的响应,以Pttubulin基因作为内参基因,利用荧光定量RT-PCR技术分析了NaCl胁迫下星星草根中PtLIR1基因相对表达水平的变化(图 5)。NaCl胁迫下PtLIR1基因在星星草根中的表达水平先下降后上升,在胁迫24 h后达到峰值(为0 h的2.4倍),随后又下降。PtLIR1基因在NaCl胁迫24 h时上调表达,表明PtLIR1基因在胁迫晚期(12-48 h)参与星星草根对NaCl胁迫的应答,并可能对帮助星星草根抵御盐碱地逆境中的NaCl胁迫具有重要作用。
|
| 图 5 NaCl胁迫下星星草根中PtLIR1基因的表达与蔗糖含量的相关性 |
蔗糖的积累是植物抵御逆境胁迫的机制之一,而类光诱导蛋白1基因LIR1可能参与对蔗糖水平的调节[11]。LIR1基因可能通过调节蔗糖水平参与植物对盐胁迫的应答。NaCl胁迫下星星草根中的蔗糖水平在胁迫6 h后上升,在24 h后达到峰值(图 5),表明蔗糖的积累是星星草根响应盐胁迫的策略之一。在NaCl胁迫早期(0-12 h),PtLIR1基因的表达与蔗糖水平的变化趋势相反,而在胁迫晚期(12-48 h),PtLIR1基因的表达与蔗糖水平的变化趋势一致。结果表明,在NaCl胁迫晚期,PtLIR1基因的表达与蔗糖水平的变化具有正相关性,可能是参与调节蔗糖水平的主要基因,而在胁迫早期,其他蔗糖代谢调节基因可能在蔗糖水平的调节中发挥主要作用。
2.3.3 NaCl胁迫下可溶性糖与星星草根中PtLIR1基因表达的相关性可溶性糖的积累也是植物适应逆境胁迫的机制之一。在拟南芥(A. thaliana)和黑麦草(L. perenne)中都发现了类光诱导蛋白1基因LIR1的表达会受到可溶性糖的抑制[11, 12]。盐胁迫下星星草根中LIR1的表达可能也受到可溶性糖的调节。NaCl胁迫下,星星草根中可溶性糖的水平在胁迫6 h后达到峰值,但随后呈现下降的变化趋势(图 6),表明可溶性糖的积累是星星草根响应早期盐胁迫的策略之一。在NaCl胁迫早期(0-12 h),PtLIR1基因的表达与可溶性糖水平的变化趋势相反。而在胁迫晚期(12-48 h),PtLIR1基因的表达与可溶性糖水平的变化趋势一致,但可溶性糖的水平低于胁迫早期(0-12 h)(图 6)。结果表明,在NaCl胁迫早期星星草根中可溶性糖的积累与PtLIR1基因的表达水平负相关,这与拟南芥(Arabidopsis thaliana)类光诱导蛋白1基因的表达受到可溶性糖抑制的研究结果相一致[16],星星草根中PtLIR1基因的表达可能受到可溶性糖积累的负调控。但在胁迫晚期(12-48 h),低于早期的可溶性糖水平与PtLIR1基因的表达未呈现出负相关性。
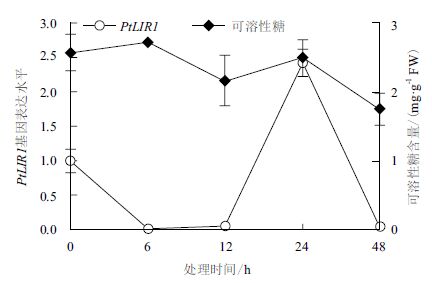
|
| 图 6 NaCl胁迫下星星草根中PtLIR1基因的表达与可溶性糖含量的相关性 |
盐胁迫是影响植物生长发育,导致农作物减产的重要环境因子之一。植物具有一定的调节离子平衡、渗透平衡以及氧化还原平衡的能力,可以在一定程度上适应盐胁迫逆境。盐胁迫下可溶性糖和蔗糖的积累是植物适应逆境胁迫的机制之一[17, 18]。可溶性糖是重要的小分子渗透调节物质,是合成其它有机物质的碳架与能量来源。蔗糖可以作为渗透调节物质调节渗透平衡,可以维持细胞膜的稳定性、保护细胞内的分子结构,是植物生长发育的重要碳源和能源,此外还具有信号功能,可以调节相关基因的表达[18]。可溶性糖和蔗糖代谢水平常被用来衡量逆境胁迫的程度以及植物对逆境胁迫的适应性。
类光诱导蛋白1基因LIR1与植物的抗逆性密切相关,参与植物对盐胁迫、寒冷胁迫和病害胁迫的应答[10, 11, 12, 13]。该基因可能协助光合作用的进行或光合作用产物的加工[14]。LIR1基因在盐碱地星星草根中表达[10],在400 mmol/L NaHCO3胁迫6 h后在星星草叶中上调表达[11],在100 mmol/L NaCl处理后在虎尾草中上调表达[12]。因此,LIR1基因可能在植物对盐胁迫的适应性中发挥作用。本研究发现在NaCl胁迫晚期,星星草根中的PtLIR1基因响应盐胁迫上调表达。由于类光诱导蛋白1基因可能与蔗糖水平的调节有关sup>[15],因此LIR1基因可能通过调节蔗糖水平参与植物对盐胁迫的应答。在NaCl胁迫晚期,星星草根中的蔗糖积累至最高水平,PtLIR1基因的表达水平与蔗糖水平的变化趋势一致,说明在盐胁迫晚期PtLIR1基因可能是调节蔗糖水平的主要基因。盐胁迫下PtLIR1基因可能通过调节蔗糖水平,以使蔗糖行使渗透调节、保护分子结构,提供碳源和能量,以及通过信号传导调节下游基因表达的功能从而响应盐胁迫。植物LIR1基因的表达会受到可溶性糖的抑制[15, 16]。植物在抵御寒冷胁迫的过程中往往伴随着可溶性糖的积累,黑麦草类光诱导蛋白1基因LpLIR1在寒冷胁迫下先上调表达,然后下降到本底水平,表达水平的下降可能与可溶性糖的积累有关[15]。在NaCl胁迫早期,星星草根中的可溶性糖迅速积累响应盐胁迫,星星草根中PtLIR1基因的表达水平与蔗糖和可溶性糖积累的水平均呈现出相反的变化趋势。一方面提示我们,在盐胁迫早期可溶性糖的积累可能对PtLIR1基因的表达产生了负调控; 另一方面也说明,在NaCl胁迫早期蔗糖合成酶(sucrosesynthase,SS)、蔗糖磷酸合成酶(sucrose phosphatesynthase,SPS)和蔗糖转化酶(invertase,Ivr)等其他蔗糖代谢相关酶基因可能在蔗糖水平的调节中发挥了更主要的作用。我们在前期的研究[13]和本研究中都发现了盐碱地星星草根中PtLIR1基因的高水平表达,这提示PtLIR1基因的表达对星星草根适应盐碱地逆境的重要性,以及蔗糖在此过程中可能执行的重要功能。综合文献内容和我们的研究结果,将PtLIR1基因在星星草根逆境胁迫应答中的可能发挥的作用总结如图 7所示。为了深入了解PtLIR1基因在盐胁迫应答中的作用,将通过进一步的研究明确PtLIR1基因是否通过调节蔗糖水平帮助星星草抵御盐胁迫逆境,以及可溶性糖是否对PtLIR1基因具有负调控作用。
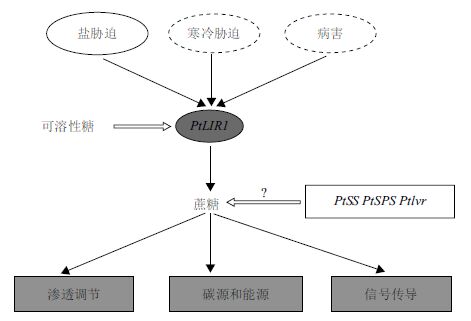
|
| 图 7 PtLIR1基因在星星草根逆境胁迫应答中的作用 |
类光诱导蛋白1基因PtLIR1参与与星星草根对盐胁迫的应答反应。在盐胁迫早期,星星草根中PtLIR1基因的表达水平与可溶性糖水平的变化趋势相反,可溶性糖的积累与PtLIR1基因的表达呈现出负相关性,但蔗糖却迅速积累,其他蔗糖代谢相关基因可能在蔗糖水平的调节中发挥主要作用。在盐胁迫晚期,星星草根中的PtLIR1基因响应盐胁迫上调表达,蔗糖积累水平进一步上升,PtLIR1基因的表达水平与蔗糖水平的变化趋势一致,在盐胁迫晚期PtLIR1基因可能是参与调节蔗糖水平的主要基因。
| [1] | Vij S, Tyagi AK. Emerging trends in the functional genomics of the abiotic stress response in crop plants[J]. Plant Biotechnol J, 2007, 5(3):361-380. |
| [2] | Apse MP, Aharon GS, Snedden WA, et al. Salt tolerance conferred by overexpression of a vacuolar Na+/H+ antiport in Arabidopsis[J]. Science, 1999, 285(5431):1256-1258. |
| [3] | Zhang HX, Hodson JN, Williams JP, et al. Engineering salt-tolerant Brassica plants:Characterization of yield and seed oil quality in transgenic plants with increased vacuolar sodium accumulation[J]. Proc Natl Acad Sci USA, 2001, 98(22):12832-12836. |
| [4] | Zhou S, Chen X, Zhang X, et al. Improved salt tolerance in tobacco plants by co-transformation of a betaine synthesis gene BADH and a vacuolar Na+/H+ antiporter gene SeNHX1[J]. Biotechnol Lett, 2008, 30(2):369-376. |
| [5] | Guo ZJ, Chen XJ, Wu XL, et al. Overexpression of the AP2/EREBP transcription factor OPBP1 enhances disease resistance and salt tolerance in tobacco[J]. Plant Mol Biol, 2004, 55(4):607-618. |
| [6] | Zheng X, Chen B, Lu G, et al. Overexpression of a NAC transcription factor enhances rice drought and salt tolerance[J]. Biochem Bioph Res Commun, 2009, 379(4):985-989. |
| [7] | Kumari S, Sabharwal VP, Kushwaha H, et al. Transcriptome map for seedling stage specific salinity stress response indicates a specific set of genes as candidate for saline tolerance in Oryza sativa L.[J]. Funct Integr Genomics, 2009, 9(1):109-123. |
| [8] | 石德成, 殷立娟. 盐(NaCl)与碱(Na2CO3)对星星草胁迫作用的差异[J]. 植物学报, 1993, 35(2):144-149. |
| [9] | 阎秀峰, 孙国荣. 星星草生理生态学的研究[M]. 北京:科学出版社, 2000. |
| [10] | 朱冬梅. 盐碱胁迫下星星草根部转录应答的初步分析[D]. 哈尔滨:哈尔滨师范大学, 2010:67. |
| [11] | Wang Y, Yang C, Liu G, et al. Microarray and suppression subtractive hybridization analyses of gene expression in Puccinellia tenuiflora after exposure to NaHCO3[J]. Plant Sci, 2007, 173(3):309-320. |
| [12] | 梁涵, 张欣欣, 管清杰, 等. 盐胁迫下虎尾草上调基因的筛选和鉴定[J]. 分子植物育种, 2008, 6(5):886-892. |
| [13] | 喻修道, 屈志鹏, 郭军, 等. 小麦与条锈菌亲和互作的差减文库构建及初步分析[J]. 中国农业科学, 2008, 41:1267-1273. |
| [14] | Abied MA, Holland D. Two newly isolated genes from citrus exhibit a different pattern of diurnal expression and light response[J]. Plant Mol Biol, 1994, 26(1):165-173. |
| [15] | Ciannamea S, Jensen CS, Agerskov H, et al. A new member of the LIR gene family from perennial ryegrass is cold-responsive, and promotes vegetative growth in Arabidopsis[J]. Plant Science, 2007, 172:221-227. |
| [16] | Bläsing OE, Gibon Y, Günther M, et al. Sugars and circadian regulation make major contributions to the global regulation of diurnal gene expression in Arabidopsis[J]. Plant Cell, 2005, 17:3257-3281. |
| [17] | Sheen J, Zhou L, Jang JC. Sugar as signaling molecules[J]. Curr Opin Plant Biol, 1999, 2(5):410-418. |
| [18] | Gibson SI. Plant sugar-response pathways. Part of a complex regulatory web[J]. Plant Physiol, 2000, 124(4):1532-1539. |




