2.中国科学院大学,北京 100049;
3. 江苏省农业科学院农业生物技术研究所 省农业生物学重点实验室,南京 210014;
4. 上海市能源作物育种及应用重点实验室, 上海 200444
2. University of Chinese Academy of Sciences,Beijing 100049;
3. Provincial Key Laboratory of Agrobiology,Institute of Agro-biotechnology,Jiangsu Academy of Agricultural Sciences,Nanjing 210014;
4. Shanghai Key Laboratory of Bio-Energy Crops,Shanghai 200444
钾(K)、钠(Na)是植物生长过程中重要的营养元素,对维持细胞的渗透压、调节叶片和气孔运动以及细胞伸长等具有重要作用[1],K+/Na+比是衡量植物是否具有耐盐特性的重要指标之一[2],植物细胞质中的Na+是重要的毒性决定因子,在盐渍条件下,过量Na+对植物是有害的[3],大量的Na+会限制K+的吸收[4],从而影响K+/ Na+比,因此保持细胞内相对较高的K+含量对植物的生长至关重要。HKT类蛋白是一种高亲和钾离子转运载体,广泛存在于植物、真菌、真细菌和古细菌中,对于维持生物体内K+/ Na+比具有重要作用[5]。
Schachtman等[6]从小麦中克隆得到第一个HKT基因,以后陆续在水稻、拟南芥、冰叶日中花、海篷子等多种植物中克隆了其同源蛋白基因。大量研究表明,HKT类蛋白根据其功能可以分为两类,一种是以小麦TaHKT1、水稻OsHKT2、冰叶日中花McHKT1等为代表,为K+/Na+共转运体,其对两种离子具有高选择性,当两种离子都存在且Na+浓度较低时,同时转运这两种离子,是一种Na+偶联的K+吸收载体,当K+浓度较低而Na+浓度较高时,介导低亲和的Na+转运而高亲和K+吸收受到阻遏[7],Su等[8]利用蛋白免疫定位技术发现McHKT1在叶片的液泡处有较强的表达信号,这一发现表明该蛋白在盐生植物中起调节细胞质内离子平衡的作用; 另一种则是以拟南芥为代表的AtHKT1蛋白,其主要在根中表达[9],越来越多的证据表明HKT类蛋白不是作为一种高亲和K+吸收载体在起作用,可能作为一种重要的Na+内流系统在起作用,且对Na+吸收起调控作用[10]。然而,目前对于HKT类蛋白在盐生植物耐盐特性上的机制仍然研究较少,本研究以盐生植物盐角草为材料,利用RACE技术克隆K+离子相关的转运载体SeHKT1基因全长cDNA 序列,结合生物信息学方法分析其可能的跨膜特性和亲和特性,且利用qRT-PCR技术对SeHKT1在不同胁迫条件下的表达情况进行初步分析,为在分子水平上揭示盐生植物Na+、K+选择性吸收、耐盐机理及通过基因工程手段培育耐盐作物新品种,改良盐碱地提供更多依据。
1 材料与方法 1.1 材料盐角草(Salicornia europaea L.)种子由中国科学院阜康荒漠生态站提供。
1.2 方法 1.2.1 材料的处理和培养盐角草种子播种于灭菌的蛭石培养基中,在温室中培养,昼夜温度为25℃/20℃,Hoagland全营养液浇灌,光照16 h,光强度200 μmol/(m2·s),相对湿度是60%-70%,盐角草长至3 cm左右时进行疏苗,每盆保留20株左右,生长2个月后分别用0、10、200、500和800 mmol/L NaCl进行胁迫处理,适时补充溶液,200 mmol/L NaCl处理的植株在6、24、48、72和120 h进行地上部和根部分别取样,其余4个处理均在72 h后取样,液氮速冻,-80℃保存备用。
1.2.2 SeHKT1基因部分编码序列的获得TIANGEN RNA试剂盒提取盐角草0和200 mmol/L NaCl处理的地上部及根部总RNA,将两种RNA混合作为模板反转为cDNA,根据盐角草转录组Unigene测序结果(由华大基因测转录组数据)用软件Primer 5.0设计引物SeHKT1:正向引物:5'-AATGGAATGGAGT-AATGAAGGA-3'; 反向引物:5'-TATGTGGGAAAT-GAATGGGCTA-3'。PCR扩增程序:95℃变性4 min; 95℃ 30 s,56℃ 45 s,72℃ 1 min,36个循环; 72℃延伸10 min。将PCR扩增产物纯化、连接pMD18-T vector(TaKaRa),经大肠杆菌转化及阳性菌落筛选后,提取阳性质粒送华大基因公司测序,测序结果与转录组序列进行比对分析。
1.2.3 5'-RACE及3'-RACE利用SMARTer RACE cDNA Amplification kit试剂盒(Clontech),以盐角草0和200 mmol/L NaCl处理的地上部及根部混合RNA为模板合成5'-Ready cDNA和3'-Ready cDNA。根据所获得的SeHKT1基因部分cDNA序列,设计RACE特异性引物。5'端SeHKT1(5):5'-ACGACCAACATAGCTGGTGCAATGGAGG-3'及 5'端的巢式引物SeHKT1(Nest 5):5'-CCAACATAGCTGGTGCAATGGAGGAAAG-3'; 3'端SeHKT1(3):5'-TGGTATGGATTTGCAGGGAGGTGGAGTG-3'及3'端的巢式引物SeHKT1(Nest 3):5'-GGATTTGCAGGGAGGTGGAGTGACCAAG-3'。第一轮反应条件是:95℃ 4 min; 95℃ 30 s,68℃ 45 s,72℃ 2 min,5个循环; 95℃ 30 s,66℃ 45 s,72℃ 2 min,5个循环; 95℃ 30 s,64℃ 45 s,72℃ 2 min,35个循环; 72℃ 10 min。通过第二轮巢式PCR获得末端扩增产物,分离纯化此PCR产物后连接pMD19-T载体并测序。
1.2.4 盐角草SeHKT1基因cDNA全长的获得 测序结果用DNAMAN和NCBI网站上的Blast比对工具进行分析和拼接得到SeHKT1全长序列1 761 bp。从全长序列两端设计引物:SeHKT1-S:5'-AAAATCCT-CTTGTACTCCATTAAGC-3',SeHKT1-A:5'-GTTTT-AGAGGAGTTTCCAAGCTTTG-3'。按照程序95℃ 4 min; 95℃ 30 s,54℃ 45 s,72℃ 2 min,35个循环; 72℃ 10 min进行cDNA全长扩增,分离纯化PCR产物后连接pMD19-T载体并测序,进一步确认测序拼接结果。
1.2.5 生物信息学分析通过MEGA5.1对HKT类蛋白序列进行系统进化树聚类分析,利用ProtParam tool(http://www.expasy.ch/tools/protparam.html)分析SeHKT1蛋白分子量和理论等电点,利用TMHMM Serverv 2.0及TMpred(http://ch.embnet.org/software/TMPRED_form.html)分析该蛋白序列跨膜区和跨膜方向,利用Protscale(http://web.expasy.org/protscale/)分析氨基酸序列疏水性,利用InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)分析蛋白质结构域。
1.2.6 SeHKT1基因的实时荧光定量表达分析利用RNA植物提取试剂盒(QIANGEN)分别提取200 mmol/L NaCl处理6、24、48、72、120 h及0、10、500、800 mmol/L NaCl处理地上部和根部总RNA,0 mmol/L NaCl处理作为对照,以盐角草Tubulin基因作为内参,引物序列为:SeTubulin-S:5'-AGGTCAA-CAAAGACAGCA-3'; SeTubulin-A:5'-AGCGAAAAT-GAGAGAGTG-3'。根据上述获得的SeHKT1基因全长序列设计定量引物:SeHKT1r-F:5'-CTATCTTGCCA-TCTTCACCATTATT-3',SeHKT1r-R:5'-GTCACTCC-ACCTCCCTGC-3'。按照SYBR Premix Ex Taq II试剂盒(TaKaRa)操作:95℃ 30 s; 95℃ 5 s,60℃ 30 s,40个循环反应,添加熔解曲线程序(从65℃-95℃,每5 s增加0.5℃并读取荧光值一次)。反应混合液在Bio-Rad荧光定量96孔PCR仪中进行反应,所有的样品都设置3个重复。反应结束后获得SeHKT1和内参基因SeTubulin在各个样品的Ct值(threshold cycle),按照2-△△ Ct方法[11]对实验数据进行处理,计算基因在各个样品中的相对表达量。
2 结果 2.1 SeHKT1基因部分序列的获得根据前期本实验室盐角草转录组测序结果获得的片段设计正反向引物,利用高保真酶扩增获得800 bp左右的PCR产物(图 1-A),克隆并测序后提交NCBI网站进行Blast分析,发现SeHKT1基因与毕氏海篷子SbHKT1基因、盐地碱蓬SsHKT1基因氨基酸序列相似性较高,分别为95%、76%、62%和52%,故推测该片段为盐角草HKT1基因的部分序列,故命名为SeHKT1。
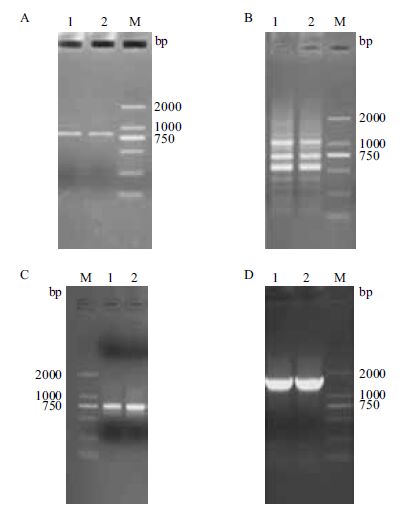
|
| A:SeHKT1基因部分序列PCR; B:5' RACE PCR; C:3' RACE PCR; D:全长序列扩增PCR。1,2:基因扩增片段; M:DL2000 DNA Marker 图 1 SeHKT1基因的PCR扩增 |
5' RACE PCR扩增出3条带(图 1-B),测序后通过BLAST及DNAMAN软件分析表明第一条1.2 kb左右的条带是SeHKT1 5'端序列,3' RACE巢式PCR扩增出750 bp左右的预期产物(图 1-C)。
2.3 SeHKT1基因完整开放阅读框(ORF)的获得利用软件DNAMAN将SeHKT1 cDNA的5'端和3'端测序结果,与已知的SeHKT1基因部分编码序列拼接,利用NCBI中的Open Reading Frame Finder 工具分析得到1 653 bp完整的ORF(GenBank登录号为KP739261),编码550个氨基酸。从ORF两端设计引物对cDNA扩增并测序,进一步确认SeHKT1的全长序列(图 1-D)。
2.4 SeHKT1基因生物信息学分析 2.4.1 SeHKT1基因蛋白同源性分析NCBI中Blast分析显示该蛋白与毕氏海篷子SbHKT1、盐地碱蓬SsHKT1、冰叶日中花McHKT2、赤桉EcHKT1、EcHKT2氨基酸序列同源性分别为95%、76%、62%和52%。应用MEGA5.1软件对HKT类蛋白质序列进行系统进化树聚类分析也表明,SeHKT1与毕氏海篷子SbHKT1、盐地碱蓬SsHKT1、冰叶日中花McHKT2、赤桉EcHKT1、EcHKT2、大麦HvHKT1; 5、水稻OsHKT7属于一组,同源性较高; 水稻OsHKT1、OsHKT3、OsHKT9与小麦TaHKT1、芦苇PhaHKT1一组,OsHKT4、OsHKT6与HsHKT4为一组(图 2)。
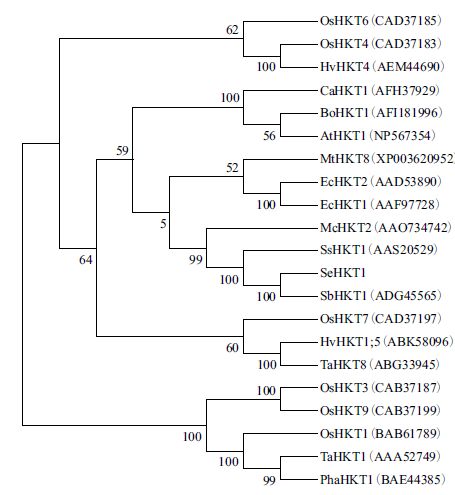
|
| Os:水稻; Ca:辣根; Bo:甘蓝; At:拟南芥; Mt:苜蓿; Ec:赤桉; Mc:冰叶日中花; Ss:盐地碱蓬; Sb:毕氏海篷子; Hv:大麦; Ta:小麦; Pha:芦苇 图 2 SeHKT1蛋白与其他物种HKT类蛋白系统进化树分析 |
利用ProtParam tool分析SeHKT1蛋白分子量为62.41 kD,理论等电点为9.35,带正电荷的氨基酸残基(天冬氨酸和谷氨酸)总数为36个,带负电荷的氨基酸残基(精氨酸和赖氨酸)总数为54个,该蛋白的稳定指数为34.89,说明该蛋白较稳定。经TMHMN(图 3-A)及TMpred(图 3-B)共同分析推测SeHKT1具有10个跨膜区(12-34,44-66,117-139,209-231,244-266,281-303,337-359,440-462,477-499),跨膜区长度均为23个氨基酸残基。InterProScan软件分析SeHKT1具有一个信号肽序列(1-35)和一个TrkH家族特征序列(199-539),具有跨膜运输活性和阳离子转运载体结合位点,因此推测SeHKT1为跨膜运输蛋白。通过ProtScale工具分析(图 4)显示,该蛋白在N端、C端及跨膜区均存在明显的疏水区(11-67,8-143,200-236,241-265,271-306,334-357,390-414,446-467,477-493,521-540),ProtParam tool分析该蛋白氨基酸的平均疏水性为0.376,表明该蛋白具有强烈的疏水性,符合载体蛋白的特点。
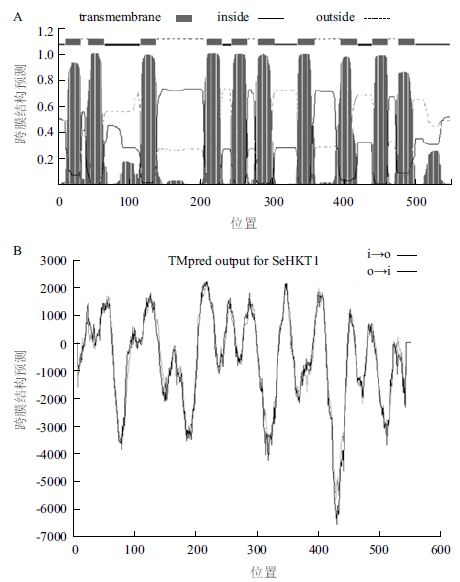
|
| 图 3 SeHKT1 跨膜分析 |
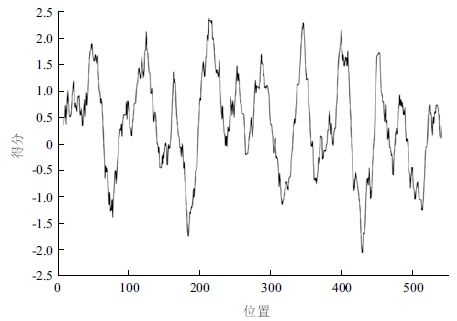
|
| 图 4 SeHKT1 疏水性分析 |
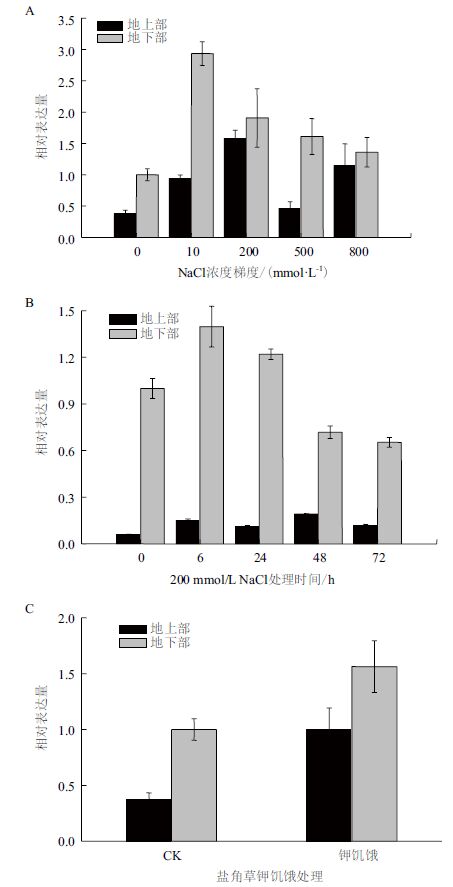
由图 5-A显示,SeHKT1基因主要在根部表达,在地上部表达相对较低,无盐条件下在地上部表达量极低。NaCl处理条件下该基因在地上部和地下部的表达均明显增加,说明该基因受盐诱导表达; 随着NaCl处理浓度的增加,SeHKT1在根部表达量在10 mmol/L浓度下最高,随NaCl浓度升高,表达量逐渐降低。而在地上部,SeHKT1在200 mmol/L NaCl处理下表达量最高,呈现先上升后下降的趋势(图 5-A)。地下部直接接触外界盐胁迫,因此SeHKT1的响应更为敏感和快速,而在地上部响应则相对迟钝。在200 mmol/L NaCl处理下(图 5-B),SeHKT1主要在地下部表达,处理6 h该基因在地下部表达量达到最高峰,24 h后开始下降; 而地上部SeHKT1表达量缓慢提高,直到48 h达到最高值。此外,该基因在地上部的表达量在200 mmol/L NaCl处理时最高(图 5-A)。在钾饥饿的条件下该基因在根部和地上部的表达均明显升高,在地下部的表达量仍高于地上部,组织的差异表达结果与上文一致(图 5-C)。
|
| 图 5 SeHKT1 在不同处理下不同组织中的相对表达量分析 |
盐角草是已知最耐盐的高等植物之一,不仅能够忍受1 000 mmol/L的NaCl[12],而且需要一定的盐分来维持其最适生长[13]。盐角草需要具备一定的细胞及分子水平的调控策略以适应长期生长的盐渍环境。因此,研究盐角草的耐盐机制及挖掘耐盐基因具有潜在的应用价值。
目前已经有研究证明在许多植物物种中HKT(High-affinity K+ Transporter)基因家族成员在调控细胞免受Na+毒害机制上发挥重要作用[14]。盐角草具备有效的离子转运能力并将其区隔化到液泡中[1],以避免Na+对细胞器的损害。SeHKT基因家族能够调控Na+和K+转运及细胞内K+/ Na+稳态,可推测该基因家族对盐角草的耐盐性具有重要贡献。
本研究利用盐角草转录组Unigene序列及RACE扩增技术成功获得了一条HKT全长基因,通过系统进化树聚类分析暂命名为SeHKT1。利用生物信息学手段预测,SeHKT1蛋白具有10个跨膜域,一个信号肽序列(1-35)和一个TrkH家族特征序列(199-539),具有跨膜运输活性和阳离子转运载体结合位点,在N端、C端及跨膜区均存在明显的疏水区,符合跨膜运输载体的特点。通过生物信息学分析,可预测基因的功能,编码蛋白的特性,为后续基因功能的验证实验指明了方向。
本研究通过荧光定量PCR技术,对盐角草SeHKT1基因在不同组织,不同处理时间,不同浓度NaCl及K+缺失条件下的表达特性进行了分析。结果显示,SeHKT1主要在根中表达,高盐和钾饥饿诱导条件下,表达量均升高,尤其是在根部能对NaCl胁迫和K+缺失快速响应,通过提高基因表达量来增强K+/Na+运输载体功能。200 mmol/L NaCl浓度是盐角草生长的最适浓度[15],该条件下根部组织被处理6 h时SeHKT1基因被快速诱导表达,但在24 h后表达量开始逐渐下降,在48 h后该基因相对表达量低于对照,表明SeHKT1基因能够短时间内响应外界环境Na+离子的变化而上调表达,吸收一定量的Na+离子进入盐角草细胞内,以维持一定的渗透压平衡。大量的研究也表明,在长期进化过程中盐生植物演化出一套完整的适应高盐环境机制[16],其内外的渗透压主要靠大量Na+来调节[17],因此一定浓度的NaCl会促进盐生植物的生长,在高盐环境下,其细胞中K+含量及胞质中K+/Na+比几乎不受影响[10],推测盐生植物细胞内离子平衡调节与HKT类蛋白密切相关。低盐条件下(10 mmo/L)SeHKT1 在根部的相对表达量较高,这可能与吸收更多Na+维持体内渗透势有关; 在高盐条件下(500和800 mmol/L),该基因表达量有所下降,可能与Na+限制K+吸收机制相关,因此,HKT类蛋白对于盐角草细胞内离子平衡调节具有重要作用[18]。
综上所述,盐角草SeHKT1基因全长的获得及系统的表达分析有助于我们进一步研究该基因在盐生植物中调节离子平衡,维持细胞渗透压,揭示真盐生植物的耐盐机制,利用基因工程手段将其运用于培育新的耐盐品种具有一定的意义[19]。
4 结论
真盐生植物盐角草SeHKT1基因氨基酸序列中具有一个信号肽序列(1-35 aa),一个TrKH家族特征序列; 该基因编码的蛋白具有跨膜载体蛋白活性,共10个跨膜域,N端、C端及中部多个跨膜区呈现明显的疏水性,符合载体类蛋白特点,因此推测SeHKT1蛋白为跨膜运输蛋白。qRT-PCR分析表明SeHKT1基因主要在根部表达,其表达能够响应外界NaCl和钾离子浓度变化,编码蛋白可能参与Na+和K+的吸收和运输。
| [1] | Britto DT, Ebrahimi-Ardebili S, Hamam AM, et al. 42K analysis of sodium-induced potassium efflux in barley:mechanism and relevance to salt tolerance[J]. New Phytologist, 2010, 186(2):373-384. |
| [2] | 张科, 张道远, 王雷, 等. 自然生境下盐角草的离子吸收一运输特征[J]. 干旱区研究, 2007, 24(4):80-486. |
| [3] | Ding L, Zhu JK. Reduced Na+ uptake in the NaCl-hypersensitive sos1 mutant of Arabidopsis thaliana[J]. Plant Physiology, 1997, 113(3):795-799. |
| [4] | Aghaleh M, Niknam V, Ebrahimzadeh H, et al. Effect of salt stress on physiological and antioxidative responses in two species of Salicornia(S. persica and S. europaea)[J]. Acta Physiologiae Plantarum, 2010, 33(4):1261-1270. |
| [5] | 高媛媛, 张保龙, 杨郁文, 等. 海蓬子中高亲和钾离子转运体 SbHKT1 基因的克隆, 表达及生物信息学分析[J]. 基因组学与应用生物学, 2010, 29(4):646-652. |
| [6] | Schachtman DP, Schroeder JI. Structure and transport mechanism of a high-affinity potassium uptake transporter from higher plants[J]. Nature, 1994, 370:655-658. |
| [7] | 邵群, 丁同楼, 韩宁, 等. 高亲和 K+ 转运载体(HKT)与植物抗盐性[J]. 植物生理学通讯, 2006, 42(2):175-181. |
| [8] | Su H, Balderas E, Vera-Estrella R, et al. Expression of the cation transporter McHKT1 in a halophyte[J]. Plant Molecular Biology, 2003, 52(5):967-980. |
| [9] | Rus A, Yokoi S, Sharkhuu A, et al. AtHKT1 is a salt tolerance deter-minant that controls Na+ entry into plant roots[J]. Proceedings of the National Academy of Sciences, 2001, 98(24):14150-14155. |
| [10] | 邵群. 盐地碱蓬阳离子转运载体 SsHKT1 的基因克隆及其功能分析[D]. 济南:山东师范大学, 2007. |
| [11] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 2001, 25(4):402-408. |
| [12] | Lv S, Jiang P, Chen X, et al. Multiple compartmentalization of sodium conferred salt tolerance in Salicornia europaea[J]. Plant Physiology and Biochemistry, 2011, 51:47-52. |
| [13] | 韩和平. 盐角草(Salicornia europaea L.)SePSY和SeLCY基因克隆及功能分析[D]. 北京:中国科学院植物研究所, 2006. |
| [14] | Horie T, Hauser F, Schroeder JI. HKT transporter-mediated salinity resistance mechanisms in Arabidopsis and monocot crop plants[J]. Trends in Plant Science, 2009, 14(12):660-668. |
| [15] | 郑青松, 华春, 董鲜, 等. 盐角草幼苗对盐离子胁迫生理响应的特性研究(简报)[J]. 草业学报, 2008, 17(6):164-168. |
| [16] | 蒋永超, 刘斌, 郭欢, 等. 稀盐盐生植物盐角草对盐胁迫生理响应的初步研究[J]. 新疆农业科学, 2012, 49(4):694-700. |
| [17] | Blumwald E. Sodium transport and salt tolerance in plants[J]. Current Opinion in Cell Biology, 2000, 12(4):431-434. |
| [18] | Flowers TJ, Colmer TD. Salinity tolerance in halophytes[J]. New Phytologist, 2008, 179(4):945-963. |
| [19] | Munns R, Tester M. Mechanisms of salinity tolerance[J]. Plant Biology, 2008, 59:651-681. |
| [20] | 顾峰雪, 潘晓玲. 中国西北干旱荒漠区盐生植物资源与开发利用[J]. 干旱区研究, 2002, 19(4):17-20. |



