2. 巴彦淖尔市农牧业科学研究院,巴彦淖尔 015000
2. Bayannaoer Academy of Agricultural and Animal Science,Bayannaoer 015000
AP2/ERF(APETALA2/ethylene response factor)是植物中特有的一类转录因子大家族,因含有由60-70个氨基酸组成的AP2/ERF结构域而得名,包含AP2、RAV、ERF、DREB和其他类别共5个亚家族[1]。1994年第一个AP2基因从拟南芥(Arabidopsis thaliana)中分离[2],1995年从烟草中分离出乙烯响应元件结合蛋白(Ethylene-inducible DNA binding proteins)ERF1、2、3、4,含有保守的ERF结构域[3]。研究发现AP2/ERF家族中的成员参与植物生长、发育以及多种胁迫下的多种生物学过程,包括花发育[4]、果实发育[5]、种子发育[6]、病菌防御[7]、高盐和干旱[8]等环境胁迫响应。同时,发现AP2/ERF转录因子家族成员参与乙烯、脱落酸、茉莉酸、水杨酸等多种信号转导途径,并且一些家族成员是逆境信号交叉途径中的连接因子[9, 10]。目前,拟南芥[11]、水稻[12]、甘蓝型油菜[13]、番茄[14]、桃[15]等多种植物ERF家族基因的cDNA被克隆。Mizuno等[16]利用酵母单杂交技术从甜瓜中分离出与CM1-ACS2启动子上游GCCGAC序列相互作用的3个转录因子——CMe-DREBl、CMe-ERF1、CMe-ERF2。高峰等[17]从甜瓜中克隆获得CMeERF1、CMeERF2的cDNA。Ma等[18]对甜瓜AP2/ERF转录因子家族基因进行了鉴定,得到136个成员,包括AP2亚家族13个、ERF亚家族119个、RAV亚家族4个;对ERF亚家族的119个成员进行聚类分析,将其分为11个亚群。本研究选取其第三亚群的第一个成员CmERFIII-1,其在甜瓜基因组数据库(https://melonomics.net)中的登录号为MELO3C005465,克隆其cDNA,分析不同组织中的表达情况,旨在为进一步研究该基因的功能提供参考。
1 材料与方法 1.1 材料植物材料为甜瓜品种“河套蜜瓜”原种,由本实验室保存,种植于大田,采摘授粉后30 d的甜瓜果实,取中果皮组织于液氮中速冻,-80℃保存,用于基因克隆。采摘授粉后0、10、20、30和40 d的甜瓜果实,取中果皮组织速冻于液氮中,-80℃保存,用于不同发育阶段果实中基因的表达分析。分别取培养于1/2 MS培养基35日龄河套蜜瓜无菌苗的根、茎和叶片组织,速冻于液氮中,-80℃保存,用于不同组织器官中基因的表达分析。
大肠杆菌E.coli DH5α由本实验室保存。pEASY®-Blunt克隆载体购自北京全式金公司。总RNA提取试剂盒RNAiso Plus、反转录试剂盒PrimeScriptTM II 1st Strand cDNA Synthesis Kit、PrimeScriptTM Master Mix Perfect Real Time 及实时荧光定量PCR试剂盒SYBR® Premix Ex Taq II、高保真DNA聚合酶PrimeSTAR® GXL DNA Polymeras、dNTPs、氨苄青霉素、DNA Marker等购自宝生物工程(大连)有限公司;PCR产物纯化试剂盒购自上海生工生物工程有限公司;其余试剂为进口或国产分析纯。
1.2 方法 1.2.1 总 RNA 提取使用RNAiso Plus总RNA提取试剂盒,按照说明书提取甜瓜根、茎、叶和不同发育阶段甜瓜果实的总RNA。
1.2.2 引物设计与合成根据已报道的CmERFIII-1基因cDNA序列,使用Primer Premier 5.0软件设计特异性引物。上游引物P1序列:5'-CATAACCAATCAACCAATTCC-3',下游引物P2序列:5'-TTATATGACTGTGCATTGAGGAT-3',引物由上海生工生物工程有限公司合成。
1.2.3 cDNA克隆取采摘授粉后30 d甜瓜果实的总RNA 5 μg,按照反转录试剂盒PrimeScriptTM II 1st Strand cDNA Synthesis Kit说明书进行反转录反应,得到cDNA第一链。
以合成的cDNA第一链为模板,用高保真DNA聚合酶PrimeSTAR® GXL DNA Polymeras 进行PCR扩增。PCR反应条件为:98℃预变性10 s,49℃退火15 s,68℃延伸1 min,扩增30个循环;68℃延伸10 min。PCR产物经1.2%的琼脂糖凝胶电泳检测。扩增片段用San-prep 型柱式PCR产物纯化试剂盒纯化。纯化产物与pEASY®-Blunt载体连接。连接物转化到大肠杆菌DH5α中,在Amp抗性培养基上筛选出阳性克隆并经PCR鉴定,随机选取3个独立的重组克隆由上海生工生物工程有限公司测序。利用DNAStar软件比对测序序列与甜瓜基因组数据库中目的基因cDNA序列(登录号:MELO3C005465)。
1.2.4 编码蛋白质分析利用NCBI的ORF Finder软件(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)对CmERFIII-1序列进行开放阅读框分析。利用ExPaSy ProtParam(http://web.expasy.org/ protparam/)、ExPASy ProtScale(http:// web.expasy.org/cgi-bin/protscale/protscale.pl)、PredictProtein(http://www.predictprotein.org/)、SWISS-MODEL(http://swwiss-model.expasy.org/)对编码蛋白质进行理化性质、亲疏水性、蛋白质二级结构、蛋白质结构域的空间结构分析。利用NCBI的protein blast程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行同源比对分析,查找与预测的CmERFIII-1蛋白序列一致性较高的蛋白质序列,利用MEGA5.1软件中的Neighbor-Joining算法进行聚类分析。
1.2.5 基因表达分析利用PrimeScriptTM Master Mix Perfect Real Time反转录试剂盒,分别以河套蜜瓜根、茎、叶和授粉后0、10、20、30和40 d的甜瓜果实总RNA为模板进行反转录反应,将合成的cDNA第一链用SYBR® Premix Ex Taq II实时荧光定量PCR试剂盒进行。根据测序结果设计CmERFIII-1基因的实时荧光定量PCR引物,上下游引物分别为5'-TACTCTGTAGGATCGGCGGTT-3'和5'-GCGGTA-TCATAAGTTCCAAGCC-3'。以甜瓜GAPDH(登录号:MELO3C002342T1)基因为内参基因,上下游引物分别为5'-CGTGTTCCTACCGTTGATGTCTCT-3'和5'-TCAGTGTACCCCAAAATTCCCTTC-3'。以反转录产物为模板建立25 µL PCR扩增体系:加入SYBR® Premix Ex Taq II(Tli RNaseH Plus)(2×)12.5 µL,上游、下游引物各1 µL,模板2 µL,ddH2O 8.5 µL,混匀。PCR反应条件:95℃预变性30 s;95℃5 s,60℃30 s,40个循环。数据采用2-△△CT[19]方法进行分析,将叶片中CmERFIII-1基因表达量设定为1。标准误(SE)来自3个独立生物学样本,每个生物学样品进行3次技术重复。
2 结果 2.1 cDNA的克隆及序列分析 2.1.1 RT-PCR产物分析RT-PCR扩增后得到约750 bp的特异性条带(图 1),与预期相符。
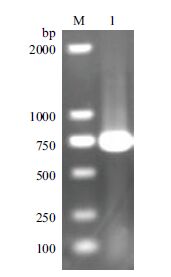
|
| 图 1 甜瓜CmERFIII-1 基因cDNA 扩增 |
将克隆得到的cDNA序列与甜瓜基因组数据库中MELO3C005465序列比对,两条序列一致性达100%。克隆到的CmERFIII-1 cDNA序列为711个核苷酸,ORF Finder软件分析结果(图 2)表明,该基因编码由236个氨基酸组成的蛋白质。编码蛋白质的分子量为25.99 kD,理论等电点为8.39,对该蛋白质的亲疏水性分析表明其为亲水性蛋白质。

|
| 实线框内的氨基酸是预测的CmERFIII-1 转录因子的蛋白结合区;虚线框内的氨基酸是预测的CmERFIII-1 转 录因子的DNA 结合区;下划线氨基酸序列为该转录因子AP2/ERF 结构域区域 图 2 河套蜜瓜CmERFIII-1 基因cDNA ORF 及推测的氨基酸序列 |
对CmERFIII-1基因cDNA所编码蛋白质的二级结构分析,发现α螺旋占12.29%,无规则卷曲占80.51%,链接部分占 7.21%。推测该蛋白质序列中第52、77、79、92、107、163、165-167、178和182位氨基酸是该转录因子的蛋白质结合区(protein-binding domain);第84-86、85-89、90-95和97位氨基酸是该转录因子的DNA结合区(DNA-binding domain)(图 2)。对蛋白质GO功能注释分析发现该蛋白质具有与DNA结合的分子功能可信度为65%,具有转录因子活性的可信度为49%,具有与蛋白质结合的分子功能可信度为21%。亚细胞定位分析表明该蛋白质定位于细胞核内。
2.1.4 CmERFIII-1中AP2/ERF结构域的空间结构预测对CmERFIII-1的AP2/ERF结构域(图 2中下划线氨基酸序列)分析发现,该结构域与拟南芥中AtERF1[17]的AP2/ERF结构域一致性为80.33%。以拟南芥中AtERF1的AP2/ERF结构域(图 3-B)作为模板,预测CmERFIII-1的AP2/ERF结构域的空间结构(图 3-A)。
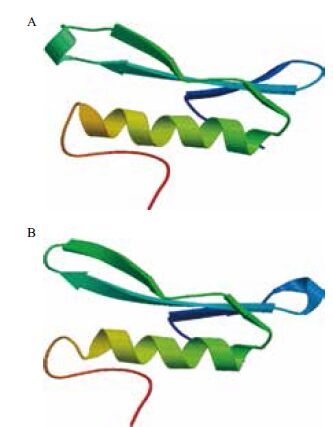
|
| A :CmERFIII-1 中AP2/ERF 结构域的预测空间结构;B :AtERF1 中AP2/ ERF 结构域的空间结构 图 3 CmERFIII-1 中AP2/ERF 结构域的预测空间结构 |
通过对CmERFIII-1蛋白质与不同物种中同源蛋白质的比对分析发现,该蛋白氨基酸序列中AP2/ERF结构域与多种植物的ERF亚家族成员相似。筛选序列一致性在40%以上的12条序列,利用MEGA5.1分析并构建聚类关系图(图 4)。12条序列分别来自:甜瓜(Cucumis melo)、黄瓜(Cucumis sativus)、大豆(Glycine max)、樱桃番茄(Solanum lycopersicum)、苹果(Malus domestica)、油菜(Brassica napus)、拟南芥(Arabidopsis thaliana)、葡萄(Vitis vinifera)、甘薯(Coffea canephora)、猕猴桃(Actinidia deliciosa)和柑橘(Citrus clementina)。克隆到的河套蜜瓜CmERFIII-1与GenBank中登录号为XP_008436974.1的氨基酸序列一致性为100%,与黄瓜的2条氨基酸序列(GenBank登录号为XP_004143677.1和XP_004158119.1)序列一致性分别为92%和88%。
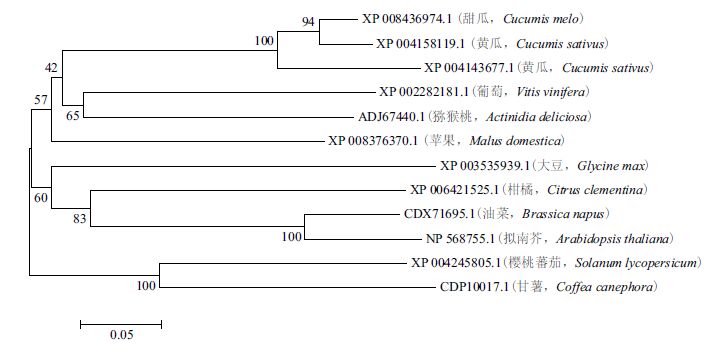
|
| 图 4 CmERFIII-1 系统进化聚类 |
利用实时荧光定量PCR对CmERFIII-1基因在甜瓜不同组织和果实发育不同时期的表达情况进行检测。结果(图 5)显示,CmERFIII-1基因在根、茎、叶以及果实不同发育时期均有不同程度的表达,在叶片中的表达量最高。
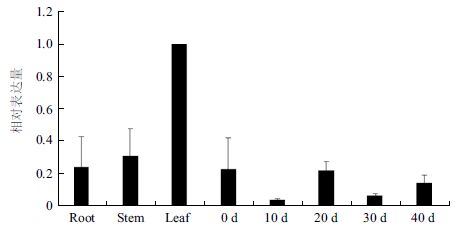
|
| 图 5 CmERFIII-1 基因表达分析 |
ERF是一类植物特有的乙烯应答因子家族,位于乙烯信号转导途径的下游,含有高度保守的DNA结合结构域,与乙烯诱导基因启动子的GCC-box结合来调节一系列乙烯应答基因的转录[3]。拟南芥中AtERF1的AP2/ERF结构域可以与下游基因启动子中GCC-box相互作用,从而实现对下游基因的表达调控[20]。本研究对河套蜜瓜CmERFIII-1编码蛋白的结构推测发现,该转录因子的DNA结合区位于其AP2/ERF结构域内;进一步对CmERFIII-1的AP2/ERF结构域分析发现,该结构域与拟南芥中AtERF1的AP2/ERF结构域一致性较高,为80.33%;由此推测该转录因子可能与AtERF1具有相似的功能,即与乙烯诱导基因启动子的GCC-box发生相互作用,调控下游基因的表达。目前对ERF亚家族成员研究发现,不同的ERF成员在不同植物中表达部位不同。在苹果(Malus domestica)中ERF亚家族成员MdERF1主要在成熟果实中表达,在其它组织中表达量很低[21]。而从李(Prunus salicina)中分离得到的PsERF2a和PsERF2b在果实发育过程中则没有呈现递增或递减的表达规律,而是在花中高水平表达[15]。猕猴桃(Actinidia deliciosa)AdERF4、AdERF6随果实的发育成熟表达量逐渐下降,而AdERF5在根、茎、叶、花和果实中的表达量均很低,但在花和成熟果实中的表达量高于其他组织[22]。甜瓜中CMeERF1和CMeERF2两个基因的表达与内源乙烯生成量显著相关[17]。本研究通过对河套蜜瓜CmERFIII-1基因在根、茎、叶和不同发育时期果实进行实时荧光定量PCR检测,发现该基因在根、茎、叶及果实的不同发育时期均有不同程度表达,在叶片中表达量最高,推测该蛋白在叶片中发挥重要作用。AP2/ERF类转录因子在植物整个生命周期中参与多种生理生化和生长发育调控过程,因此对该类转录因子基因的分离及其表达和功能的研究有重要意义。
4 结论从河套蜜瓜果实中克隆得到CmERFIII-1基因711 bp cDNA,编码一个由236个氨基酸组成的蛋白质,该cDNA序列与甜瓜基因组数据库中ERF基因(登录号MELO3C005465)的核苷酸序列一致性达100%。由该cDNA所编码的蛋白质氨基酸序列与黄瓜ERF亚家族2个成员的氨基酸序列(GenBank登录号:XP_004143677.1和XP_004158119.1)一致性较高。实时荧光定量PCR检测显示,该基因在河套蜜瓜叶片中表达量最高。
| [1] | Sakuma Y, Liu Q, Dubouzet JG, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration-and cold-inducible gene expression[J]. Biochemical and Biophysical Research Communications, 2002, 290(3):998-1009. |
| [2] | Jofuku KD, Den Boer BG, Van Montagu M, et al. Control of Arabidopsis flower and seed development by the homeotic gene APETALA2[J]. Plant Cell, 1994, 6(9):1211-1225. |
| [3] | Ohme-Takagi M, Shinshi H. Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element[J]. Plant Cell, 1995, 7(2):173-182. |
| [4] | Kunst L, Klenz JE, Martinez-Zapater J, et al. Ap2 gene determines the identity of perianth organs in flowers of Arabidopsis thaliana[J]. Plant Cell, 1989, 1(12):1195-1208. |
| [5] | Bartley GE, Ishida BK. Digital fruit ripening:data mining in the TIGR tomato gene index[J]. Plant Molecular Biology Reporter, 2002, 20(2):115-130. |
| [6] | Niu X, Helentjaris T, Bate NJ. Maize ABI4 binds coupling element1 in abscisic acid and sugar response genes[J]. Plant Cell, 2002, 14(10):2565-2575. |
| [7] | Park JM, Park CJ, Lee SB, et al. Overexpression of the tobacco Tsi1 gene encoding an EREBP/AP2-type transcription factor enhances resistance against pathogen attack and osmotic stress in tobacco[J]. Plant Cell, 2001, 13(5):1035-1046. |
| [8] | Kasuga M, Liu Q, Miura S, et al. Improving plant drought, salt, and freezing tolerance by gene transfer of a single stress-inducible transcription factor[J]. Nat Biotech, 1999, 17(3):287-291. |
| [9] | Zhang G, Chen M, Chen X, et al. Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean(Glycine max L.)[J]. Journal of Experimental Botany, 2008, 59(15):4095-4107. |
| [10] | Zhang G, Chen M, Li LC, et al. Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco[J]. Journal of Experimental Botany, 2009, 60(13):3781-3796. |
| [11] | Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors:Genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290(5499):2105-2110. |
| [12] | 祝传书, 武莹, 李威, 等. 水稻转录因JAMYB和ERF3的克隆及遗传转化[J]. 西北植物学报, 2012, 32(10):1935-1941. |
| [13] | 庄静, 周熙荣, 孙超才, 等. 甘蓝型油菜中一类AP2/ERF转录因子的克隆和生物信息学分析[J]. 中国生物工程杂志, 2008, 28(5):29-40. |
| [14] | Zhang HX, Zhu BZ, Yu BY. Cloning and DNA-binding properties of ethylene response factor, LeERF1 and LeERF2, in tomato[J]. Biotechnology Letters, 2005, 27(6):423-428. |
| [15] | El-Sharkawy I, Sherif S, Mila I, et al. Molecular characterization of seven genes encoding ethylene-responsive transcriptional factors during plum fruit development and ripening[J]. Journal of Experimental Botany, 2009, 60(3):907-922. |
| [16] | Mizuno S, Hirasawa Y, Sonoda M, et al. Isolation and characteriza-tion of three DREB/ERF-type transcription factors from melon(Cucumis melo L.)[J]. Plant Science, 2006, 170(6):1156-1163. |
| [17] | 高峰, 怡荣, 郝金凤, 等. 甜瓜乙烯应答因子基因在果实发育成熟过程中的表达特性[J]. 西北植物学报, 2012, 32(5):886-889. |
| [18] | Ma Y, Zhang FY, BaDe RG, et al. Genome-Wide identification and phylogenetic analysis of the ERF gene family in melon[J]. Journal of Plant Growth Regulation, 2014, DOI 10. 1007/s0034 4-014-9443-z(on line). |
| [19] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△CT method[J]. Methods, 2001, 25(4):402-408. |
| [20] | Hao D, Ohme-Takagi M, Sarai A. Unique mode of GCC box recognition by the DNA-binding domain of ethylene-responsive element-binding factor(ERF domain)in plants[J]. Journal of Biological Chemistry, 1998, 273(41):26857-26861. |
| [21] | Wang A, Tan D, Takahashi A, et al. MdERFs, two ethylene-response factors involved in apple fruit ripening[J]. Journal of Experimental Botany, 2007, 58(13):3743-3748. |
| [22] | Yin XR, Allan AC, Chen KS, et al. Kiwifruit EIL and ERF genes involved in regulating fruit ripening[J]. Plant Physiology, 2010, 153(3):1280-1292. |




