YABBY基因家族属于锌指蛋白超家族(Zinc finger super-family)的亚家族,在调控植物叶和花器官发育过程中起着重要的作用,是一类植物特有的转录因子家族,与植物形态建成有关[1, 2, 3, 4]。该基因家族有两个保守的结构域,位于N端的C2C2锌指结构域和位于C端的YABBY结构域,又称与HMG box相似的“螺旋-环-螺旋”结构域[5, 6, 7]。
目前,在很多植物中都有YABBY基因家族的相关报道,在模式生物拟南芥中研究的较为清楚。拟南芥YABBY基因家族有6个成员:FIL(FILAMEN-TOUS FLOWER)、CRC(CRABSCLAW)、INO(INN-ER NO OUTER)(YABBY4)、YAB2(YABBY2)、YAB3(YABBY3)和YAB5(YABBY5)[8, 9, 10, 11, 12],拟南芥YABBY基因家族的6个成员在其侧生器官发育过程中发挥着不同的作用。FIL参与花器官和叶的发育[10, 13];CRO和INO基因有一定的组织特异性,CRC参与蜜腺和心皮远轴端的发育,INO基因则在外部珠被的发育中起着重要的作用[11, 14, 15, 16];YAB2、YAB3和YAB5基因主要在拟南芥的营养组织中特异表达[3, 17]。
棉花作为重要的经济作物,其纤维是纺织工业重要的天然纤维来源。随着基因组学和测序技术的发展,二倍体D组雷蒙德氏棉(Gossypium raimondii L.)、A组亚洲棉石系亚1号(G. arboreum)以及陆地棉遗传标准系TM-1(G. hirsutum L. acc. TM-1)相继完成了测序工作[18, 19, 20, 21, 22],从而为全基因组范围内分析基因家族奠定了一定的数据基础。当前关于棉花YABBY基因家族的研究,鲜有报道。本研究利用公布的陆地棉TM-1的基因组数据,对YABBY基因家族进行全基因组扫描,并进一步分析其亚细胞定位、染色体分布、基序、进化关系和组织表达情况等,旨在为进一步深入探索YABBY基因家族的功能提供理论依据。
1 材料与方法 1.1 材料两份陆地棉标准系TM-1(G. hirsutum L.)蛋白和CDS数据:中国农业科学院棉花研究所(http://cgp.genomics.org.cn/page/species/index.jsp)和Pytozome 网站(http://www.phytozome.net/);拟南芥YABBY 基因家族的氨基酸蛋白序列和陆地棉EST数据来自NCBI(http://www.ncbi.nlm.nih.gov/protein/)数据库;衣藻(Chlamydomonas reinhardtii)YABBY基因家族的氨基酸序列来自PlantTFDB(http://planttfdb.cbi.pku.edu.cn/)数据库。
1.2 方法 1.2.1 棉花YABBY基因家族的鉴定与进化树分析将下载的拟南芥6条YABBY基因家族成员在SMART网站进行结构域预测,然后在Pfam(http://pfam.janelia.org/)网站下载鉴定到的YABBY结构域的种子文件(PF04690.9),最终利用HMMER3.1b1程序在TM-1蛋白数据库中搜索棉花YABBY基因家族成员。
利用MEGA 5.1生物信息软件对鉴定的TM-1基因组中YABBY基因家族氨基酸序列和拟南芥中6条YABBY基因家族的氨基酸序列进行多重序列比对,采用邻接(Neighbor-Joining,NJ)算法构建进化树,进行1 000次Boot strap抽样。
1.2.2 棉花YABBY基因家族的染色体定位和亚细胞定位结合Perl语言编程,在TM-1基因组数据中的gff3文件中提取YABBY基因家族成员的染色体起始和结束位置,然后利用MapInspect软件绘制YABBY基因家族的染色体物理分布图;利用在线软件CELLO:Subcellular Localization Predictive System预测棉花YABBY基因家族成员的亚细胞定位。
1.2.3 棉花YABBY基因家族的基序分析利用MEME软件分析棉花YABBY基因家族成员的motif类型和排列顺序。参数设置如下:基序的最大发现数目是10个,其它参数为默认值。
1.2.4 棉花的YABBY基因家族的组织表达分析在NCBI数据库下载330 520条陆地棉的EST序列。利用Blastn程序,搜索棉花YABBY基因家族成员CDS序列与陆地棉EST序列的匹配,分析棉花YABBY基因家族成员的组织表达类型,参数设定标准为E≤10-10,其他参数为默认值。
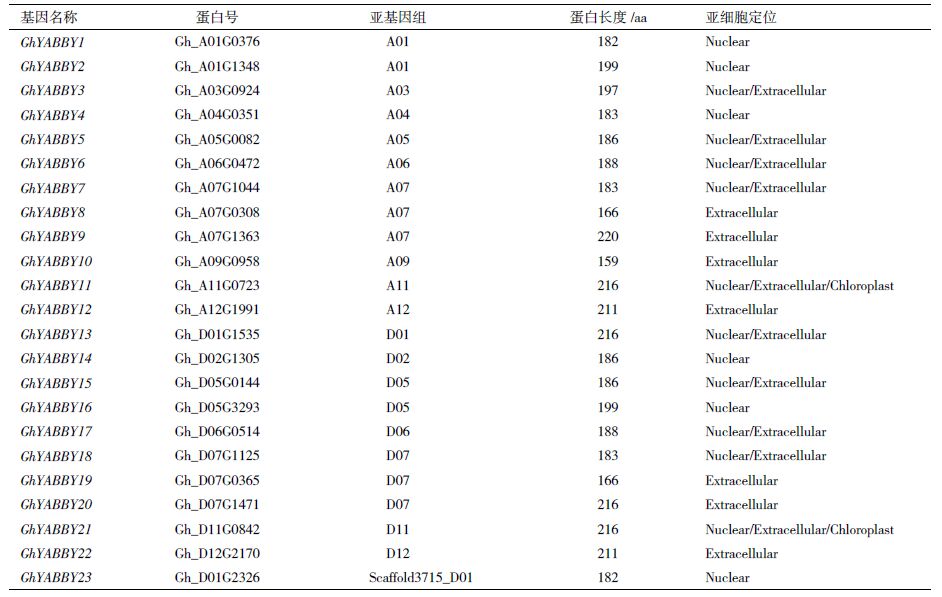
2 结果 2.1 棉花YABBY基因家族的鉴定通过结构域预测,发现拟南芥的6条YABBY蛋白的氨基酸序列都含有YABBY结构域。因此,利用Pfam网站中下载的YABBY结构域的种子文件在陆地棉标准系TM-1蛋白数据库中搜索,又在SMART网站进行进一步确认和人工校对,最终在TM-1基因组中鉴定到23条YABBY蛋白。根据一般植物蛋白的命名方法,对鉴定到的23条棉花YABBY蛋白进行了命名,并统计了它们对应的蛋白ID号、所在的亚基因组以及对应的蛋白长度,从159个氨基酸到220个氨基酸不等(表 1)。
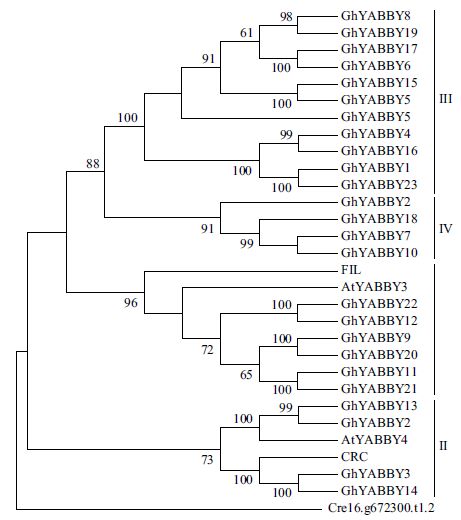
我们以衣藻(Chlamydomonas reinhardtii)作为外类群,利用陆地棉标准系TM-1基因组中鉴定的23条YABBY蛋白和拟南芥的6条YABBY蛋白的氨基酸序列构建了系统进化树(图 1)。与拟南芥类似,棉花的YABBY基因家族也分为4个亚组(Ⅰ-Ⅳ),且每个亚组都有与拟南芥同源的基因:GhYABBY15、GhYABBY5、GhYABBY17、GhYABBY6、GhYABBY8、GhYABBY19、GhYABBY4、GhYABBY16、GhYABBY1和GhYABBY23属于Ⅲ亚组,与拟南芥AtYABBY5属于一个亚组;GhYABBY10、GhYABBY18和GhYABBY7属于亚组Ⅳ,与拟南芥AtYABBY2属于一个亚组;GhYABBY22、GhYABBY12、GhYABBY9、GhYABBY20、GhYABBY11和GhYABBY21属于亚组Ⅰ,与拟南芥FIL和AtYABBBY3属于一个亚组;GhYABBY3、GhYABBY14、GhYABBY13和GhYABBY2属于亚组Ⅱ,与拟南芥CRC和AtYABBY4属于一个亚组。
|
| 图 1 陆地棉TM-1 和拟南芥YABBY 基因家族的进化树 |
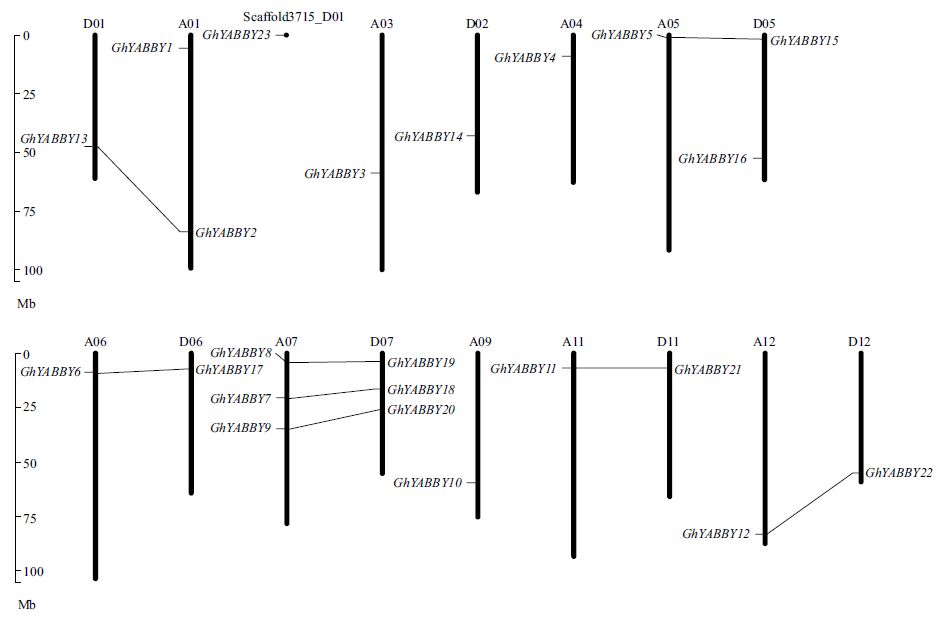
陆地棉标准系TM-1基因组中23个YABBY基因家族成员分布在16条染色体和1条Scaffold上,分别为A01、D01、D02、A03、A04、A05、D05、A06、D06、A07、D07、A09、A11、D11、A12、D12和Scaffold3715_D01(图 2);除了A07和D07染色体上分别有3个成员外,其他染色体上只有1-2个成员(图 2);在TM-1的A亚组和D亚组具有成对的平行进化同源YABBY基因,分布在D01、A01、Scaffold3715_D01、A05、D05、A07、D07、A11、D11、A12和D12染色体上;共有9对共线性基因,分别为GhYABBY13和GhYABBY2、GhYABBY1和GhYABBY23、GhYABBY5和GhYABBY15、GhYABBY6和GhYABBY17、GhYABBY7和GhYABBY18、GhYABBY8和GhYABBY19、GhYABBY9和GhYABBY20、GhYABBY11和GhYABBY21、GhYABBY12和 GhYABBY22。
|
| 灰线表示共线性同源基因 图 2 陆地棉TM-1 基因组上YABBY 基因家族的染色体分布 |
TM-1基因组中23个YABBY基因家族成员的亚细胞定位预测结果(表 1)表明,13个YABBY基因家族成员具有1个亚细胞定位,定位到细胞核或胞外;8个成员具有2个亚细胞定位,定位到细胞核和胞外;2个成员具有3个亚细胞定位,定位到细胞核、胞外和线粒体上。
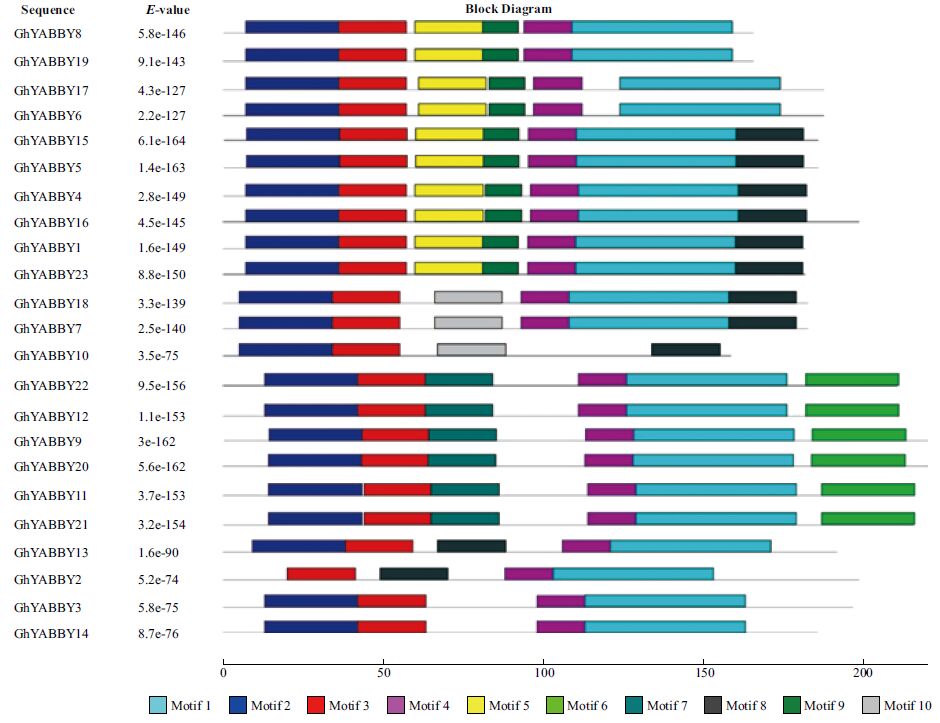
2.4 棉花YABBY基因家族的基序(motif)分析棉花的YABBY基因组家族分为4个亚组(Ⅰ-Ⅳ),23个YABBY基因家族成员的基序分析结果显示,每个亚组的成员具有相同或类似的基序类型和排列顺序,而且9对同线性基因具有相同的基序类型和排列顺序(图 3)。GhYABBY15、GhYABBY5、GhYABBY17、GhYABBY6、GhYABBY8、GhYABBY19、GhYABBY4、GhYABBY16、GhYABBY1和GhYABBY23具有相似的motifs(7-8个),motif排列顺序也基本相同;GhYABBY18、GhYABBY10和GhYABBY7具有类似的motifs(4-6个),排序顺序也类似;GhYABBY22、GhYABBY12、GhYABBY9、GhYABBY20、GhYABBY11和GhYABBY21具有完全相同的6个motifs,而且排列顺序完全相同;GhYABBY3、GhYABBY14、GhYABBY13和GhYABBY2具有相似的4-5个motifs,排序顺序类似。
|
| 图 3 陆地棉TM-1 基因组上YABBY 基因家族的基序类型 |
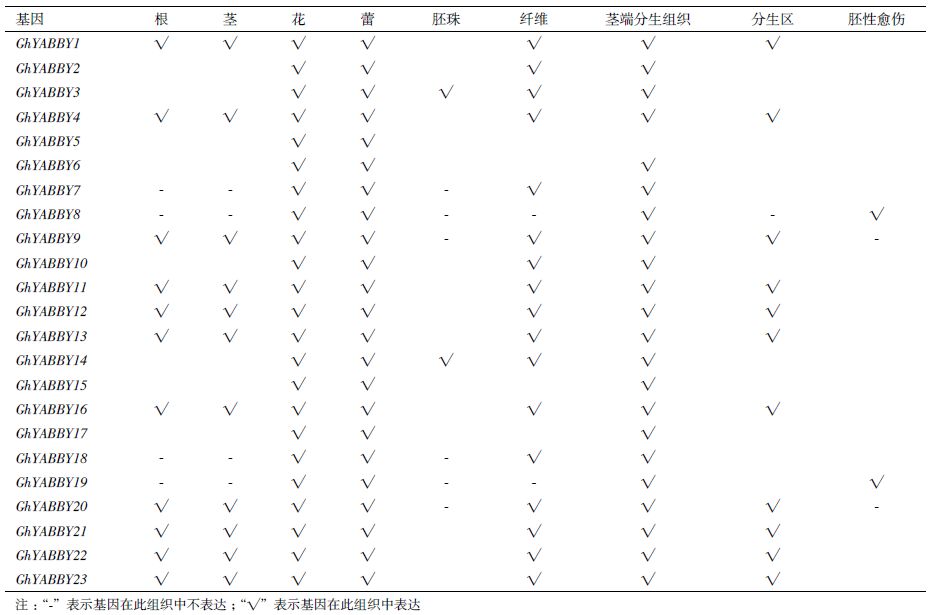
TM-1全基因组中的23个YABBY基因家族成员在棉花的根、茎、叶、花、蕾、幼胚珠、胚珠、纤维、茎端分生组织、分生区和胚性愈伤组织中广泛表达(表 2)。进一步分析发现,所有的23个YABBY基因家族成员在2个及两个以上的组织中表达;23个YABBY基因家族成员全部在花、蕾和茎端分生组织中表达,大部分成员在根、茎和分生区域表达,还有部分基因在胚珠和胚性愈伤组织表达。
本研究通过生物信息学和基因组学手段在陆地棉标准系TM-1全基因组中共鉴定了23条YABBY基因家族成员,其中有9对为共线性基因(A亚组和D亚组),只有5个基因不是共线性基因,这与以往的报道一致,二倍体A组亚洲棉和D组雷蒙德氏棉基因组之间存在很强的共线性[22];9对共线性基因中,除了GhYABBY9和GhYABBY20、GhYABBY13和GhYABBY2以外,其余7对共线性基因的氨基酸长度完全相同;除了GhYABBY13和GhYABBY2这一对共线性基因外,其余的每对共线性基因具有完全相同的亚细胞定位;9对共线性基因在进化上非常保守,在进化树上分布在相同的分支上;motif分析表明,除GhYABBY13和GhYABBY2外,其余的每对共线性基因具有完全相同的motif类型和排列顺序。
拟南芥YABBY基因家族有6个成员FIL、CRC、AtYABBY4、AtYABBY2、AtYABBY3和AtYABBY5,其中每个成员的功能都已研究得较为清楚[8, 9, 10, 11, 12]。进化树分析表明,本研究在陆地棉标准系TM-1基因组中鉴定的23个YABBY基因家族成员与拟南芥一样,分为4个亚组,且每一个亚组都有与拟南芥同源的基因,由此推测,棉花和拟南芥YABBY基因家族成员在功能上可能具有一定的相似性。因此,研究棉花YABBY基因家族成员的功能,在一定程度上可以参考拟南芥的研究结果。
组织表达分析(表 2)表明,TM-1全基因组中的23个YABBY基因家族成员具有较为广泛的组织表达类型。其中,23个YABBY基因家族成员全部在花、蕾和茎端分生组织中表达,这在一定程度上暗示棉花的YABBY基因家族成员可能参与了花、蕾和茎端分生组织的发育;亚组Ⅰ的6个成员和亚组Ⅲ的部分成员都在根、茎和分生区域表达,这在一定程度上暗示这些基因可能参与了根、茎和分生区域的发育。
4 结论本研究从陆地棉标准系TM-1(Gossypium hirsu-tum L. acc. TM-1)基因组中鉴定到23个YABBY基因家族成员,具有不同的亚细胞定位和染色体定位,且具有较为广泛的组织表达类型。这些成员分为4个亚组,每个亚组成员间具有相似的基序类型和排列顺序。
| [1] | Bowman JL. The YABBY gene family and abaxial cell fate[J]. Curr Opin Plant Biol, 2000, 3(1):17-22. |
| [2] | Reinhardt D, Frenzm M, Mandel T, et al. Microsurgical and laser ablation analysis of leaf positioning and dorsoventral patterning in tomato[J]. Development, 2005, 132(1):15-26. |
| [3] | Eckardtn A. YABBY genes and the development and origin of seed plant leaves[J]. Plant Cell, 2010, 22(7):2103. |
| [4] | Yamada T, Yokota S, Hirayama Y, et al. Ancestral expression patterns and evolutionary diversification of YABBY genes in angiosperms[J]. Plant J, 2011, 67(1):26-36. |
| [5] | Kanaya E, Nakajima N, Okada K. Non-sequence-specific DNA binding by the FILAMENTOUS FLOWER protein from Arabidopsis thaliana is reduced by EDTA[J]. J Biol Chem, 2002, 277(14):11957-11964. |
| [6] | Sieber P, Petrascheck M, Barberis A, et al. Organ polarity in Arabidopsis NOZZLE physically interacts with members of the YABBY family[J]. Plant Physiol, 2004, 135(4):2172-2185. |
| [7] | Golz JF, Roccaro M, Kuzoff R, et al. GRAMINIFOLIZ promotes growth and polarity of Antirrhinum leaves[J]. Development, 2004, 131(15):3661-3670. |
| [8] | Siegfried KR, Eshed Y, Baum SF, et al. Members of the YABBY gene family specify abaxial cell fate in Arabidopsis[J]. Development, 1999, 126(18):4117-4128. |
| [9] | Villanueva JM, Broadhvest J, Hayser BA, et al. INNER NO OUTER regulates abaxial-adaxial patterning in Arabidopsis ovules[J]. Genes Dev, 1999, 13(23):3160-3169. |
| [10] | Sawa S, Watanabe K, Goto K, et al. FILAMENTOUS FLOWER, a meristem and organ identity gene of Arabidopsis, encodes a protein with a zinc finger and HMG-related domains[J]. Genes Dev, 1999, 13(9):1079-1088. |
| [11] | Bowman JL, Smyth DR. CRABS CLAW, a gene that regulates carpel and nectary development in Arabidopsis, encodes a novel protein with zinc finger and helix-loop-helix domains[J]. Development, 1999, 126(11):2387-2396. |
| [12] | Lee JY, Baum SF, Oh SH, et al. Recruitment of CRABS CLAW to promote nectary development within the eudicot clade[J]. Development, 2005, 132:5021-5032. |
| [13] | Stahle MI, Kuehlich J, Staron L, et al. YABBYs and the transcriptional corepressors LEUNIG and LEUNIG_HOMOLOG maintain leaf polarity and meristem activity in Arabidopsis[J]. Plant Cell, 2009, 21(10):3105-3118. |
| [14] | Zhang XL, Yang ZP, Zhang J, et al. Ectopic expression of BraYAB1-702, a member of YABBY gene family in Chinese cabbage, causes leaf curling, inhibition of development of shoot apical meristem and flowering stage delaying in Arabidopsis thaliana[J]. J Mol Sci, 2013, 14(7):14872-14891. |
| [15] | Shamimuzzaman M, Vodkin L. Genome-wide identification of bin-ding sites for NAC and YABBY transcription factors and coregulated genes during soybean seedling development by ChIPSeq and RNA-Seq[J]. BMC Genomics, 2013, 14(1):477. |
| [16] | Ha CM, Jun JH, Fletcher JC. Control of Arabidopsis leaf morphogen-esis through regulation of the YABBY and KNOX families of transcription factors[J]. Genetics, 2010, 186(1):197-206. |
| [17] | Sarojam R, Sappl PG, Goldshmidt A, et al. Differentiating Arabidopsis shoots from leaves by combined YABBY activities[J]. Plant Cell, 2010, 22(7):2113-2130. |
| [18] | Wang KB, Wang ZW, Li FG, et al. The draft genome of a diploid cotton Gossypium raimondii[J]. Nat Genet, 2012, 44:1098-1103. |
| [19] | Parerson AH, Wendel JF, Gundlach H, et al. Repeated polyploidiz-ation of Gossypium genomes and the evolution of spinnable cotton fibres[J]. Nature, 2012, 492:423-427. |
| [20] | Li FG, Fan GY, Wang KB, et al. Genome sequence of the cultivated cotton Gossypium arboreum[J]. Nature Genetics, 2014, 46(6):562-574. |
| [21] | Li FG, Fan GY, Lu CR, et al. Genome sequence of cultivated Upland cotton(Gossypium hirsutum TM-1)provides insights into genome evolution[J]. Nature Biotechnology, 2015, doi:10. 1038/nbt. 3208. |
| [22] | Zhang TZ, Hu Y, Jiang WK, et al. Sequencing of allotetraploid cotton(Gossypium hirsutum L. acc. TM-1)provides a resource for fiber improvement[J]. Nature Biotechnology, 2015, doi:10.1038/nbt.3207 |