水稻黄单胞水稻致病变种(Xanthomonas oryzae pv. oryzae,Xoo)引起的白叶枯病是世界水稻生产上重要的细菌性病害之一[1]。由于经济重要性以及遗传可操作性,水稻-Xoo互作已成为植物-病原物互作研究的模式系统之一[2]。随着KACC10331(韩国菌株)、MAFF311018(日本菌株)和PXO99A(菲律宾菌株)全基因组测序的完成[3, 4, 5],为深入研究Xoo致病性的信号调控机理奠定了基础。
环二鸟苷酸(c-di-GMP)是一种存在广泛的细菌小分子第二信使,调控了毒性、生物膜形成和运动性等诸多生物学功能[6, 7, 8]。含GGDEF结构域的鸟苷酸环化酶(DGC)和含EAL或HD-GYP结构域的磷酸二酯酶(PDE)分别控制了c-di-GMP的合成和降解[9]。GGDEF、EAL和HD-GYP结构域蛋白编码基因在细菌基因组中广泛存在,形成一个复杂的信号调控网络[10]。
本研究比较分析上述菌株中GGDEF、EAL和HD-GYP结构域蛋白的种类、数量及其序列特征,鉴别基序保守的DGC或PDE、基序退化的c-di-GMP信号受体。此外,发现感知外界环境信号、调控输出结构域活性的信号输入结构域,旨在为从Xoo中鉴定DGC和PDE编码基因并研究其功能奠定基础。
1 材料与方法 1.1 材料供试菌株为PXO99A、MAFF311018和 KACC-10331,其核酸和氨基酸序列来源于NCBI(http://www.ncbi.nlm.nih.gov/nuccore/CP000967.1,http://www.ncbi.nlm.nih.gov/nuccore/NC_007705.1,http://www.ncbi.nlm.nih.gov/nuccore/AE013598.1)。
1.2 方法 1.2.1 蛋白保守结构域的预测利用SMART(http://smart.embl-heidelberg.de/)和MEME(http://meme.nbcr.net/meme/tools/meme)程序(参数设置motif宽度为最小3,最大50)预测蛋白保守结构域。
1.2.2 结构域蛋白基因的分布运用CGView Server作图,分析结构域蛋白基因在基因组的分布(参数设置为blastx,expect = 0.00001,Bacterial and Plant Plastid,filter = yes,alignment_cutoff = 85,identity_cutoff = 85)。
1.2.3 系统进化分析利用MEGA5构建系统进化树[11],比较所有GGDEF、EAL和HD-GYP结构域蛋白的相似性;采用基于JTT矩阵基础模型最大相似法[12],构建进化模型(bootstrap replication=1 000)[13]。
1.2.4 基因功能验证基因功能验证包括DGC/PDE酶活性、c-di-GMP结合作用以及细菌毒性相关表型等测定[14, 15, 16, 17]。
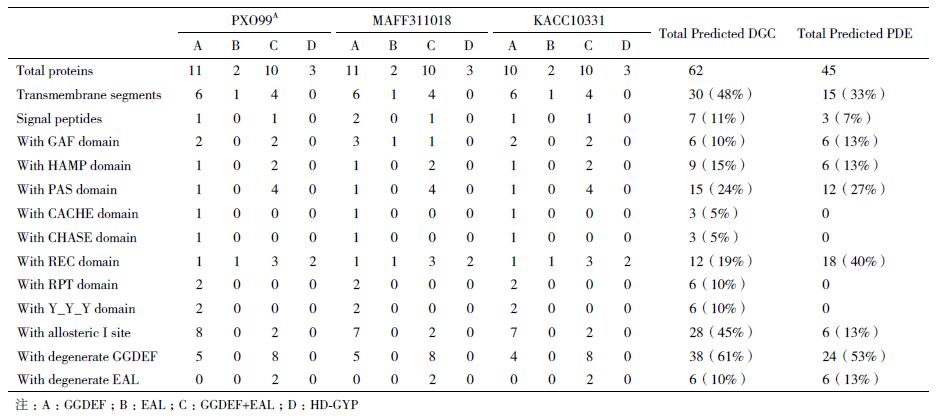
2 结果 2.1 GGDEF、EAL和HD-GYP结构域蛋白基因的鉴定PXO99A、MAFF311018和 KACC10331分别有26、26 和25个基因编码了GGDEF、EAL和HD-GYP结构域(表 1)。PXO99A和MAFF311018基因数量和类型相同,而KACC10331则比前两种菌株少一个GGDEF结构域基因(表 1和表 2)。
鉴定了几个菌株特异的、只编码GGDEF结构域基因(表 2)。PXO99A独有PXO_02615基因,但缺少与XOO2206(MAFF311018)和XOO2330(KACC10331)同源的基因;KACC10331缺少与PXO_04147(PXO99A)和XOO3786(MAFF311018)同源的基因。
2.2 GGDEF、EAL和HD-GYP结构域蛋白的同源性分析GGDEF、EAL和HD-GYP结构域蛋白基因分散于Xoo整个基因组中,但也存在一定的集聚(图 1)。同源蛋白具有高达90%以上的序列相似性,同组聚集在一个内部节点上,如单独GGDEF结构域聚集在蓝区,单独EAL结构域在绿区,GGDEF和EAL结构域杂合蛋白在黄区,HD-GYP结构域在红区。所有这些GGDEF和EAL蛋白编码基因分散在Xoo基因组各部分,但在1 800-2 900 kb的位置相对集中(图 2)。结果表明,3个菌株尤其是MAFF311018菌株和KACC10331菌株在进化上的亲缘关系很近。
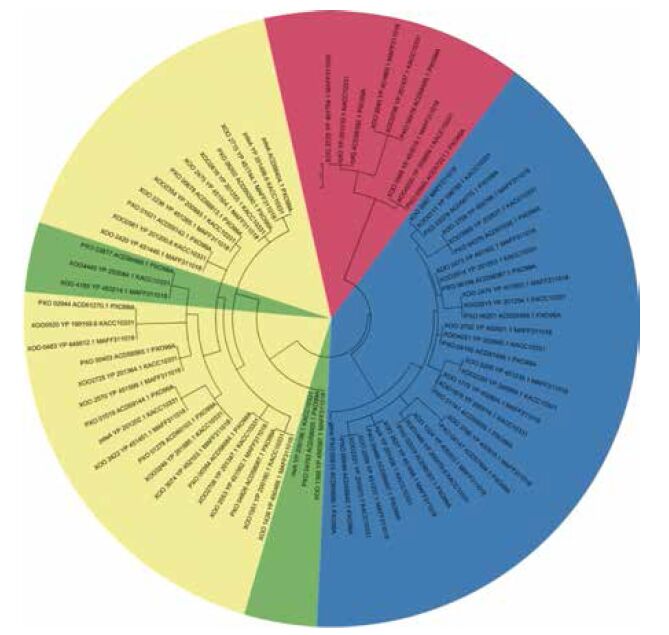
|
| 由77 条GGDEF、EAL 和HD-GYP 结构域的氨基酸序列构建而成 图 1 采用最大似然法构建水稻白叶枯病菌系统进化树 |
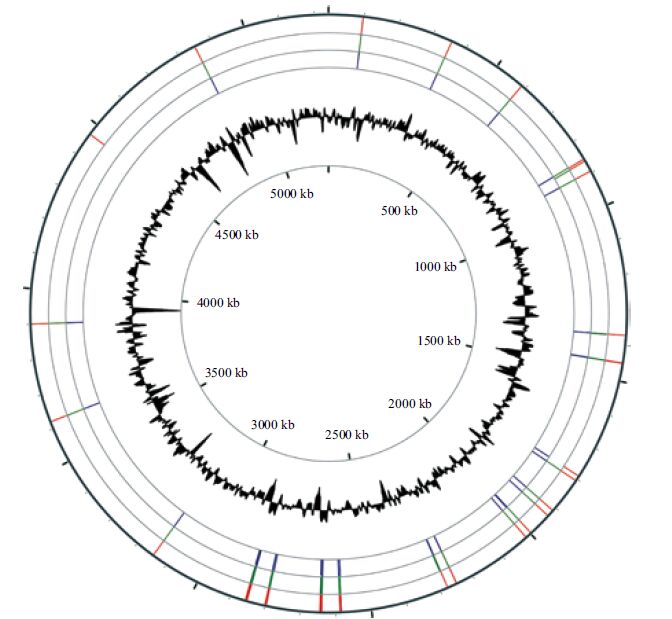
|
| 红色、绿色和蓝色线分别代表PXO99A,MAFF311018 和KACC10331 基因组中c-di-GMP 代谢相关基因。最内层标尺标出基因组的具体位置,内一层是G+C 含量 图 2 水稻白叶枯病菌c-di-GMP 代谢相关基因在基因组的分布 |
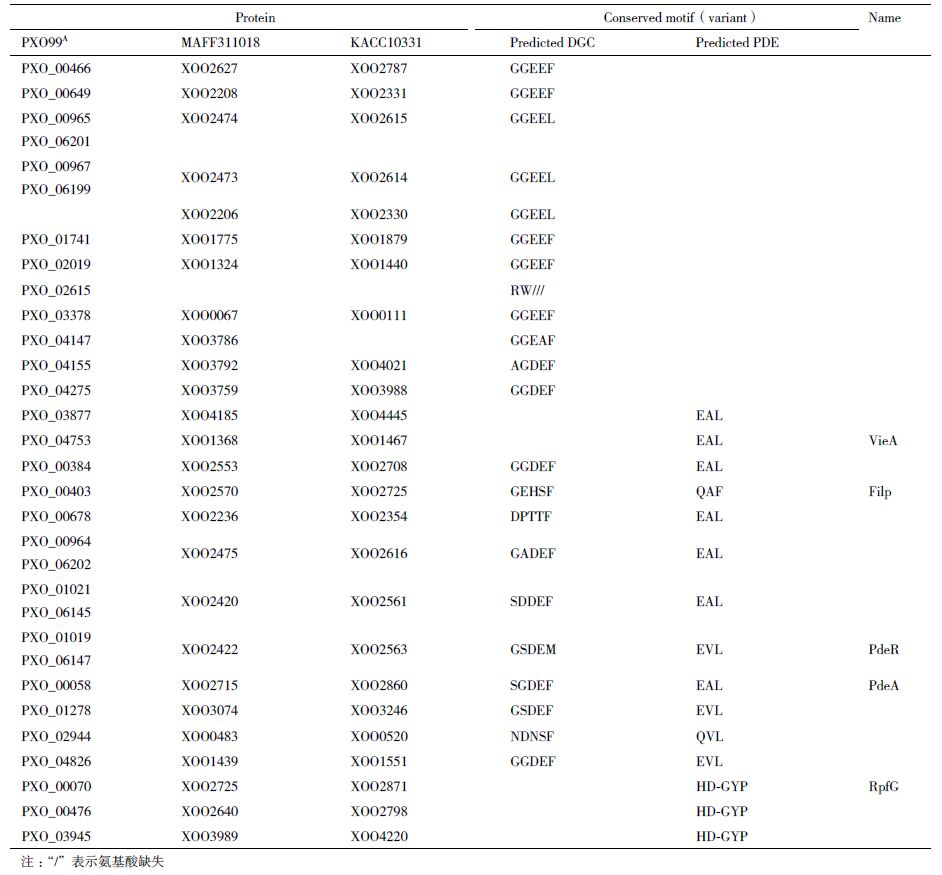
39%的GGDEF结构域含有GGEEF或GGDEF保守基序(其中GGEEF占62.5%),87%的含有EAL或EVL以及HD-GYP保守基序(表 1和表 2)。每个菌株有19个GGDEF、EAL和HD-GYP基序保守的蛋白,预测它们可能是有活性的DGC或PDE;其余退化的蛋白是c-di-GMP信号受体(表 2)。
24%的GGDEF结构域蛋白具有DGC酶活性的保守结构,包括保守的GGDEF或GGEEF基序和I位点(RXXD基序)、GTP和Mg2++结合序列、GTP结合KXXXR保守序列(图 3)。PDE包含EAL和HD-GYP结构域蛋白两种类型。
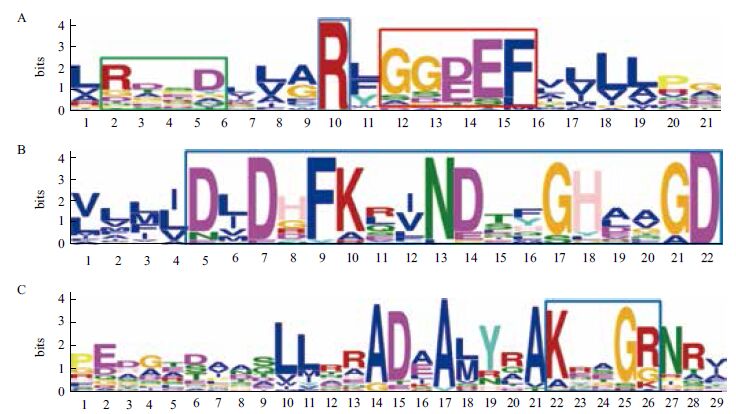
|
| A :I-site、GGDEF motif 和其他位点;B :GTP/Mg2+ 结合的保守位点;C :GTP 结合的保守位点 图 3 MEME 分析水稻白叶枯病菌DGC 的保守序列 |
含EAL结构域的PDE具有对活性非常重要的EAL/EVL和DDFGTG保守基序;RocR的R179和Q161是c-di-GMP结合关键位点,而D295、N233、E265、E175、E352、E268、K316和Q372是Mg2++结合关键位点[18],Mg2++为PDE活性所必须。MEME分析(图 4)表明,Xoo存在相同的保守位点。含HD-GYP结构域的PDE具有保守的HD和GYP残基,中间间隔60或61位氨基酸,HD残基对酶催化的影响非常关键。

|
| A :EAL 基序和c-di-GMP 结合保守位点;B :DDFGTG 基序和Mg2++ 结合保守位点 图 4 MEME 分析水稻白叶枯病菌PDE 的保守序列 |
已经验证了部分不同结构域蛋白的生物学功能(表 2)。GGDEF/EAL结构域保守的PXO_01019(PdeR)是具有c-di-GMP信号降解活性的PDE[14];GGDEF/EAL结构域退化的Filp(PXO_00403)是c-di-GMP信号受体[15];EAL单一结构域蛋白PXO_04753(VieAxoo)突变体生物膜形成明显增多[16];HD-GYP结构域蛋白PXO_00070(RpfCxoo)突变体毒性、EPS产生、生物膜形成能力等有显著变化[17]。
2.4 信号输入结构域的鉴定信号输入结构域通过感知外部环境信号变化,调节下游输出结构域的活性。GGDEF、EAL和HD-GYP蛋白具有多种信号接受结构域(表 1)。19%的DGC和40%的PDE具有REC结构域,接收磷酸化信号[19]。24%的DGC和27%的PDE具有PAS结构域,能感应环境氧、光、氧化还原电位、小配基以及细胞总能级的变化[20]。10%的DGC和13%的PDE包含GAF结构域,与cGMP结合后能调节催化活性[21]。HAMP仅存在于GGDEF蛋白中,与配基结合后具有调节同源二聚体受体磷酸化和甲基化的作用[22]。3个同源蛋白(PXO_02019、XOO1324和XOO1440)具有Cache_1结构域,参与细菌趋化性的小分子识别[23]。3个同源蛋白(PXO_01741、XOO1775和XOO1879)具有CHASE3结构域,涉及跨膜受体的信号接收[24]。 此外,11%的DGC和7%的PDE具有信号肽,48%的DGC和33%的PDE具有跨膜结构(表 1),表明这些信号肽可以透过或者锚定在细胞膜上,发生DGC和PDE活性的调节[25, 26]。
3 讨论本研究发现在Xoo中存在大量的c-di-GMP信号相关的基因,反映了其在细菌中具有重要的作用。其中一部分基因编码了DGC和PDE代谢酶,控制了c-di-GMP的胞内含量,另一部分结构域发生退化,可能编码了受体或结合蛋白,与c-di-GMP信号分子结合后,进而调控细菌包括毒性、生物膜形成、EPS产生和运动性等在内的诸多生物学功能。然而,c-di-GMP信号途径是一个非常复杂的调控网络,可能存在反馈抑制c-di-GMP合成的调节作用。此外,它还与双组份系统、群体感应信号系统等共同发挥作用,介导了下游的调控反应。
在其它一些与Xoo近缘关系相近的黄单胞细菌中,c-di-GMP信号基因的功能已获得了验证。橘黄单胞菌(X. citri subsp. citri)XAC0610(PXO_04155、XOO_3792和XOO_4021同源蛋白)具有DGC酶活性,虽然它并不具有GGDEF/GGEEF保守基序,但具有与Mg2++和GTP结合的保守位点[27]。水稻细菌性条斑病菌(X. oryzae pv. oryzicola)RpfG(PXO_00070、XOO_2236和XOO_2871的同源蛋白)和野油菜黄单胞菌(X. campestris)RavR具有PDE酶活性[28, 29]。磷酸化后的RavR杂合结构域蛋白具有PDE活性,去磷酸化后具有DGC酶活性[30],这更加说明c-di-GMP代谢调控网络的复杂性。
另外,虽然已验证Xoo四个GGDEF、EAL和HD-GYP 结构域蛋白的功能,但其它大多数的功能尚有待试验分析。目前,正在对其余蛋白进行DGC/PDE活性、c-di-GMP结合作用以及细菌毒性相关表型等测定。
4 结论Xoo GGDEF、EAL和HD-GYP 结构域蛋白参与了c-di-GMP信号途径,包括作为DGC或PDE合成或降解了c-di-GMP,或作为c-di-GMP信号受体调控了细菌生物学功能。此外,多数蛋白具有感知环境信号变化的输入结构域,调控输出结构域的活性。
| [1] | Mew TW. Current status and future prospects of research on bacterial blight of rice[J]. Annual Review of Phytopathology, 2003, 25:359-382. |
| [2] | Nino-Liu Do, Ronald PC, Bogdanove AJ. Xanthomonas oryzae pathovars:model pathogens of a model crop[J]. Molecular Plant Pathology, 2006, 7:303-324. |
| [3] | Lee BM, Park YJ, Park DS, et al. The genome sequence of Xanthomonas oryzae pathovar oryzae KACC10331, the bacterial blight pathogen of rice[J]. Nucleic Acids Research, 2005, 33(2):577-586. |
| [4] | Ochiai, H, Inoue Y, Takeya M, et al. Genome sequence of Xanthom-onas oryzae pv. oryzae suggests contribution of large numbers of effector genes and insertion sequences to its race diversity[J]. JARQ, 2005, 39:275-287. |
| [5] | Salzberg SL, Sommer DD, Schatz MC, et al. Genome sequence and rapid evolution of the rice pathogen Xanthomonas oryzae pv. oryzae PXO99A[J]. BMC Genomics, 2008, 9:204. |
| [6] | Jenal U, Malone J. Mechanisms of cyclic-di-GMP signaling in bacteria[J]. Annual Review of Genetics, 2006, 40:385-407. |
| [7] | 管文静, 吴茂森, 何晨阳. c-di-GMP信号途径对细菌致病性的调控作用[J]. 微生物学通报, 2009, 36(3):427-431. |
| [8] | Römling U, Galperin MY, Gomelsky M. Cyclic di-GMP:the first 25 years of a universal bacterial second messenger[J]. Microbiology and Molecular Biology Reviews, 2013, 77(1):1-52. |
| [9] | Hengge R. Principles of c-di-GMP signaling in bacteria[J]. Nature Reviews Microbiology, 2009, 7(4):263-273. |
| [10] | Schirmer T, Jenal U. Structural and mechanistic determinants of c-di-GMP signaling[J]. Nature Reviews Microbiology, 2009, 7:724-735. |
| [11] | Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10):2731-2739. |
| [12] | Felsenstein J. Confidence limits on phylogenies:An approach using the bootstrap[J]. Evolution, 1985, 39:783-791. |
| [13] | Jones DT, Taylor WR, Thornton JM. The rapid generation of mutation data matrices from protein sequences[J]. Computer Applications in the Biosciences, 1992, 8:275-282. |
| [14] | Yang FH, Tian F, Sun L, et al. A novel two-component system PdeK/PdeR regulates c-di-GMP turnover and virulence of Xanthomonas oryzae pv. oryzae[J]. Molecular Plant-Microbe Interactions, 2012, 25(10):1561-1569. |
| [15] | Yang FH, Tian F, Li XT, et al. The degenerate EAL-GGDEF domain protein Filp functions as a cyclic di-GMP receptor and specifically interacts with the PilZ-domain protein PXO_02715 to regulate viru-lence in Xanthomonas oryzae pv. oryzae[J]. Molecular Plant-Microbe Interactions, 2014, 27(6):578-589. |
| [16] | 梁士敏, 杨凤环, 管文静, 等. 水稻白叶枯病菌EAL结构域蛋白VieAxoo基因缺失突变和功能分析[J]. 微生物学报, 2011, 51:29-34. |
| [17] | 孙蕾, 吴茂森, 陈华民, 等. 水稻白叶枯病菌Δrpfxoo基因缺失突变体DSF信号产生和毒性表达[J]. 微生物学报, 2010, 50:717-723. |
| [18] | Rao F, Yang Y, Qi Y, et al. Catalytic mechanism of cyclic di-GMP-specific phosphodiesterase:a study of the EAL domain-containing RocR from Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2008, 190(10):3622-3631. |
| [19] | Skerker JM, Prasol MS, Perchuk BS. Two-component signal transduction pathways regulating growth and cell cycle progression in a bacterium:a system-level analysis[J]. PLoS Biology, 2005, 3(10):e334. |
| [20] | Taylor BL, Zhulin IB. PAS domains:internal sensors of oxygen, redox potential, and light[J]. Microbiology and Molecular Biology Reviews, 1999, 63(2):479-506. |
| [21] | Hughes J. Phytochrome three-dimensional structures and functions[J]. Biochemical Society Transactions, 2010, 38(2):710-716. |
| [22] | Parkinson JS. Signaling mechanisms of HAMP domains in chemoreceptors and sensor kinases[J]. Annual Review of Microbiology, 2010, 64:101-22. |
| [23] | Anantharaman V, Aravind L. Cache-a signaling domain common to animal Ca(2+)-channel subunits and a class of prokaryotic chemotaxis receptors[J]. Trends in Biochemical Sciences, 2000, 25(11):535-537. |
| [24] | Zhulin IB, Nikolskaya AN, Galperin MY. Common extracellular sensory domains in transmembrane receptors for diverse signal transduction pathways in bacteria and archaea[J]. Journal of Bacteriology, 2003, 185:285-294. |
| [25] | Walter P, Johnson AE. Signal sequence recognition and protein targeting to the endoplasmic reticulum membrane[J]. Annual Review of Cell Biology, 1994, 10:87-119. |
| [26] | Martoglio B, Dobberstein B. Signal sequences:more than just greasy peptides[J]. Trends in Cell Biology, 1998, 8(10):410-415. |
| [27] | Oliveira MC, Teixeira RD, Andrade MO, et al. Cooperative substrate binding by a diguanylate cyclase[J]. Journal of Molecular Biology, 2015, 427(2):415-432. |
| [28] | Zhang YB, Wei C, Jiang WD, et al. The HD-GYP domain protein RpfG of Xanthomonas oryzae pv. oryzicola regulates synthesis of extracellular polysaccharides that contribute to biofilm formation and virulence on rice[J]. PLoS One, 2013, 8(3):e59428. |
| [29] | He YW, Boon C, Zhou L, et al. Co-regulation of Xanthomonas campestris virulence by quorum sensing and a novel two-component regulatory system RavS/RavR[J]. Molecular Microbiology, 2009, 71(6):1464-1476. |
| [30] | Tao J, Li C, Luo C, et al. RavA/RavR two-component system regulates Xanthomonas campestris pathogenesis and c-di-GMP turnover[J]. FEMS Microbiology Letters, 2014, 358(1): 81-90. |