2. 中国科学院上海高等研究院可持续技术研究中心,上海 201210
2. Shanghai Advanced Research Institute,Chinese Academy of Sciences Sustainable Technology Research Center,Shanghai 201210
微生物自从被发现直到20世纪60年代,人们一直认为细菌是单细胞生物,不会表现出多细胞生物的性状。20世纪60年代,研究人员发现作为单细胞生物的细菌个体间有交流能力,并能表现出一些多细胞生物才有的性状,研究人员把这种信号交流方式称作群体感应(Quorum sensing,QS)[1]。首次发现群体感应现象的细菌是一种海洋细菌费氏弧菌(Vibrio fischeri),它能够通过群体感应来控制自身发光现象[2],群体感应能让细菌细胞感知周围细胞密度变化,从而引发其在高细胞密度下特有的、多样的细胞行为模式[3]。
群体感应细菌产生、释放、检测并能应答一种称为“自诱导物(AI)”的小分子物质,也称为信号分子。细菌在生长过程中不断产生并释放信号分子,随着细菌密度增加,信号分子浓度也在增加,当浓度达到阈值时,信号分子会与细菌中表达某些特定功能蛋白的基因结合,启动相应功能基因的表达,从而使细菌表现出另一些表型特征。
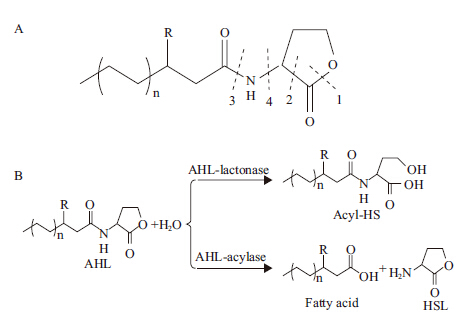
群体感应现象在许多已知的细菌中都存在,从自然界中游离生活的细菌到寄生于高级生物体内的细菌(共生体和病原菌),不同的细菌细胞产生的信号分子不同,革兰氏阴性细菌产生酰基高丝氨酸内酯(Acyl-homoserine lactones,AHL)类分子(图 1-A);革兰氏阳性细菌则是利用一种小分子多肽(Autoinducing peptides,AIP)作为信号分子;还有一种自诱导物质称为AI-2,结构为呋喃酮酰硼酸二酯,在革兰氏阴性菌和阳性菌中均存在,用于两种细菌的种间信息交流[4]。
 |
| 图 1 酰基高丝氨酸内酯(AHL)分子结构及两种酶的作用位点 |
群体感应控制着细菌的很多活动,如抗生素的产生及对抗生素的抗性、接合作用、毒力因子的产生、胞外酶合成、群集、生物膜形成及生物发光等[5, 6, 7, 8, 9],具有群体感应系统的细菌很多都是植物病原菌,植物的细菌性病害是威胁农业生产的主要自然灾害之一。病害的发生往往造成作物大面积减产,并在农产品贮藏、加工、运输过程中进一步造成更大的损失。通常对这类致病菌繁殖是采用化学药物来防治,虽然化学药物的使用能显著减轻病害的危害程度。但大量化学药物的使用,会导致环境污染,而且细菌耐药性增加,就需要增加用药量,从而导致更严重的环境污染,再者残留的化学药物也会危害人体健康等。所以,寻找更干净、更健康的方法来抑制植物的细菌性病害成为一种迫切需求。干扰QS系统则成为一种新型的病害防治策略,群体淬灭应运而生,群体淬灭是利用某种方法干扰或者降解群体感应产生的信号分子,使信号分子浓度低于启动致病因子表达所需要的阈值,从而使致病菌的致病因子无法启动表达,致病菌不表现出致病力,病害不再发生。群体淬灭主要有如下3个方式:(1)干扰信号分子的合成;(2)降解信号分子,使信号分子浓度低于阈值;(3)阻止信号分子与受体蛋白结合,使之不能行使转录调节功能[10]。其中降解信号分子是研究较多的一种淬灭方式,这种方式主要是由细菌产生一种酶称为群体淬灭酶,这种酶能降解信号分子,保持信号分子浓度低于阈值,使病原菌无法表达致病因子,不对宿主产生病害作用。这种方式对外界没有副作用,而且它并不作用于致病菌本身,而是作用于致病菌产生的信号分子,因而不会使致病菌产生抗药性,能作为一种高效、长久的药物选择。
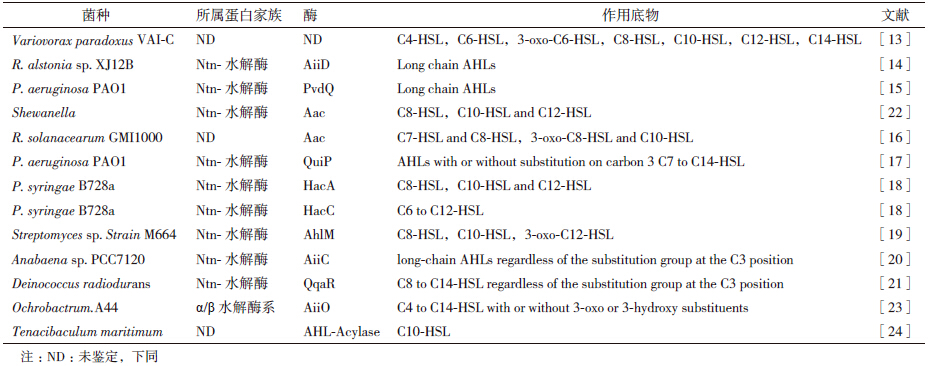
3 群体淬灭酶群体淬灭酶能利用群体感应中的信号分子AHL作为底物,通过酶促反应使AHL分解。第一个鉴定出来的群体淬灭酶是由一株革兰氏阳性菌芽孢杆菌中的基因aiiA编码的酰基高丝氨酸内酯酶(Acyl-homoserine lactonase)[7],随后,Leadbetter and Gree-nberg[11]报道争论贪噬菌(Variovorax para-doxus VAI-C)能利用信号分子AHL作为唯一能源和氮源物质,其降解AHL分子的酶属于酰胺酶。在这两株菌中发现的群体淬灭酶几乎代表了群体淬灭酶中两个重要的分支。
根据AHL的分子结构(图 1-A),可能至少有4种酶能够降解AHL分子,内酯酶和脱羧酶能从1号位和2号位水解AHL,而酰基转移酶和脱氨基酶能从3号位和4号位裂解AHL,但到目前为止,只发现了两种AHL降解酶:酰基高丝氨酸内酯酶和酰基高丝氨酸内酯酰基转移酶(Acyl-homoserine lactone acylase)[12]。酰基高丝氨酸内酯酶主要作用于高丝氨酸内酯环,从五环中酯键碳与氧之间打开内酯环(图 1-B),使得信号分子失去活性;高丝氨酸酰基转移酶则作用于与内酯五环相连的酰基侧链,切断酰胺键,生成脂肪酸和高丝氨酸内酯环(图 1-B),信号分子即失去活性。
3.1 AHL酰基转移酶第一种酰基转移酶的发现来自于争论贪噬菌VAI-C,当生长环境中有AHL存在时,这种菌会向培养基中分泌水解产物高丝氨酸内酯,而AHL的侧链脂肪酸则用来分解产生能量[13]。其他有代表性的酰基转移酶有来自青枯菌XJ12B(Ralstonia sp. XJ12B)中由aiid编码的酰基转移酶AiiD[14];铜绿假单胞菌PAO1中存在两种酰基转移酶PvdQ 和QuiP[12, 15, 17],二者间在氨基酸序列上只有21%的同源性;放线菌链霉菌M664菌株(Streptomyces sp.)中的酰基转移酶AhlM[19]。淡水藻类中也发现了具有AHL降解活性的菌,鱼腥藻属PCC7120(Anabaena sp. PCC7120)中由基因all3924编码的酰基转移酶AiiC,这种藻类的细胞原液能够降解相当一部分的AHLs,尤其是长链AHLs,侧链3号碳上的取代基对它的活性没有影响[20]。最近,有人在极端微生物耐辐射奇球菌(Deinococcus radiodurans)中发现了能降解AHL信号分子的酶,进一步分析确定这是一种酰基转移酶[21]。其他AHL酰基转移酶,见表 1。
在已经发现的酰基转移酶中,序列分析显示几乎所有AHL酰基转移酶都属于N末端亲核(Ntn)水解酶,如AiiD、PvdQ、QuiP、AhlM、AiiC等,这类酶经相应基因编码后翻译形成一个保守区域,该区域分成两个亚单位:一个信号肽,后面接一个α亚单位,然后是间隔序列,跟着一个β亚单位。β亚单位中熟化和催化作用所需要得N末端亲核在AHL酰基转移酶中是高度保守的。定点突变显示区域中保守的甘氨酸-丝氨酸对与AHL酰基转移酶活性至关重要[22]。但是Robert Czajkowsk等[23]在对一株苍白杆菌Ochrobactrum.A44研究发现,它所产生的酶AiiO虽然属于酰基转移酶,结构分析却表明这种酶没有其他酰基转移酶所具有的Ntn水解酶结构,而是属于α/β水解酶折叠酶系。
由于不同菌产生的酰基转移酶不同,所以这些酶能作用的底物信号分子也有所不同,青枯菌XJ12B合成的酰基转移酶AiiD能降解长链AHL,铜绿假单胞菌PAO1中的酰基转移酶QuiP,能够降解侧链长度在11-14个碳的AHL信号分子,而侧链3号碳上的取代基并不影响其活性,说明这种酶具有范围较广的AHL淬灭活性[17],链霉菌中AhlM能够降解范围较广的AHLs,还能降解盘尼西林G,说明AhlM的底物选择性较广[19],Czajkowsk等[23]从苍白杆菌(Ochrobactrum.A44)中发现的AiiO能降解酰基侧链长度在C4-C14的AHL,侧链3位碳上无论是氢取代还是氧取代对它的活性都没有影响,而且该酶更易降解长链的AHL。还有人发现,一种来自海洋鱼类致病菌的黄杆菌(Tenacibaculum maritimum),它能够利用短链AHL(C4-HSL)形成细胞膜,同时也表现酰基转移酶活性能够降解长链AHLs(C10-HSL)[24]。
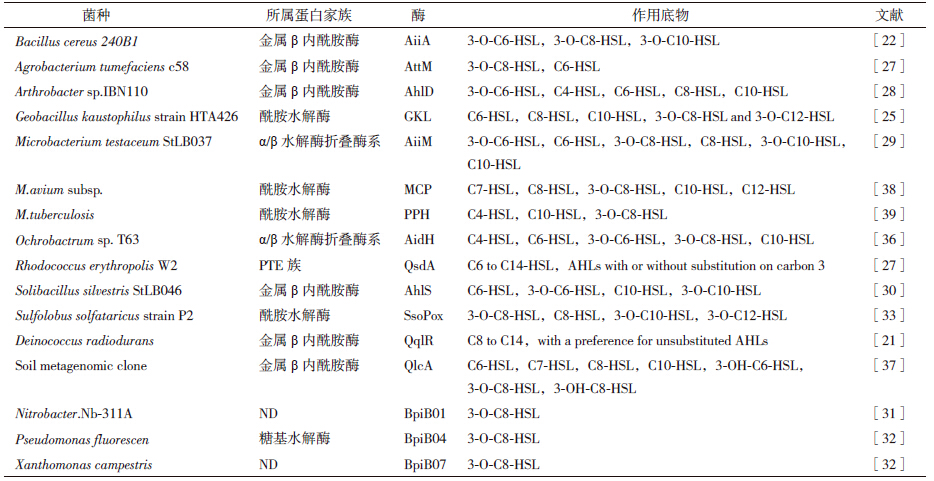
3.2 AHL内酯酶第一种AHL内酯酶AiiA是从芽孢杆菌240B1中发现的,由aiiA基因编码,芽孢杆菌中很多菌能产生AiiA同系物,其中研究较多的属苏云金芽孢杆菌中的AiiA。在一些芽孢杆菌中还发现了能产生耐热内酯酶,如嗜热地芽孢杆菌HTA426(Geobacillus kaustophilus HTA426)[25]和土芽孢杆菌YS-8(G. ca-ldoxylosilyticus YS-8)[26]。除了芽孢杆菌外,在其他一些细菌中也发现了AHL内酯酶,如根癌农杆菌中attM编码的AHL内酯酶AttM、aiiB编码的AiiB[27]、节杆菌(Arthrobacter)中的AhlD[28]、克雷伯肺炎杆菌(Klebsiella pneumonia)中的AhlK[28]、细杆菌(Microbacterium testaceum)中的AiiM[29]、土壤芽孢杆菌(Solibacillus silvestris)中的AhlS[30]及红球菌(Rhodococcus)中的QsdA[27]等。近几年,通过元基因组方法鉴定出了一组编码内酯酶的基因-bpiB[31],已经从硝化细菌(Nitrobacter)Nb-311A、荧光假单胞菌(Pseudomonas fluorescen)和黄单胞菌(Xanthomonas campestris)中检测出了BpiB01、BpiB04和BpiB07三种同源基因编码的酶,这3种酶能够降解3-O-C8-HSL,序列分析显示它们与已知的AHL降解基因没有相似性[31, 32]。其他内酯酶,见表 2。
对已知内酯酶的序列分析,几乎所有的内酯酶都属于金属β内酰胺酶,根据核苷酸序列水平上的同源性差异,系统分析进一步分为两类,首先是AiiA类,由所有芽孢杆菌中的AHL内酯酶组成,由于它们在氨基酸水平上与芽孢杆菌Bacillus sp. 240B1同源性达90%以上;第二类是AttM类,包括AttM、AiiB和AhlD,由于产生这一类酶的菌在系统分类上处于同一个分支系统下,故将这些酶统一分类为AttM类,这类酶在多肽序列上只有30%-58%的相似性。在AttM和AiiA两类内酯酶中都发现了一个高度保守的Zn2+结合域HXHXDH,但AttM和AiiA两类酶的同源性却还不到25%,该区域已经证明了对于AHL内酯酶活性是必需的,该结构域类似一些金属水解酶的Zn2+结合区域,然而它又与已知的金属水解酶Zn2+结合区域有所不同,属于一种新型的结构[7]。在已经发现的内酯酶中,几乎所有内酯酶在酶活性中心都含有一个Zn2+结合位点,而且实验也证明,Zn2+能增加内酯酶的活性,甚至有的酶中若没有Zn2+则没有内酯酶活性,说明Zn2+对于内酯酶活性比较重要,不同内酯酶Zn2+结合的区域有所不同。
尽管几乎所有内酯酶都属于金属β内酰胺酶,但仍有一些与内酰胺酶完全没有关系的内酯酶。第一种QsdA,从一株红串红球菌R.erythropolis W2中鉴定出来的AHL内酯酶[33],这是一种磷酸三酯酶(PTE),能够降解C6到C14的AHLs,PTE酶也是一种金属依赖性蛋白,Zn2+结合在PTE区域,能裂解磷酸三酯键[34],同时具有内酯酶活性和酰胺水解酶活性。土芽孢杆菌属中的嗜热菌也能产生PTE型AHL内酯酶,这种嗜热菌产生的酶由于耐热性在生物技术上具有非常大的应用潜力,如G. kaustophilus产生的GKL[35]。
除了β内酰胺酶和PTE之外,第三类发现AHL内酯酶的是α/β水解酶折叠酶系,比如细杆菌StLB037中的AiiM[29]和苍白杆菌Ochrobactrum anthropi ATCC 49188中的AidH[36],α/β水解酶是一类结构上相关,具有多种不同催化功能的酶,这类酶有两个共同特征:一个亲核酸性组氨酸催化三分子和一个序列为Gly-X-Nuc-X-Gly的亲核基团,这两个区域对AHL降解活性至关重要[36]。
此外,还有一些其他的内酯酶,如通过土壤元基因组文库分析得到的锌依赖型金属水解酶QlcA[37],与发现的其他内酯酶无太大联系;还有BpiB01、BpiB04和BpiB07[31, 32]等,氨基酸序列上也与其他内酯酶没有明显的相似性。
通常,根据细菌代谢方式不同,一种菌只产生一种酶,但是红串红球菌中却报道了能产生不同的AHL降解酶[40]。比对基因组显示,在同一菌中能同时存在编码AHL酰基转移酶和内酯酶的基因,这类菌还有耐辐射奇球菌R1(D.radiodurans),生丝单胞菌ATCC15444(Hyphomonas neptunium)和发光杆菌TTO1(Photorhabdus luminescens)[41],其中耐辐射奇球菌R1已经被证明其基因组中含有编码AHL内酯酶和酰基转移酶的基因qqlR和qqaR,并且实验中检测到耐辐射奇球菌能同时产生这两种酶,它们都能降解侧链为长链的AHL信号分子[21]。能编码两种酶的细菌很有可能是为了能在环境中取得生存优势,一种酶不足以分解环境中其他菌产生的信号分子,因而进化出了能产生两种酶的基因;还有一种可能是一些寄生菌为了防止自身数量过大而进化出的一种机制,其中一种酶用以分解其他菌产生的信号分子,取得生存优势,而另一种酶则在种群数量达到某个值时表达产生,分解自身所产信号分子,使自身数量控制在一定范围内不足以威胁寄主安全,从而使细菌与寄主达到共生的目的。
3.3 群体淬灭酶的应用由于AHL内酯酶和AHL酰基转移酶能够分解AHL信号分子,并且所有这些酶都来自微生物细胞,有越来越多的证据表明这些酶在细菌与细菌间相互作用中起重要作用。
在含有aiiA基因的苏云金芽孢杆菌中,表达产生的AHL内酯酶AiiA能抑制具有群体感应的植物病原菌软腐欧文氏菌(Erwinia carotovora),软腐欧文氏菌能通过信号分子控制毒性基因的表达,苏云金芽孢杆菌能显著抑制毒力因子的产生,降低软腐欧文氏菌带来的致病性[7];从一株布鲁氏菌(Bru-cella melitensis)中发现的酰基转移酶AibP在体外条件下能够降解自身产生的内生AHL分子并在巨噬细胞的侵染期间减少AHL的积累[42];含有attM基因的根癌农杆菌能抑制Ti质粒的接合转移[27]等,这些数据表明,菌株在自然界中取得竞争优势,群体淬灭酶具有重要作用。
另一方面,淬灭酶在植物病害防治方面也已有进展,aiiA基因表达编码的AHL内酯酶能减少病原菌感染烟草和线虫,而且,转基因植物所表达的AHL内酯酶能有效淬灭细菌群体感应信号分子并减少细菌依赖于群体数量的侵染[43],一些能产生AHL内酯酶的野生菌或者工程菌如芽孢杆菌(B. thuringiensis)、节杆菌、假单胞菌(P. fluorescens)等都能显著降低致病菌软腐欧文氏菌引起的马铃薯软腐病[22, 28, 32],因此,这些基因编码的群体淬灭酶在植物抗病性和生物制药方面有非常广阔的前景。
群体淬灭酶在环境方面同样有所应用,废水处理中,处理系统会由于微生物产生的生物膜出现膜生物淤积,从而导致处理效率的显著降低,膜生物淤积的传统处理方法是利用物理方法和化学抗菌剂或者抗生素来减少膜生物淤积,这些方法旨在消除生物膜或者杀死产生膜的微生物来达到减少淤积,因而会产生抗药性和二次污染问题,而群体淬灭并不对微生物生长产生影响,成为了一种非常有应用前景的防止膜生物淤积技术[44]。
随着分子生物学技术的发展,有人对群体淬灭酶进行改造并取得了一定的进展。在酰基转移酶PvdQ中,将其中的两个氨基酸Leuα146Trp和Pheβ24Tyr替换后得到的突变酶活性比原野生型酶显著提高;再将两种突变酶重组得到另一突变酶PvdQLα146W,Fβ24Y,它能显著减少伯克霍尔德氏菌(Burkholderia cenocepacia)中信号分子的量并抑制群体感应调控的表型产生[45];其他突变型酶还有从嗜热地芽孢杆菌中得到的一种耐热内酯酶,属于重组磷酸三酯酶类,这种酶是通过分子技术改进的耐热性工程酶,它的催化效率更高并且能催化的底物信号分子范围更广,能显著减少鲍氏不动杆菌(A.baumannii)生物膜的量,鲍氏不动杆菌是人体病原菌,说明这种重组酶在药物应用上比较有潜力[46]。重组AiiA酶则能减少栖息于铜绿假单胞菌调节的生物膜结构中浮游细胞的数量[47],而固定化的SsoPox酶(GKL)能够抑制铜绿假单胞菌中许多毒力因子的产生[48],关于淬灭酶的固定化应用,Byoungsoo等[49]将群体淬灭酰基转移酶用磁分离介孔二氧化硅固定化以后发现,在200 r/min剧烈摇晃下,固定化酶与游离酶失去90%活性所需时间分别为10 d和1 d,将固定化酶应用到水处理上后,能有效减少生物膜的形成,说明淬灭酶固定化在水处理的膜过滤方面有很大的应用前景。
4 小结前面已经讨论过根据信号分子的结构特征,至少有4种酶能降解AHL分子,但只发现两种,另外两种酶也可能存在,已经有文献报道了一种氧化还原酶,这种氧化还原酶本身并不降解信号分子,而只是氧化或还原信号分子的酰基侧链来对信号分子进行修饰,使信号分子不能与受体蛋白结合[50],还需要对这些酶进行更多的研究。群体淬灭酶的广泛存在意味着这类酶在微生物之间以及微生物和宿主之间相互作用中的重要作用,它不直接作用于微生物本身,因而不会使目标微生物产生抗性,对于病害防治是一种高效措施,而且群体淬灭酶不会污染环境,不会带来环境问题,将成为非常有潜力的农药替代品,但是目前要使群体淬灭酶能商业化运用还存在很多困难,如酶在自然环境中的稳定性,以及这类新型“农药”的运输、贮存和使用等问题,因此还需要大量的研究来解决这些问题。
| [1] | Suga H, Kristina M, Smith KM. Molecular mechanisms of bacterial quorom sensing as a new drug target[J].Curr Opin Chem Biol, 2003, 7(5):586-591. |
| [2] | Nealson KH, Platt T, Hastings JW. Cellular control of the synthesis and activity of the bacterial luminescent system[J].J Bacteriol, 1970, 104(1):313-322. |
| [3] | Fuqua WC, Winans SC, Greenberg EP. Quorum-sensing in bacteria:the LuxR-LuxI family of cell density-responsive transcriptional regulatiors[J].J Bacteriol, 1994, 176(2):269-275. |
| [4] | Bassler BL. How bacteria talk to each other:regulation of gene exp-ression by quorum sensing[J].Curr Opin Microbiol, 1999, 2(6):582-587. |
| [5] | Bruhn JB, Dalsgaard I, Nielsen KF, et al. Quorum sensing signal molecules(acylated homoserine lactones)in gram-negative fish pathogenic bacteria[J].Dis Aquat Org, 2005, 65(1):43-52. |
| [6] | Kalia VC, Purohit HJ. Quenching the quorum sensing system:potential antibacterial drug targets[J].Crit Rev Microbiol, 2011, 37(2):121-140. |
| [7] | Dong YH, Xu JL, Li XZ, et al. AiiA, an enzyme that inactivates the acylhomoserine lactone quorum-sensing signal and attenuates the virulence of Erwinia carotovora[J].Proc Natl Acad Sci USA, 2000, 97(7):3526-3531. |
| [8] | Swift S, Downie JA, Whitehead NA, et al. Quorum sensing as a popu-lation-density-dependent determinant of bacterial physiology[J].Adv Microb Physiol, 2001, 45(11):199-270. |
| [9] | Williams P. Quorum sensing, communication and cross-kingdom signalling in the bacterial world[J].Microbiology, 2007, 153(12):3923-3938. |
| [10] | Bentley SD, Chater KF, Cerdeno-Tarraga AM, et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2)[J].Nature, 2002, 417(9):141-147. |
| [11] | Leadbetter JR, Greenberg EP. Metabolism of acylhomoserine lactone quorum-sensing signals by Variovorax paradoxus[J].J Bacteriol, 2000, 182:6921-6926. |
| [12] | Dong YH, Zhang LH. Quorum sensing and quorum-quenching enzymes[J].The Journal of Microbiology, 2005, 43(S):101-109. |
| [13] | Czajkowski R, Jafra S. Quenching of acyl-homoserine lactone-dependent quorum sensing by enzymatic disruption of signal molecules[J].Acta Biochim Pol, 2009, 56(1):1-16. |
| [14] | Lin YH, Xu JL, Hu J, et al. Acyl-homoserine lactone acylase from Ralstonia strain XJ12B represents a novel and potent class of quorum-quenching enzymes[J].Mol Microbiol, 2003, 47(3):849-860. |
| [15] | Huang JJ, Han JI, Zhang LH, Leadbetter JR. Utilization of acyl-homoserine lactone quorum signals for growth by a soil pseudomonad and Pseudomonas aeruginosa PAO1[J].Appl Environ Microbiol, 2003, 69(10):5941-5949. |
| [16] | Chen CN, Chen CJ, Liao CT, Lee CY. A probable aculeacin A acylase from the Ralstonia solanacearum GMI1000 is N-acyl-homoserine lactone acylase with quorum-quenching activity[J].BMC Microbiol, 2009, 9(89):1-11. |
| [17] | Huang JJ, Petersen A, Whiteley M, Leadbetter JR. Identification of QuiP, the product of gene PA1032, as the second acyl-homoserine lactone acylase of Pseudomonas aeruginosa PAO1[J].Appl Environ Microbiol, 2006, 72(2):1190-1197. |
| [18] | Shepherd RW, Lindow SE. Two dissimilar N-acyl-homoserine lactone acylases of Pseudomonas syringae influence colony and biofilm morphology[J].Appl Environ Microbiol, 2009, 75:45-53. |
| [19] | Park SY, Kang HO, Jang HS, et al. Identification of extracellular N-acylhom-oserine lactone acylase froma Streptomyces sp. and its application to quorum quenching[J].Appl Environ Microbiol, 2005, 71(5):2632-2641. |
| [20] | Romero M, Diggle SP, Heeb S, et al. Quorum quenching activity in Anabaena sp. PCC 7120:identification of aiiC, a novel AHL-acylase[J].FEMS Microbiol Lett, 2008, 280(1):73-80. |
| [21] | Koch G, Nadal-Jimenez P, Cool RH, Quax WJ. Deinococcus radiodurans can interfere with quorum sensing by producing an AHL-acylase and an AHL-lactonase[J].FEMS Microbiology Letters, 2014, 356(1):62-70. |
| [22] | Morohoshi T, Nakazawa S, Ebata A, et al. Identification and characterization of N-acylhomoserine lactone-acylase from the fish intestinal Shewanella sp. strain MIB015[J].Biosci Biotechnol Biochem, 2008, 72(7):1887-1893. |
| [23] | Czajkowski R, Krzyzanowska D, Karczewska J, et al. Inactivation of AHLs by Ochrobactrum sp. A44 depends on the activity of a novel class of AHL acylase[J].Environmental Microbiology Reports, 2011, 3(1):59-68. |
| [24] | Romero M, Avenda?o-Herrera R, Magari?os B, et al. Acylhomose-rine lactone production and degradation by the fish pathogen Tenacibaculum maritimum, a member of the Cytophaga-Flavobact-erium-Bacteroides(CFB)group[J].FEMS Microbiol Lett, 2010, 304(2):131-139. |
| [25] | Chow JY, Xue B, Lee KH, et al. Directed evolution of a thermost-able quorum-quenching lactonase from the amidohydrolase superfa-mily[J].Biol Chem, 2010, 285:40911-40920. |
| [26] | Seo MJ, Lee BS, Pyun YR, Park H. Isolation and characterization of N-acylhomoserine lactonase from the thermophilic bacterium, Geobacillus caldoxylosilyticus YS-8[J].Biosci Biotechnol Biochem, 2011, 75(9):1789-1795. |
| [27] | Hong KW, Koh CL, Sam CK, et al. Quorum quenching revisited—from signal decays to signalling confusion[J].Sensors, 2012, 12(4):4661-4696. |
| [28] | Park SY, Lee SJ, Oh TK, et al. AhlD, an N-acylhomoserine lactonase in Arthrobacter sp. , and predicted homologues in other bacteria[J].Microbiology, 2003, 149(6):1541-1550. |
| [29] | Wang WZ, Morohoshi T, Ikenoya M, et al. AiiM, a novel class of N-acylhomoserine lactonase from the leaf-associated bacterium Microbacterium testaceum[J].Appl Environ Microbiol, 2010, 76(8):2524-2530. |
| [30] | Morohoshi T, Tominaga Y, Someya N, Ikeda T. Complete genome sequence andcharacterization of the N-acylhomoserine lactone-degrading gene of the potato leaf-associated Solibacillus silvestris[J].J Biosci Bioeng, 2012, 113(1):20-25. |
| [31] | Schipper C, Hornung C, Bijtenhoorn P, et al. Metagenome-derived clones encoding two novel lactonase family proteins involved in biofilm inhibition in Pseudomonas aeruginosa[J].Appl Environ Microbiol, 2009, 75(1):224-233. |
| [32] | Amara N, Krom BP, Kaufmann GF, Meijler MM. Macromolecular inhibition of quorum sensing:enzymes, antibodies, and beyond[J].Chem Rev, 2011, 111(1):195-208. |
| [33] | Elias M, Dupuy J, Merone L, et al. Structural basis for natural lactonase and promiscuous phosphotriesterase activities[J].J Mol Biol, 2008, 379(5):1017-1028. |
| [34] | Uroz S, Oger PM, Chapelle E, et al. A Rhodococcus qsdA-encoded enzyme defines a novel class of large-spectrum quorum-quenching lactonases[J].Applied and envirmental Microbiology, 2008, 74(5):1357-1366. |
| [35] | Elias M, Tawfik DS. Divergence and convergence in enzyme evolution:parallel evolution of paraoxonases from quorum-quenching lactonases[J].The Journal of Biological Chemistry, 2011, 287(1):11-20. |
| [36] | Mei GY, Yan XX, Turak A, et al. AidH, an alpha/beta-hydrolase fold family member from an Ochrobactrum sp. strain, is a novel N-acylhomose-rine lactonase[J].Appl Environ Microbiol, 2010, 76(15):4933-4942. |
| [37] | Riaz K, Elmerich C, Raffoux A, et al. Metagenomics revealed a quorum quenching lactonase QlcA from yet unculturable soil bacteria[J].Commun Agric Appl Biol Sci, 2008, 73(2):3-6. |
| [38] | Chow JY, Wu L, Yew WS. Directed evolution of a quorum-quenching lactonase from Mycobacterium avium subsp. paratuberculosis K-10 in the amidohydrolase superfamily[J].Biochemistry, 2009, 48(20):4344-4353. |
| [39] | Afriat L, Roodveldt C, Manco G, Tawfik DS. The latent promiscuity of newly identified microbial lactonases is linked to a recently diverged phosphotriesterase[J].Biochemistry, 2006, 45(46):13677-13686. |
| [40] | Park SY, Hwang BJ, Shin MH, et al. N-acyl homoserine lactone producing Rhodococcus spp. with different AHL-degrading activities[J].FEMS Microbiol Lett, 2006, 261(1):102-108. |
| [41] | Kalia VC, Purohit HJ. Quenching the quorum sensing system:potential antibacterial drug targets[J].Crit Rev Microbiol, 2011, 37(2):121-140. |
| [42] | Terwagne M, Mirabella A, Lemaire J, et al. Quorum sensing and self-quorum quenching in the intracellular pathogen Brucella melitensis[J].PLoS One, 2013, 8(12):e82514. |
| [43] | Dong YH, Wang LH, Xu JL, et al. Quenching quorum-sensing-dependent bacterialinfection by an N-acyl homoserine lactonase[J].Nature, 2001, 411:813-817. |
| [44] | Lade H, Paul D, Kweon JH. Quorum quenching mediated approaches for control of membrane biofouling[J].International Journal of Biological Sciences, 2014, 10(5):550-565. |
| [45] | Koch G, Nadal-Jimenez P, Reis CR, et al. Reducing virulence of the human pathogen Burkholderia by altering the substrate specificity of the quorum-quenching acylase PvdQ[J].National Academy of Sciences, 2014, 111(4):1568-1573. |
| [46] | Chow YJ, Yang YY, Tay SB, et al. Disruption of Biofilm Formation by the Human Pathogen Acinetobacter baumannii using engineered quorum-quenching lactonases[J].Antimicrobial Agents and Chemotherapy, 2014, 58(3):1802-1805. |
| [47] | Igarashi J, Suga H. Custom synthesis of autoinducers and their analogues[M].Methods Mol Biol, 2011, 692:265-274. |
| [48] | Hawwa R, Aikens J, Turner RJ, et al. Structural basis for thermost-ability revealed through the identification and characterization of a highly thermostable phosphotriesterase-like lactonase from Geobac-illus stearothermophilus[J].Arch Biochem Biophys, 2009, 488(2):109-120. |
| [49] | Lee B, Yeon KM, Shim J, et al. Effective antifouling using quorum-quenching acylase stabilized in magnetically-separable mesoporous silica[J].American Chemical Society, 2014, 15(4):1153-1159. |
| [50] | Romero M, Mayer C, Muras A, et al. Silencing bacterial communi-cation through enzymatic quorum-sensing inhibition[M] //Quo-rum Sensing vs Quorum Quenching:A Battle with No End in Sight. Springer India, 2015:219-236. |