2. 贵阳医学院附属医院 儿科学教研室,贵阳 550004 ;
3. 山东省荷泽市市立医院 口腔科,荷泽 274000
2. Guiyang Medical College Affiliated Hospital Department of Pediatrics, Guiyang 550004;
3. Department of Stomatology, Heze Municiple Hospital, Heze 274000
同源异型盒基因(homeobox genes)又称HOX基因,是一类含有同源框、参与动物早期胚胎发育。HOX基因是高度保守的转录调控因子,按照胚胎发育期的前—后轴(anterior-posterior axis)方式表达[1]。更重要的是过表达Hoxb基因可以促进造血干细胞(HSC)的更新及增殖[2, 3, 4]。目前已鉴定的哺乳类动物HOX基因共有39个,是控制胚胎发育和细胞分化的调节基因,其分为分为4簇,分别位于7(HOXA)、17(HOXB)、12(HOXC)、2(HOXD)号染色体上,根据簇间基因序列的相似性及基因在染色体上的位置又分为13组。HOXB4基因编码一类具有同源盒 DNA 依赖的结构域核蛋白,在进化过程中高度保守,HOX蛋白调节许多细胞途径,如细胞分裂,细胞粘附、增殖、细胞凋亡,HOX基因的异常表达已被证实与多种成人恶性肿瘤相关[1]。作为一种特异性的转录因子,此核蛋白是造血细胞自我更新和分化的重要调控因子,并且在狗及小鼠中若提高Hoxb4基因的表达量可显著促进HSC的自我更新能力。以往研究表明过表达Hoxb4基因可以显著地增加移植后小鼠的造血干细胞的再生,并且可以提高人类的严重联合免疫缺陷(SCID)再植细胞的再增殖能力;与其他Hox基因簇不同的是在移植了HOXB4细胞的1-3个月后的小鼠中并没有发生白血病等异常[5]。本研究利用CRISPR/Cas9系统对斑马鱼的hoxb4基因进行基因组编辑,并筛选及测序验证出其阳性突变体,为初步观察敲除了hoxb4基因的斑马鱼的表型变化提供实验材料,为研究hoxb4基因对斑马鱼早期造血发育的影响机制及其作用的下游靶基因作铺垫。
1 材料与方法 1.1 材料野生型斑马鱼:Tuebingen(本课题组饲养),质粒pSP6-2sNLS-spCas9,质粒pT7-gRNA(由上海交通大学医学院附属瑞金医院上海血液病研究所 袁浩博士惠赠),DH5α感受态(本课题组制备)。
根据靶点预测网站http://zifit.partners.org/ZiFiT/CSquare9Nuclease.aspx 预测符合以下设计条件的hoxb4基因的sgRNA,设计原则如下:(1)靶序列需符合5'-GG-N(20)-NGG-3'的设计原则,可以位于正义链,也可以位于反义链。(2)将靶序列在ncbi上比对,与pam区相连的碱基与其他基因组里比对后相同的碱基数不能超过12个。(3)靶序列的5'端添加atag,3'端添加gt,靶序列互补链的5'端添加taaaac,使其与质粒经BbsⅠ酶切后形成粘性互补末端。靶序列如下表 1。(4)在靶序列的上下游各选择约300 bp,共600 bp左右,设计引物并PCR鉴定。
将化学合成好的寡核苷酸单链经退火形成互补的双链:将上下游靶序列稀释为工作浓度10 μmol/L,各取0.5 μL,加入kod plus buffer 2.5 μL,加入双蒸水21.5 μL。然后95℃,5 min;58℃,10 min,自然冷却至室温。用BbsⅠ单酶切线性化质粒pT7-gRNA,跑电泳形成单一的条带后割胶回收后测其浓度约40 ng/μL。加入以下连接体系:T4 ligase:1 μL,10×T4 buffer:1.5 μL,线性化质粒pT7-gRNA:2.5 μL,靶序列寡核苷酸双链:10 μL。22℃反应2 h。使用DH5α感受态进行转化,涂板,培养过夜后挑取单克隆,进行菌液PCR鉴定,目的条带大小约130 bp,抽提质粒并用通用引物M13-47及RV-M序列测序验证(北京奥维森公司完成)。
1.2.3 体外转录Cas9 mRNA使用XbaⅠ单酶切线性化质粒pSP6-2sNLS-spCas9,酚氯仿回收线性化产物,sp6启动子体外转录得到Cas9 mRNA,加样体系如下:2× NTP/CAP:10 μL 10× Reaction Buffer:2 μL,Linearized template DNA:1 μg,SP6 Enzyme Mix:2 μL,补加双蒸水至20 μL。混匀,瞬离,在37℃恒温反应2h后取0.5 μL,2%的琼脂糖凝胶跑电泳15 min,转录出来的mRNA长度约1 300 bp,然后加入1 μL的 TURBO DNase,37℃反应15 min以去除DNA模板。然后取0.5 μL电泳验证模板DNA是否去除干净。将转录出来的Cas9的mRNA进行添加polyA序列,其反应体系如下:SP6 转录体系:21 μL,双蒸水:36 μL,5XE-PAP buffer:20 μL,25 mmol/L MnCl2:10 μL,10 mmol/L ATP:10 μL。吸出1 μL作为此反应电泳时的对照组。然后加入4 μL E-PAP,混匀,瞬离,放入37℃恒温反应1 h;2%的琼脂糖凝胶跑电泳30 min。添加polyA序列后的mRNA长约1 500 bp。将所得mRNA经试剂盒纯化回收,使用分光光度计测浓度并分装:300 ng/μL。
1.2.4 体外转录制备gRNA以构建好的gRNA质粒1 μL(浓度为5 ng/ μL)为模板,T7-cr fwd和tracr rev各1 μL作为上下游的引物,加入kod plus酶1 μL进行PCR,94℃,5 min;94℃,15 s,30个循环;60℃,30 s,30个循环;68℃,30 s,30个循环;68℃,10 min;4℃,保存。取3 μL的PCR产物在2%的琼脂糖凝胶中跑电泳30 min,目的条带大小约130 bp。然后酚氯仿回收纯化PCR产物。以此纯化的产物作为模板体外转录gRNA,体系如下:10 mmol/L NTP:各1 μL;10×Reaction Buffer:2 μL;Template DNA:1 μg;T7 Enzyme Mix:2 μL;DEPC H2O:4 μL。混匀,瞬离,放入37℃恒温反应1 h,取1 μL 反应液最后作对照,加入1 μL TURBO DNase,混匀,瞬离,37℃恒温反应15 min以去除DNA模板。取1 μL在2%琼脂糖凝胶中电泳15 min,确认转录的条带约130 bp。采用试剂盒纯化回收体外转录产物。紫外分光光度计测浓度并分装:70 ng/ μL。
1.2.5 显微注射并检测敲除效率显微注射上述两者的RNA,其浓度比约为Cas9∶gRNA=3∶1。将上述体外转录的产物按比例混匀显微注射入单细胞期的野生型斑马鱼Tuebinggen胚胎中。此步骤严格要求RNase-free。每个靶点注射300枚胚胎,同时留一部分未注射的同一批胚胎作为对照。28℃恒温培养箱中培养2 d,分别收集2 d的注射有靶点的胚胎作为实验组及未注射靶点的胚胎作为对照组,5个/epp管。提取基因组,并以此为模板,ExonⅠ的sense及antisense作为引物,PCR自己的目的基因:长度为415 bp。将PCR产物采用酚氯仿纯化回收。T7EⅠ酶切检测敲除效率:纯化的PCR产物:minimum 200 ng;加入NEB buffer 1 μL,95℃,5 min;ramp down to 85℃ at -2c/s;ramp down to 25℃ at -0.1c/s;4℃保存。然后加入0.5 μL的T7EⅠ酶,37℃反应30 min,2%的琼脂糖凝胶电泳30 min。突变条带的百分比用Biorad GelDoc XR 凝胶成像软件分析灰度值。
1.2.6 连入T载体并测序以确定碱基变化情况将PCR产物连入pMD19-T simple载体中,挑取单克隆,过夜摇菌,取适量菌液做菌落PCR,电泳鉴定条带大小以排除空载,将有目的条带的PCR产物送测序,测序引物为通用引物:RV-M及M13-47。测序结果与原基因组DNA用chromos软件进行序列比对。检测是否成功靶向斑马鱼的hoxb4基因。
2 结果 2.1 构建的质粒hoxb4-gRNA的测序结果插入序列与预期相符(图 1),说明质粒构建成功。
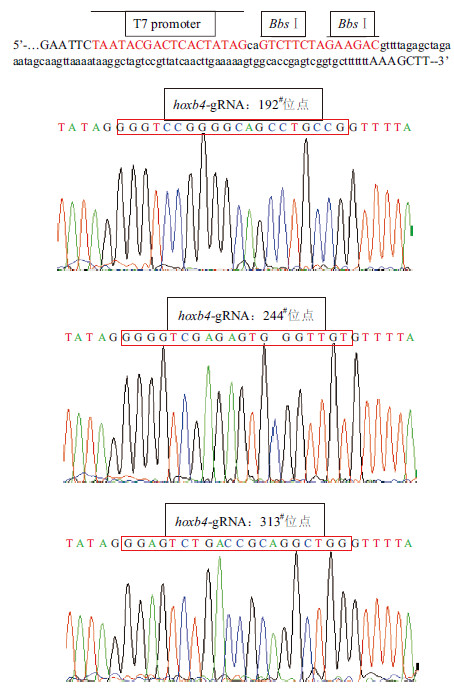 |
| 图 1 质粒构建测序图 |
ExonⅠ的192#位点、244#位点及313#三个位点均有阳性结果,突变条带所占的百分比用Biorad GelDoc XR 凝胶成像软件分析灰度值。切出的多的条带的亮度可大致反应突变效率,3个位点的突变率分别为:6.5%、10.2%和26.5%(图 2)。
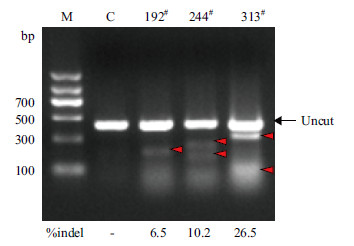 |
| M :DNA Marker ;C :control ;灰色三角指示预期的T7E Ⅰ酶切出的片段,黑色箭头指示未突变的条带图 2 T7E Ⅰ酶切测效图 |
将突变率高的313#靶点的PCR产物连入T载体并使用一代测序(Sanger测序),测序结果与ExonⅠ原基因组序列比对进一步确认碱基的变化,如图 3。将同一批注射了sgRNA的斑马鱼恒温培养至受精后3-4 d,显微镜下观察其表型变化(图 4),可以看到实验组有发育迟缓的表现,并且可以看到约10%的斑马鱼有轻微的心包增大,心腔肥大血流减少的表现。
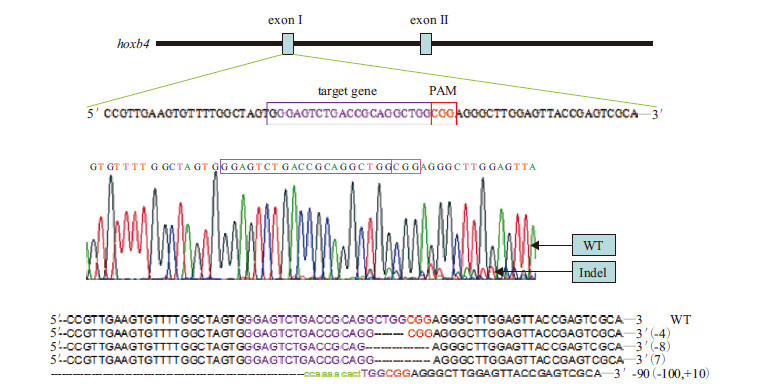 |
| + :插入的碱基数,- :缺失的碱基数图 3 Sanger 测序验证靶向Exon Ⅰ sgRNA 的敲除效果及4 个阳性的碱基插入或缺失的突变体 |
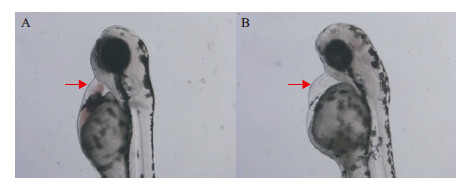 |
| A :对照组;B :注射了313# gRNA 后2d的斑马鱼(红色箭头所示为心包, 放大3 倍)图 4 48 h 的斑马鱼心腔变化 |
RISPR/Cas9系统作为一种新兴的基因定点编辑技术,其功能自1987年日本科学家的首次报道后逐渐成熟,并在多个物种中得到广泛应用如酵母[6]、斑马鱼[7]、人类细胞[8]、果蝇[9]、线虫[10]、水稻等[11]。这种sgRNA介导的Cas9酶切作用能够在多种类型的细胞和生物体内正常地行使功能,在完整基因组上的特定位点完成切割反应。
相比基因组定点编辑技术,如ZFNs[12, 13]及TALENs[14, 15],ZNFs成本高昂,效率低,而每一对TALENs都需要重新合成。CRISPR/Cas9系统的优点为:设计更简单,仅需要20 bp的碱基配对就可以靶向目的基因[16, 17, 18, 19, 20, 21],在人类基因组中每8个碱基就会出现一个PAM序列(NGG);实验周期短;效率高效;sgRNA全长不超过100 bp,构建更容易。本实验采用Ⅱ型Cas9蛋白介导基因组靶序列的双链断裂(Double strand breaksk,DSBs),从而引发机体的非同源末端连接(Non-homologous ending-joining)NHEJ机制[22],这样就为获得多种表型提供可能性。现在国内外研究已经将CRISPR/Cas9系统改造尽量使其的效率更高,不仅可以达到基因敲除的目的,也可以使基因在转录水平沉默(dCas9)[23, 24],而且还可以实现基因敲入[25]。以上3种方法的难度及复杂度存在于后期筛选其阳性突变体及是否可遗传给子代。而2014年Dong等[26]发表的文章提到,将带有YFP-nanos3基因的mRNA、Cas9的mRNA以及单链的sgRNA三者共注射入单细胞期的斑马鱼胚胎中,如果此黄色荧光蛋白能够到达PGCs(原始生殖细胞)区域,就可以直接通过荧光显微镜观察到黄色荧光蛋白,从而快速、简便地筛选出可以遗传的突变体,可以说通过YFP来筛选阳性突变体是一个突破性的的进展。
在测定CRISPR/Cas9系统的敲除效率时,有两种方法可供选择,一种是在靶位点选择一个限制性的单一的酶切位点,此酶切位点用于后期测效简便、时间短;另一种是如果所选的靶位点没有合适的单一酶切位点,那么就需要用T7EI来检测敲除效率。本实验采用的是第二种,在后期测序鉴定时就需要挑取更多的单克隆来筛选鉴定,此方法耗时,费力。所以在选择靶位点时最好能筛选出一个单一的限制性酶切位点以便检测敲除效率。
本实验成功通过CRISPR/Cas9系统在基因组水平编辑斑马鱼hoxb4基因,发现敲除了hoxb4基因的少数斑马鱼有心包增大的表型,并且在显微镜下肉眼可以观察到其心腔动静脉有增大表现,其心肌泵血弱而无力,这有两种可能:(1)hoxb4基因可能是导致其发育迟缓的重要原因之一;(2)本身显微注射RNA后,往往会出现胚胎发育迟缓的现象。显微注射过程中可能造成了对胚胎较大的伤害(技术欠熟练等原因造成的),因而部分胚胎中出现了心包增大等现象。但以往的研究不可否认hoxb4基因对胚胎的发育起到很重要的作用。
通过CRISPR/Cas9系统敲除hoxb4基因的方法在本课题组成功建立并获得了hoxb4突变的嵌合体F0(Founder),若想获得纯合突变的hoxb4基因的斑马鱼模型,需经过外交并对F1、F2逐条筛选,最终得到可遗传的突变体模型,本实验为筛选出纯合突变体奠定了基础,为后续观察在敲除了hoxb4的斑马鱼体内检测与其造血相关的基因如Irx5a(伊洛魁同源框蛋白5a)、Rap1b(RAS的致癌基因)、Cdh5(血管内皮)等可以得到更直观的表型;可以为研究hoxb4基因调控的下游靶基因及其如何调控造血干细胞的增殖提供实验模型。
4 结论将靶向斑马鱼hoxb4 的外显子一的靶序列克隆到载体pT7-gRNA质粒中,构建gRNA的体外转录载体并通过体外转录得到靶位点的gRNA,线性化质粒pSP6-2sNLS-spCas9并通过体外转录获得Cas9的mRNA,将混合好的两种RNA经显微注射并T7EⅠ酶切检测靶位点敲除效率,将突变率高的313#靶点的PCR产物连入T载体经PCR鉴定、Sanger测序后证明成功通过CRISPR/Cas9系统编辑斑马鱼hoxb4基因。
致谢:感谢上海交通大学医学院附属瑞金医院上海血液病研究所袁浩博士惠赠质粒pSP6-2sNLS-spCas9及质粒pT7-gRNA。
| [1] | Hur H, Lee JY, Yun HJ, et al. Analysis of HOX gene expression patterns in human breast cancer[J]. Springer Molecular Biotechnology, 2013, 56:64-71. |
| [2] | Miyake N, Brun AC, Magnusson M, et al. HOXB4-induced self-renewal of hematopoietic stem cells is significantly enhanced by p21 deficiency[J]. Stem Cells, 2006, 24:653-661. |
| [3] | Pearson JC, Lemons D, McGinnis W. Modulating Hox gene functions during animal body patterning[J]. Nat Rev Genet, 2005, 12:893-904. |
| [4] | Shah N, Sukumar S. The Hox genes and their roles in oncogenesis[J]. Nature , 2010, 10(10):361-370. |
| [5] | Zhang XB, Schwartz JL, Humphries RK, Kiem HP . Effects of HOXB4 overexpression on ex vivo expansion and immortalization of hematopoietic cells from different species[J]. Stem Cells, 2007, 25:2074-2081. |
| [6] | DiCarlo JE, Norwille JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J]. Nucleic Acids Res, 2013, 41(7):4336-4343. |
| [7] | Chang NN, Sun CH, Gao L, Zhu D, et al. Genome editing with RNA-guided Cas9 nucleasein zebrafish embryos[J]. Cell Res, 2013, 23(4):465-472. |
| [8] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121):823-826. |
| [9] | Gratz SJ, Cummings AM, Nguyen JN, et al. Genome engineering of Drosophila with the CRISPR RNA-guided Cas9 nuclease[J]. Genetics, 2013, 194(4):1029-1035. |
| [10] | Friedland AE, Tzur YB, Esvelt KM, et al. Heritable genome editing in C. elegans via a CRISPR-Cas9 system [J]. Nat Methods, 2013, 10(8):741-743. |
| [11] | Shan QW, Wang Y Li J, Zhang Chen KL, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31(8):686-688. |
| [12] | Bibikova M, Golic M, Golic KG, et al. Targeted chromosomal cleav-age and mutagenesis in Drosophila using zinc-finger nucleases[J]. Genetics, 2002, 161(3):1169-1175. |
| [13] | Dreier B, Fuller RP, Segal DJ, et al. Development of zinfinger domains for recognition of the 5’-CNN-3 family DNA sequences and their use in the construction of artificial transcrirltion factors [J]. J Biol Chem, 2005, 280(42):5588-5597. |
| [14] | Hockemeyer D, Wang HY, Kiani S, et al. Genetic engineering of human pluripotent cells using TALE nucleases[J]. Nat Biotechnol, 2011, 29(8):731-734. |
| [15] | Huang P, Xiao A, Zhou MG, et al. Heritable gene targeting in zebrafish using customized TALENS[J]. Nat Biotechnol, 2011, 29(8):699-700. |
| [16] | Gaj T, Gersbach CA, Barbas CF 3rd, et al. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Cell, 2013, 1073:1-9. |
| [17] | Hwang W Y, Fu YF, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, (3):227-229. |
| [18] | Mali P, Yang L, Esvelt K M, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 6121:823-826. |
| [19] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 6121:819-823. |
| [20] | Ding Q, Regan SN, Xia Y, et al. Enhanced Efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs[J]. Cell, 2013, 12:393-394. |
| [21] | Ramalingam S, Annaluru N, et al. A CRISPR way to engineer the human genome[J]. Genome Biol, 2013, 14(2):107-110. |
| [22] | Sander JD, Joung JK . CRISPR -Cas systems for editing, regulating and targeting genomes[J]. Nature Biotechnology, 2014, 1-9. |
| [23] | González F, Zhu ZR, Shi ZD, et al. An iCRISPR platform for rapid, multiplexable, and inducible genome editing in human pluripotent stem cells[J]. Cell, 2014, 15:1-12. |
| [24] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152:1173-1183. |
| [25] | Kimura Y, Hisano Y, Kawahara A, et al. Efficient generation of knock-in transgenic zebrafish carrying reporter/driwer genes by CRISPR/Cas9-medeated genome engineering[J]. Scientific Reports, 2014, 6545:1-7. |
| [26] | Dong ZJ, Dong XH, Jia WS. Improving the efficiency for generation of genome-edited zebrafish by labeling primordial germ cells[J]. Biochem Cell Biol, 2014, 5:329-334. |
| [27] | Taghon T, Thys K, De Smedt M, et al. Homeobox gene expression profile in human hematopoietic multipotent stem cells and T-cell progenitors:implications for human T-cell development. [J]. Leukemia, 2003, 17:1157-1163. |





