生物表面活性剂是微生物在特定培养条件下分泌的具有表面活性的代谢产物,与化学合成表面活性剂相比,具有结构多样性、高效和生物可降解性等优点,有潜力替代化学合成表面活性剂的地位[1]。
许多种类的微生物都可分泌生物表面活性剂,包括细菌、酵母菌和丝状真菌。生物表面活性剂种类包括糖脂类、脂肽类、磷脂类和脂肪酸类等。其中,细菌生产的表面活性剂一般都是糖脂类[2]。鼠李糖脂是糖脂类表面活性剂中一种重要类型,一般由鼠李糖基和长链脂肪酸组成。合成鼠李糖脂的微生物主要是铜绿假单胞菌,通常可以合成含一个鼠李糖基的单鼠李糖脂和含两个鼠李糖基的双鼠李糖脂,且以双鼠李糖脂为主[3]。
鼠李糖脂可以利用可再生及经济的底物生产,例如餐饮废油、油厂废水和油饼粉等[4]。目前的文献多以排油圈直径或表面张力等指标来评价生物表面活性剂的性能,较少文献以产量为出发点评价细菌生产鼠李糖脂的能力[5]。
本研究采用CTAB亚甲基蓝平板法从实验室保存菌株和环境样品中筛选能够生产鼠李糖脂的菌株,并通过蒽酮硫酸法定量检测每株菌的鼠李糖脂产量。利用分子生物学方法鉴定分离菌株的种类,研究不同碳源、发酵温度对菌株高产鼠李糖脂的作用,并对鼠李糖脂进行薄层色谱分析和红外光谱分析。
1 材料与方法 1.1 材料 1.1.1 菌株实验室保存假单胞菌属菌株。来源于天津科技大学校园内不同环境分离菌株,样品包括草地土壤、污泥、河水以及餐余富油垃圾等共计40份。
1.1.2 培养基与溶液CTAB固体培养基:KH2PO4 0.7 g/L、Na2HPO4 0.9 g/L、NaNO3 2 g/L、MgSO4 0.4 g/L、CaCl2 0.1 g/L、甘油20 g/L、CTAB 0.2 g/L、亚甲基蓝0.005 g/L、微量元素溶液1 mL/L、琼脂粉15 g/L[6],用于产鼠李糖脂菌株的筛选。
LB培养基:蛋白胨10 g/L、NaCl 10 g/L、酵母浸粉5 g/L。用于一般细菌培养及发酵中种子培养基。
无机盐培养基:NaCl 1 g/L、KCl 1 g/L、NaNO3 6 g/L、KH2PO4 3 g/L、Na2HPO4 0.3 g/L、MgSO4 2.5 g/L、CaCl2 1 g/L、微量元素溶液1 mL/L。
微量元素溶液:FeCl3 0.16 g/L、ZnSO4 1.5 g/L、CuSO4 0.15 g/L、MnSO4 1.5 g/L、H3BO3 1.5 g/L。
发酵培养基是在无机盐培养基中添加不同的碳源制成,本研究所用碳源包括甘油、菜籽油、花生饼粉和葵花籽饼粉。
蒽酮硫酸溶液:称取0.2 g蒽酮,加入到100 mL 98%硫酸中,用于定量测定鼠李糖脂的浓度。
1.2 方法 1.2.1 CTAB平板法筛选鼠李糖脂生产菌株鼠李糖脂是一种阴离子型表面活性剂,它能与培养基中的阳离子溴化盐形成不溶的复合物,然后通过亚甲基蓝的显色作用使产鼠李糖脂的菌落周围出现深蓝色的晕圈。晕圈的大小在一定程度上反映了鼠李糖脂的分泌水平。因此,CTAB平板法可区分产鼠李糖脂与不产鼠李糖脂的菌株[7]。
向环境样品加入无菌生理盐水稀释,振荡后静止10 min,取上清液涂布在CTAB平板上,挑选其上生长的菌株三区划线纯化保存。将获得的分离菌株与实验室保藏的假单胞菌属菌株单菌落点种在CTAB平板培养基上,37℃培养72 h。观察菌落周围是否出现深蓝色晕圈,并测量菌落直径与晕圈直径,晕圈直径与菌落直径的比值大致与鼠李糖脂的产量呈正比[8]。
1.2.2 鼠李糖脂产量的测定鼠李糖基团在强酸下被水解生成甲基糠醛,然后与蒽酮生成蓝绿色化合物,它在625 nm波长处有最大吸收,其吸光度与浓度呈线性关系。根据鼠李糖与鼠李糖脂分子量的关系,鼠李糖脂浓度=3.4×鼠李糖浓度[9]。
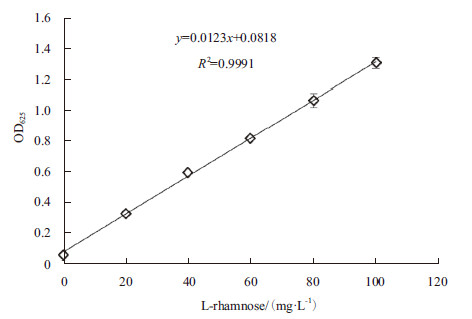
配制20、40、60、80和100 mg/L的鼠李糖标准溶液,每个浓度3个平行。分别取1.25 mL标准溶液与2.5 mL蒽酮硫酸溶液冰浴中混合,沸水浴中反应15 min。待反应液温度下降到室温,测定625 nm波长下的吸光度值,绘制吸光度值与鼠李糖浓度的标准曲线。得到吸光度值与鼠李糖浓度之间的关系:y=0.0123x+0.0818,y为吸光度值,x为L-鼠李糖(mg/L),线性相关系数为0.999 1,线性关系很强,可以用来计算鼠李糖浓度(图 1)。
 |
| 图 1 L-鼠李糖标准曲线 |
将单菌落接种到LB培养基中振荡培养过夜,转接3%种子液到发酵培养基中,35℃、200 r/min培养72 h。将发酵液12 000 r/min离心10 min,上清液用蒸馏水稀释合适倍数,取1.25 mL稀释液于冰浴中缓缓加入2.5 mL蒽酮硫酸溶液,沸水浴15 min。冷却至室温后测定625 nm波长处的吸光度值。根据标准曲线的函数关系及其他相关系数计算鼠李糖脂产量。
1.2.3 鼠李糖脂乳化活性将发酵液12 000 r/min离心10 min,上清液用蒸馏水稀释5倍后,取2 mL与等体积的液体石蜡加入透明玻璃试管(10 mm×10 cm)中,于涡旋振荡器上准确振荡1 min混匀。室温静置3 d后测量乳化层高度和发酵液稀释液层高度,并计算乳化指数。乳化指数(%)=乳化层高度/(乳化层高度+发酵液稀释液高度)×100%。
1.2.4 菌株鉴定及系统发育树构建挑取单菌落接种到LB培养基中振荡培养过夜,按照文献[10]中的方法提取细菌的基因组DNA。
以细菌的基因组DNA为模板,使用通用引物27F和1 492R[11]对16S rRNA基因进行扩增。PCR扩增程序:94℃预变性5 min;95℃变性1 min,53℃退火1 min,72℃延伸1.5 min,30个循环;72℃终延伸10 min。
采用引物27F和1 492R对扩增产物进行测序,使用NCBI网站上的BLAST工具进行序列同源性比对,确定菌株的种属[12]。利用MEGA5.0软件中的邻接法(Neighbor Joining,NJ)进行系统发育分析,构建系统发育树[13]。
1.2.5 产鼠李糖脂的碳源及发酵温度的优化对高产菌株进行碳源种类的优化,将高产菌株接种到种子培养基(LB培养基)中振荡培养过夜,转接到发酵培养基中(3%接种量),碳源分别为甘油、菜籽油、花生饼粉、葵花籽饼粉(榨油副产物打磨成的粉末),碳源含量为30 g/L。35℃,200 r/min振荡培养72 h,测定生物量(OD600)和鼠李糖脂产量。
对高产菌株进行发酵温度的优化,将高产菌株接种到种子培养基中过夜培养后,以3%接种量转接到30 g/L发酵培养基中,分别在35℃、37℃和40℃,200 r/min振荡培养72 h,分别测定生物量(OD600)和鼠李糖脂产量。
1.2.6 鼠李糖脂纯化及性质分析将发酵液以4 000 r/min的转速离心30 min,上清液用6.0 mol/L HCl调节pH至2.0,用等体积的有机溶剂(氯仿∶甲醇=2∶1,V/V)萃取2次后合并有机相,于45℃旋转蒸发得到棕黄色固体或黏稠液体,用0.05 mol/L NaHCO3溶液溶解后即得鼠李糖脂溶液[14]。
实验层析板为青岛化工硅胶G板,将纯化的鼠李糖脂溶液用毛细管滴加在距层析板一端约1 cm处的起点线上,风干后将薄层板置于盛有展开剂(氯仿∶甲醇∶醋酸=65∶15∶2,V/V/V)的展开槽中,待展开剂上升到距离顶端1 cm左右时取出层析板,干燥后喷洒显色剂(苯酚∶硫酸∶酒精=3 g∶10 mL∶25 mL),100℃烘烤显色。迁移率=待测物质迁移距离/溶剂展开距离。
红外光谱(Fourier Transform Infrared Spectrome-ter,FTIR)可以通过鉴定化学键和功能基团,从而确定混合物中的某种组分。将冷冻干燥的表面活性剂粉末与KBr(样品∶KBr=1∶100-300)10 MPa压片1 min形成半透明薄片,进行红外吸收光谱扫描。
2 结果 2.1 CTAB平板法筛选产鼠李糖脂菌株将经过处理的样品在CTAB平板上涂布并培养,共得到38个菌落大于2 mm且菌落周围具有深蓝色晕圈的菌株。另外,将实验室保存173株微生物点接在CTAB平板上,发现有125株细菌的菌落周围具有深蓝色晕圈,其余42株菌可以在CTAB平板上生长但不产生深蓝色晕圈,还有6株菌不能在CTAB平板上生长。共计有163株细菌菌落周围形成了深蓝色晕圈,显示这些菌株能够合成鼠李糖脂。
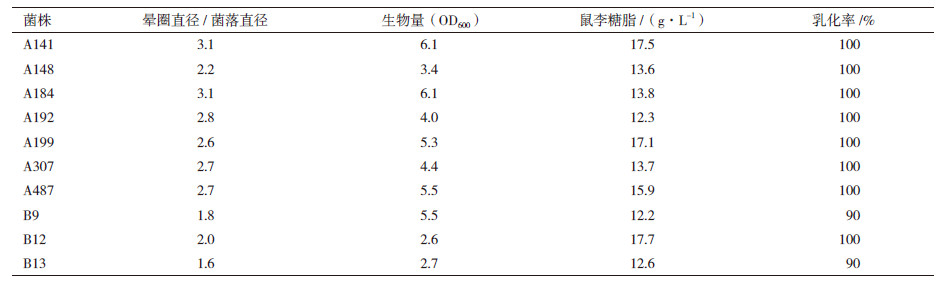
2.2 定量筛选高产鼠李糖脂菌株将初筛阳性的163株细菌分别接种到5 mL发酵培养基(20 g/L甘油为碳源)中,振荡培养72 h,测定鼠李糖脂产量。结果(表 1)显示,有10株细菌鼠李糖脂产量在12.2-17.7 g/L。其中,来源于土壤样品的菌株B12鼠李糖脂产量最高,为17.7 g/L。
分别提取上述10株细菌基因组DNA,扩增16S rRNA基因并进行序列测定。使用NCBI网站上BLASTN工具进行序列比对,10株细菌与铜绿假单胞菌Pseudomonas aeruginosa GF(KM259920)的同源性均为100%,确定分离菌株都属于铜绿假单胞菌。将序列上传至GenBank数据库,各菌株序列接收号见系统进化树(图 2),10株分离菌株同源性很高,位于同一个进化枝(clade)。与他们最相似的参比菌株中,P. aeruginosa FR2是从泰国南部温泉中分离的,能够分泌表面活性剂,且具有耐热的性质;P. aeruginosa GF是从印度被污染土壤中分离的菌株,可以降解氯酚等有毒化合物;P. aeruginosa JS-11最初分离于小麦根际土壤,能够降解苯脲类除草剂异丙隆[15]。
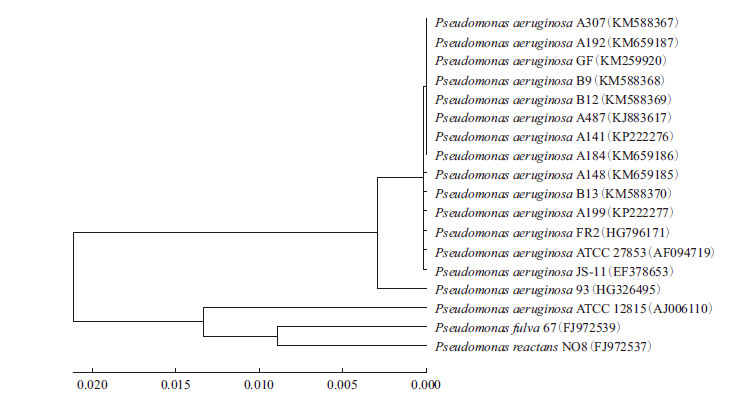 |
| 图 2 分离菌株的系统发育树 |
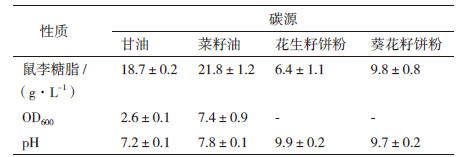
分别以甘油、菜籽油、花生饼粉和葵花籽饼粉为碳源,分析菌株B12在不同培养基中的鼠李糖脂产量、OD600和pH。结果(表 2)显示,分别以甘油、菜籽油、花生饼粉和葵花籽饼粉为碳源的培养基,鼠李糖脂产量依此为21.8、18.7、9.8和6.4 g/L,菜籽油是菌株B12合成鼠李糖脂最适宜的碳源。
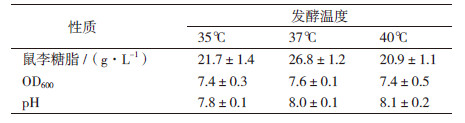
以30 g/L菜籽油为碳源,分别测定35℃、37℃和40℃条件下,菌株B12鼠李糖脂的产量。结果(表 3)显示,37℃发酵后获得的鼠李糖脂产量最高,为26.8 g/L。
通过碳源种类和发酵温度的优化,菌株B12发酵的条件确定为以菜籽油为碳源,发酵温度为37℃。
2.6 菌株B12发酵产鼠李糖脂分析将种子液以3%的接种量接种到含30 g/L菜籽油的发酵培养基中,37℃,200 r/min振荡培养。每12 h取样1 mL,分别测定样品的pH值、OD600和鼠李糖脂产量。时间曲线(图 3)显示,发酵液的pH值从初始的6.8上升到8.0左右。OD600值一般能够代表细菌的浓度,在本次发酵中,OD600值在12-24 h之间迅速从1.8攀升到了19.3,之后一直保持下降的趋势,到72 h发酵终点OD600值下降到了13.2。但是从摇瓶表观来看,一开始形成菜籽油的乳浊液,在之后的发酵中菜籽油逐渐被消耗,乳浊液逐渐变清。因此在本实验中OD600值并不能代表细菌的生长。鼠李糖脂的产量开始增长缓慢,后来开始大量合成,到发酵终点72 h时,鼠李糖脂产量为26.8 g/L。
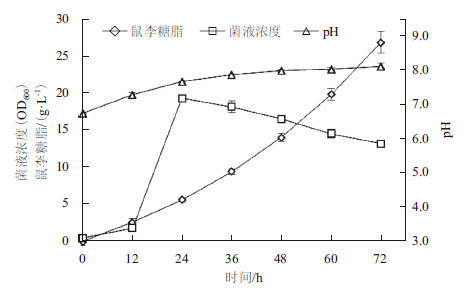 |
| 图 3 菌株P. aeruginosa B12 摇瓶发酵 |
对菌株B12发酵合成的鼠李糖脂进行萃取纯化,然后将鼠李糖脂溶液点在硅胶G板上进行薄层色谱分析。结果(图 4)显示,硅胶板上出现两个棕黄色的斑点,斑点的颜色证明了样品的糖脂类特性,斑点RL1(Rhamnolipid)和RL2的迁移率Rf分别为0.71和0.95。
 |
| 图 4 薄层色谱分析菌株B12 的鼠李糖脂产物 |
在薄层色谱分析中,硅胶吸附剂对样品中各成分的吸附能力不同,展开剂对各成分的吸附能力也不同,从而使混合物品得以分离。铜绿假单胞菌生产的鼠李糖脂可能含有一个或两个鼠李糖分子[16],其中,含有一个鼠李糖分子的糖脂极性较小,Rf值较大;含有两个鼠李糖分子的糖脂极性较大,其Rf值较小。可以看出,菌株B12合成的鼠李糖脂是混合物,包括单鼠李糖脂和双鼠李糖脂,且主要成分是双鼠李糖脂。
2.8 发酵产物鼠李糖脂的红外光谱分析溶液冷冻干燥成粉末状,与KBr混匀研磨后,测定400-4 000 cm-1波长处的吸收峰。菌株B12所合成的鼠李糖脂的红外吸收光谱图(图 5)显示,3 000-3 650 cm-1处有很宽的吸收峰,为大量的-OH和-CHX不对称伸缩振动;1 030-1 230 cm-1处的峰是C-O-C键伸缩振动形成;1 634 cm-1的峰可能是酰胺Ⅰ带,即C=O伸缩振动吸收峰,也可能是酰胺N-H面内弯曲振动吸收带即酰胺Ⅱ带缔合态,或二者重叠。从以上的各个峰的波数可以确定肽链和烷基链,与鼠李糖脂分子式能够相对应。
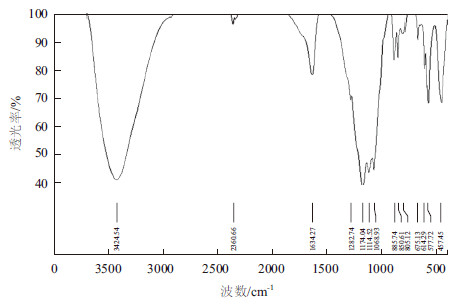 |
| 图 5 P. aeruginosa B12 合成的鼠李糖脂的红外光谱 |
生产成本是生物表面活性剂产品工业化的限制性因素,降低生产成本的方法包括筛选高产鼠李糖脂的菌株,或者选择价格低廉的底物进行生产。Thavasi等[17]以花生油饼为底物,将库彻氏棒状杆菌(Corynebacterium kutscheri)在3 L发酵罐中发酵132 h,得到了6.4 g/L鼠李糖脂。George等[18]菌株P. aeruginosa D以煎炸椰子油为底物,合成了3.6 g/L鼠李糖脂。菌株P. aeruginosa GIM32利用糖蜜酒厂废水为原料,发酵64 h,得到2.6 g/L鼠李糖脂[19]。从矿物切削油中筛选到菌株P. aeruginosa NCAIM,能够利用煎炸废油、葵花籽油和油厂废水为原料进行发酵,鼠李糖脂产量分别达到1.30、0.43和0.40 g/L[20]。菌株P. aeruginosa 67分离于原油污染土壤,该菌株利用葵花籽油能够生产3.4 g/L鼠李糖脂[20]。
Wadekar等[21]报道,铜绿假单胞菌在葡萄糖、甘油、石蜡和菜籽油为碳源的不同培养基中,鼠李糖脂的产量分别为0.5、18.9、2.3和13.5 g/L,从经济性考虑,菜籽油是大规模生产生物表面活性剂鼠李糖脂的首选碳源。菜籽油富含油酸,是适宜生物表面活性剂生产的碳源,与本研究的结果相同。
铜绿假单胞菌合成的鼠李糖脂,其鼠李糖环数量为1个或2个,由于疏水性差异,可以采用薄层色谱法分离,得到迁移率不同的两个斑点。且两种鼠李糖脂的红外吸收图谱相似,具有相同的化学基团[5]。大部分由铜绿假单胞菌生产的都是双鼠李糖脂,很少一部分单鼠李糖脂占主要部分。
4 结论本研究通过CTAB平板法初步筛选和定量测定,获得高产鼠李糖脂菌株P. aeruginosa B12。以菜籽油为碳源条件下,菌株B12的鼠李糖脂产量为26.8 g/L,是工业生产鼠李糖脂的候选菌株。
| [1] | Thavasi R, Nambaru VR, Jayalakshmi S. Biosurfactant production by Pseudomonas aeruginosa from renewable resources[J]. Indian Journal of Microbiology, 2011, 51(1):30-36. |
| [2] | Markus MM, Barbara H, Michaela K, et al. Evaluation of rhamnolipid production capacity of Pseudomonas aeruginosa PAO1 in comparison to the rhamnolipid over-producer strains DSM 7108 and DSM 2874[J]. Applied Microbiology Biotechnology, 2011, 89(7):585-592. |
| [3] | Rahman KS, Rahman TJ, McClean S, et al. Rhamnolipid biosurfactant production by strains of Pseudomonas aeruginosa using low-cost raw materials[J]. Biotechnology Progress, 2002, 18:1277-1281. |
| [4] | Makkar RS, Cameotra SS. Biosurfactant production by microorganisms on unconventional carbon sources-a review[J]. J Surf Det, 1999, 2:237-241. |
| [5] | 包木太, 张金秋, 张娟, 等. 产糖脂类生物表面活性剂菌株鉴定及发酵条件优化[J]. 环境工程学报, 2013, 7(1):365-370. |
| [6] | Nur AM, Amirul AA, Mohamad NM. Rhamnolipid produced by Pseudomonas aeruginosa USM-AR2 facilitates crude oil distillation[J]. The Journal of General and Applied Microbiology, 2012, 58:153-161. |
| [7] | Siegmund I, Wagner F. New method for detecting rhamnolipids excreted by Pseudomonas species during growth on mineral agar[J]. Biotechnology Technology, 1991, 5:265-268. |
| [8] | Pinzon N, Ju LK. Improved detection of rhamnolipid production using agar plates containing methylene blue and cetyl trimethylammonium bromide[J]. Biotechnology Letter, 2009, 31:1583-1588. |
| [9] | Benincasa M, Accorsini FR. Pseudomonas aeruginosa LBI production as an integrated process using the wastes from sunflower-oil refining as a substrate[J]. Bioresource Technology, 2008, 99:3843-3849. |
| [10] | Sambrook J, Fritsch EF, Maniatis T. Molecular cloning:A laboratory manual[M]. 2nd ed. New York:Cold Spring Harbor Laboratory Press, 1989:20-25. |
| [11] | Kutter E. Bacteriophages:biology and applications[J]. Boca Raton, FL:CRC Press, 2004:528. |
| [12] | Zhang JH, Madden TL. Power BLAST:A new network BLAST application for interactive or automated sequence analysis and annotation[J]. Genome Research, 1997, 7(6):649-656. |
| [13] | Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28:2731-2739. |
| [14] | Zhao JF, Wu YJ, Akateh TA, et al. Chemical structures and biological activities of rhamnolipid biosurfactants produced by Pseudomonas aeruginosa M14808[J]. Journal of Chemical and Pharmaceutical Research, 2013, 5(12):177-182. |
| [15] | Dwivedi S, Singh BR, Al KA, et al. Biodegradation of isoproturon using a novel Pseudomonas aeruginosa strain JS-11 as a multi-functional bioinoculant of environmental significance[J]. Journal of Hazardous Materials, 2011, 185(2-3):938-944. |
| [16] | Abalos A, Pinazo A, Infante MR, et al. Physicochemical and antimicrobial properties of new rhamnolipids produced by Pseudomonas aeruginosa AT10 from soybean oil refinery wastes[J]. Langmuir, 2001, 17(5):1367-1371. |
| [17] | Thavasi R, Jayalakshmi S, Balasubramanian T, et al. Biosurfactant production by Corynebacterium kutscheri from waste motor lubricant oil and peanut oil cake[J]. Letters in Applied Microbiology, 2007, 45(1):686-691. |
| [18] | George S, Jayachandran K. Production and characterization of rhamnolipid biosurfactant from waste frying coconut oil using a novel Pseudomonas aeruginosa D[J]. Journal of Applied Microbiology, 2013, 114(2):373-383. |
| [19] | Li AH, Xu MY, Sun W, et al. Rhamnolipid production by Pseudomonas aeruginosa GIM 32 using different substrates including molasses distillery wastewater[J]. Applied Biochemistry Biotechnology, 2011, 163(5):600-611. |
| [20] | Milena GR, Ahmad MA, Eric DZ. Comparative analysis of rhamno- lipids from novel environmental isolates of Pseudomonas aeruginosa [J]. Journal of Surfactants and Detergents, 2013, 16(3):673-682. |
| [21] | Wadekar SD, Kale SB, Lali AM, et al. Microbial synthesis of rhamnolipids by Pseudomonas aeruginosa(ATCC 10145)on waste frying oil as low cost carbon source[J]. Preparative Biochemistry and Biotechnology, 2012, 42(3):249-266. |