卵黄原蛋白(Vitellogenin,Vg)是一种特异存在于非哺乳类性成熟卵生雌性动物血液中的磷糖脂蛋白质,是几乎所有卵生动物卵黄磷蛋白的前体[1]。Vg是Telfer和Williams[2]于1953年用免疫化学方法在惜古比天蚕蛾(Hyalophora cecropia)的血液中首先发现,当时称为雌性特异血蛋白。Pan等[3]将此雌性血蛋白特称Vg,后来把这种在卵生动物的卵黄形成过程中,雌性个体血浆中大量存在的含糖、磷、脂的大分子蛋白定义为卵黄原蛋白。卵黄原蛋白是卵黄发生的关键性物质,是一种重要的生殖蛋白,在卵生动物的生殖、发育等生命过程起重要作用。该蛋白是一类大分子量的糖脂复合蛋白,含有1%-14%的糖类,6%-16%的脂类和大约84%的氨基酸[4]。在大多数昆虫中,卵黄原蛋白在脂肪体中合成,通过受体介导的胞吞作用被发育中的卵母细胞所摄取,为正在发育的胚胎提供氨基酸、碳水化合物、维生素、脂肪等营养和功能性物质,是胚胎发育的重要物质基础[5],能影响昆虫产卵的数量和质量。Vg作为存储蛋白,一直被认为只有营养存储的功能,但是近年来,科学家也发现Vg不仅参与卵母细胞成熟和给胚胎发育提供营养物质外,还有一些其它重要生理功能[6],例如意蜂中Vg参与免疫防御、锌离子转运、寿命调控等多种生物学功能和复杂的新陈代谢过程[7, 8],同时卵黄原蛋白也是检测环境激素的生物指标[9]。
天敌昆虫在生物防治中的应用受到越来越多的关注,生物防治既可以降低农药对环境造成的污染,保护生态坏境,又可以提供安全的农产品,避免高毒农药对人体健康可能产生的某些不良影响。但是由于天敌昆虫繁殖基础等方面的研究薄弱,天敌昆虫在害虫生物防治中的应用受到了限制。卵黄原蛋白对昆虫生殖繁育至关重要,因此卵黄原蛋白研究对天敌昆虫的繁育以及促进害虫生物防治的应用意义重大。
鉴于昆虫卵黄原蛋白的重要性,该蛋白研究成为昆虫生物学和生物化学的研究热点[10, 11, 12, 13],自20世纪60年代以来,对昆虫Vg的研究持续深入,目前已有7个目50多种昆虫的Vg基因序列被克隆,研究主要集中在鳞翅目、双翅目、膜翅目和半翅目[14]。大眼长蝽(Geocoris pallidipennis)隶属半翅目长蝽科,是一种重要的捕食性天敌昆虫,主要捕食蚜虫、螨类、蓟马、鳞翅目的卵和初孵幼虫,是很多农业害虫的重要天敌[15],在害虫生物防治中具有广阔的应用前景。但是国内外对大眼长蝽的研究主要集中在食物、温度、光周期对其发育的影响[16, 17],生物学特性,捕食功能[18, 19]等领域,对其繁殖关键因子——卵黄原蛋白的分子生物学基础研究未见报道。本研究首先利用RT-PCR方法,对大眼长蝽的Vg基因进行克隆并测序,在获取cDNA片段基础上,采用RACE技术得到Vg基因的全长序列,并利用生物信息学方法对Vg的氨基酸序列组成成分、理化性质、信号肽、结构域等方面进行分析,揭示其分子特性,旨在为Vg基因的功能及其在昆虫生殖中的作用机理研究奠定基础。
1 材料与方法 1.1 材料 1.1.1 昆虫材料实验所用大眼长蝽采自北京市小月河,在实验室用人工饲料饲养多代。饲养条件为:温度(25±1)℃,相对湿度(RH)为(70±5)%,光周期L∶D=16 h∶8 h,人工饲料[20]为本实验室研制,分装到石蜡膜制成的袋子中,-20℃保存备用。
1.1.2 菌种、质粒与试剂大肠杆菌DH5α购自北京江晨生物公司;克隆载体pMD19-T、DNA分子量Marker购自TaKaRa公司;琼脂糖凝胶DNA回收试剂盒购自天根公司;反转录试剂盒、Taq聚合酶(HiFi)购自TransGen Biotech公司;SMARTTM RACE cDNA Amplification Kit购自Clontech公司。其他试剂均为分析纯。
1.2 方法 1.2.1 虫体总RNA的提取和cDNA第一链的合成取羽化后7 d的大眼长蝽雌虫放入液氮预冷的1.5 mL离心管中,充分研磨虫体直至研磨成粉末状,加入1 mL的Trizol试剂,按照试剂说明书提取总RNA。提取样品的完整性用1%的琼脂糖凝胶电泳检测。以反转录(TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix)试剂盒合成cDNA第一链;利用3'和5'RACE(SMARTTM RACE cDNA Amplification Kit)试剂盒合成 3'和5'RACE cDNA第一链。
1.2.2 引物设计本实验设计的引物根据NCBI(美国国家生物技术信息中心)数据库中点蜂缘椿象(Riptortus clavatus,U97277)、赤须盲蝽(Trigonotylus caelestialium,AB600673)、大田鳖(Lethocerus deyr-ollei,AB425334)、褐飞虱(Nilaparvata lugens,AB-353856)、琉璃叶蝉(Homalodisca coagulata,DQ11-8408)、斯氏珀蝽(Plautia stali,AB033500)、斯氏珀蝽(Plautia stali,AB033499)、斯氏珀蝽(Plautia stali,AB033498)、油蝉(Graptopsaltria nigrofuscata,AB026848)共9个物种的卵黄原蛋白基因保守序列设计简并引物,根据简并引物扩增结果设计特异性引物,引物序列见表 1。所用引物由北京生物工程有限公司合成。
以cDNA为模板进行PCR扩增,反应体系:10×Buff-erII(Mg2+)5 μL,GpVg1/GpVg2正反向引物(10 μmol/L)各1 μL,dNTPs(2.5 mmol/L)4 μL,Taq-polymerase(HiFi)2.5 U,cDNA模板2 μL,加ddH2O至50 μL。PCR反应条件为:94℃ 3 min;94℃ 30 s,53.8℃/44.3℃ 30 s,72℃ 1 min,35个循环;72℃ 10 min。扩增产物进行1%琼脂糖凝胶电泳检测。根据获得的Vg基因序列设计的GpVg3-GpVg9一系列引物,利用PCR扩增将两目的片段之间的序列克隆并测序。
1.2.3.2 卵黄原蛋白基因cDNA3'末端快速扩增以3'RACE cDNA为模板进行PCR扩增,反应体系:3'RACE cDNA 2.5 μL,GpVg-F 1 μL,UPM 5 μL,10×Advantage 2 PCR Buffer(Clontech)5 μL,dNTP Mix 1 μL,50×Advantage 2 Polymerase Mix(Clontech)1 μL,加ddH2O至50 μL。PCR反应条件为:94℃ 3 min;94℃ 30 s,65℃ 30 s,72℃ 2 min,35个循环;72℃ 10 min。扩增产物进行1%琼脂糖凝胶电泳检测。
1.2.3.3 卵黄原蛋白基因cDNA 5'末端快速扩增以5' RACE cDNA为模板进行PCR扩增,反应体系:5' RACE cDNA 2.5 μL,GpVg-R 1 μL,UPM 5 μL,10×Advantage 2 PCR Buffer(Clontech)5 μL,dNTP Mix 1 μL,50×Advantage 2 Polymerase Mix(Clontech)1 μL,加ddH2O至50 μL。PCR反应条件为:94℃ 3 min;94℃ 30 s,65℃ 30 s,72℃ 1 min,35个循环;72℃ 10 min。扩增产物进行1%琼脂糖凝胶电泳检测。
1.2.3.4 PCR扩增产物的克隆及测序PCR扩增产物用1%琼脂糖凝胶电泳分离,在紫外凝胶成像仪下将目的条带切下,采用琼脂糖凝胶DNA回收试剂盒回收纯化。回收产物与pMD19-T载体连接,连接体系包括:PCR产物 4 μL,pMD19-T载体1 μL,solution I 5 μL,16℃连接过夜,转化大肠杆菌DH5α感受态细胞,涂布于含有Amp的LB培养平板上,于37℃恒温培养过夜。通过蓝白斑和菌落PCR筛选阳性克隆,并送北京生物工程有限公司测序。
1.2.4 序列测定及大眼长蝽卵黄原蛋白系统进化分析利用美国国家生物技术信息中心的Blast工具与GenBank数据库中的其它物种进行同源性序列比对,确定目标产物的可靠性。蛋白质分子量和等电点采用ExPASy:http://www.expasy.org/tools/pi_tool.html软件进行预测;信号肽序列预测采用SignalP 4.0 Server[21]:http://www.cbs.dtu.dk/services/SignalP/软件进行;功能结构域预测采用NCBI的CCD:http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi软件进行;序列比对使用Clustal W软件,系统发育树的构建采用MEGA 5 软件。
1.2.5 卵黄原蛋白在大眼长蝽中的表达分析 1.2.5.1 卵黄原蛋白样品的制备参照龚和等[22]的方法制备,收集大眼长蝽新鲜的卵,用蒸馏水清洗干净,滤纸吸干后用0.4 mol/L NaCl溶液研磨粉碎,4℃ 12 000 r/min离心30 min,取上清,重复此步骤一次,收集上清,按1∶10的比例加入冷重蒸水,使卵黄原蛋白沉淀。置于4℃过夜,5 000 r/min离心20 min,弃上清,沉淀溶于0.4 mol/L NaCl中,低速离心,取上清按照1∶10加入冷重蒸水使卵黄原蛋白沉淀。用考马斯亮蓝法测定卵黄原蛋白的浓度。
1.2.5.2 ELISA法制备标准曲线和测定卵黄原蛋白在大眼长蝽中的表达量ELISA法制备标准曲线 将测定浓度的卵黄原蛋白提取液用包被液按照100、250、500、1 000和2 000倍稀释,每个酶标孔加100 μL,4℃包被过夜,洗涤3次,加300 μL封闭液,37℃温育2 h,洗涤,加入1∶8 000稀释的一抗[23]100 μL,37℃温育2 h,洗涤,加入1∶8 000稀释的酶标二抗100 μL,37℃温育1 h,洗涤,反应孔中加入刚配制的TMB(2 mg TMB 溶于1 mL乙醇中,配制成 2 mg/mL TMB 溶液,取 0.05 mL TMB 溶液、0.95 mL底物缓冲液和 0.001 mL 30% H2O2配成 1 mL TMB 显色液)底物缓冲液100 μL,37℃暗反应15 min,之后加入50 μL 的2 mol/L 硫酸终止反应,读取450 nm下各反应孔的OD值。
ELISA法测定卵黄原蛋白在大眼长蝽中的表达量分别将羽化后3、5、8、10、14、18、22、26和30 d的大眼长蝽雌虫用匀浆缓冲液在冰上研磨粉碎后,4℃,12 000 r/min离心30 min,取上清,再次离心,取上清即为待测样品。按照上述标准曲线制备的步骤测定大眼长蝽羽化后不同时间段卵黄原蛋白的表达量。
2 结果 2.1 大眼长蝽卵黄原蛋白基因cDNA全长克隆PCR扩增产物经琼脂糖凝胶电泳检测,在约250 bp和500 bp处分别有一明亮的条带(图 1-A),PCR产物经连接转化后测序,结果得到大小为248 bp和445 bp的cDNA片段,经Blastx分析,两目的片段与点蜂缘椿象(Riptortus clavatus)的相似性分别达到76%和54%,表明克隆的cDNA片段是大眼长蝽卵黄原蛋白基因的部分序列。根据已经获得的卵黄原蛋白基因序列设计一系列引物,扩增获得两目的片段之间的cDNA序列(图 1-B)。
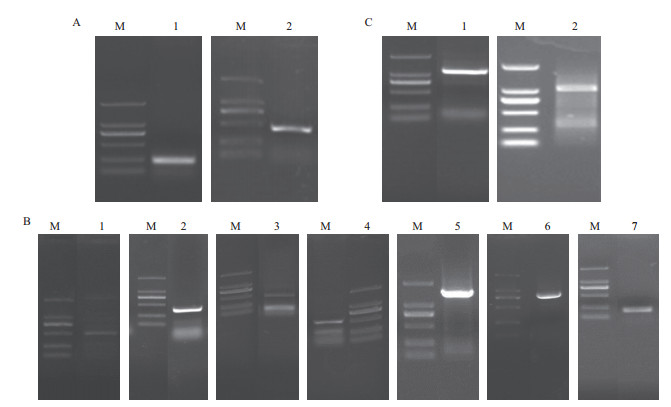 |
| M:DNA Marker DL2000;A:1:简并引物GpVg1扩增产物;2:简并引物GpVg2扩增产物;B:1-7:分别代表引物GpVg3-GpVg9扩增产物;C:1:3' RACE PCR扩增产物;2:5' RACE PCR扩增产物图 1 PCR扩增产物 |
在扩增得到大眼长蝽卵黄原蛋白基因cDNA序列的基础上,采用RACE技术分别获得卵黄原蛋白基因的3'端序列1 049 bp和5'端序列828 bp(图 1-C)。利用DNAman软件进行序列拼接,获得大眼长蝽卵黄原蛋白基因5 667 bp全长序列。卵黄原蛋白cDNA序列的5'起始密码子ATG前有一段长30 bp的非编码区,3'末端有Poly A 多聚核苷酸的尾巴和90 bp的3'非翻译区,表明该cDNA序列的完整性。
2.2 大眼长蝽卵黄原蛋白cDNA序列分析利用DNAman软件对卵黄原蛋白基因序列进行分析,其开放读码框(ORF)为5 547 bp,编码1 848个氨基酸残基(图 2)。根据在线软件SignalP 4.0分析其氨基酸信号肽序列,得到大眼长蝽卵黄原蛋白的N-端从起始密码子开始前19个氨基酸为信号肽,最可能信号肽切割位点在第18位与19位氨基酸之间。预测的分子质量为210.56 kD,等电点为7.02。通过同源性比对,大眼长蝽卵黄原蛋白氨基酸序列与点蜂缘椿象的Vg序列相似度达59%,与斯氏珀蝽的相似度达46%。用Blast分析该卵黄原蛋白氨基酸序列,在N-末端含有Vitellogenin-N结构域,在C-末端含有一个Willerbrand factor type D(VWD)结构域,这些结构域都是卵黄原蛋白所特有的。分析该卵黄原蛋白的氨基酸序列可能在位点RIRR(第387-390位氨基酸)处被切割成两个亚基,在切割位点上下游分别有一个保守的多聚丝氨酸区域。C-末端有5个保守的半胱氨酸残基,在其上游有GLAG保守氨基酸基序,DGYR基序位于GLAG基序上游第18个氨基酸残基处。
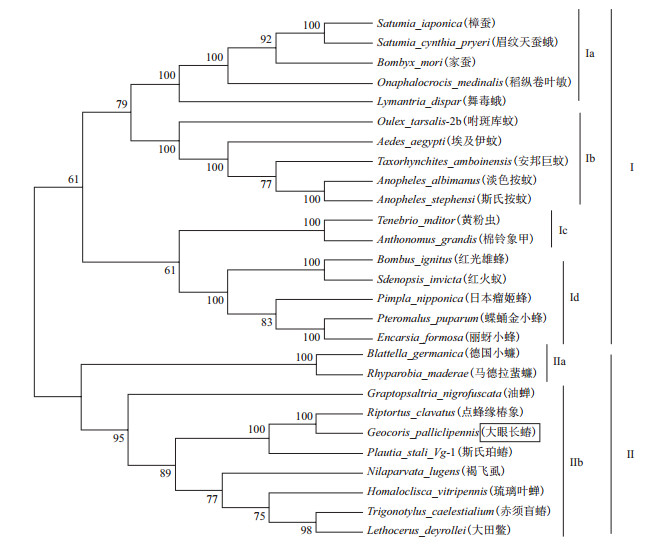 |
| 图 2 不同物种间卵黄原蛋白的系统进化分析 |
在GenBank中搜索昆虫纲已克隆的Vg序列,应用MEGA5.0软件采用邻接法对27个物种的卵黄原蛋白进行系统进化分析,以1 000次回抽的置信度作置信分析,结果(图 2)显示,27个物种的卵黄原蛋白分为I、II两组,分别代表全变态昆虫、半变态昆虫,第一组又分为4个亚组Ia、Ib、Ic和Id,分别代表双翅目、鳞翅目、鞘翅目和膜翅目;第二组分为2个亚组IIa、IIb,分别代表网翅目、半翅目。大眼长蝽卵黄原蛋白基因属于亚组IIb,与点蜂缘椿象的亲缘关系最近。
2.4 ELISA法制备标准曲线和测定卵黄原蛋白在大眼长蝽中的表达量 2.4.1 ELISA方法制作的标准曲线图 3显示,卵黄原蛋白浓度与OD值成线性关系(R2=0.950 3),可以用于测定羽化后不同时间卵黄原蛋白表达量。
 |
| 图 3 卵黄原蛋白标准曲线 |
羽化后不同时间的大眼长蝽雌虫卵黄原蛋白如图 4所示,卵黄原蛋白表达在羽化后第3天就可以检测到,随着发育时间的增加,卵黄原蛋白含量也随之增加,在羽化后第22天达到最大值,然后开始下降。
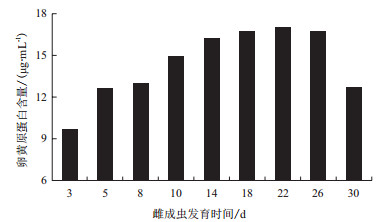 |
| 图 4 大眼长蝽雌虫羽化后不同时期卵黄原蛋白的含量 |
现有研究表明,卵黄原蛋白是一种重要的营养物质,在卵生雌性动物胚胎发育中起关键性作用,是卵母细胞生长发育所需营养物质的主要来源。卵黄原蛋白因其所具有的特性,备受昆虫生物学和生物化学研究领域的关注。但是目前在GenBank数据库中公布的半翅目卵黄原蛋白基因序列,还未见大眼长蝽卵黄原蛋白(Vg)基因报道。本研究克隆获得了大眼长蝽卵黄原蛋白基因,该基因是半翅目昆虫中的一种新基因,填补了半翅目长蝽科Vg分子特性的信息,有助于揭示Vg基因在长蝽科昆虫代谢、生殖及进化中的生理功能。
本研究利用RT-PCR及RACE技术,得到了大眼长蝽卵黄原蛋白基因cDNA全序列,该序列全长5 667 bp,包括30 bp的5'非翻译区(UTR),编码1 848个氨基酸残基的开放阅读框,90 bp的3'非翻译区(UTR)。预测卵黄原蛋白的分子质量为210.56 kD,等电点为7.02。氨基酸保守结构域分析该蛋白在488-822氨基酸残基处有一个Vitellogenin-N结构域,C-末端的1 598-1 762位氨基酸残基处有VWD结构域,这些结构域为卵黄原蛋白基因所特有的,也与脂质转移活动密切相关[24]。
同时,大眼长蝽卵黄原蛋白基因序列分析表明,从甲硫氨酸开始的前19个氨基酸为信号肽,而一般昆虫卵黄原蛋白N-末端的15-18个氨基酸为信号肽[25]。本研究结果也揭示大眼长蝽Vg氨基酸序列中有RIRR的酶切位点,将卵黄原蛋白切割成大小两个亚基,该酶切位点识别的氨基酸序列在昆虫中较为保守,一般为(R/K)X(R/K)R或者RXXR[26]。尹志亮等[27]研究柳蚕卵黄原蛋白也发现该氨基酸残基在RSRR酶切位点处被切割成大小两个亚基,但Nose等[28]研究寄生蜂卵黄原蛋白基因得出其保守的RXXR位点突变为LYRR,并且不被切割。大眼长蝽卵黄原蛋白氨基酸序列C-末端区域存在保守的GLAG基序,该基序不同于大多数昆虫的GL/ICG[29, 30],而与点蜂缘椿象[31]的保守基序完全一致。其他学者研究表明红光雄蜂[13]和大草蛉[32]的Vg氨基酸序列在GL/ICG基序的下游有9个保守的半胱氨酸残基,天蚕、蓖麻蚕中GL/ICG基序下游的保守半胱氨酸残基数为6,而家蚕中为7,而本研究结果显示大眼长蝽卵黄原蛋白氨基酸序列在GLAG基序的下游只有5个保守的半胱氨酸残基,可见不同物种保守的半胱氨酸残基数目有差异。此外,大眼长蝽DGYR位于GLAG基序上游18个氨基酸残基处,这与目前已知的大部分昆虫的DGXR基序相同[33]。
Blast比对和系统发育树的分析发现,大眼长蝽卵黄原蛋白基因氨基酸序列与昆虫卵黄原蛋白有较高的相似度,它与半翅目昆虫相似度达到33%-59%。其中与点蜂缘椿象(Riptortus clavatus)、斯氏珀蝽(Plautia stali)、琉璃叶蝉(Homalodisca vitripennis)、褐飞虱(Nilaparvata lugens)等的相似度分别为:59%、46%、37%和34%。大眼长蝽卵黄原蛋白与点蜂缘椿象具有较高的相似度,与这两种昆虫同属于半翅目有关。
用ELISA方法对大眼长蝽雌虫卵黄原蛋白的表达分析显示,大眼长蝽雌虫中卵黄原蛋白含量在羽化后第3天即可检测到,并随着发育时间的延长,卵黄原蛋白含量逐渐升高,在羽化后第22天达到高峰,而后逐渐下降。观察也发现大眼长蝽一般在羽化后7-10 d开始产卵,可见,大眼长蝽雌虫并不是在卵黄原蛋白合成的初期开始产卵,只有卵黄原蛋白累积到一定程度才开始产卵,产卵量会随着卵黄原蛋白含量的增加而增加,该结果表明大眼长蝽雌虫卵黄原蛋白的表达量与大眼长蝽产卵量紧密相关。
昆虫的生殖力是衡量种群生存与繁殖的重要指标[34],卵黄原蛋白是昆虫繁殖的关键因子,尽管不同昆虫卵黄原蛋白合成的调控方式有差异,而卵黄原蛋白的合成与激素[35]及相关基因特别是Vg基因紧密关联。本研究克隆得到的大眼长蝽卵黄原蛋白基因,将有利于进一步研究基因对天敌昆虫大眼长蝽卵黄原蛋白的合成调控及促进大眼长蝽大量繁殖的关键技术。
4 结论本研究成功克隆了大眼长蝽卵黄原蛋白基因,该基因包含有高度保守的Vg-N和VWD结构域,同时系统进化树分析结果表明大眼长蝽Vg与点蜂缘椿象的Vg亲缘关系密切。本研究结果也揭示了大眼长蝽雌虫卵黄原蛋白的表达量与大眼长蝽产卵量紧密相关。
| [1] | Utarabhand P, Bunlipatanon P. Plasma vitellogenin of grouper(Epinephelus malabaricus):isolation and properties[J]. Comparative Biochemistry Physiology, 1996, 115(2):101-110. |
| [2] | Telfer WH. Immunological studies of insect metamorphosis. Ⅱ. The role of a sex-limited blood protein in egg formation by the Cecropia silkworm[J]. Journal of General Physiology, 1954, 37(4):539-558. |
| [3] | Pan ML, Bell WJ, Telfer WH. Vitellogenic blood protein synthesis by insect fat body[J]. Science, 1969, 165(3891):393-394. |
| [4] | 董胜张, 叶恭银, 刘朝良. 昆虫卵黄原蛋白分子进化的研究进展[J]. 昆虫学报, 2008, 11:1196-1209. |
| [5] | 张年国, 张颖, 孙大江, 曲秋芝. 卵黄蛋白原的发生、结构及功能研究现状[J]. 水产学杂志, 2007, 20(1):97-106. |
| [6] | Piulachs MD, Guidugli KR, Barchuk AR, et al. The vitellogenin of the honey bee, Apis mellifera:structural analysis of the cDNA and expression studies[J]. Insect Biochemistry and Molecular Biology, 2003, 33:459-465. |
| [7] | Amdam GV, Simões ZLP, Hagen A, et al. Hormonal control of the yolk precursor vitellogenin regulates immune function and longevity in honeybees[J]. Experimental Gerontology, 2004, 39:767-773. |
| [8] | Gro VA, Kari N, Arne H, et al. Social exploitation of vitellogenin[J]. Proceeding of the National Academy of Scienses of the United States of America, 2003, 100:1799-1802. |
| [9] | 汝少国, 潘宗保, 田华. 鱼类卵黄原蛋白研究进展[J]. 海洋湖沼通报, 2012(2):22-32. |
| [10] | Hatakeyama M, Sawa M, Oishi K. Ovarian development and vitellogenesis in the sawfly, Athalia rosae ruficornis Jakovlev(Hymenoptera, Tenthredinidae)[J]. Invertebrate Reproduction and Development, 1990, 17(3):237-245. |
| [11] | Raikhel AS, Brown MR, Belles X. Hormonal control of reproductive process[M]. Oxford:Comprehensive Molecular Insect Science, 2005, 433-491. |
| [12] | Swevers L, Raikhel AS, Sappington TW, et al. Vitellogenesis and post-vitellogenic maturation of the insect ovarian follicle[M]. Oxford:Comprehensive Molecular Insect Science, Elsevier Ltd., 2005:87-155. |
| [13] | Shaul R, Shmuel P, Carmen S, et al. Complete sequence of Litopenaeus vannamei(Crustacea:Decapoda)vitellogenin cDNA and its expression in endocrinologically induced sub-adult females[J]. General and Comparative Endocrinology, 2006, 145(1):39-50. |
| [14] | 李继莲, 吴杰, 彩万志, 彭文君. 红光熊蜂卵黄原蛋白基因的cDNA全长序列克隆和表达分析[J]. 应用昆虫学报, 2012, 49(5):1132-1139. |
| [15] | 彩万志, 庞雄飞, 花保祯. 普通昆虫学[M]. 北京:中国农业大学出版社, 2001:413. |
| [16] | Decker KB, Yeargan KV. Seasonal phenology and natural enemies of the squash bug(Hemiptera:Coreidae)in Kentucky[J]. Environmental Entomology, 2008, 37(3):670-678. |
| [17] | Mansfield S, Scholz B, Armitage S, et al. Effects of diet, temperature and photoperiod on development and survival of the big eyed bug, Geocoris lubra[J]. Biology, 1951, 28(2):173-180. |
| [18] | Tillman G, Lamb M, Mullinix JB. Pest insects and natural enemies in transitional organic cotton in Georgia[J]. Journal of Entomological Science, 2009, 44:11-23. |
| [19] | Torres JB, Silva-Torres CS, Ruberson JR. Effect of two prey types on life-history characteristics and predation rate of Geocoris floridanus(Heteroptera:Geocoridae)[J]. Environmental Entomology, 2004, 33:964-974. |
| [20] | Liu FJ, Zeng FR. The influence of nutritional history on the functional response of Geocoris pallidipennis to its prey, Myzus persicae[J]. Bulletin of Entomological Research, 2014, 7:1-5. |
| [21] | Petersen TN, Brunak S, Von Heijne G, et al. SignalP 4. 0:discriminating signal peptides from transmembrance regions[J]. Nature Methods, 2011, 8:785-786. |
| [22] | 龚和, 翟启慧, 魏定义. 七星瓢虫的卵黄发生:卵黄原蛋白的发生和取食代饲料的影响[J]. 昆虫学报, 1980, 23:252-259. |
| [23] | 刘丰姣. 食物对大眼长蝽营养生理生化及行为影响的研究[D]. 北京:中国农业科学院, 2013. |
| [24] | Smolenaars MMW, Madsen O, Rodenburg KW, et al. Molecular diversity and evolution of the large lipid transfer protein superfamily[J]. The Journal of Lipid Research, 2007, 48(3):489-502. |
| [25] | 刘朝良, 董胜张, 王磊, 等. 昆虫卵黄原蛋白(Vg)分子生物学研究进展[C]. 食品安全的理论与实践——安徽食品安全博士科技论坛论文集, 2005. |
| [26] | Matthews DJ, Goodman LJ, Gorman CM, et al. A survey of furin substrate specificity using substrate phage display[J]. Protein Science, 1994, 3:1197-1205. |
| [27] | 尹志亮, 刘朝良, 邵引刚, 李锋. 柳蚕卵黄原蛋白cDNA的克隆及序列分析[J]. 激光生物学报, 2007, 16(5):552-558. |
| [28] | Nose Y, Lee JM, Ueno T, et al. Cloning of cDNA for vitellogenin of the Parasitoid Wasp. Pimpla nipponica(Hymenopetera:Apocrita:Ichneumonidae):Vitellogenin Primary Structure and Evolutionary Considerations[J]. Insect Biochemistry and Molecular Biology, 1997, 27(1):47-56. |
| [29] | Lee JM, Nishimori Y, Hatakeyama M, et al. Vitellogenin of the cicada Graptopsaltria nigrofuscata(Homoptera):analysis of its primary structure[J]. Insect Biochemistry and Molecular Biology, 2000, 30(1):1-7. |
| [30] | Piulachs MD, Guidugli KR, Barchuk AR, et al. The vitellogenin of the honey bee, Apis mellifera:structural analysis of the cDNA and expression studies[J]. Insect Biochemistry and Molecular Biology, 2003, 33(4):459-465. |
| [31] | Hirai M, Watanabe D, Kiyota A, et al. Nucleotide sequence ofvitellogeninmRNA in the bean bug, Riptortusclavatus:analysis of processing in the fat body and ovary[J]. Insect Biochemistry and Molecular Biology, 1998, 28(8):537-547. |
| [32] | 刘昌燕. 大草蛉卵黄原蛋白基因功能研究[D]. 北京:中国农业科学院, 2013. |
| [33] | Tufail M, Takeda M. Molecular characteristics of insect vitellogenins[J]. Journal of Insect Physiology, 2008, 54:1447-1458. |
| [34] | 王利娜. 龟纹瓢虫幼虫人工饲料的研究[D]. 北京:中国农业科学院, 2008. |
| [35] | Zeng F, Shu S, Park YI, et al. Vitellogenin and egg production in the moth, Heliothis virescens[J]. Archives of Insect Biochemistry and Physiology, 1997, 34(3):287-300. |





