b. Key Laboratory of Plant Resources and Biodiversity of Jiangxi Province, Jingdezhen University, Jingdezhen 333400, Jiangxi, China;
c. Germplasm Bank of Wild Species & Yunnan Key Laboratory of Crop Wild Relatives Omics, Kunming Institute of Botany, Chinese Academy of Sciences, Kunming 650201, Yunnan, China;
d. Department of Biology, Faculty of Science, Yasouj University, Yasouj 75918-74934, Iran;
e. University of Chinese Academy of Sciences, Beijing 100049, China;
f. Department of Forestry and Environmental Resources, North Carolina State University, Raleigh, NC 27695, United States;
g. Department of Forestry and Natural Resources, Faculty of Agriculture, Kabul University, Kabul D-1006, Afghanistan;
h. National Academy of Science of the Kyrgyz Republic, Jalal-Abad Scientific Center, Jalal-Abad 715600, Kyrgyzstan;
i. Laboratory of Molecular Genetics, Institute of Plant Biology and Biotechnology, Almaty 050040, Kazakhstan
Genetic diversity, a key component of biodiversity, provides essential information on adaptation and the evolutionary capacity of plant species (Schaberg et al., 2008; Jump et al., 2009; Potter et al., 2017). The genetic diversity of forest trees is severely affected by increased harvesting (nuts, barks, timbers, etc.) and human interference (overexploitation, overgrazing, etc.), resulting in habitat fragmentation and considerable reduction of forestland area (MacDougall et al., 2013; Gaisberger et al., 2020). Consequently, genetic variation provides significant insights into conservation efforts and efficient utilization of plant resources (Loo et al., 2014). To date, the genetic diversity of endangered species has been widely studied, e.g., Taxus wallichiana (Liu et al., 2013), Bertholletia excelsa (Baldoni et al., 2020), and Pinus koraiensis (Wei et al., 2022). However, few studies have examined genetic diversity in tree species that face long-term and persistent anthropogenic pressures, particularly those planted in forest and agroforestry systems (Dawson et al., 2009; Gaisberger et al., 2020).
Juglans regia L. (Juglandaceae), popularly known as the common walnut, is a deciduous tree species typically distributed and/or cultivated in mountainous areas across subtropical and temperate regions (Lu et al., 1999; Yan et al., 2024). Its main distribution contains several countries from the Balkans eastward to Asia, including China, Iran, Kazakhstan, Afghanistan, and Pakistan (McGranahan and Leslie, 2012; Zohary et al., 2012). Presently, J. regia is cultivated in more than 60 countries, China being the leading world producer (with 31% of the total harvested) (FAOSTAT, 2023), and is one of the top three most-consumed nuts in the world (Avanzato et al., 2014; Ebrahimi et al., 2016). However, many ecosystems with walnut tree forests are seriously impacted by human activity (Beer et al., 2008; Lenda et al., 2017; Liu et al., 2023; Yan et al., 2024). In addition, post-destruction recovery of walnut forests can be challenging due to the trees' slow growth and reproduction, and late fruit ripening, which result in complex regeneration under natural conditions. Conservation efforts, including the effective use of plant germplasm, rely on a clear understanding of the population dynamics of a species, including its distribution, differentiation, as well as the factors that affect its genetic diversity (McNeely et al., 1990; Salgotra and Chauhan, 2023).
Due to the unique and fragile biodiversity in Central Asia, the region is considered as Mountains of Central Asia biodiversity hotspot (Mittermeier et al., 2004; Foggin et al., 2021; Liu et al., 2022). Because of the specific geomorphologic landscapes caused by the several Asian mountains (Tien-Shan, Western Himalaya, Hindu-Kush, and Karakoram Mountains) and Pamir Plateaus (Liu et al., 2022), Central Asia has an arid and semi-arid climate (Lioubimtseva and Henebry, 2009; Wang and Zhang, 2022). It is suggested that the diversity and spread of different species in this area were associated with the climatic transition characterized by enhanced aridification and global cooling since the late Eocene (Lioubimtseva and Henebry, 2009). In addition, Central Asia is also recognized as a center of origin of numerous fruit tree species, like the common walnut (Juglans regia) (Vavilov et al., 1992; Gaisberger et al., 2020), apricot (Prunus armeniaca) (Groppi et al., 2021), and wild apple (Malus sieversii) (Zhang et al., 2020). Regarding to walnut, previous studies have evaluated both genetic diversity and population structure of J. regia in some regions of Central Asia (Pollegioni et al., 2014; Aradhya et al., 2017; Roor et al., 2017; Mapelli, 2018; Torokeldiev et al., 2018; Gaisberger et al., 2020; Magige et al., 2022; Khan et al., 2023; Yan et al., 2024). However, despite the extensive distribution of J. regia in this area, there has been no comprehensive investigation into the genetic variation of this species across Central Asia. This is particularly true for walnuts in Afghanistan and the Xinjiang Autonomous Region of China.
In this study, we characterized the genetic diversity and population structure of 46 populations of Juglans regia across Central Asia. Specifically, we used 31 polymorphic microsatellite loci to genotype a total of 1082 individuals. Our findings will help develop genetically informed germplasm protection and management strategies as well as effective utilization of J. regia.
2. Materials and methods 2.1. Sample collectionFrom 2016 to 2018, we formed a walnut research network (see the author list) to collect walnut samples across Central Asia. This collection, which encompasses the whole range of walnuts, consists of 1082 Juglans regia individuals from 46 natural populations (Fig. 1 and Table S1) from six countries in the region: Afghanistan (4), China (21), India (3), Kyrgystan (4), Kazakhastan (2), and Pakistan (12). We collected a minimum of ten individuals per population, except in population 1 (from Ziarat in Pakistan). To avoid consanguinity, we only sampled neighboring trees that were at least 50 m apart. Within each population, fresh, healthy leaves were sampled and preserved in silica gel for DNA isolation. Voucher specimens have been deposited at the Herbarium of Kunming Institute of Botany, Chinese Academy of Sciences (KUN). Information on these populations can be found in Table S1.
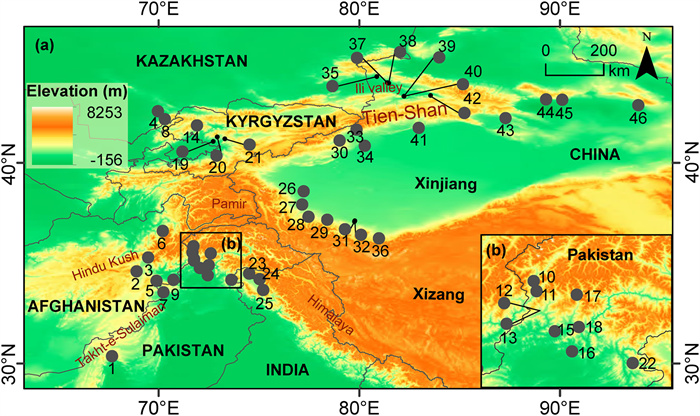
|
| Fig. 1 Geographical location of the 46 Juglans regia populations (see Table S1 for detailed information). (a) Geographical location of all populations in China, Kazakhstan, Kyrgyzstan, Afghanistan, Pakistan, and India. (b) Right-lower inset shows the geographical distribution of nine populations in Pakistan. |
Genomic DNA was extracted from approximately 20 mg of silica gel dried leaves of Juglans regia using a modified cetyltrimethylammonium bromide (CTAB) method (Doyle and Doyle, 1987; Liu and Gao, 2011). Total DNA was dissolved in 50–100 mL 1 × TAE (pH = 8.0) and assessed by electrophoresis on 1.0% agarose gels. DNA concentration and quality were evaluated using a NanoDrop-1000 Spectrophotometer (Thermo Fisher Scientific, Wilmington, DE, USA). All DNA samples were diluted to a final concentration of 30–50 ng/uL pending PCR procedures.
2.3. PCR amplification and SSR genotypingA total of 1082 individuals from the 46 populations was genotyped using 31 nuclear microsatellite primer pairs following methods from our previous studies (Xu et al., 2020; Wambulwa et al., 2022; Xiahou et al., 2023). Genotype data of 12 populations from Pakistan and three populations from India were retrieved from our previous studies (Magige et al., 2022; Yan et al., 2024). The genotype data of both studies are publicly available from the publisher's website. For new samples, oligonucleotide tails were attached to the 5′ ends of all the forward primers and were labeled with FAM, HEX, or TAMRA fluorescent dyes (Optimus Bio., Kunming, China). PCR amplification was carried out in a final volume of 20 μL reaction containing 60–100 ng genomic DNA, 0.5 μM of each primer, 10 μL 2× Taq PCR MasterMix [Tiangen, 0.1 U Taq Polymerase/μL, 0.5 mM dNTP each, 20 mM Tris–HCl (pH = 8.3), 100 mM KCl, 3 mM MgCl2]. PCR reaction conditions were as follows: an initial denaturation for 5 min at 95 ℃, followed by 30 cycles of denaturation at 95 ℃ for 30 s, renaturation at the specified annealing temperature (Tm) of each primer for 30 s, elongation at 72 ℃ for 40 s, and a final extension step at 72 ℃ for 5 min. To genotype the samples, a multiplex PCR system was used on a Veriti® 96-Well Thermo-Cycler (Applied Biosystems, Foster City, California, USA), following Xiahou et al. (2023). Amplified fragments were initially visualized and assessed on 1.0% agarose gels with 1× TAE buffer, and the successful amplification products were separated on an ABI3730XL DNA Analyzer (Applied Biosystems, Foster City, CA, USA) using GeneScan 500 LIZ (Applied Biosystems, Foster City, CA, USA) as an internal size standard.
2.4. Data analysisAlleles of diploid genotypes were scored manually using GeneMarker v.4.0 (SoftGenetics, State College, Pennsylvania, USA). An allelic ladder of walnuts was used to calibrate the size of alleles among different batches of PCR products (Xiahou et al., 2023). Micro-Checker v.2.2.3 (Van Oosterhout et al., 2004) was employed to examine the frequencies of null alleles at each locus in each population with 1000 randomizations. We detected Hardy–Weinberg equilibrium (HWE) and linkage disequilibrium (LD) between loci in Arlequin v.3.5 (Excoffier and Lischer, 2010) using Bonferroni correction. We used GenAlEx v.6.5 (Peakall and Smouse, 2006) to estimate the following genetic diversity parameters: number of alleles (NA), the effective number of alleles (NE), observed heterozygosity (HO), expected heterozygosity (HE), Shannon's information index (I), fixation index (F), gene flow (Nm), inbreeding coefficient (FIS), unbiased expected heterozygosity (uHE) across loci, Nei's genetic distances between populations, percentage of polymorphic loci (PPL), and private alleles (NP). Allelic richness (AR) and the polymorphic information content (PIC) were calculated by the hierfstat R package (Goudet, 2005) and PICcalc v.0.6 (Nagy et al., 2012), respectively. Principal co-ordinates analysis (PCoA) and analysis of molecular variance (AMOVA) (1000 permutations) were conducted in GenAlEx (Peakall and Smouse, 2006).
BayesAss v.3.0.4 (Wilson and Rannala, 2003) was used to compute recent migration rate between populations. In addition, the presence of population bottlenecks was computed using BOTTLENECK v.1.2.02 (Piry et al., 1999). A Two-Phase Mutation Model (TPM) was applied for the Wilcoxon (Cornuet and Luikart, 1996) and standardized differences tests. The parameter settings included 30% multi-step changes and a probability of 70%. The analysis was performed with 1000 permutations.
A Bayesian clustering algorithm in STRUCTURE v.2.3.4 (Pritchard et al., 2000) was utilized to assign each individual to a specific genetic cluster, determined by the pre-defined number of clusters (K). Each K value ranged from 1 to 10 and was repeated for 20 simulations. For each run, the burn-in period and Markov chain Monte Carlo (MCMC) generations were 100, 000 and 1, 200, 000, respectively. All iterations were performed with the admixture model and correlated allele frequencies. The optimal K value was identified using the delta K method in STRUCTURE HARVESTER (Earl and vonHoldt, 2012). According to Wambulwa et al. (2016), an individual is assigned to a specific genetic cluster that is classified as a pure genetic group when the membership percentage is more than 80% (Q ≥ 0.80), whereas those exhibiting a low membership coefficient (0.20 < Q < 0.80) are defined as 'admixture.' Genetic relationships among individuals were determined in Populations v.1.2.31 (Langella, 1999) using the unrooted neighbor-joining (NJ) tree method based on the DA genetic distances calculated by Microsatellite Analyzer (MSA) v.4.05 (Dieringer and Schlötterer, 2003). The results were visualized using the ggtree R package (Yu et al., 2016). Pairwise Wright's FST values between populations were computed using Arlequin v.3.5 (Excoffier and Lischer, 2010), and a heatmap of DA and FST estimates between various populations was visualized by the ggplot2 R package (Wickham, 2016).
To analyze the impact of geographical isolation on patterns of genetic differentiation (FST), we conducted a Mantel test with the whole data and each group using the vegan R package (Oksanen et al., 2013). P-value was assessed through 1000 permutations. The geographic distance and FST matrix were generated using the geosphere R package (Hijmans et al., 2022) and Arlequin v.3.5 (Excoffier and Lischer, 2010), respectively. Moreover, to investigate correlations between environmental variables and genetic diversity (HE) of Juglans regia, we evaluated the relationship between genetic diversity of walnut populations and annual mean temperature, annual mean precipitation, and elevation. Climatic data were downloaded from the WorldClim v.2.0 dataset (http://www.worldclim.org/bioclim), encompassing recent climate data (ca. 2000–2018), with a grid size of 30 arc-seconds (Fick and Hijmans, 2017).
Finally, the existence of genetic barriers among populations was evaluated using Monmonier's maximum difference algorithm implemented in Barrier v.2.2 (Manni et al., 2004). Microsatellite Analyzer (MSA) generated the pairwise genetic distance (DA) with a bootstrap of 100 and geographical coordinates from Table S1. These matrices were then used to create a Delaunay triangulation net for linking neighboring populations and subsequently projecting the corresponding Voronoi tessellation. Every edge of the Voronoi polygon was connected based on Nei's genetic distance (DA).
3. Results 3.1. Genetic diversity across SSR markers and populationsOur findings revealed that the 31 SSR markers were polymorphic. Micro-Checker analysis did not detect any evidence of scoring errors attributable to stuttering or significant allele dropout for any of the loci. A total of 39 out of 465 possible pairwise comparisons exhibited significant linkage disequilibrium (P < 0.05) (Table S2). A random association (linkage equilibrium) between all the pairs of alleles was observed using Bonferroni correction. Only three loci (JR05, JM5446, and JS02) showed a departure from HW equilibrium, likely due to monomorphisms in some populations (Table S3). Hence, all data was used for downstream analyis. Genetic diversity parameters are presented for each locus in Table S4. All 31 nuclear SSR loci yielded 226 alleles overall. The average number of alleles throughout marker loci was 7.00, ranging from 5 for loci JR06, JR07, JR08, JR09, and CUJRD102 to 12 alleles for locus JS12 (Table S4). At the same time, the effective number of alleles (NE) varied from 1.03 (JM5446) to 4.94 (BFU-Jr38), with an average of 2.62. The mean observed heterozygosity (HO) and expected heterozygosity (HE) were 0.44 and 0.56, respectively. The average Shannon's information index (I) and unbiased expected heterozygosity (uHE) were 1.09 and 0.56, respectively. The inbreeding coefficient (FIS) showed negative and significant values in loci JR02, JR03, JR04, JR06, JR07, JR08, JS02, JS04, JS14, and JS22 (Table S4), indicating heterozygosity excess.
At the population level, the genetic diversity parameters differed across populations of Juglans regia (Table 1). Total genetic diversity indices were typically moderate (NE = 2.22, HO = 0.46, HE = 0.48). The average number of observed alleles (NA), the effective number of alleles (NE), and allelic richness (AR) values for the whole dataset were 3.33, 2.22, and 3.04, respectively. The mean gene flow (Nm) between populations was 2.08. The maximum level of Nm was observed between populations 31 and 32 (244.85), while the minimum level was found between populations 39 and 45 (0.32) (Table S5). Populations 1 and 39 were varied considerably across all genetic diversity values. Particularly, populations 1, 15, 18, 22, and 25 exhibited high values of genetic diversity, whereas populations 39 and 40 showed relatively low values (Table 1). The fixation index (F) ranged from −0.09 (population 1) to 0.16 (population 36), with a mean of 0.03. The low value of the fixation index infers the deficiency of heterozygosity in the populations of J. regia.
| Code | PID | N | NP | NA | NE | I | HO | HE | uHE | F | PPL (%) | AR | FIS | TPM | Standardized difference test | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ZTR | 1 | 9 | 2 | 3.61 | 2.71 | 1.05 | 0.64 | 0.59 | 0.62 | -0.09 | 1.00 | 3.65 | -0.03 | 2.89 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KPR | 2 | 22 | 0 | 3.71 | 2.39 | 0.94 | 0.51 | 0.52 | 0.54 | 0.02 | 0.97 | 3.31 | 0.05 | 2.28 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PBR | 3 | 22 | 2 | 3.81 | 2.44 | 0.95 | 0.53 | 0.52 | 0.54 | 0.01 | 0.97 | 3.31 | 0.02 | 1.70 | 0.04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| TUR | 4 | 20 | 0 | 3.84 | 2.61 | 0.98 | 0.47 | 0.52 | 0.54 | 0.09 | 0.97 | 3.56 | 0.13 | 1.90 | 0.03 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| UKR | 5 | 19 | 0 | 3.87 | 2.31 | 0.95 | 0.52 | 0.52 | 0.53 | -0.02 | 0.97 | 3.62 | 0.02 | 0.54 | 0.30 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| BKR | 6 | 22 | 0 | 3.68 | 2.28 | 0.90 | 0.48 | 0.49 | 0.50 | 0.00 | 0.97 | 3.32 | 0.03 | 0.62 | 0.27 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KMR | 7 | 19 | 1 | 3.74 | 2.48 | 1.00 | 0.56 | 0.56 | 0.58 | -0.01 | 0.97 | 3.54 | 0.02 | 2.92 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SUR | 8 | 20 | 0 | 3.52 | 2.50 | 0.94 | 0.50 | 0.53 | 0.54 | 0.04 | 0.97 | 3.33 | 0.07 | 3.45 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NSR | 9 | 22 | 1 | 4.19 | 2.59 | 1.03 | 0.55 | 0.56 | 0.57 | 0.02 | 0.97 | 3.58 | 0.03 | 1.42 | 0.08 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CLR | 10 | 20 | 1 | 3.55 | 2.57 | 0.96 | 0.57 | 0.54 | 0.55 | -0.06 | 0.94 | 3.31 | -0.03 | 3.62 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HCR | 11 | 10 | 1 | 3.94 | 2.71 | 1.06 | 0.57 | 0.58 | 0.61 | 0.02 | 0.97 | 3.92 | 0.06 | 1.37 | 0.09 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| DUR | 12 | 20 | 3 | 4.52 | 2.64 | 1.08 | 0.58 | 0.57 | 0.59 | -0.01 | 0.97 | 3.80 | 0.01 | 0.45 | 0.33 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| DIR | 13 | 20 | 0 | 3.19 | 2.33 | 0.90 | 0.51 | 0.52 | 0.53 | 0.03 | 0.97 | 3.00 | 0.05 | 3.72 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| TYR | 14 | 10 | 0 | 3.16 | 2.24 | 0.86 | 0.50 | 0.49 | 0.52 | -0.04 | 0.97 | 3.10 | 0.03 | 1.65 | 0.05 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HDR | 15 | 20 | 4 | 4.35 | 2.78 | 1.13 | 0.55 | 0.61 | 0.62 | 0.08 | 0.97 | 4.06 | 0.12 | 2.30 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HSR | 16 | 22 | 3 | 4.19 | 2.57 | 1.04 | 0.51 | 0.57 | 0.58 | 0.10 | 0.97 | 3.78 | 0.13 | 1.64 | 0.05 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| STR | 17 | 11 | 0 | 3.65 | 2.64 | 1.02 | 0.56 | 0.57 | 0.61 | 0.00 | 0.97 | 3.62 | 0.10 | - | - | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SHR | 18 | 14 | 1 | 3.94 | 2.78 | 1.07 | 0.61 | 0.58 | 0.60 | -0.02 | 0.97 | 3.78 | -0.01 | 2.59 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KTR | 19 | 10 | 0 | 2.61 | 1.94 | 0.69 | 0.39 | 0.42 | 0.44 | 0.05 | 0.87 | 2.70 | 0.11 | 2.11 | 0.02 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ASR | 20 | 20 | 0 | 2.81 | 2.04 | 0.71 | 0.41 | 0.42 | 0.43 | 0.01 | 0.90 | 2.60 | 0.03 | 2.26 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KLR | 21 | 20 | 0 | 2.58 | 1.96 | 0.70 | 0.45 | 0.43 | 0.44 | -0.04 | 0.94 | 2.46 | -0.04 | 3.22 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| KAR | 22 | 20 | 0 | 4.13 | 2.79 | 1.09 | 0.63 | 0.59 | 0.61 | -0.06 | 1.00 | 3.68 | -0.04 | 2.79 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| AJR | 23 | 19 | 1 | 3.68 | 2.51 | 0.96 | 0.53 | 0.54 | 0.55 | 0.01 | 1.00 | 3.28 | 0.04 | 2.49 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| BR | 24 | 27 | 0 | 4.13 | 2.59 | 1.04 | 0.48 | 0.56 | 0.57 | 0.11 | 0.97 | 3.68 | 0.15 | 1.60 | 0.06 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PR | 25 | 26 | 2 | 4.32 | 2.82 | 1.12 | 0.59 | 0.59 | 0.60 | 0.03 | 1.00 | 4.04 | 0.02 | 2.55 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SYR | 26 | 30 | 0 | 3.13 | 2.11 | 0.79 | 0.43 | 0.46 | 0.47 | 0.08 | 0.87 | 2.69 | 0.10 | 3.08 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SCR | 27 | 30 | 1 | 3.10 | 1.95 | 0.72 | 0.41 | 0.42 | 0.43 | 0.03 | 0.90 | 2.64 | 0.05 | 1.21 | 0.11 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| YCR | 28 | 30 | 0 | 3.03 | 2.05 | 0.75 | 0.40 | 0.44 | 0.45 | 0.09 | 0.87 | 2.62 | 0.11 | 2.83 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PSR | 29 | 30 | 0 | 3.16 | 2.07 | 0.76 | 0.39 | 0.44 | 0.45 | 0.09 | 0.87 | 2.72 | 0.12 | 2.39 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| AWSR | 30 | 29 | 1 | 3.13 | 1.98 | 0.76 | 0.40 | 0.44 | 0.45 | 0.10 | 0.90 | 2.72 | 0.12 | 1.97 | 0.02 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HTCR | 31 | 49 | 0 | 3.10 | 2.04 | 0.77 | 0.45 | 0.45 | 0.46 | 0.02 | 0.90 | 2.69 | 0.02 | 3.26 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HTR | 32 | 30 | 1 | 3.23 | 2.02 | 0.77 | 0.44 | 0.44 | 0.44 | 0.00 | 0.90 | 2.91 | 0.01 | 1.64 | 0.05 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| TMR | 33 | 30 | 0 | 2.94 | 1.96 | 0.71 | 0.38 | 0.43 | 0.43 | 0.12 | 0.87 | 2.54 | 0.13 | 2.53 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WKMR | 34 | 30 | 0 | 3.16 | 1.97 | 0.75 | 0.40 | 0.44 | 0.44 | 0.08 | 0.90 | 2.68 | 0.11 | 1.75 | 0.04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| GZR | 35 | 20 | 1 | 3.03 | 1.93 | 0.70 | 0.40 | 0.41 | 0.42 | 0.04 | 0.97 | 2.72 | 0.06 | 0.28 | 0.39 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HCLR | 36 | 30 | 0 | 3.13 | 1.94 | 0.73 | 0.35 | 0.42 | 0.43 | 0.16 | 0.94 | 2.84 | 0.19 | 1.12 | 0.13 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PJR | 37 | 30 | 0 | 2.81 | 1.92 | 0.71 | 0.39 | 0.42 | 0.42 | 0.06 | 0.97 | 2.72 | 0.08 | 2.24 | 0.01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| TLR | 38 | 14 | 0 | 2.71 | 1.85 | 0.66 | 0.39 | 0.39 | 0.40 | 0.00 | 0.81 | 2.54 | 0.04 | 1.16 | 0.12 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| DMR | 39 | 30 | 0 | 1.74 | 1.47 | 0.37 | 0.21 | 0.25 | 0.25 | 0.13 | 0.55 | 1.74 | 0.15 | 4.15 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| DM2R | 40 | 23 | 1 | 1.74 | 1.49 | 0.38 | 0.25 | 0.26 | 0.26 | 0.01 | 0.55 | 1.71 | 0.03 | 4.22 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| YXR | 41 | 30 | 0 | 3.06 | 1.94 | 0.70 | 0.36 | 0.41 | 0.42 | 0.15 | 0.90 | 2.61 | 0.14 | 1.24 | 0.11 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ALR | 42 | 29 | 0 | 3.35 | 2.10 | 0.80 | 0.44 | 0.45 | 0.46 | 0.01 | 0.94 | 2.94 | 0.04 | 1.44 | 0.07 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WSR | 43 | 30 | 1 | 2.97 | 1.80 | 0.64 | 0.33 | 0.37 | 0.38 | 0.11 | 0.90 | 2.53 | 0.13 | -0.23 | 0.41 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| QQR | 44 | 30 | 0 | 2.74 | 1.72 | 0.60 | 0.35 | 0.36 | 0.37 | 0.05 | 0.94 | 2.33 | 0.04 | 0.57 | 0.28 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PZR | 45 | 30 | 0 | 2.39 | 1.71 | 0.57 | 0.37 | 0.35 | 0.36 | -0.04 | 0.81 | 2.18 | -0.05 | 2.63 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| YZR | 46 | 30 | 0 | 2.97 | 2.03 | 0.76 | 0.41 | 0.45 | 0.46 | 0.08 | 0.97 | 2.78 | 0.11 | 2.62 | 0.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Mean | - | 23 | - | 3.33 | 2.22 | 0.84 | 0.46 | 0.48 | 0.49 | 0.03 | 0.92 | 3.04 | 0.06 | - | - | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | - | - | 28 | - | - | - | - | - | - | - | - | - | - | - | - | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Notes: PID, Population ID; N, Sample size; NP, Private alleles; NA, Number of alleles; NE, Effective number of alleles; I, Shannon's information index; HO, Observed heterozygosity; HE, Expected heterozygosity; uHE, Unbiased expected heterozygosity; F, Fixation index; PPL, Percentage of polymorphic loci; AR, Allelic richness; FIS, Inbreeding coefficient; TPM, Two phase mutation model. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Our analyses grouped all walnut populations into three clusters (Fig. 2, Fig. 3). Bayesian structure analysis revealed that the optimal number of K value was 3 (Fig. 2a). At K = 3, the 1082 individuals from the 46 populations of Juglans regia were classified into three principal genetic clusters (Fig. 2c). Specifically, Group 3 consists of 53 individuals from two populations in Gongliu Wild Walnut Valley in Xinjiang; Group 2 comprises 418 individuals from 22 populations from the western Himalaya, and Group 1 contains 611 individuals from the 22 remaining populations from the southern piedmont of Tien-Shan (Figs. 2c and S1). PCoA indicates that PCoA 1, PCoA 2, and PCoA 3 explained 8.52%, 5.54%, and 4.73% of the total genetic variation, respectively (Fig. 3a), categorizing individuals of J. regia into three major groups. NJ analysis also grouped all 1082 individuals into three principal clades (Fig. 3b). The three walnut population clusters identified by our analyses correspond to the geographical distribution of J. regia populations (For more details, see Fig. S1). The genetic divergence between the three groups is moderate to high (G1 vs G2, 0.137; G1 vs G3, 0.349; G2 vs G3, 0.304). Notably, our analyses indicated that Group 3 (populations 39 and 40) were genetically distinct from Group 1 and Group 2. However, some individuals from Group 1 and Group 2 exhibited admixed genetic composition from two groups, suggesting potential gene flow or limited genetic differentiation.
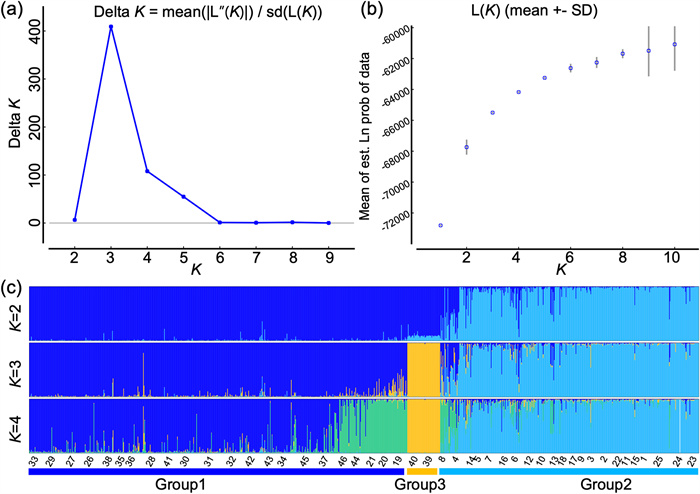
|
| Fig. 2 Bayesian inference clustering STRUCTURE analysis of 1082 individuals from 46 populations of Juglans regia. (a) The optimal K value using the delta K (ΔK) method, maximum number of populations (K), was inferred at K = 3. (b) Mean log-likelihood [Ln(K) ± SD] of the data against the number of K. (c) A Bayesian clustering of 1082 individuals of J. regia from K = 2 to K = 4. Different colors indicate different genetic groups: blue for Group 1 (G1), light blue for Group 2 (G2), and yellow for Group 3 (G3). |

|
| Fig. 3 Clustering relationship of 1082 Juglans regia individuals. (a) Principal coordinates analysis (PCoA). The first and second coordinates explain 8.52% and 5.54% of the total genetic variation, respectively. (b) A radial distance neighbor-joining tree of all J. regia individuals. The color scheme and group designation correspond to Fig. 2c. |
Genetic differentiation between the 46 walnut populations was moderate (0.05 < FST < 0.15). Nei's genetic distance values (DA) were similar to genetic differentiation (FST) (Fig. S2 and Table S6). Measures of both genetic distance and genetic differentiation indicate that populations 39 and 40 diverged genetically from the remaining walnut populations (Fig. S2 and Table S6). Migration rates were low among walnut populations (Table S7). The Wilcoxon and/or standardized difference tests indicated that 28 out of 46 populations exhibit substantial bottlenecks (P < 0.05), suggesting excess heterozygosity and a recent decrease in effective population size at the population level (Table 1). AMOVA indicated that 74.23% of the overall variation was associated with differences within populations. The remaining 25.77% of variation resided among populations (Table 2), possibly due to the substantial gene flow (Nm = 2.08) between populations of J. regia. Furthermore, AMOVA indicated that most (82.03%) of the observed genetic diversity within groups was partitioned within populations, whereas 17.97% could be due to population differences (Table 2). A hierarchical AMOVA based on the findings of the genetic diversity analysis revealed that at the group level genetic diversity was higher within populations than among populations. Specifically, the genetic diversity among the populations from Gongliu Wild Walnut Valley (Group 3) was greater (93.18%) than that within the populations of the southern piedmont of Tien-Shan (Group 2) (83.92%) and of the western Himalaya (Group 1) (81.12%) (Table 2).
| Scale | Source | d.f. | Sum of squares | Mean squares | Percentage of variation (%) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | Among Pops. | 45 | 6621.79 | 147.15 | 25.77 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Within Pops. | 1036 | 16666.55 | 16.09 | 74.23 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | 1081 | 23288.34 | 100.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Groups | Among Pops. | 2 | 2431.95 | 1215.98 | 17.97 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Within Pops. | 1079 | 20856.39 | 19.33 | 82.03 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | 1081 | 23288.34 | 100.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G1 | Among Pops. | 22 | 2408.26 | 109.47 | 18.88 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Within Pops. | 608 | 9039.73 | 14.87 | 81.12 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | 630 | 11447.99 | 100.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G2 | Among Pops. | 20 | 1755.04 | 87.75 | 16.08 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Within Pops. | 377 | 7161.20 | 19.00 | 83.92 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | 397 | 8916.24 | 100.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| G3 | Among Pops. | 1 | 26.53 | 26.53 | 6.82 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Within Pops. | 51 | 465.62 | 9.13 | 93.18 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Total | 52 | 492.15 | 100.00 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Note: d.f., degree of freedom. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
The Mantel test showed a weak positive correlation between genetic and geographic distance among populations in the whole investigated dataset of Juglans regia (r2 = 0.011, P = 1e-04) as well as among populations in each specific group (Group 1, r2 = 0.133, P = 1e-04; Group 2, r2 = 0.039, P = 1e-04). The contribution of altitude to genetic differentiation was found to be statistically insignificant (Figs. 4 and S3). Correlation analysis between genetic diversity and environmental distances indicated a non-significant correlation between annual mean temperature, annual mean precipitation, and genetic diversity in the overall populations (r2 = 0.098, P = 2e-04; r2 = 0.135, P = 1e-04), respectively (Fig. S3). This relationship was identified within groups, except for the populations from the Gongliu Wild Walnut Valley (Group 3), where no correlation was detected due to small sample size (Fig. 4). Nevertheless, according to the Barrier analysis, two primary genetic barriers were determined, with a bootstrap support of > 90% (Figs. 5 and S4). The first geographical barrier (B1) separated population 1 from all other populations of J. regia in Central Asia. The second barrier (B2) isolated populations 10 and 11 from the remaining populations (Fig. 5 and S4). These two barriers align with the presence of the Takht-e-Sulaiman Mountains and Hindu Kush Range, respectively (Figs. 5 and S1).
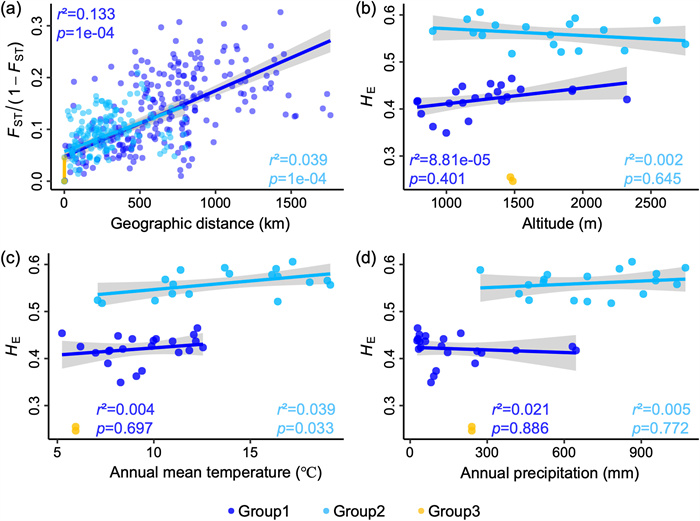
|
| Fig. 4 The relationship between genetic component and environmental variables at the group level: (a) Mantel test of genetic distance (pairwise FST) and geographic distance (in kilometers), (b–d) correlations between genetic diversity (HE) and altitude, annual mean temperature, and annual mean precipitation within three groups. The group designation corresponds to genetic structure analysis results (Fig. 2c). |
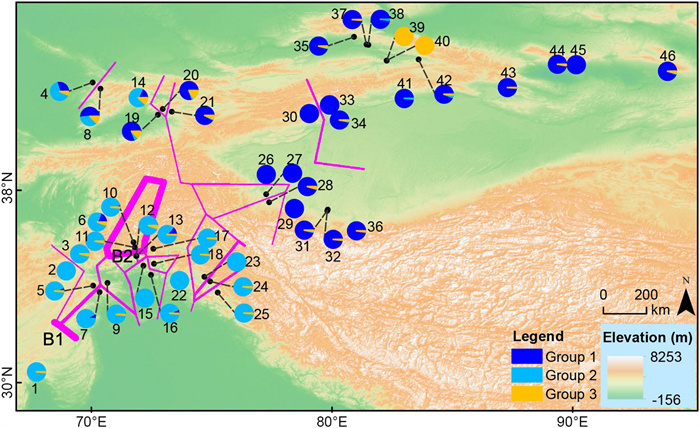
|
| Fig. 5 Geographical distribution of genetic structure of walnuts in Central Asia. Colors and proportions in the pie charts correspond to the results of genetic structure analysis at K = 3 (Fig. 2c). Solid and thick lines represent the location of the two most probable barriers detected by Barrier analysis (B1 and B2). Population identifiers align with those found in Table S1. |
We found that Juglans regia populations across Central Asia have moderate levels of genetic diversity. These levels of genetic diversity are comparable to that reported for J. regia from the Iranian Plateau (Shahi Shavvon et al., 2023), although our estimates are generally lower than those from previous studies (Table 3). Low levels of genetic diversity may be attributed to habitat fragmentation and/or human interference. Previous studies have shown that isolated habitat patches decrease population sizes, which in turn decreases genetic diversity owing to genetic drift, inbreeding, and bottleneck events (Bijma et al., 2000; González et al., 2020). In addition, human introduced populations may show low genetic diversity, as proved in J. regia populations from Xizang and Northern China (Qi et al., 2023; Yan et al., 2024). Alternatively, these moderate levels of genetic diversity might be ascribed to our use of SSR markers from J. sigillata. Primers transferred from closely related species, which are likely to produce more conservative results than those developed from the focal species. Although we believe the use of primers from J. sigillata has not greatly affected our estimations of J. regia genetic diversity in Central Asia, we recommend that future studies use population genomics with extensive geographical sampling.
| Empty Cell | Current study | Pollegioni et al. (2014) | Roor et al. (2017) | Gaisberger et al. (2020) | Magige et al. (2022) | Shahi Shavvon et al. (2023) | Yan et al. (2024) |
| HO | 0.46 | 0.56 | 0.62 | 0.57 | 0.56 | 0.44 | 0.42 |
| HE | 0.48 | 0.59 | 0.64 | 0.59 | 0.56 | 0.44 | 0.47 |
| NA | 3.33 | 4.78 | 7.54 | 5.10 | 3.79 | 3.14 | 4.52 |
Several genetic diversity parameters (i.e., HE, HO, NE, and I) indicated that the highest values of genetic diversity were found in the J. regia population (population 25) located in mountainous areas of Jammu and Kashmir in the western Himalaya, followed by four populations from Pakistan. This is consistent with previous studies that found walnut populations from these areas have high levels of genetic diversity (Ahmed et al., 2012; Gaisberger et al., 2020; Magige et al., 2022; Yan et al., 2024). Hence, the western Himalaya (e.g., Jammu and Kashmir) can be considered core centers of genetic diversity. Pollen records at these sites date back to the Upper Pleistocene, nearly 30, 000 years ago (Kotlia et al., 2000), suggesting that these sites might serve as refugia for walnuts during the LGM. This geographic area is situated at the intersection of Eurasia, with its boundaries extending towards Xizang and Xinjiang Autonomous Region of China in the northeast and Afghanistan and Pakistan in the northwest (Fig. 1). In addition, this area serves as the critical center for commercial walnut production (Shah et al., 2019). The high genetic diversity of four populations in Pakistan (i.e., 15, 22, 1, and 18) may be attributed to artificial seed selection and dispersal driven by the enduring economic and cultural significance of walnut cultivation in Asia, as suggested by Gunn et al. (2010) and Magige et al. (2022). Furthermore, we found multiple private alleles in three of these populations (i.e., populations 15, 18, and 12) (Table 1), which have important conservation implications (See below 4.3).
Our analysis indicates that the lowest genetic variation was typically found in populations from Xinjiang, China, indicating that the populations may have experienced a bottleneck event during the human-mediated introduction. This is consistent with previous work that suggested the low genetic diversity of walnut populations in northern China is attributable to a founder effect caused by the introduction of individuals from West Asia (Ding et al., 2022; Qi et al., 2023). However, two walnut populations in Gongliu, Xinjiang (i.e., populations 39 and 40) are assumed to be a relic communities from the late Tertiary (Xi and Zhang, 1996), which do show distinct genetic structure and clear genetic differentiation (Fig. 2, Fig. 3; Table S6). Furthermore, the low genetic diversity of these populations might have been caused by glaciation-driven bottleneck events. Our finding that a significant proportion of walnut populations in Central Asia have experienced genetic bottlenecks and a decrease in effective population size is likely due to farmer activities such as seed exchange (Alvarez et al., 2005; Shahi Shavvon et al., 2023).
Regarding populations in Ili valley, genetic diversity was higher in populations from the humid Ili valley than in populations of Gongliu Wild Walnut Valley (populations 39 and 40) (Fig. S1 and Table 1). Geographically, the distance is close between these populations. This genetic pattern may be the result of processes such as expanded quaternary glaciation and long-standing aridification in Central Asia. Researchers have also speculated that high genetic diversity is preserved, and isolated, in the moist valley areas of Tien-Shan, which are surrounded by areas subject to long-term aridification (Zhang et al., 2020). Hence the possible explanation for the genetic diversity of populations from Gongliu Wild Walnut Valley (populations 39 and 40) is long genetic isolation due to historical climatic oscillations. In contrary, the populations outside of Gongliu Wild Walnut Valley might be introduced from multiple resources by human being, which resulted in high genetic diversity.
4.2. Population structure and geographic barriersThe evolutionary potential and adaptability of a species to environmental changes are not only dependent on genetic diversity but also on population genetic structure (Stebbins, 1950). Population structure is determined primarily by genetic differentiation among populations (Frankham et al., 2002). As predicted for woody tree species, we observed a moderate level of genetic differentiation (FST = 0.156) across the 46 walnut populations (Table S6). This level of genetic differentiation in Juglans regia is consistent with results for most out-crossing and wind-pollinated species (e.g., Quercus semecarpifolia, Taxus wallichiana, and Liquidambar formosana) (Liu et al., 2013; Sun et al., 2016; Ginwal et al., 2023). Seeds of J. regia are spread mainly by rodents (Lenda et al., 2017; Zhang et al., 2017). Previous studies have shown that pollination and seed dispersal mechanisms of J. regia contribute to increased gene flow within and among populations, probably decreasing genetic differentiation among populations (Nybom, 2004).
Our analyses consistently categorized the 46 populations of Juglans regia in this study into three clusters: populations from the southern piedmont of Tien-Shan (Group 1), populations from the western Himalaya (Group 2), and populations from Gongliu Wild Walnut Valley (Group 3) (Fig. 2, Fig. 3). Significant genetic differentiation was detected between the three groups. Meanwhile, consistent with previous studies (Torokeldiev et al., 2018; Shahi Shavvon et al., 2023; Yan et al., 2024), most genetic variation resides within populations of the three groups (Table 2). Moreover, we also found that geographical distance had a weak influence on genetic differentiation (Fig. 4). Although mountain ranges are known restrict gene flow between different populations in Central Asia (Zhang et al., 2020), our findings indicated that the distribution of genetic clusters is only weakly correlated with geographical barriers. In other words, although these populations are separated by the Pamir Plateau and Tien-Shan, these geographical barriers do not completely prevent gene flow between the populations. Consistent with this, non-significant correlation between genetic diversity and environmental variables might also indicate human activity reshape the genetic landscape (Fig. 4). One possible reason for this finding is that the extensive germplasm exchange among farmers and consumers from similar environmental regions may lead to the movement of plant material, thus aiding species dispersal. A similar case for human-mediated movement of germplasm of walnut was recently reported in the Himalaya (Yan et al., 2024).
Our finding that populations from Gongliu Wild Walnut Valley (Group 3) diverged from the other groups of walnut populations suggest restricted gene flow between Gongliu populations and those of southern Tien-Shan and of the western Himalaya (Table S5 and Table S7). Barrier analysis identified no distinct geographical barriers between Gongliu populations and those of other groups (Fig. 5). Thus, it can be inferred, as previously mentioned, that this separation might be due to the group originating from a distinct ancestry population with long isolation. Despite the geographical distances between populations, mixtures were observed in most populations within the populations of southern Tien-Shan (Group 1) and those of the western Himalaya (Group 2), but not those of Gongliu (Group 3) (Fig. 2c). This phenomenon may be associated with factors such as a shared historical gene pool, dispersal of seeds facilitated by humans, and prior historical contact within the geographic range.
4.3. Management implicationsEffective conservation strategies rely on assessing spatial and temporal changes in genetic diversity and population structure (Frankham, 2010; Hvilsom et al., 2022). For example, one key goal of conservation genetics is to identify and prioritize the protection of populations with high genetic variation and allele diversity values (Petit et al., 1998; Xiao et al., 2020). Our analyses indicate that populations of wild walnuts in the western Himalaya have high levels of genetic variation (Fig. 1 and Table 1). Thus, we recommend that these populations be prioritized for in situ conservation to prevent fragmentation of suitable walnut habitats and/or restore fragmented areas. Furthermore, because the Himalaya have been shown to undergo rapid environmental changes (Liu et al., 2018; Wambulwa et al., 2021), conservation efforts in this region should be adjusted in response to climate change. We also recommend that the western Himalayan accessions (particularly populations 1, 15, 16, 22, and 25) should be subject to ex situ conservation and breeding plans.
Walnut populations from Gongliu Wild Walnut Valley (Group 3), Xinjiang, China have low levels of genetic diversity (Table 1), although this region has populations with distinct genetic components, including Gongliu populations (39 and 40) (Fig. 5). We recommend that these populations be conserved using both in situ and ex situ approaches. Furthermore, the presence of two barriers limits natural genetic exchange and migration in several populations (1, 10, 11, and other populations). To limit the erosion of genetic diversity mediated by global climate change, we recommend that artificial assistant dispersal strategies be used within and between these populations.
5. ConclusionsThis study provides comprehensive data on the genetic diversity and broad-scale population structure of Juglans regia in different eco-geographic regions across Central Asia. J. regia exhibits moderate genetic diversity and limited genetic differentiation, which can be attributed to both the breeding system and the human-mediated movement of plant material. The populations in Xinjiang, China may historically be introduced by human being except the two from Gongliu Wild Walnut Valley. Walnut populations from the western Himalaya showed high genetic diversity and therefore may be a rich genetic resource for future improvement programs and conservation actions. These populations should be protected by in situ and ex situ conservation measures. Special attention is needed for genetically distinct populations from Gongliu Wild Walnut Valley of Xinjiang, China. Our study paves the way for exploring the germplasm and conservation of common walnuts and serves as an exemplar for studies on other fruit trees in Central Asia.
AcknowledgementsWe are grateful to Mrs. Tao Liu, Xue-Wen Liu, Guang-Fu Zhu, Hao Deng, Song Tu, Zong-Rong He and other volunteers for their assistance in the field survey. We thank our collaborators in Pakistan and India who were involved in our previous studies to generate the genotype data publicly retrieved in this study. We thank Dr. Jian-Wen Zhang and two anonymous reviewers for their constructive comments and suggestions, which greatly improve the manuscript. We thank Dr. Moses Wambulwa, Miss Winnie Mambo and Mrs. Amos Kipkoech and Raymond Porter to read over and comment the manuscript. We also thank Professors De-Zhu Li, Lian-Ming Gao, and Wen-Jiang Liu for their help with the project. This study was supported by grants from the National Natural Science Foundation of China (32170398, 42211540718, 32260149, 41971071), the Top-notch Young Talents Project of Yunnan Provincial "Ten Thousand Talents Program" (YNWR-QNBJ-2018-146), CAS "Light of West China" Program, and Natural Science Foundation of Yunnan (202201AT070222), the Fund of Yunnan Key Laboratory of Crop Wild Relatives Omics (CWR-2024-04), the Jiangxi Provincial Natural Science Foundation (20224BAB215012), the Science and Technology Research Project of Jiangxi Provincial Department of Education (GJJ2202401), Key Research Program of Frontier Sciences, CAS (ZDBSLY-7001), Yunnan Fundamental Research Projects (202201BC070001). Molecular experiments were performed at the Laboratory of Molecular Biology at the Germplasm Bank of Wild Species, Kunming Institute of Botany, Chinese Academy of Sciences.
Data availability
The datasets used in this study are available in Supplementary Material Table S8. Notably, the genotype data of 31 SSR loci of 12 populations from Pakistan and three populations in India were publicly available in previous studies (
CRediT authorship contribution statement
Linjiang Ye: Formal analysis, Funding acquisition, Writing – original draft – review & editing. Robabeh Shahi Shavvon: Formal analysis, Writing– original draft – review & editing. Hailing Qi: Data curation, Methodology, Formal analysis, Writing – review & editing. Hongyu Wu: Methodology, Formal analysis. Pengzhen Fan: Methodology, Formal analysis. Mohammad Nasir Shalizi: Investigation, Writing – review & editing. Safiullah Khurram: Investigation, Writing – review & editing. Mamadzhanov Davletbek: Investigation, Writing – review & editing. Yerlan Turuspekov: Investigation, Writing – review & editing. Jie Liu: Conceptualization, Investigation, Validation, Funding acquisition, Writing– original draft – review & editing.
Declaration of competing interest
The authors declare that they have no known competing financial interests or personal relationships that could have appeared to influence the work reported in this paper.
Appendix A. Supplementary data
Supplementary data to this article can be found online at https://doi.org/10.1016/j.pld.2024.06.001.
Ahmed, N., Mir, J.I., Mir, R.R., et al., 2012. SSR and RAPD analysis of genetic diversity in walnut (Juglans regia L.) genotypes from Jammu and Kashmir, India. Physiol. Mol. Biol. Plants, 18: 149-160. DOI:10.1007/s12298-012-0104-z |
Alvarez, N., Garine, E., Khasah, C., et al., 2005. Farmers' practices, metapopulation dynamics, and conservation of agricultural biodiversity on-farm: a case study of sorghum among the Duupa in sub-sahelian Cameroon. Biol. Conserv., 121: 533-543. DOI:10.1016/j.biocon.2004.05.021 |
Aradhya, M., Velasco, D., Ibrahimov, Z., et al., 2017. Genetic and ecological insights into glacial refugia of walnut (Juglans regia L.). PLoS ONE, 12: e0185974. DOI:10.1371/journal.pone.0185974 |
Avanzato, D., McGranahan, G.H., Vahdati, K., et al., 2014. Following Walnut Footprints (Juglans regia L.): Cultivation and Culture, Folklore and History, Traditions and Uses. International Society for Horticultural Science (ISHS), Leuven, Belgium.
|
Baldoni, A.B., Ribeiro Teodoro, L.P., Eduardo Teodoro, P., et al., 2020. Genetic diversity of Brazil nut tree (Bertholletia excelsa Bonpl.) in southern Brazilian Amazon. For. Ecol. Manage., 458: 117795. DOI:10.1016/j.foreco.2019.117795 |
Beer, R., Kaiser, F., Schmidt, K., et al., 2008. Vegetation history of the walnut forests in Kyrgyzstan (Central Asia): natural or anthropogenic origin?. Quat. Sci. Rev., 27: 621-632. DOI:10.1016/j.quascirev.2007.11.012 |
Bijma, P., Van Arendonk, J.A.M., Woolliams, J.A., 2000. A general procedure for predicting rates of inbreeding in populations undergoing mass selection. Genetics, 154: 1865-1877. DOI:10.1093/genetics/154.4.1865 |
Cornuet, J.M., Luikart, G., 1996. Description and power analysis of two tests for detecting recent population bottlenecks from allele frequency data. Genetics, 144: 2001-2014. DOI:10.1093/genetics/144.4.2001 |
Dawson, I.K., Lengkeek, A., Weber, J.C., et al., 2009. Managing genetic variation in tropical trees: linking knowledge with action in agroforestry ecosystems for improved conservation and enhanced livelihoods. Biodivers. Conserv., 18: 969-986. DOI:10.1007/s10531-008-9516-z |
Dieringer, D., Schlötterer, C., 2003. Microsatellite analyser (MSA): a platform independent analysis tool for large microsatellite data sets. Mol. Ecol. Notes, 3: 167-169. DOI:10.1046/j.1471-8286.2003.00351.x |
Ding, Y.M., Cao, Y., Zhang, W.P., et al., 2022. Population-genomic analyses reveal bottlenecks and asymmetric introgression from Persian into iron walnut during domestication. Genome Biol., 23: 145. DOI:10.1186/s13059-022-02720-z |
Doyle, J.J., Doyle, J.L., 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem. Bull, 19: 11-15. |
Earl, D.A., vonHoldt, B.M., 2012. Structure HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conserv. Genet. Resour., 4: 359-361. DOI:10.1007/s12686-011-9548-7 |
Ebrahimi, A., Zarei, A., Lawson, S., et al., 2016. Genetic diversity and genetic structure of Persian walnut (Juglans regia) accessions from 14 European, African, and Asian countries using SSR markers. Tree Genet. Genomes, 12: 114. DOI:10.1007/s11295-016-1075-y |
Excoffier, L., Lischer, H.E., 2010. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Resour., 10: 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
FAOSTAT, 2023. Crops and livestock products. Food and Agriculture Organization, Rome, Italy.
|
Fick, S.E., Hijmans, R.J., 2017. WorldClim 2: new 1-km spatial resolution climate surfaces for global land areas. Int. J. Climatol., 37: 4302-4315. DOI:10.1002/joc.5086 |
Foggin, J.M., Lechner, A.M., Emslie-Smith, M., et al., 2021. Belt and Road Initiative in Central Asia: anticipating socioecological challenges from large-scale infrastructure in a global biodiversity hotspot. Conserv. Lett., 14: e12819. DOI:10.1111/conl.12819 |
Frankham, R., 2010. Challenges and opportunities of genetic approaches to biological conservation. Biol. Conserv., 143: 1919-1927. DOI:10.1016/j.biocon.2010.05.011 |
Frankham, R., Briscoe, D.A., Ballou, J.D., 2002. Introduction to Conservation Genetics. Cambridge, UK: Cambridge University Press.
|
Gaisberger, H., Legay, S., Andre, C., et al., 2020. Diversity under threat: connecting genetic diversity and threat mapping to set conservation priorities for Juglans regia L. populations in Central Asia. Front. Ecol. Evol., 8: 171. DOI:10.3389/fevo.2020.00171 |
Ginwal, H.S., Rawat, A., Shekhar, C., et al., 2023. Population genetic structure of a timberline oak (Quercus semecarpifolia Sm.) of western Himalayas and conservation implications. Conserv. Genet., 25: 133-147. |
González, A.V., Gómez-Silva, V., Ramírez, M.J., et al., 2020. Meta-analysis of the differential effects of habitat fragmentation and degradation on plant genetic diversity. Conserv. Biol., 34: 711-720. DOI:10.1111/cobi.13422 |
Goudet, J., 2005. HIERFSTAT, a package for R to compute and test hierarchical F-statistics. Mol. Ecol. Notes, 5: 184-186. DOI:10.1111/j.1471-8286.2004.00828.x |
Groppi, A., Liu, S., Cornille, A., et al., 2021. Population genomics of apricots unravels domestication history and adaptive events. Nat. Commun., 12: 3956. DOI:10.1038/s41467-021-24283-6 |
Gunn, B.F., Aradhya, M., Salick, J.M., et al., 2010. Genetic variation in walnuts (Juglans regia and J. sigillata; Juglandaceae): species distinctions, human impacts, and the conservation of agrobiodiversity in Yunnan, China. Am. J. Bot., 97: 660-671. DOI:10.3732/ajb.0900114 |
Hijmans, R.J., Karney, C., Williams, E., et al., 2022. Package 'geosphere' Version 1.5-18. Spherical trigonometr.
|
Hvilsom, C., Segelbacher, G., Ekblom, R., et al., 2022. Selecting Species and Populations for Monitoring of Genetic Diversity. IUCN SSC Conservation Genetics Specialist Group, Gland, Switzerland.
|
Jump, A.S., Marchant, R., Penuelas, J., 2009. Environmental change and the option value of genetic diversity. Trends Plant Sci., 14: 51-58. DOI:10.1016/j.tplants.2008.10.002 |
Khan, H., Ullah, I., Woeste, K., et al., 2023. Population genetics informs new insights into the phytogeographic history of Juglans regia L. Genet. Resour. Crop Evol., 70: 2263-2278. DOI:10.1007/s10722-023-01597-6 |
Kotlia, B.S., Sharma, C., Bhalla, M.S., et al., 2000. Palaeoclimatic conditions in the late Pleistocene Wadda lake, eastern Kumaun Himalaya (India). Palaeogeogr. Palaeoclimatol. Palaeoecol., 162: 105-118. DOI:10.1016/S0031-0182(00)00107-3 |
Langella, O., 1999. Populations version 1.2.31: population genetic software (individuals or populations distances, phylogenetic trees). http://bioinformatics.org/?tryphon/populations.
|
Lenda, M., Knops, J.H., Skórka, P., et al., 2017. Cascading effects of changes in land use on the invasion of the walnut Juglans regia in forest ecosystems. J. Ecol., 106: 671-686. |
Lioubimtseva, E., Henebry, G.M., 2009. Climate and environmental change in arid Central Asia: impacts, vulnerability, and adaptations. J. Arid Environ., 73: 963-977. DOI:10.1016/j.jaridenv.2009.04.022 |
Liu, J., Gao, L.M., 2011. Comparative analysis of three different methods of total DNA extraction used in Taxus. Guihaia 31, 244: 238. |
Liu, J., Magige, E.A., Fan, P.Z., et al., 2023. Genetic imprints of grafting in wild iron walnut populations in southwestern China. BMC Plant Biol., 23: 423. DOI:10.1186/s12870-023-04428-z |
Liu, J., Milne, R.I., Cadotte, M.W., et al., 2018. Protect Third Pole's fragile ecosystem. Science, 362: 1368. DOI:10.1126/science.aaw0443 |
Liu, J., Milne, R.I., Zhu, G., et al., 2022. Name and scale matter: clarifying the geography of Tibetan Plateau and adjacent mountain regions. Global Planet. Change, 215: 103893. DOI:10.1016/j.gloplacha.2022.103893 |
Liu, J., Moller, M., Provan, J., et al., 2013. Geological and ecological factors drive cryptic speciation of yews in a biodiversity hotspot. New Phytol., 199: 1093-1108. DOI:10.1111/nph.12336 |
Loo, J., Souvannavong, O., Dawson, I.K., 2014. Seeing the trees as well as the forest: the importance of managing forest genetic resources. For. Ecol. Manage., 333: 1-8. DOI:10.1016/j.foreco.2014.08.014 |
Lu, A., Stone, D., Grauke, L., 1999. Juglandaceae. In: Wu, Z.Y., Peter, R.H. (Eds.), Flora of China. Science Press and Missouri Botanical Garden Press, Beijing, China and St. Louis, Missouri, USA, pp. 277–285.
|
MacDougall, A.S., McCann, K.S., Gellner, G., et al., 2013. Diversity loss with persistent human disturbance increases vulnerability to ecosystem collapse. Nature, 494: 86-89. DOI:10.1038/nature11869 |
Magige, E.A., Fan, P.Z., Wambulwa, M.C., et al., 2022. Genetic diversity and structure of Persian walnut (Juglans regia L.) in Pakistan: implications for conservation. Plants, 11: 1652. DOI:10.3390/plants11131652 |
Manni, F., Guerard, E., Heyer, E., 2004. Geographic patterns of (genetic, morphologic, linguistic) variation: how barriers can be detected by using monmonier's algorithm. Hum. Biol., 76: 173-190. DOI:10.1353/hub.2004.0034 |
Mapelli, S., 2018. Spatial genetic structure of common walnut (Juglans regia L.) in central Asia. Acta Hortic., 1190: 27-34. DOI:10.17660/actahortic.2018.1190.5 |
McGranahan, G., Leslie, C., 2012. Walnut. In: Badenes, M.L., Byrne, D.H. (Eds.), Fruit
Breeding. Springer US, Boston, MA, pp. 827-846.
|
McNeely, J.A., Miller, K.R., Reid, W.V., et al., 1990. Conserving the world's biological diversity. International Union for Conservation of Nature and Natural Resources (Gland, Switzerland).
|
Mittermeier, R.A., Gil, P.R., Hoffman, M., et al., 2004. Hotspots Revisited: Earth's
Biologically Richest and Most Endangered Ecoregions, Second ed. CEMEX,
Mexico City, Mexico.
|
Nagy, S., Poczai, P., Cernák, I., et al., 2012. PICcalc: an online program to calculate polymorphic information content for molecular genetic studies. Biochem. Genet., 50: 670-672. DOI:10.1007/s10528-012-9509-1 |
Nybom, H., 2004. Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants. Mol. Ecol., 13: 1143-1155. DOI:10.1111/j.1365-294X.2004.02141.x |
Oksanen, J., Blanchet, F., Kindt, R., et al., 2013. Package 'vegan'. Community ecology package, version 2. https://cran.r-project.org/web/packages/vegan.
|
Peakall, R.O.D., Smouse, P.E., 2006. Genalex 6: genetic analysis in Excel. Population genetic software for teaching and research. Mol. Ecol. Notes, 6: 288-295. DOI:10.1111/j.1471-8286.2005.01155.x |
Petit, R.J., El Mousadik, A., Pons, O., 1998. Identifying populations for conservation on the basis of genetic markers. Conserv. Biol., 12: 844-855. DOI:10.1111/j.1523-1739.1998.96489.x |
Piry, S., Luikart, G., Cornuet, J.M., 1999. Computer note. BOTTLENECK: a computer program for detecting recent reductions in the effective size using allele frequency data. J. Hered., 90: 502-503. DOI:10.1093/jhered/90.4.502 |
Pollegioni, P., Woeste, K.E., Chiocchini, F., et al., 2014. Landscape genetics of Persian walnut (Juglans regia L.) across its Asian range. Tree Genet. Genomes, 10: 1027-1043. DOI:10.1007/s11295-014-0740-2 |
Potter, K.M., Jetton, R.M., Bower, A., et al., 2017. Banking on the future: progress, challenges and opportunities for the genetic conservation of forest trees. New For., 48: 153-180. DOI:10.1007/s11056-017-9582-8 |
Pritchard, J.K., Stephens, M., Donnelly, P., 2000. Inference of population structure using multilocus genotype data. Genetics, 155: 945-959. DOI:10.1093/genetics/155.2.945 |
Qi, H., Fan, P., Wang, Y., et al., 2023. Genetic diversity and population structure of Juglans regia from six provinces in northern China. Biodivers. Sci., 31: 23120. DOI:10.17520/biods.2023120 |
Roor, W., Konrad, H., Mamadjanov, D., et al., 2017. Population differentiation in common Walnut (Juglans regia L.) across major parts of its native range—insights from molecular and morphometric data. J. Hered., 108: 391-404. DOI:10.1093/jhered/esw122 |
Salgotra, R.K., Chauhan, B.S., 2023. Genetic diversity, conservation, and utilization of plant genetic resources. Genes, 14: 174. DOI:10.3390/genes14010174 |
Schaberg, P.G., DeHayes, D.H., Hawley, G.J., et al., 2008. Anthropogenic alterations of genetic diversity within tree populations: implications for forest ecosystem resilience. For. Ecol. Manage., 256: 855-862. DOI:10.1016/j.foreco.2008.06.038 |
Shah, U.N., Mir, J.I., Ahmed, N., et al., 2019. Genetic diversity analysis of walnut (Juglans regia L.) from Kashmir valley using RAPD and ISSR markers. Agrotechnology, 8: 185. |
Shahi Shavvon, R., Qi, H.L., Mafakheri, M., et al., 2023. Unravelling the genetic diversity and population structure of common walnut in the Iranian Plateau. BMC Plant Biol., 23: 201. DOI:10.1186/s12870-023-04190-2 |
Stebbins, G.L., 1950. Variation and Evolution in Plants. New York, USA: Columbia University Press.
|
Sun, R., Lin, F., Huang, P., et al., 2016. Moderate genetic diversity and genetic differentiation in the relict tree Liquidambar formosana Hance revealed by genic simple sequence repeat markers. Front. Plant Sci., 7: 1411. |
Torokeldiev, N., Ziehe, M., Gailing, O., et al., 2018. Genetic diversity and structure of natural Juglans regia L. populations in the southern Kyrgyz Republic revealed by nuclear SSR and EST-SSR markers. Tree Genet. Genomes, 15: 5. |
Van Oosterhout, C., Hutchinson, W.F., Wills, D.P.M., et al., 2004. MICRO-CHECKER: software for identifying and correcting genotyping errors in microsatellite data. Mol. Ecol. Notes, 4: 535-538. DOI:10.1111/j.1471-8286.2004.00684.x |
Vavilov, N.I., Vavylov, M.I., Vavílov, N.Í., et al., 1992. Origin and Geography of Cultivated Plants. Cambridge, UK: Cambridge University Press.
|
Wambulwa, M.C., Fan, P.Z., Milne, R., et al., 2022. Genetic analysis of walnut cultivars from southwest China: implications for germplasm improvement. Plant Divers., 44: 530-541. DOI:10.1016/j.pld.2021.08.005 |
Wambulwa, M.C., Meegahakumbura, M.K., Chalo, R., et al., 2016. Nuclear microsatellites reveal the genetic architecture and breeding history of tea germplasm of East Africa. Tree Genet. Genomes, 12: 11. DOI:10.1007/s11295-015-0963-x |
Wambulwa, M.C., Milne, R., Wu, Z.Y., et al., 2021. Spatiotemporal maintenance of flora in the Himalaya biodiversity hotspot: current knowledge and future perspectives. Ecol. Evol., 11: 10794-10812. DOI:10.1002/ece3.7906 |
Wang, Q., Zhang, H.X., 2022. Population genetic structure and biodiversity conservation of a relict and medicinal subshrub Capparis spinosa in arid central Asia. Diversity, 14: 146. DOI:10.3390/d14020146 |
Wei, J., Li, X., Xu, H., et al., 2022. Evaluation of the genetic diversity of Pinus koraiensis by EST-SSR and its management, utilization and protection. For. Ecol. Manage., 505: 119882. DOI:10.1016/j.foreco.2021.119882 |
Wickham, H., 2016. Ggplot2: Elegrant Graphics for Data Analysis. New York, USA: Springer.
|
Wilson, G.A., Rannala, B., 2003. Bayesian inference of recent migration rates using multilocus genotypes. Genetics, 163: 1177-1191. DOI:10.1093/genetics/163.3.1177 |
Xi, R.T., Zhang, Y.P., 1996. Walnut flora. In: Zhao, H.C., Tian, B.F. (Eds.), China FruitPlant Monograph. China Forestry Publishing House, Beijing, China.
|
Xiahou, Z.Y., Wambulwa, M.C., Xu, Z.C., et al., 2023. A multiplex PCR system of novel microsatellite loci for population genetic application in walnuts. Plants, 12: 4101. DOI:10.3390/plants12244101 |
Xiao, S., Ji, Y., Liu, J., et al., 2020. Genetic characterization of the entire range of Cycas panzhihuaensis (Cycadaceae). Plant Divers., 42: 7-18. DOI:10.1016/j.pld.2019.10.001 |
Xu, Z.C., Jin, Y.C., Milne, R.I., et al., 2020. Development of 32 novel microsatellite loci in Juglans sigillata using genomic data. Appl. Plant Sci., 8: e11328. DOI:10.1002/aps3.11328 |
Yan, L.J., Fan, P.Z., Wambulwa, M.C., et al., 2024. Human-associated genetic landscape of walnuts in the Himalaya: implications for conservation and utilization. Divers. Distrib., 30: e13809. DOI:10.1111/ddi.13809 |
Yu, G., Smith, D.K., Zhu, H., et al., 2016. GGTREE: an R package for visualization and annotation of phylogenetic trees with their covariates and other associated data. Methods Ecol. Evol., 8: 28-36. |
Zhang, H., Chu, W., Zhang, Z., 2017. Cultivated walnut trees showed earlier but not final advantage over its wild relatives in competing for seed dispersers. Integr. Zool., 12: 12-25. DOI:10.1111/1749-4877.12242 |
Zhang, H.X., Li, X.S., Wang, J.C., et al., 2020. Insights into the aridification history of Central Asian Mountains and international conservation strategy from the endangered wild apple tree. J. Biogeogr., 48: 332-344. |
Zohary, D., Hopf, M., Weiss, E., 2012. Domestication of Plants in the Old World: the Origin and Spread of Domesticated Plants in Southwest Asia, Europe, and the Mediterranean Basin. Oxford University Press, Oxford, UK, p. 149.
|



