扩展功能
文章信息
- 屠培培, 黄彬, 路景涛, 薛绍礼, 夏觅珍, 吴琳梅, 张耀方, 李明刚
- DNA和组蛋白表观遗传修饰在2型糖尿病发病中作用的研究进展
- Research progress in effects of DNA and histone epigenetic modifications in pathogenesis of type 2 diabetes
- 吉林大学学报(医学版), 2018, 44(04): 853-858
- Journal of Jilin University (Medicine Edition), 2018, 44(04): 853-858
- 10.13481/j.1671-587x.20180431
-
文章历史
- 收稿日期: 2017-09-18
2. 淮北矿工总医院集团骨科, 安徽 淮北 235000;
3. 天津农学院基础科学学院生物制药系, 天津 300384;
4. 南开大学生命科学学院分子生物学研究所, 天津 300071
糖尿病是一类由遗传和环境因素相互作用导致胰岛素分泌障碍,致靶组织细胞对胰岛素敏感性降低而引发的代谢紊乱综合征,发病率高且难以治愈。然而,2型糖尿病的确切发病机制迄今仍未被阐明。糖尿病主要是由遗传因素和环境因素相互作用引起的,而表观遗传又是连接基因与环境的分子桥梁。研究[1]表明:表观遗传学在糖尿病的发生发展及持续存在的糖尿病并发症中起重要作用,与遗传多态性类似,表观修饰可改变基因的转录活性进而导致2型糖尿病的各种表型特征。目前,对糖尿病表观遗传学机制的研究主要是在全基因组扫描与糖尿病相关DNA甲基化改变,或者是检测某个已知功能的糖尿病相关基因的启动子区组蛋白修饰改变。表观遗传学机制在糖尿病及其一系列慢性并发症中的应用情况国内已有报道[2]。Rorbach-Dolata等[3]系统阐述了表观遗传修饰变化在糖尿病(1型和2型)及其并发症发生发展中的作用机制。DNA甲基化和组蛋白修饰是重要的表观遗传学现象。本文作者围绕DNA甲基化修饰、组蛋白乙酰化和甲基化修饰等表观遗传学修饰机制在2型糖尿病发病中的作用展开分析和讨论,为其预防和治疗提供新策略。
1 DNA甲基化与2型糖尿病 1.1 DNA甲基化DNA的甲基化修饰是目前为止科学家们研究最为透彻的表观遗传学标记。绝大多数哺乳动物和人类基因的启动子区、多数功能基因编码区和转座子中均存在大量富含CG碱基序列的区域,该区域被称为CpG岛。DNA甲基化修饰主要受DNA甲基转移酶(DNA methyl-transferase,DNMT)家族蛋白的调控,其分子机制[4]见图 1。具体过程:在DNA甲基转移酶的作用下,以S-腺苷甲硫氨酸作为甲基供体使CpG岛上的胞嘧啶5′端发生甲基化。通常情况下基因的CpG岛处于非甲基化状态,但当CpG二核苷酸发生甲基化修饰时,可抑制该基因的表达,可能是因为甲基化阻碍了关键的转录因子与启动子的相互靠近和结合导致。越来越多的证据[5]表明:甲基化的发生由氧化酶类引起,甲基化水平的调节与饮食、生理活动和高糖等有关联,说明甲基化是个动态的过程。有证据[6]表明:组织特异性启动子区CpG岛的密度以及甲基化水平在控制相关基因的表达中起重要作用。
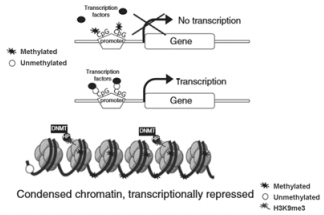
|
| 图 1 DNA甲基化的分子机制 Figure 1 Molecular mechanism of DNA methylation |
|
|
在糖尿病表观遗传隔代效应中DNA甲基化修饰发挥了重要作用,环境和饮食均可导致基因组中某些印迹基因和转座因子表观修饰的改变,进而影响疾病的发生发展[7]。研究[8]显示:2型糖尿病患者的糖代谢功能紊乱与外周血白细胞的DNA低甲基化有关。在糖尿病前期向2型糖尿病发展的进程所涉及的糖代谢、氧化应激和炎症等基因中,共出现694个CpG位点低甲基化和174个位点高甲基化[9]。胰岛素生长因子2(insulin-like growth factor-2,IGF-2)有促进胰岛细胞增殖的作用,1944-1945年荷兰大饥荒时的调查数据[10]显示:饥荒发生60年后,在出生前遭受饥荒人群的IGF-2甲基化水平较其兄弟姐妹严重偏低,而怀孕期间营养过剩也会影响后代的胰岛β细胞的功能。近年来研究[11]显示:启动子区DNA甲基化改变了新疆维吾尔族糖尿病患者内脏脂肪组织中肿瘤坏死因子α(TNF-α)和脂联素(ADIPOQ)等基因的表达。糖尿病患者的DNA甲基化异常,主要表现在患者外周血白细胞中与心血管病相关的基因DNA甲基化水平比正常人明显升高,在1型和2型糖尿病肾病患者的唾液中DNA甲基化水平明显高于普通人[12-13]。另外有研究[14]显示:长期胰岛素和葡萄糖的暴露环境会严重改变骨骼肌的DNA甲基化状况,表明DNA甲基化是一种快速适应的表观遗传标记。DNA甲基化可干扰胰岛的正常发育和降低胰岛素分泌水平,同时也使能量代谢通路上的甲基化增高,进而减弱胰岛素重要靶器官代谢葡萄糖的能力。
1.2.2 DNA甲基化与炎症反应胰岛素抵抗(insulin resistance,IR)和(或)胰岛素分泌不足一直是2型糖尿病发病的关键因素,炎症在2型糖尿病的发病过程中起媒介作用,2型糖尿病可能是一种先天的免疫性疾病,是由细胞因子介导的炎症反应。多数炎症因子在2型糖尿病的发生发展进程中起重要作用,TNF-α、C反应蛋白(C reactive protein, CRP)和白细胞介素6 (interleukin,IL-6)等不但参与IR,而且与糖尿病血管并发症有密切关联。肥胖人群处于慢性的低剂量炎症状态从而导致的代谢损伤是2型糖尿病公认的发病因素,而DNA甲基化与肥胖及2型糖尿病患者的IR有关[15-16]。Simar等[15]研究显示:与正常人比较,2型糖尿病患者的B细胞和NK细胞甲基化水平升高,而B细胞高甲基化程度与IR和空腹胰岛素水平有关,表明DNA甲基化异常可能与肥胖和2型糖尿病患者的代谢失调及免疫功能改变有关。此外,机体内的高糖和游离脂肪酸可以调控DNA的甲基化修饰,提示营养过剩可能导致异常的甲基化修饰,进而改变肥胖和糖尿病易感基因的表达水平[17]。
1.2.3 DNA甲基化与胰岛素分泌受损胰岛β细胞分泌胰岛素受损是2型糖尿病发病的重要原因。Kuroda等[18]发现:胰岛素分泌调节基因INS的甲基化水平与2型糖尿病的发病关系密切,2型糖尿病患者胰岛β细胞中INS启动子呈去甲基化状态可能影响胰岛β细胞的发育成熟,进而影响胰岛素的分泌。Yang等[19]研究2型糖尿病患者胰岛候选基因的DNA甲基化和表达水平发现:胰腺转录因子PDX1启动子区域的DNA高甲基化水可抑制其转录活性,导致胰岛β细胞功能紊乱。随着高通量技术的不断发展,全基因组表观遗传的应用研究也越来越广泛。全基因组甲基化研究[20]显示:与正常人比较,2型糖尿病患者胰岛的853个基因(包括Tcf7l2、Irs1、Fto、Cdkn1a和Kcnq1等)中共1649 CpG位点出现明显的甲基化水平变化。Ling等[21]利用候选基因法证实:在2型糖尿病患者胰岛组织中PPARGC1A基因高甲基化与胰岛素分泌减少有密切关联。Hall等[22]发现:经棕榈酸盐处理的2型糖尿病患者胰岛组织与未处理的胰岛组织间共1860个基因甲基化存在明显差异,致使其表达异常,损伤胰岛β细胞分泌功能。已知胰岛DNA甲基化水平改变与2型糖尿病发病有关[23],而外周IR的基因甲基化水平改变是否会增加罹患糖尿病的风险目前尚不明确。已有相关研究[24]显示:外周组织(肝脏、肌肉和脂肪)中糖尿病相关基因的DNA甲基化水平变化与糖尿病发病存在必然联系,而外周组织中部分DNA的甲基化水平变化可通过生理运动干预。
2 组蛋白修饰与2型糖尿病 2.1 组蛋白修饰组蛋白修饰是表观遗传学的重要组成部分。染色质结构中的核心组蛋白会受到一系列不同化学基团的共价修饰[包括乙酰化、甲基化、磷酸化和类泛素蛋白修饰分子(SUMO)化等],形成一个修饰级联,称为组蛋白密码。组蛋白密码可被一系列蛋白复合物识别、翻译成特定的染色质状态,具有非常重要的基因调控转录功能[25]。染色质上接近转录因子结合位点的难易程度由组蛋白的位置和包裹程度决定,而核小体压缩程度由一系列酶的活性决定,这些修饰酶负责组蛋白特异氨基酸的各种共价修饰(例如乙酰化、甲基化和磷酸化等)[26]。目前研究最多的组蛋白修饰类型是发生在组蛋白H3和H4的一些特定氨基酸残基上的乙酰化和甲基化修饰。组蛋白修饰的分子机制[27]见图 2。
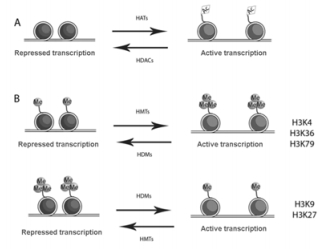
|
| 图 2 组蛋白修饰的分子机制 Figure 2 Molecular mechanisms of histone modification |
|
|
组蛋白密码假说提出在基因表达调控过程中不同的组蛋白修饰类型发挥了不同的作用。组蛋白的乙酰化过程由组蛋白乙酰转移酶(histone acetyltransferases,HATs)和组蛋白去乙酰化酶(histone deacetylases,HDACs)协调催化完成[27]。已知HATs能够催化组蛋白发生乙酰化修饰,导致染色质结构松弛,从而容易招募转录因子与之相结合并促进基因的转录表达;相反,HDACs的作用是使组蛋白去乙酰化,导致染色质压缩,抑制基因转录。HATs和HDACs共同参与组蛋白乙酰化的动态平衡过程,精确调控基因的转录,例如组蛋白H3第9、14、18及23位上的赖氨酸残基发生乙酰化可激活染色体,利于转录因子的结合,从而促进转录[28]。组蛋白的乙酰化和去乙酰化的动态平衡状态一旦被打破就可能引起疾病。近年来研究[29]显示:胰岛素受体底物2(IRS2)启动子区H3K9乙酰化水平与该基因的表达水平呈正相关关系,H3K9的乙酰化水平上调会间接促使胰岛β细胞增殖。组蛋白去乙酰化酶7(HDAC7)的高表达使大鼠胰岛细胞分泌能力受损,且2型糖尿病患者胰岛HDAC7的表达水平明显增加[30]。以上研究结果均证实组蛋白乙酰化机制的失调与糖尿病的发生有密切关联。
2.1.2 组蛋白甲基化组蛋白甲基化异常通常可导致基因的转录水平异常,进而促使了相关疾病的发生发展。组蛋白的甲基化修饰经常发生在组蛋白H3和H4的赖氨酸或者精氨酸的残基上,由组蛋白甲基转移酶(histone methyltransferases,HMTs)和组蛋白去甲基化酶1(lysine demethylase 1,LSD1)协同催化完成,表明组蛋白甲基化修饰过程也是可逆的[31]。与组蛋白乙酰化修饰不同,组蛋白甲基化更加稳定和持久。组蛋白甲基化的形式、位点以及被修饰的氨基酸种类不同可导致基因转录表达的激活或者抑制[32]。组蛋白甲基化的形式包括单甲基化、双甲基化和三甲基化。组蛋白赖氨酸上的甲基化修饰相对稳定,常发生在H3K4、H3K9、H4K20、H3K27、H3K36和H3K79位点,如组蛋白H3第9位、第27位的赖氨酸二甲基化(H3K9me2、H3K27me2)或三甲基化(H3K9me3、H3K27me3)通过抑制转录因子的结合使染色质压缩;相反,组蛋白H3第4位赖氨酸上的甲基化(H3K4me)却促进基因的转录激活[33]。
2.2 组蛋白修饰在2型糖尿病发病中的作用 2.2.1 组蛋白修饰与炎症研究[34]表明:组蛋白的各种共价修饰在糖尿病及其并发症中起重要作用,而这些修饰并非一成不变,组蛋白动态的修饰状态可导致转录复合体的变化,并在DNA和其他蛋白因子间高效地调节染色质的沉默或激活,进而发挥协同或拮抗作用。HATs和HDAC已被证明可调控与糖尿病相关的基因表达,HATs和HDAC可通过调节核因子κB(NF-κB)的转录活性导致下游相关炎性因子表达水平的改变[35]。Miao等[36]发现:机体在高糖状态下,单核细胞分泌的炎症因子NF-κB和HATs呈现高乙酰化和转录活性,致使淋巴细胞有关炎症和免疫的信号通路中的H3上的第9位赖氨酸发生甲基化(H3K9)修饰;文献[37]报道:在机体血管平滑肌细胞中,H3K9位点甲基化可以促进炎症因子的表达上调。此外,体外实验[38]表明:高糖培养条件可以引起环氧合酶2(COX-2)和TNF-α等炎症基因启动子区域的组蛋白乙酰化水平升高,结果导致COX-2和TNF-α基因表达水平提高。
2.2.2 组蛋白修饰与胰岛β细胞发育组蛋白修饰在胰岛β细胞的分化、增殖和凋亡中发挥重要作用,已知在胚胎干细胞定向分化和发育的过程中,关键基因的转录必须受到严格控制,组蛋白修饰是该过程中必不可少的监管机制之一[39]。研究[40]显示:胰十二指肠同源盒1(PDX-1)作为一个自主的重编码因子,通过影响募集胰岛β细胞周围特定的组蛋白甲基转移酶SET7/9特异位点发生表观遗传改变,完成胰岛从α细胞到β细胞的转换。多梳基因(polycomb group genes,PcG)是一类关键的与发育有关的基因,PcG基因家族通常以表观遗传修饰来维持基因的转录抑制状态。已知B细胞特异的莫洛尼白血病毒插入位点1(B cell-specific MLV integration site-1,Bmi-1)是PcG家族核心成员之一,在STZ诱导的糖尿病小鼠模型中被发现,Bmi-1在年轻、可再生的胰岛β细胞中表达水平增加,引起胰岛β细胞全基因组的H3K4三甲基化水平降低、H2A泛素化和H3K27三甲基化水平升高,胰岛β细胞的复制能力提高;随着年龄增长,Bmi-1表达水平逐渐降低,H3K4三甲基化增加,H2A泛素化减少,胰岛β细胞的增殖能力下降[41]。Zeste基因增强子同源物2(enhancer of zeste homolog2,Ezh2)也是多梳基因家族成员之一,具有组蛋白甲基化酶、组蛋白去乙酰化酶和多基因表达的调控功能。研究者[42]发现:Ezh2基因缺失导致抑癌基因Ink4a/Arf组蛋白甲基化水平下降,同时伴有胰岛β细胞增殖受损,而Ezh2 KO小鼠可出现胰岛β细胞数量减少及轻度糖尿病。
2.2.3 组蛋白修饰与胰岛素表达糖尿病发病过程中关键基因胰岛素的表达也受组蛋白修饰的影响。已知组蛋白H3乙酰化(H3Ac)和组蛋白H3赖氨酸4甲基化(H3K4Me)水平对于染色质维持特定的疏松状态起重要作用,而胰岛素基因激活的标志就是其启动子区高水平的H3Ac和H3K4Me状态[43]。此外,当胰岛素基因的启动子区大量募集组蛋白甲基转移酶Set7/9时,可使H3K4Me始终维持较高水平,进而上调胰岛素基因的表达[44]。研究[45]显示:胰岛素受体底物分子在IR状态时常表现出表达不足或磷酸化水平异常,胰岛素受体底物分子IRS-2磷酸化减少是组蛋白去乙酰化酶SIRT1受到抑制导致,使胰岛素的敏感性增加,因此SIRT1将有望成为治疗2型糖尿病的新靶点。肝细胞核转录因子Foxa2水平受肝细胞核因子6(HNF6)稳定性的影响,HNF6受组蛋白乙酰化酶CBP的调控,而Foxa2活性的改变会引发糖脂代谢的变化,因此CBP可能同时调控HNF6与Foxa2转录因子的表达[46]。近年来研究[47]显示:机体内循环脂肪酸也可产生“代谢记忆”现象,其机制可能是改变了糖异生基因调节因子FOXO1启动子区的H3K36me2和H3K27me3的水平。高脂饮食会导致FOXO1基因及其靶基因的持续激活,利用染色质免疫共沉淀技术(ChIP)检测,FOXO1启动子中H3K36me2和H3K27me3分别表现出持续增加和持续降低[48]。通过以上研究可进一步推测组蛋白修饰在2型糖尿病的发病过程中可能起着重要作用。
3 展望表观遗传学修饰可在不改变DNA序列的前提下使基因的转录和表达水平发生变化,进而影响机体多种生理病理过程。表观遗传学中DNA甲基化和组蛋白修饰参与了2型糖尿病的发病过程,但其在糖尿病发生发展中的具体机制尚未完全阐明,仍有许多问题亟待解决。糖尿病是一种多基因多因素疾病,研究单个基因的表观遗传修饰改变,难以全面揭示疾病发生的分子机制,多基因表观修饰的失衡也许在糖尿病发病过程中扮演重要角色。
综上所述,随着表观遗传学研究的不断进展,将会对DNA甲基化和组蛋白甲基化、乙酰化等修饰方式有更加深入的了解,多基因的表观遗传修饰水平的研究能进一步阐明2型糖尿病发病的分子机制。利用表观遗传干预DNA甲基化和组蛋白修饰的方法在未来有望成为治疗糖尿病的关键技术手段,进一步开发各种表观修饰酶抑制剂以及有相似作用的药物,通过改变表观遗传的方式影响糖尿病进程,可成为治疗糖尿病药物开发的新方向。
| [1] | Raciti GA, Nigro C, Longo M, et al. Personalized medicine and type 2 diabetes:lesson from epigenetics[J]. Epigenomics, 2014, 6(2): 229–238. DOI:10.2217/epi.14.10 |
| [2] | 燕艳, 刘仲祥, 张秀娟. 表观遗传学在糖尿病及其慢性并发症中的应用[J]. 中国实验诊断学, 2017, 21(9): 1669–1671. |
| [3] | Rorbach-Dolata A, Kubis A, Piwowar A. Epigenetic modifications:An important mechanism in diabetic disturbances[J]. Postepy Hig Med Dosw (Online), 2017, 71: 960–974. |
| [4] | Bansal A, Pinney SE. DNA methylation and its role in the pathogenesis of diabetes[J]. Pediatr Diabetes, 2017, 18(3): 167–177. DOI:10.1111/pedi.2017.18.issue-3 |
| [5] | Wicklow BA, Sellers EA. Maternal health issues and cardio-metabolic outcomes in the offspring:a focus on Indigenous populations[J]. Best Pract Res Clin Obstet Gynaecol, 2015, 29(1): 43–53. DOI:10.1016/j.bpobgyn.2014.04.017 |
| [6] | Ponnaluri VK, Ehrlich KC, Zhang G, et al. Association of 5-hydroxymethylation and 5-methylation of DNA cytosine with tissue specific gene expression[J]. Epigenetics, 2017, 12(2): 123–138. DOI:10.1080/15592294.2016.1265713 |
| [7] | Jirtle RL, Skinner MK. Environmental epigenomics and disease susceptibility[J]. Nat Rev Genet, 2007, 8(4): 253–262. DOI:10.1038/nrg2045 |
| [8] | Toperoff G, Kark JD, Aran D, et al. Premature aging of leukocyte DNA methylation is associated with type 2 diabetes prevalence[J]. Clin Epigenetics, 2015, 7(1): 35. DOI:10.1186/s13148-015-0069-1 |
| [9] | VanderJagt TA, Neugebauer MH, Morgan M, et al. Epigenetic profiles of pre-diabetes transitioning to type 2 diabetes and nephropathy[J]. World J Diabetes, 2015, 6(9): 1113–1121. DOI:10.4239/wjd.v6.i9.1113 |
| [10] | Barres R, Zierath JR. The role of diet and exercise in the transgenerational epigenetic landscape of T2DM[J]. Nat Rev Endocrinol, 2016, 12(8): 441–451. DOI:10.1038/nrendo.2016.87 |
| [11] | Zhang J, Wang C, Ha X, et al. DNA methylation of tumor necrosis factor-α, monocyte chemoattractant protein-1, and adiponectin genes in visceral adipose tissue is related to type 2 diabetes in the Xinjiang Uygur population[J]. J Diabetes, 2017, 9(7): 699–706. DOI:10.1111/1753-0407.12478 |
| [12] | Rönn T, Ling C. DNA methylation as a diagnostic and therapeutic target in the battle against Type 2 diabetes[J]. Epigenomics, 2015, 7(3): 451–460. DOI:10.2217/epi.15.7 |
| [13] | Babu M, Durga Devi T, Makinen P, et al. Differential promoter methylation of macrophage genes is associated with impaired vascular growth in ischemic muscles of hyper-lipidemic and type 2 diabetic mice:a genome-wide promoter methylation study[J]. Circ Res, 2015, 117(3): 289–299. DOI:10.1161/CIRCRESAHA.115.306424 |
| [14] | Mudry JM, Lassiter DG, Nylén C, et al. Insulin and glucose alter death-associated protein kinase 3(DAPK3) DNA methylation in human skeletal muscle[J]. Diabetes, 2017, 66(3): 651–662. DOI:10.2337/db16-0882 |
| [15] | Simar D, Versteyhe S, Donkin I, et al. DNA methylation is altered in B and NK lymphocytes in obese and type 2 diabetic human[J]. Metab Clin Exp, 2014, 63(9): 1188–1197. DOI:10.1016/j.metabol.2014.05.014 |
| [16] | Zhao J, Goldberg J, Bremner JD, et al. Global DNA methylation is associated with insulin resistance:a monozygotic twin study[J]. Diabetes, 2012, 61(2): 542–546. DOI:10.2337/db11-1048 |
| [17] | Yang BT, Dayeh TA, Kirkpatrick CL, et al. Insulin promoter DNA methylation correlates negatively with insulin gene expression and positively with HbA(1c) levels in human pancreatic islets[J]. Diabetologia, 2011, 54(2): 360–367. DOI:10.1007/s00125-010-1967-6 |
| [18] | Kuroda A, Rauch TA, Todorov I, et al. Insulin gene expression is regulated by DNA methylation[J]. PLoS One, 2009, 4(9): e6953. DOI:10.1371/journal.pone.0006953 |
| [19] | Yang BT, Dayeh TA, Volkov PA, et al. Increased DNA methylation and decreased expression of PDX-1 in pancreatic islets from patients with type 2 diabetes[J]. Mol Endocrinol, 2012, 26(7): 1203–1212. DOI:10.1210/me.2012-1004 |
| [20] | Dayeh T, Volkov P, Salö S, et al. Genome-wide DNA methylation analysis of human pancreatic islets from type 2 diabetic and non-diabetic donors identifies candidate genes that influence insulin secretion[J]. PLoS Genet, 2014, 10(3): e1004160. DOI:10.1371/journal.pgen.1004160 |
| [21] | Ling C, Del Guerra S, Lupi R, et al. Epigenetic regulation of PPARGC1A in human type 2 diabetic islets and effect on insulin secretion[J]. Diabetologia, 2008, 51(4): 615–622. DOI:10.1007/s00125-007-0916-5 |
| [22] | Hall E, Volkov P, Dayeh T, et al. Effects of palmitate on genome-wide mRNA expression and DNA methylation patterns in human pancreatic islets[J]. BMC Med, 2014, 12: 103. DOI:10.1186/1741-7015-12-103 |
| [23] | Gillberg L, Ling C. The potential use of DNA methylation biomarkers to identify risk and progression of type 2 diabetes[J]. Front Endocrinol (Lausanne), 2015, 6: 43. |
| [24] | Lindholm ME, Marabita F, Gomez-Cabrero D, et al. An integrative analysis reveals coordinated reprogramming of the epigenome and the transcriptome in human skeletal muscle after training[J]. Epigenetics, 2014, 9(12): 1557–1569. DOI:10.4161/15592294.2014.982445 |
| [25] | Sims RJ 3rd, Reinberg D. Is there a code embedded in proteins that is based on post-translationalmodifications?[J]. Nat Rev Mol Cell Biol, 2008, 9(10): 815–820. DOI:10.1038/nrm2502 |
| [26] | Berger SL. The complex language of chromatin regulation during transcription[J]. Nature, 2007, 447(7143): 407–412. DOI:10.1038/nature05915 |
| [27] | Ahmed M, de Winther MPJ, Van den Bossche J. Epigenetic mechanisms of macrophage activation in type 2 diabetes[J]. Immunobiology, 2017, 222(10): 937–943. DOI:10.1016/j.imbio.2016.08.011 |
| [28] | Zimmet P, Alberti KG, Shaw J. Global and societal implications of the diabetes epidemic[J]. Nature, 2001, 414(6865): 782–787. DOI:10.1038/414782a |
| [29] | Kawada Y, Asahara SI, Sugiura Y, et al. Histone deacetylase regulates insulin signaling via two pathways in pancreatic β cells[J]. PLoS One, 2017, 12(9): e0184435. DOI:10.1371/journal.pone.0184435 |
| [30] | Daneshpajooh M, Bacos K, Bysani M, et al. HDAC7 is overexpressed in human diabetic islets and impairs insulin secretion in rat islets and clonal beta cells[J]. Diabetologia, 2017, 60(1): 116–125. DOI:10.1007/s00125-016-4113-2 |
| [31] | Jenuwein T. The epigenetic magic of histone lysine methylation[J]. FEBS J, 2006, 273(14): 3121–3135. DOI:10.1111/ejb.2006.273.issue-14 |
| [32] | Marmorstein R, Trievel RC. Histone modifying enzymes:structures, mechanisms, and specificities[J]. Biochim Biophys Acta, 2009, 1789(1): 58–68. DOI:10.1016/j.bbagrm.2008.07.009 |
| [33] | Watson M, Chow S, Barsyte D, et al. The study of epigenetic mechanisms based on the analysis of histone modification patterns by flow cytometry[J]. Cytometry A, 2014, 85(1): 78–87. DOI:10.1002/cyto.a.22344 |
| [34] | Du J, Johnson LM, Jacobsen SE, et al. DNA methylation pathways and their crosstalk with histone methylation[J]. Nat Rev Mol Cell Biol, 2015, 16(9): 519–532. DOI:10.1038/nrm4043 |
| [35] | Reddy MA, Natarajan R. Epigenetic mechanisms in diabetic vascular complications[J]. Cardiovasc Res, 2011, 90(3): 421–429. DOI:10.1093/cvr/cvr024 |
| [36] | Miao F, Chen Z, Zhang L, et al. Profiles of epigenetic histone post-translational modifications at type 1 diabetes susceptible genes[J]. J Biol Chem, 2012, 287(20): 16335–16345. DOI:10.1074/jbc.M111.330373 |
| [37] | Greiβel A, Culmes M, Napieralski R, et al. Alternation of histone and DNA methylation in human atherosclerotic carotid plaques[J]. Thromb Haemost, 2015, 114(2): 390–402. |
| [38] | Miao F, Gonzalo IG, Lanting L, et al. In vivochromatin remodeling events leading to inflammatory gene transcription under diabetic conditions[J]. J Biol Chem, 2004, 279(17): 18091–18097. DOI:10.1074/jbc.M311786200 |
| [39] | Reddy MA, Zhang E, Natarajan R. Epigenetic mechanisms in diabetic complications and metabolic memory[J]. Diabetologia, 2015, 58(3): 443–455. DOI:10.1007/s00125-014-3462-y |
| [40] | Guo XR, Wang XL, Li MC, et al. PDX-1 mRNA-induced reprogramming of mouse pancreas-derived mesenchymal stem cells into insulin-producing cells in vitro[J]. Clin Exp Med, 2015, 15(4): 501–509. DOI:10.1007/s10238-014-0319-0 |
| [41] | Avrahami D, Kaestner KH. Epigenetic regulation of pancreas development and function[J]. Semi Cell Develop Biol, 2012, 23(6): 213–223. |
| [42] | Chen H, Gu X, Su IH, et al. Polycomb protein Ezh2 regulates islets pancreatic beta-cell in diabetes mellitus[J]. Genes Dev, 2009, 23(8): 975–985. DOI:10.1101/gad.1742509 |
| [43] | Chakrabarti SK, Francis J, Ziesmann SM, et al. Covalent histone modifications underlie the developmental regulation of insulin gene transcription in pancreatic beta cells[J]. J Biol Chem, 2003, 278(26): 23617–23623. DOI:10.1074/jbc.M303423200 |
| [44] | Francis J, Chakrabarti SK, Garmey JC, et al. Pdx-1 links histone H3-Lys-4 methylation to RNA polymerase Ⅱ elongation during activation of insulin transcription[J]. J Biol Chem, 2005, 280(43): 36244–36253. DOI:10.1074/jbc.M505741200 |
| [45] | Zhang J. The direct involvement of SirT1 in insulin-induced insulin receptor substrate-2 tyrosine phosphorylation[J]. J Biol Chem, 2007, 282(47): 34356–34364. DOI:10.1074/jbc.M706644200 |
| [46] | Rausa FM 3rd, Hughes DE, Costa RH. Stability of the hepatocyte nuclear factor 6 transcription factor requires acetylation by the CREB-binding protein coactivator[J]. J Biol Chem, 2004, 279(41): 43070–43076. DOI:10.1074/jbc.M407472200 |
| [47] | Kumar S, Pamulapati H, Tikoo K. Fatty acid induced metabolic memory involves alterations in renal histone H3K36me2 and H3K27me3[J]. Mol Cell Endocrinol, 2016, 422: 233–242. DOI:10.1016/j.mce.2015.12.019 |
| [48] | Gupta J, Kumar S, Li J, et al. Histone H3 lysine 4 monomethylation (H3K4me1) and H3 lysine 9 monomethylation (H3K9me1):distribution and their association in regulating gene expression under hyperglycaemic/hyperinsulinemic conditions in 3T3 cells[J]. Biochimie, 2012, 94(12): 2656–2664. DOI:10.1016/j.biochi.2012.08.011 |
 2018, Vol. 44
2018, Vol. 44


