扩展功能
文章信息
- 许文舒, 胡正, 杨永广, 王晓丹
- XU Wenshu, HU Zheng, YANG Yongguang, WANG Xiaodan
- 人抑癌基因p53与PTEN的CRISPR-Cas9导向RNA靶点筛选与效率验证
- Screening and efficiency verification of CRISPR-Cas9 single-guide RNA against human tumor suppressor gene p53 and PTEN
- 吉林大学学报(医学版), 2018, 44(02): 235-242
- Journal of Jilin University (Medicine Edition), 2018, 44(02): 235-242
- 10.13481/j.1671-587x.20180206
-
文章历史
- 收稿日期: 2018-01-17
与正常细胞比较,癌细胞无限增殖、转化并易转移的特征严重威胁人体的健康与生存。而在癌细胞的转化与产生过程中,往往伴随着原癌基因或抑癌基因的突变或异常。肿瘤抑制基因p53是抑癌基因家族中至关重要的一员,恶性肿瘤中半数以上可检测到该基因突变,其编码的正常P53蛋白作为检验点调控细胞周期,监察细胞状态,进而在细胞基因损伤或变异时诱导细胞周期阻滞、修复及凋亡的发生。p53已广泛应用于结直肠癌、胃癌和肝癌等常见恶性肿瘤的形成机制探索及诊疗中[1-2]。抑癌基因PTEN的突变同样会导致多种肿瘤的发生。该基因编码的磷酸酶蛋白PTEN的作用与P53相似,能够通过细胞周期对细胞状态进行监控,防止细胞生长分裂过速,同时发挥抑制细胞迁移及血管生发等功能以减缓癌细胞的转移与扩散[3]。因此,在原代细胞中如能同时对p53与PTEN这对关键抑癌基因进行敲除性突变将会有效诱导原代细胞癌变。规律性成簇的间隔短回文重复序列结合核酸酶系统(CRISPR-Cas9)是一种细菌和古生菌中抵御外源病毒或质粒入侵的获得性免疫系统。来源于外源病毒或质粒序列片段的导向RNA(single-guide RNA,sgRNA)可介导Cas9特异性识别相应病原DNA分子并进行双链切割,保护细菌及古生菌自身。2013年,人们开始利用CRISPR-Cas9系统的这一特性在哺乳动物中实现了特定靶基因的定点编辑,开启了基因编辑的新纪元[4-7]。
利用基因编辑途径构建的肿瘤动物模型与其他肿瘤诱导模型相比更接近正常病程的发生与发展,为药物及治疗方法的探索提供了更为适合的研究对象[8-11]。此前已有报道通过尾静脉高压注射将表达小鼠p53和PTEN的sgRNA与Cas9质粒注射至小鼠体内,成功诱导小鼠肝脏原位肿瘤发生[12-13]。本研究设计靶向人p53及PTEN基因的sgRNA序列,并在体外检测其诱导相应基因特异位点突变的效果,为将原代细胞向肿瘤细胞转化以及在重建人肝脏的人源化小鼠中建立人自发肝脏肿瘤模型提供肿瘤诱导工具。
1 材料与方法 1.1 细胞、主要试剂和仪器Trans1-T1感受态细胞和pEASY-Blunt zero cloning kit(中国北京全式金公司),DMEM培养基、胎牛血清和Opti-MEM (美国Gibco公司),SanPrep柱式质粒DNA小量抽提试剂盒(中国上海生工公司),Annealing Buffer(中国上海碧云天公司),转染试剂Lipofectamine2000(美国Invitrogen公司),基因提取试剂盒(美国Axygen公司),PCR所需试剂(日本TaKaRa公司),200 bp Marker(中国北京Tiangen公司),凝胶回收试剂盒(德国QIAGEN公司),CRISPR-Cas9载体pL-CRISPR.EFS.GFP(美国Addgene公司)。FasrtDigest Esp3I kit和微型离心机(美国Thermo公司),微量移液器(美国RAININ公司),PCR仪和凝胶成像系统(美国BIO RAD公司)。
1.2 p53/PTEN sgRNA-pL-CRISPR.EFS.GFP质粒构建 1.2.1 获得用于构建sgRNA表达质粒的引物将靶基因外显子序列输入在线工具(http://tools.genome-engineering.org),结合所得靶序列在外显子中的位置及其可能存在的脱靶位点预测分数,初选出2组PTEN靶序列:PTEN-1,5′-TTATCCAAACATTATTGCTA-3′;PTEN-2,5′-ACCGCCAAATTTAATTGCAG-3′。其褪火引物分别为PTEN sg1 Forward:5′-CACCGTTATCCAAACATTATTGCTA-3′,PTEN sg1 Reverse:5′-AAACTAGCAATAATGTTTGGA-TAAC-3′;PTEN sg2 Forward: 5′-CACCGACCGCCAAA TTTAATTGCAG-3′,PTEN sg2 Reverse: 5′-AAACCTGCAATTAAATTTGGCGGTC-3′2组p53靶序列如下:p53-1,5′-TCGACGCTAGGATCTGACTG-3′;p53-2,5′-CGTCGAGCCCCCTCTGAGTC-3′。其褪火引物分别为p53 sg1 Forward:5′-CACCGTCGACGC-TAGGATCTGACTG-3′,p53 sg1 Reverse:5′-AAACCAGTCAGATCCTAGCGTCGAC-3′;p53 sg2 Forward: 5′-CACCGCGTCGAGCCCCCTCTGAGTC-3′,p53 sg2 Reverse: 5′-AAACGACTCAGAGGGGGCTCGACGC-3′。
1.2.2 获取sgRNA插入片段并将其与CRISPR质粒载体连接将sgRNA引物经褪火-稀释-连接至Esp3Ⅰ酶切并胶回收的pL-CRISPR.EFS.GFP质粒载体上。褪火:20μL褪火体系包含正义链引物5 μL(200 μmol·L-1),反义链引物5 μL(200 μmol·L-1),Annealing Buffer 2 μL,ddH2O 8 μL。褪火步骤:95℃,4 min;0.1℃·s-1,-1.5℃每个循环,48个循环;22℃、∞。稀释:取2 μL褪火产物,加98 μL ddH2O混匀;再取2 μL与20 μL Annealing Buffer和178 μL ddH2O混匀。连接:20 μL连接反应体系包含10× T4 ligase buffer 2 μL,Esp3Ⅰ酶切回收的CRISPR载体10 ng,T4 ligase 1 μL,褪火产物1 μL(5 nmol·L-1),ddH2O补足20 μL。混匀后16℃连接过夜。
1.2.3 连接产物转化加连接产物于50 μL Trans1-T1感受态细胞(在感受态细胞刚刚解冻时加入连接产物)中,轻弹混匀,冰浴20~30 min。42℃水浴热激30 s,立即置于冰上2 min;加600 μL常温的无抗性LB培养基,200 r·min-1、37℃培养1 h;菌液1500× g离心1 min,弃掉上清部分,保留100~150 μL,轻弹悬浮菌体,取全部菌液涂板(Amp抗性),37℃培养过夜。
1.2.4 提取并鉴定sgRNA-Cas9共表达质粒取3 mL过夜培养的菌液,8000× g离心2 min收集菌体,弃尽培养基;在沉淀中加入250 μL Buffer P1,彻底悬浮菌体;加入250 μL Buffer P2,立即温和颠倒EP管5~10次混匀。室温静置2~4 min;加入350 μL Buffer P3立即温和颠倒EP管5~10次混匀;12000× g离心5~10 min。将上清液移入吸附柱,8000× g离心30 s,倒掉收集管中液体;加入500 μL Buffer DW1,9000× g离心30 s,倒掉收集管中液体;加入500 μL Wash Solution,9000× g离心30 s,倒掉收集管中液体;重复上一步骤;空吸附柱于9000× g离心1 min;将吸附柱放入一个干净的1.5 mL EP管中,在吸附膜中央加入30~60 μL ddH2O,室温静置1 min后,离心1 min,-20℃保存管中DNA溶液。通过DNA测序鉴定sgRNA是否成功连接进入质粒载体,选择连接正确质粒大量提取,进行转染实验。
1.3 sgRNA-Cas9共表达质粒载体转染于6孔板中培养293T细胞,待其处于对数生长期且每孔细胞覆盖率至80%左右时进行实验,并于实验前一天换无双抗DMEM完全培养基培养过夜;用微量移液器吸取0.3 mL Opti-MEM与5 μL Lipofectamine 2000于1.5 mL EP管中混匀,另取0.3 mL Opti-MEM与2 μg sgRNA质粒于另一1.5 mL EP管中混匀,二者于室温下静置5 min后加入同一EP管混匀再在室温下静置20 min;小心吸去6孔板中293T细胞原有的培养基,贴壁轻轻加入上一步的混合液,并于37℃培养箱中培养6 h;6 h后,吸出6孔板中液体并加2 mL DMEM完全培养基正常培养;24 h后采用荧光显微镜观察细胞绿色荧光蛋白(green fluoresence protein,GFP)表达情况并拍照,GPF阳性细胞即为成功转染质粒的细胞;将培养基吸出后进行细胞分选,通过流式细胞术检测分选前后GFP阳性细胞在总细胞中所占比例,将分选获得的GFP阳性细胞继续培养。待细胞扩增到一定数量后部分冻存,剩余细胞提基因组DNA。
1.4 基因组DNA提取实验分为对照组[野生型(wild type, WT)细胞]和实验组。分别提取2组细胞的基因组DNA:用2 mL EP管收集含2×106个细胞的悬浮液,2000× g离心5 min,弃尽上清,并用350 μL PBS重悬;加0.8 μL RNase A,漩涡震荡15 s,室温静置1 min;加入150 μL Buffer C-L和8 μL Proteinase K,立即漩涡振荡1 min混合均匀,短暂离心后,将EP管置于56℃水浴10 min;加入350 μL Buffer P-D,漩涡振荡30 s混合均匀,12000× g离心10 min;将DNA制备管置于2 mL EP管中,将上一步的混合液上清移至制备管中,12000× g离心1 min;弃滤液,将制备管置回到原来的2 mL EP管中,加入500 μL Buffer W1,12000× g离心1 min;弃滤液,将制备管置回到原来的2 mL EP管中,加入700 μL Buffer W2,12000× g离心1 min,以同样的方法,用700 μL Buffer W2再洗涤1次;弃滤液,将制备管置回原来的2 mL EP管中,12000× g离心1 min;将DNA制备管置于另一洁净的1.5 mL EP管中,在制备管膜中央加100 μL去离子水,室温静置1 min,12000× g离心1 min洗脱DNA。产物-20℃保存,并作为下一步的PCR模板。
1.5 包含突变位点的基因组片段PCR扩增50 μL PCR体系:5×buffer 10 μL,dNTP 4 μL,上游引物1 μL,下游引物1 μL,Primestar 1 μL,模板3 μL,ddH2O 30 μL。其中PTEN-1靶点效率检测上游引物:5′-GCCAGCTCTTAAATCTGACTTC-3′,下游引物:5′-CTGAAGTCCATTAGGTACGGT-3′,PCR产物理论长度为667 bp;PTEN-2靶点效率检测上游引物:5′-GAAAAAGGTGATCGTTGGCT-3′,下游引物:5′-GAAAAAGGTGATCGTTGGCT-3′,PCR产物理论长度为947 bp;p53-1,2靶点效率检测上游引物:5′-GAAGAGAGAATGTGAA-3′,下游引物:5′-CATTCTGGGAGCTTCATCTG-3′,PCR产物理论长度为612 bp。PCR反应条件:98℃、30 s,98℃、15 s,60℃、10 s,72℃、1 min10 s,34个循环后72℃、10 min。PCR结束后进行1%琼脂糖凝胶电泳,110 V、30 min,利用凝胶成像系统观察PCR产物片段条带。
1.6 胶回收用干净的手术刀片从琼脂糖凝胶中切下DNA片段,注意去除多余的凝胶块;称质量后每100μg凝胶中加入300μL缓冲液QG,50℃加热约10 min,直至凝胶完全溶解;向样品中加入与凝胶等体积的异丙醇并混合;将样品加到收集柱中,再放到收集管内,17900× g离心1 min;弃滤液,加500 μL QG,17900× g离心1 min;弃滤液,加750 μL缓冲液PE,17900× g离心1 min;弃滤液并17900× g空转离心1 min;将收集柱放入干净的1.5 mL EP管中,加30 μL ddH2O,室温静置1 min,17900× g洗脱离心1 min;滤液-20℃保存。
1.7 待测序回收产物克隆与突变效率检测5 μL反应体系:PCR Product 20 ng,pEASY-Blunt Zero Cloning Vector 1 μL,ddH2O补足5 μL,25℃反应30 min(PCR仪控温)。利用连接产物转化,单克隆培养8~16 h后挑单克隆,每个单克隆加3 mL Amp抗性LB培养基200 r·min-1、37℃培养过夜,并对菌液进行DNA测序。以未经转染的对照组细胞的基因序列为标准序列,通过统计测序结果中突变序列数量与全部送检样品数量比率来确定sgRNA诱导基因突变的效率。
2 结果 2.1 p53/PTEN sgRNA靶点通过数据库查询p53及PTEN基因的各个亚型mRNA,选择各基因mRNA亚型剪辑的共同外显子区域,将该区域序列输入在线工具(http://tools.genome-engineering.org)获得推荐靶点及靶点预测分数,选择针对p53及PTEN基因的CRISPR-Cas9 sgRNA靶点各2个:p53-1、p53-2、PTEN-1和PTEN-2。见表 1。
| (x±s) | |||
| Target name | Target locus | Score | Number of target sites |
| PTEN-1 | PTEN exon2 | 41 | 346(14 are in genes) |
| PTEN-2 | PTEN exon4 | 74 | 104(8 are in genes) |
| p53-1 | p53 exon2 | 91 | 33(2 are in genes) |
| p53-2 | p53 exon2 | 75 | 79(15 are in genes) |
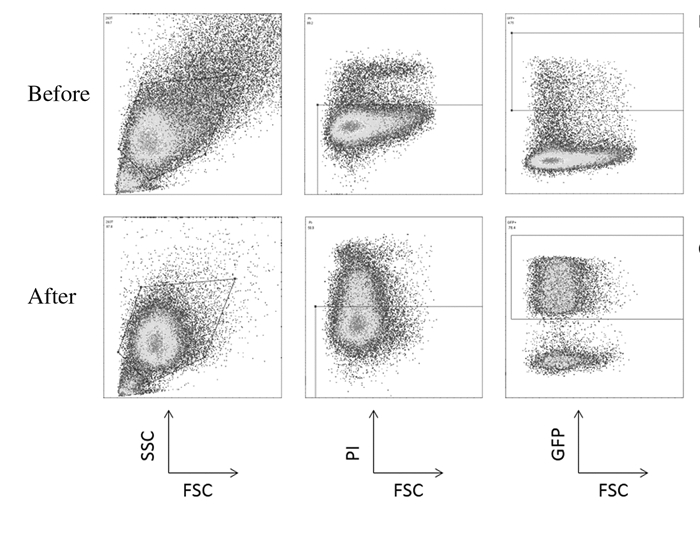
利用Lipofectamine 2000将共表达sgRNA与Cas9-P2A-GFP的CRISPR质粒转染进293T细胞内,24~48 h内通过荧光显微镜观察和流式细胞术分析均发现了3%GFP表达,证明质粒转染成功,并通过流式分选对表达GFP转染成功的细胞进行纯化。见图 1和2(插页二)。与分选前的荧光比率(4%)比较,分选后荧光比率升高至82%(P < 0.05),成功富集了质粒转染成功的293T细胞。
|
| 图 1 质粒转染流式细胞术结果 Figure 1 Results of flow cytometry of plasmid transfection |
|
|

|
| 图 2 质粒转染荧光观察结果(Bar=200 μm) Figure 2 Fluorescence observation results of plasmid transfection (Bar=200 μm) |
|
|
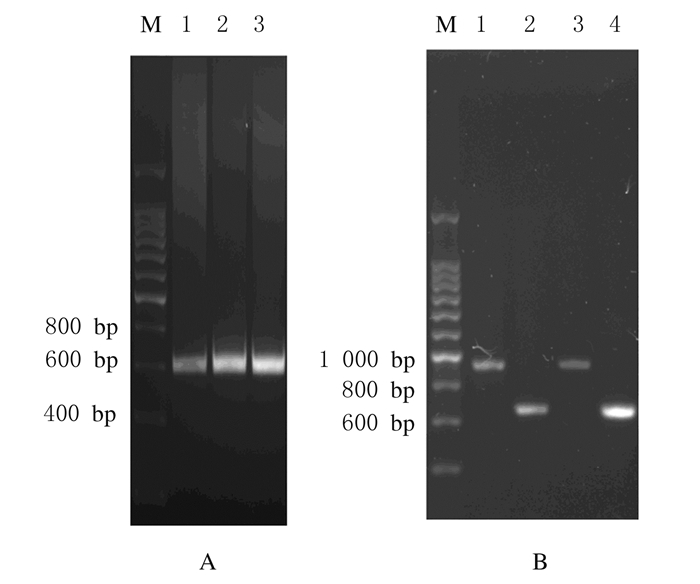
采用PCR法对含有突变区域的基因片段进行大量扩增,并利用电泳胶回收的方式对PCR产物片段进行纯化,避免非特异性扩增的片段对下一步测序产生干扰,其中p53扩增片段理论长度均为612 bp,而包含PTEN-1突变位点的扩增片段理论长度为667 bp,包含PTEN-2突变位点的扩增片段理论长度为947 bp。凝胶电泳结果显示:成功获得了预期大小的扩增片段。见图 3。
|
| A : p53 sgRNA plasmid transfected cells. M: 200 bp marker; Lane 1: p53 wt group; Lane 2: p53-1 sgRNA plasmid transfected group; Lane 3: p53-2 sgRNA plasmid transfected group. B: PTEN sgRNA plasmid transfected cells. M: 200 bp marker; Lane 1: PTEN-2 sgRNA plasmid transfected group; Lane 2: PTEN-1 sgRNA plasmid transfected group; Lane 3: PTEN-2 wt group; Lane 4: PTEN-1 wt group. 图 3 PCR产物电泳图 Figure 3 Electrophoregram of PCR products |
|
|
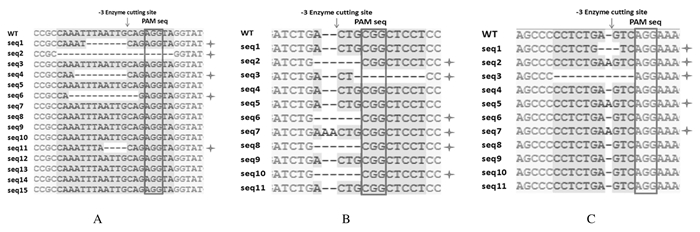
将胶回收产物与空载体进行连接,经转化扩增后进行测序,将实验组与对照组野生型基因序列进行对比,确定sgRNA质粒能否对基因进行成功编辑及编辑效率。见图 4。通过计算测序结果中突变序列数量与全部送检数量比率,可以得出靶序列p53-1、p53-2和PTEN-2诱导的突变效率分别为54.5%、45.5%和33.3%。
|
| A: PTEN-2 sgRNA; B: p53-1 sgRNA; C: p53-2 sgRNA. Wt: Wild type.╈ indicated the mutated sequences. 图 4 PCR产物的测序结果 Figure 4 Sequencing results of PCR products |
|
|
对实验整体设计思路及流程进行总结(图 5),将实验分为两大部分,上游sgRNA靶点设计与克隆构建作为步骤1(sgRNA设计与质粒构建),下游细胞操作与基因编辑功能验证部分作为步骤2(质粒转染与细胞分选及基因编辑效率分析)。

|
| 图 5 基因编辑效率的获取方案示意图 Figure 5 Schematic representations of gene editing efficiency obtaining |
|
|
构建诱发式肿瘤模型的传统方法主要分为物理方法(如射线辐照)、化学方法(化学致癌物)和生物学方法(病毒感染、肿瘤细胞接种等)。但其中大部分方法效率较低、时程较长,且难以模拟正常情况下人体自发形成肿瘤、免疫系统随之产生应答的相互作用过程。
基因编辑技术是一种通过程式化的重组核酸酶对DNA序列进行改造的基因操作技术,通过核酸内切酶对序列的靶向定位,对DNA双链进行识别与切割,产生双链断裂,在机体DNA损伤的非同源重组修复过程中,有很高概率发生核苷酸的插入或缺失,当插入或删除碱基数不为3的整数倍时,将导致修复后的基因发生移码突变,引起基因功能丧失,获得基因敲除的效果[14-15]。
目前,较为主流的基因编辑技术主要包括ZFN技术、TALEN技术及近年来发展迅猛的CRISPR-Cas9技术。CRISPR-Cas9系统的主要功能元件包括sgRNA和Cas9核酸内切酶。与前2种技术相比,CRISPR-Cas9简单、高效,且可以通过导入多个sgRNA的方法同时进行多靶点的编辑,因此本文作者选择CRISPR-Cas9技术进行p53与PTEN抑癌基因的编辑,以便应用于后期这2个基因在原代细胞乃至动物模型中的同时编辑性敲除[16-17]。
肿瘤的发生开端往往始于体内细胞增殖异常。究其根源,抑癌基因被抑制或突变是其中的重要因素。p53和PTEN是人体抑癌基因家族中至关重要的成员,广泛表达于全身不同组织器官,在阻滞细胞周期、抑制细胞增殖和诱导细胞凋亡等方面有重要作用。其作用方式是通过编码表达相关蛋白,或者直接参与代谢,或者通过影响信号通路等过程调节细胞的生理代谢途径,达到抑制肿瘤发生的作用。在肿瘤形成后p53和PTEN还会进一步阻止细胞迁移及血管生成,最终达到阻碍肿瘤发展的目的。因而当这2个基因同时缺失或抑制时,可以高效地诱导细胞向肿瘤细胞转化[18-21]。本研究针对这2个基因设计并选择了sgRNA对其进行特异性编辑,以期获得p53和PTEN协同性功能缺失型突变乃至移码突变型敲除,为在细胞水平及活体水平建立肿瘤诱导模型提供工具[22-23]。
本研究以选择p53及PTEN存在的多个亚型的共同外显子为原则进行软件分析,选择基于脱靶位点的评分数较高的sgRNA靶点。在sgRNA设计选择过程中,由于基因组DNA在生物体内的复杂折叠状态以及核小体蛋白复合物的遮蔽,软件基于脱靶位点的评分高低并不能代表靶点在生物体内的实际切割效率,因而需要进行前期体外工作来确定所选择的靶点能够介导Cas9执行基因编辑的实际效率。这也是本研究的主要工作内容。
具体操作中,切割pL-CRISPR.EFS.GFP质粒使其线性化,该质粒的sgRNA克隆位点两侧为一对Esp3Ⅰ内切酶,该酶切割位点在其识别位点的下游因而成对使用时可以产生不易导致质粒自连的黏性末端,通过合成sgRNA序列引物并褪火,可以获得具有同样黏性末端的sgRNA序列DNA双链并连接,挑取克隆测序,选择正确插入sgRNA的pL-CRISPR.EFS.GFP质粒。随后采用转染试剂Lipofectamine2000把测序鉴定过的p53/PTEN sgRNA-Cas9系统导入293T细胞,利用Cas9质粒表达GFP的特性进行分选富集,获得成功转染质粒的细胞,扩大培养后提取基因组,并以包含突变区域的序列为模板进行PCR扩增,对片段纯化后通过测序方式与对照组野生型序列进行对比来确定所选sgRNA靶点对基因靶向编辑的效果。
对于CRISPR-Cas9系统,基因组上靶序列毗邻序列需为NGG,即前间区序列毗邻基序PAM,而Cas9的理论切割位点位于PAM前3位碱基对间。根据序列对比结果分析,野生型对照组靶位点PAM周围序列与NCBI网站基因库提供的标准序列一致,而p53-1、p53-2和PTEN-2实验组靶位点PAM周围序列在预期切割位点(PAM-3位)出现了较大量的突变,说明它们能够有效介导Cas9进行p53及PTEN基因的位点特异性编辑,同时发现碱基缺失占突变的大部分,只有少数发生了碱基插入。
通过以上一系列体外实验,本文作者确定了能介导Cas9对p53和PTEN基因进行有效编辑的sgRNA序列并建立了方案,通过分选初步富集发生了基因编辑的细胞,为进一步研究p53/PTEN基因突变细胞成瘤性奠定了基础。有效sgRNA序列的确定也为在原代细胞向肿瘤细胞转化以及人源化鼠中人原位肿瘤模型的构建完成了部分前期工作。
| [1] | Agarwal S, Bell CM, Taylor SM, et al. p53 deletion or hot-spot mutations enhance mTORC1 activity by altering lysosomal dynamics of TSC2 and Rheb[J]. Mol Cancer Res, 2016, 14(1): 66–77. DOI:10.1158/1541-7786.MCR-15-0159 |
| [2] | Kruiswijk F, Labuschagne CF, Vousden KH. p53 in survival, death and metabolic health:a lifeguard with a licenceto kill[J]. Nat Rev Mol Cell Biol, 2015, 16(7): 393–405. DOI:10.1038/nrm4007 |
| [3] | Song Z, Han X, Shen L, et al. PTEN silencing enhances neuronal proliferation and differentiation by activating PI3K/Akt/GSK3β pathway in vitro[J]. Exp Cell Res, 2018, 4827(18): 30001–30006. |
| [4] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6069): 816–821. |
| [5] | Chou WC, Lin PH, Yeh YC, et al. Genes involved in angiogenesis and mTOR pathways are frequently mutated in Asian patients with pancreatic neuroendocrine tumors[J]. Int J Biol Sci, 2016, 12(12): 1523–1532. DOI:10.7150/ijbs.16233 |
| [6] | Zhang Z, Zhang Y, Gao F, et al. CRISPR-Cas9 Genome-Editing System in Human Stem Cells:Current Status and Future Prospects[J]. Mol Ther Nucleic Acids, 2017, 15(9): 230–241. |
| [7] | Cribbs AP, Perera SMW. Science and bioethics of CRISPR-Cas9 gene editing:an analysis towards separating facts and fiction[J]. Yale J Biol Med, 2017, 90(4): 625–634. |
| [8] | Sánchez-Rivera FJ, Jacks T. Applications of the CRISPR-Cas9 system in cancer biology[J]. Nat Rev Cancer, 2015, 15(7): 387–395. DOI:10.1038/nrc3950 |
| [9] | Korkmaz G, Lopes R, Ugalde AP. Functional genetic screens for enhancer elements in the human genome using CRISPR-Cas9[J]. Nat Biotechnol, 2016, 34(2): 192–198. DOI:10.1038/nbt.3450 |
| [10] | Abbasi F, Miyata H, Ikawa M. Revolutionizing male fertility factor research in mice by using the genome editing tool CRISPR-Cas9[J]. Reprod Med Biol, 2017, 17(1): 3–10. |
| [11] | Bhat SA, Malik AA, Ahmad SM, et al. Advances in genome editing for improved animal breeding:A review[J]. Vet World, 2017, 10(11): 1361–1366. DOI:10.14202/vetworld |
| [12] | Xue W, Chen SD, Yin H. CRISPR-mediated direct mutation of cancer genes in the mouse liver[J]. Nature, 2014, 514(7522): 380–384. DOI:10.1038/nature13589 |
| [13] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819–823. DOI:10.1126/science.1231143 |
| [14] | de Lecuona I, Casado M, Marfany G, et al. Gene editing in humans:towards a global and inclusive debate for responsible research[J]. Yale J Biol Med, 2017, 90(4): 673–681. |
| [15] | Wyvekens N, Tsai SQ, Joung JK. Genome editing in human cell using CRISPR/Cas nucleases[J]. Curr Protoc Mol Biol, 2015, 112: 31–33. |
| [16] | Wang X, Huang X, Fang X, et al. CRISPR-Cas9 system as a versatile tool for genome engineering in human cells[J]. Mol Ther Nucleic Acids, 2016, 5(11): 388–395. |
| [17] | Ma D, Liu F. Genome editing and its applications in model organisms[J]. Genomics Proteomics Bioinformatics, 2015, 13(6): 336–344. DOI:10.1016/j.gpb.2015.12.001 |
| [18] | Pan Y, Bush EC, Toonen JA, et al. Whole tumor RNA-sequencing and deconvolution reveal a clinically-prognostic PTEN/PI3K-regulated glioma transcriptional signature[J]. Oncotarget, 2017, 8(32): 52474–52487. |
| [19] | Sun MM, Zhang MZ, Chen Y, et al. Effect of PTEN antisense oligonucleotide on oesophageal squamous cell carcinoma cell lines[J]. J Int Med Res, 2012, 40(6): 2098–2108. DOI:10.1177/030006051204000607 |
| [20] | Contractor T, Harris CR. p53 negatively regulates transcription of the pyruvate dehydrogenase kinase Pdk2[J]. Cancer Res, 2012, 72(2): 560–567. DOI:10.1158/0008-5472.CAN-11-1215 |
| [21] | Kruiswijk F, Labuschagne CF, Vousden KH. p53 in survival, death and metabolic health:a lifeguard with a licence to kill[J]. Nat Rev Mol Cell Biol, 2015, 16(7): 393–405. DOI:10.1038/nrm4007 |
| [22] | Heckl D, Kowalczyk MS, Yudovich D, et al. Generation of mouse models of myeloid malignancy with combinatorial genetic lesions using CRISPR-Cas9 genome editing[J]. Nat Biotechnol, 2014, 32(9): 941–946. DOI:10.1038/nbt.2951 |
| [23] | Luo J. CRISPR/Cas9:from genome engineering to cancer drug discovery[J]. Trends Cancer, 2016, 2(6): 313–324. DOI:10.1016/j.trecan.2016.05.001 |
 2018, Vol. 44
2018, Vol. 44


