扩展功能
文章信息
- 闫风琴, 范如意, 王方正, 王磊, 叶智敏, 傅真富, 王建秋
- YAN Fengqin, FAN Ruyi, WANG Fangzheng, WANG Lei, YE Zhimin, FU Zhenfu, WANG Jianqiu
- 乏氧对人口腔鳞状细胞癌细胞中叉头蛋白P3表达的影响及其机制
- Effect of hypoxia on forkhead box P3 expression in human oral squamous cell carcinoma cells and its mechanism
- 吉林大学学报(医学版), 2017, 43(03): 491-495
- Journal of Jilin University (Medicine Edition), 2017, 43(03): 491-495
- 10.13481/j.1671-587x.20170306
-
文章历史
- 收稿日期: 2016-12-09
2. 杭州师范大学医学院生物化学与分子生物学教研室, 浙江 杭州 310036
2. Department of Biochemistry and Molecular Biology, School of Medical Sciences, Hangzhou Normal University, Hangzhou 310036, China
叉头蛋白P3(forkhead box P3, FOXP3) 是Fox蛋白家族成员,主要表达于造血细胞中,参与调节性T细胞的转录调节[1],研究[2]显示FOXP3也表达于多个谱系的上皮细胞和不同组织来源的非造血细胞以及肿瘤细胞中。本文作者在前期关于乏氧诱导口腔鳞状细胞癌(oral squamous cell carcinoma, OSCC)细胞侵袭和转移的研究结果显示:FOXP3基因启动子上的组蛋白修饰可能受到乏氧调节。组蛋白化学修饰的变化可调节周围染色质的结构,进而调节基因的表达[3],本文作者推测:乏氧可能调节了OSCC细胞中FOXP3的表达,目前尚未见相关报道。本研究采用人OSCC细胞株FaDu和OECM-1,以实时定量PCR和Western blotting法检测乏氧对FOXP3表达的影响,并采用染色质免疫沉淀(chromatin immunoprecipitation, ChIP)方法检测FOXP3基因启动子上组蛋白修饰H3K4乙酰化(H3K4ac)、H3K4三甲基化(H3K4me3) 和H3K27三甲基化(H3K27me3) 相对表达水平的变化,以明确乏氧对FOXP3表达的调节作用及可能的表观遗传学机制。
1 材料与方法 1.1 细胞株、主要试剂和仪器野生型人OSCC细胞株FaDu、OECM-1细胞和HDAC3基因沉默FaDu细胞株由台湾阳明大学生化暨分子生物研究所惠赠。改良型RPMI-1640培养基购自美国Thermo公司,胎牛血清购自美国Gibco公司,总RNA提取试剂RNAiso Plus、反转录试剂盒和Real-time qPCR试剂盒购自日本TaKaRa公司,HIF-1 (ab82832) 和FOXP3兔抗人多克隆抗体(ab20034) 均购自美国Abcam公司,Magna ChIPTM protein A+G magnetic beads、ChIPAb+acetyl-histone H3 (Lys4) 试剂盒、ChIPAb+ trimethyl-histone H3 (Lys4) 试剂盒和ChIPAb+ trimethyl-histone H3 (Lys27) 试剂盒均购自美国Millipore公司,蛋白A/G磁珠购自美国ThermoFisher公司。APM-30DR三气培养箱购自日本ASTEC公司,7500型荧光定量PCR仪购自美国ABI公司,Odyssey双色红外荧光扫描成像系统购自美国LI-COR公司。
1.2 细胞的常氧培养和乏氧处理常氧培养:取生长状态良好的FaDu细胞和OECM-1细胞,采用含10%胎牛血清的RPMI-1640培养基,于37℃、5%CO2条件下进行常规培养。乏氧处理:取常规培养的细胞,用含1%胎牛血清的RPMI-1640培养基换液,置于三气培养箱中,将氧浓度调整为1%后,继续培养18 h。
1.3 实时定量PCR法检测FaDu和OECM-1细胞中FOXP3 mRNA相对表达水平TRIzol法提取各组细胞总RNA,采用逆转录试剂盒将mRNA逆转录成cDNA。设计FOXP3特异性引物,上游为5′-AGGTCTGAGGCTTTGGGTG-3′,下游为5′-TTCTGTCAGTCCACTTCACCA-3′;设计18s RNA特异性引物(参照),上游为5′-GTAACCCGTTGAACCCCATT-3′,下游为5′-CCATCCAATCGGTAGTAGCG-3′,引物由上海捷瑞生物工程有限公司合成。采用SYBR-Green染料法进行定量PCR,循环条件:95℃预变性5 min; 95℃变性10 s,60℃复性30 s,72℃延伸20 s,40个循环。根据熔解曲线评估引物特异性。以18s RNA为内参,采用2-△△CT法对数据进行相对定量分析[4],每次实验设3个复孔,行3次独立重复试验。
1.4 Western blotting法检测FaDu和OECM-1细胞中FOXP3蛋白表达水平提取细胞总蛋白,Bradford法测定蛋白水平,取40μg蛋白样品行SDS聚丙烯酰胺凝胶电泳(5%积层胶,10%分离胶)。将凝胶上的蛋白转印到聚偏氟乙烯(PVDF)膜上。PVDF膜与FOXP3抗体(1:1 000) 在4℃摇床下孵育过夜后,再与结合有荧光染料的二抗孵育。采用Odyssey红外扫描系统对PVDF膜进行扫描成像。以β-actin (抗体稀释比例为1:1 000) 作为内参照。
1.5 ChIP实验检测常氧和乏氧条件下FaDu细胞中FOXP3基因启动子上的组蛋白修饰的H3K4ac、H3K4me3和H3K27me3相对表达水平1%甲醛固定不同处理因素(常氧/乏氧、HDAC3基因沉默/对照)处理的FaDu细胞10 min,依次裂解细胞膜和细胞核。超声破碎染色质DNA,使其片段长度分布于200~1 000 bp。以稀释缓冲液将染色质悬液稀释5倍,取2.5%作为对照(input),剩余部分4等分,分别加入抗H3K4ac、抗H3K4me3、抗H3K27me3抗体和兔正常IgG,于4℃摇床孵育过夜。应用蛋白A/G磁珠收集免疫沉淀复合物,充分洗涤后65℃水浴过夜。应用PCR产物提取试剂盒提取DNA,分别以总DNA(input)和各免疫沉淀DNA为模板,应用合成的FOXP3启动子特异性引物(上游为5′-TCCTCCCT-GCCCAACTACAA-3′,下游为5′-CGGCTAACA-ATCAGG GGCAT-3′),SYBR green染色法进行Real-time PCR扩增。结果以免疫沉淀DNA与总DNA(input)的相对表达水平(%input)表示,相对表达水平=2^[ct(input)-ct(chip)]×稀释倍数。以兔正常IgG沉淀DNA与input DNA的相对表达水平(%input)作为阴性对照。
1.6 统计学分析采用SPSS 20.0统计软件进行统计学分析。FaDu和OECM-1细胞中FOXP3 mRNA相对表达水平、FaDu细胞中FOXP3基因启动子组蛋白修饰H3K4ac、H3K4me3和H3K7me3相对表达水平均以x±s表示,组间均值比较采用两独立样本t检验,多组间比较采用方差分析。以α=0.05为检验水准。
2 结果 2.1 FaDu和OECM-1细胞中FOXP3 mRNA相对表达水平乏氧处理18 h后,FaDu细胞中FOXP3 mRNA相对表达水平仅为常氧条件下的(34.4±2.4)% (P<0.01),OECM-1细胞中则仅为常氧条件下的(24.3±1.6)%(P<0.01),下降幅度分别达65.6%和75.7%。见表 1。
| (n=3, x±s) | |||
| Cell line | Relative expression level of FOXP3 mRNA | ||
| Normoxia | Hypoxia | Hypoxia/normoxia (η/%) | |
| FaDu | 3.12±0.27 | 1.07±0.07* | 34.4±2.4 |
| OECM-1 | 7.72±0.59 | 1.88±0.12* | 24.3±1.6 |
| *P < 0.05 compared with normoxic condition. | |||
Western blotting结果显示:乏氧条件下FaDu和OECM-1细胞中FOXP3蛋白表达水平均明显低于常氧条件。见图 1。
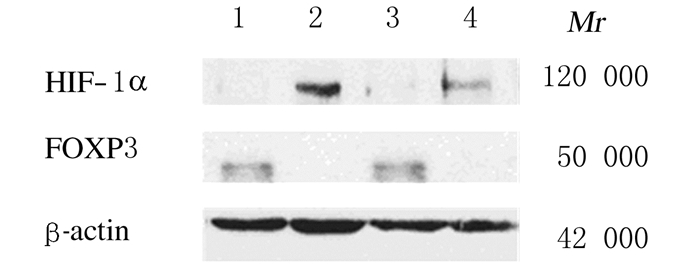
|
| Lane 1-2: FaDu cells; Lane 3-4: OECM-1 cells; Lane 1, 3: Normoxic condition; Lane 2, 4: Hypoxia condition. 图 1 常氧和乏氧条件下FaDu和OECM-1细胞中FOXP3蛋白表达电泳图 Figure 1 Electrophoregram of expressions of FOXP3 protein in FaDu and OECM-1 cells under normoxic or hypoxic conditions |
|
|
与常氧条件比较,乏氧条件下FaDu细胞中FOXP3基因启动子上H3K4ac和H3K4me3相对表达水平均明显降低(P<0.01),而H3K27me3水平无明显变化。见表 2。
| (n=3, x±s) | ||||||
| Group | α-H3K4ac | α-normal IgG | α-H3K4me3 | α-normal IgG | α-H3K27me3 | α-normal IgG |
| Normoxia | 2.05±0.24 | 0.05±0.04 | 2.74±0.18 | 0.09±0.02 | 2.20±0.14 | 0.17±0.04 |
| Hypoxia | 0.82±0.04* | 0.06±0.02 | 1.25±0.02* | 0.12±0.01 | 2.24±0.22 | 0.13±0.01 |
| * P < 0.01 compared with normoxic condition. | ||||||
qPCR法检测结果显示:与对照组FaDu细胞[(2.075±0.114)×10-3]比较,HDAC3基因沉默的FaDu细胞中HDAC3 mRNA相对表达水平[(0.485±0.024)×10-3]明显降低(P<0.01),表达抑制率为76.6%。
缺氧诱导的HDAC3基因沉默的FaDu细胞中FOXP3基因启动子上的H3K4ac和H3K4me3表达抑制率(乏氧/常氧)分别为(19.6±0.98)%和(36.5±1.6)%,与对照组FaDu细胞比较,其表达抑制率明显降低(P<0.05)。见表 3。
| (n=3, x±s) | |||||||
| Group | -H3K4ac | -H3K4me3 | |||||
| Normoxia | Hypoxia | Inhibitory rate (η/%) | Normoxia | Hypoxia | Inhibitory rate (η/%) | ||
| FaDu-control | 2.16+0.18 | 0.89±0.07 | 58.8±3.2 | 2.92±0.10 | 1.36±0.09 | 53.4±2.1 | |
| FaDu-HDAC3-si | 2.09±0.13 | 1.68±0.11 | 19.6±1.0* | 2.77±0.16 | 1.76±0.14 | 36.5±1.6* | |
| * P < 0.05 compared with FaDu-control group. | |||||||
基于HDAC3基因沉默FaDu细胞株的实时定量PCR结果显示:与无HDAC3基因沉默的FaDu细胞比较,HDAC3基因沉默的FaDu细胞中缺氧诱导的FOXP3mRNA表达抑制率明显降低(P<0.05)。见表 4。
| (n=3, x±s) | |||
| Group | Normoxia | Hypoxia | Inhibitory rate(η/%) |
| FaDu-control | 3.27±0.22 | 1.15±0.09 | 64.8±3.6 |
| FaDu-HDAC3-si | 2.92±0.59 | 2.16±0.12 | 26.0±1.4* |
| * P < 0.05 compared with FaDu-control group. | |||
OSCC是常见的上皮来源的恶性肿瘤,患者5年生存率不到50%,癌细胞转移是导致癌症患者死亡的重要因素[5]。乏氧是肿瘤微环境的基本特征之一,是促进OSCC侵袭和转移的重要因素[6]。乏氧激活缺氧诱导因子1α(hypoxia-inducible factor -1α, HIF-1α)进而诱导上皮细胞间质转化(epithelial-mesenchymal transition, EMT), 是上皮细胞来源的恶性肿瘤细胞获得迁移和侵袭能力的重要生物学过程[7]。肿瘤的生长、侵袭和转移均与肿瘤的异质性有关,而异质性又可以肿瘤细胞的表观遗传学紊乱来解释。表观遗传修饰主要通过改变染色质结构来影响基因的表达,主要包括DNA甲基化、组蛋白修饰和染色质重塑等[8]。本文作者前期[9]一项关于乏氧诱导OSCC细胞侵袭和转移的研究中通过高通量筛选发现:FOXP3基因启动子上的组蛋白修饰可能受到乏氧调节。组蛋白化学修饰的变化可调节周围染色质的结构,进而调节基因的表达[10],因此本文作者推测:FOXP3的表达可能受到乏氧的表观遗传学调节,并可能在乏氧诱导OSCC侵袭和转移中发挥作用。
本研究分别检测常氧和乏氧条件下2种OSCC细胞中FOXP3 mRNA和蛋白表达水平结果显示:乏氧处理的FaDu和OECM-1细胞中FOXP3 mRNA相对表达水平分别是常氧条件下的(34.4±2.4)%和(24.3±1.6)%,其蛋白表达水平也明显低于常氧组,表明乏氧抑制了FOXP3在OSCC细胞中的表达。本研究进一步采用ChIP-qPCR方法检测FOXP3基因启动子上的H3K4ac和H3K4me3修饰水平结果显示:两者均受到乏氧的影响而明显下调。组蛋白H3/H4的位点特异性乙酰化通常会引起基因转录激活,去乙酰化则导致转录抑制[11], 而组蛋白的甲基化和去甲基化修饰对基因的转录调节则是各不相同[12],且组蛋白H3K4位点的甲基化可调节该位点乙酰化的转录激活作用[13]。据此本文作者推断:乏氧通过对启动子上组蛋白特异性位点H3K4乙酰化和甲基化的协同调节实现了对FOXP3的转录抑制作用。组蛋白修饰H3K27me3与基因转录抑制密切关联[14],但本实验未检测到FOXP3启动子上该组蛋白修饰受到乏氧的调节,推测该修饰可能与乏氧对FOXP3的表达调节无关联。
组蛋白修饰的变化通常是由组蛋白修饰因子介导的,常见的组蛋白修饰因子主要包括一些组蛋白甲基转移酶(HMTs)/去甲基化酶(HDMs)、乙酰基转移酶(HATs)/去乙酰化酶(HDACs)等[15-16]。找到介导乏氧下调FOXP3启动子上H3K4ac水平的组蛋白修饰因子,将有助于阐明乏氧抑制FOXP3表达的表观遗传学机制。研究[17-18]显示:HIF-1α可直接激活肿瘤细胞的HDAC3,下调EMT标志基因启动子上的组蛋白修饰H3K4ac水平。本文作者利用实验室已有的HDAC3基因沉默FaDu细胞株发现:HDAC3基因沉默可明显改善乏氧诱导的FOXP3启动子上H3K4ac水平下调,并逆转乏氧对FOXP3表达的抑制作用。以上研究证实:HDAC3介导了乏氧诱导的FOXP3表达的抑制作用。
| [1] | Gerriets VA, Kishton RJ, Johnson MO, et al. Foxp3 and Toll-like receptor signaling balance Tregcell anabolic metabolism for suppression[J]. Nat Immunol, 2016, 17(12): 1459–1466. DOI:10.1038/ni.3577 |
| [2] | Triulzi T, Tagliabue E, Balsari A, et al. FOXP3 expression in tumor cells and implications for cancer progression[J]. J Cell Physiol, 2013, 228(1): 30–35. DOI:10.1002/jcp.24125 |
| [3] | Wang JQ, Wu KJ. Epigenetic regulation of epithelial-mesenchymal transition by hypoxia in cancer:targets and therapy[J]. Curr Pharm Des, 2015, 21(10): 1272–1278. DOI:10.2174/1381612821666141211145610 |
| [4] | 陆永光, 李浪, 苏强, 等. 急性冠脉综合征患者内皮细胞微粒与CD4+ CD25+ Foxp3+调节性T细胞的关系[J]. 吉林大学学报:医学版, 2016, 42(5): 963–967. |
| [5] | 张莉, 卢志远, 殷操, 等. 口腔鳞状细胞癌中上皮-间充质转化标志物E-钙黏蛋白、波形蛋白、β-联蛋白及转化生长因子β1的表达[J]. 中华口腔医学杂志, 2015, 50(4): 228–234. |
| [6] | Kaneko T, Dehari H, Sasaki T, et al. Hypoxia-induced epithelial-mesenchymal transition is regulated by phosphorylation of GSK3-β via PI3 K/Akt signaling in oral squamous cell carcinoma[J]. Oral Surg Oral Med Oral Pathol Oral Radiol, 2016, 122(6): 719–730. DOI:10.1016/j.oooo.2016.06.008 |
| [7] | Chen SY, Teng SC, Cheng TH, et al. miR-1236 regulates hypoxia-induced epithelial-mesenchymal transition and cell migration/invasion through repressing SENP1 and HDAC3[J]. Cancer Lett, 2016, 378(1): 59–67. DOI:10.1016/j.canlet.2016.05.006 |
| [8] | 翟羽. 肿瘤细胞上皮间质转化表观遗传调控机制的研究进展[J]. 中国肿瘤生物治疗杂志, 2016, 23(1): 114–118. DOI:10.3872/j.issn.1007-385X.2016.01.019 |
| [9] | 王建秋, 范如意, 谢其洋, 等. 乏氧激活人口腔鳞癌细胞DKKL1的表达[J]. 中国老年学杂志, 2016, 36(11): 2569–2571. DOI:10.3969/j.issn.1005-9202.2016.11.001 |
| [10] | Kiesslich T, Pichler M, Neureiter D. Epigenetic control of epithelial-mesenchymal-transition in human cancer[J]. Mol Clin Oncol, 2013, 1(1): 3–11. |
| [11] | Shahbazian MD, Grunstein M. Functions of site-specific histone acetylation and deacetylation[J]. Annu Rev Biochem, 2007, 76(1): 75–100. DOI:10.1146/annurev.biochem.76.052705.162114 |
| [12] | Ruthenburg AJ, Allis CD, Wysocka J. Methylation of lysine 4 on histone H3:intricacy of writing and reading a single epigenetic mark[J]. Mol Cell, 2007, 25(1): 15–30. DOI:10.1016/j.molcel.2006.12.014 |
| [13] | Guillemette B, Drogaris P, Lin HH, et al. H3 lysine 4 is acetylated at active gene promoters and is regulated by H3 lysine 4 methylation[J]. PLoS Genet, 2011, 7(3): e1001354. DOI:10.1371/journal.pgen.1001354 |
| [14] | Basenko EY, Sasaki T, Ji L, et al. Genome-wide redistribution of H3K27me3 is linked to genotoxic stress and defective growth[J]. Proc Natl Acad Sci USA, 2015, 112(46): E6339–6348. DOI:10.1073/pnas.1511377112 |
| [15] | Bueno MT, Richard S. SUMOylation negatively modulates target gene occupancy of the KDM5B, a histone lysine demethylase[J]. Epigenetics, 2013, 8(11): 1162–1175. DOI:10.4161/epi.26112 |
| [16] | Cohen I, Poreba E, Kamieniarz K, et al. Histone modifiers in cancer:friends or foes?[J]. Genes Cancer, 2011, 2(6): 631–647. DOI:10.1177/1947601911417176 |
| [17] | Wu MZ, Tsai YP, Yang MH, et al. Interplay between HDAC3 and WDR5 is essential for hypoxia-induced epithelial-mesenchymal transition[J]. Molecular Cell, 2011, 43(5): 811–822. DOI:10.1016/j.molcel.2011.07.012 |
| [18] | Wang JQ, Wu MZ, Wu KJ. Analysis of epigenetic regulation of hypoxia-induced epithelial-mesenchymal transition in cancer cells by quantitative chromatin immunoprecipitation of histone deacetylase 3(HDAC3)[J]. Methods Mol Biol, 2016, 1436: 23–29. DOI:10.1007/978-1-4939-3667-0 |
 2017, Vol. 43
2017, Vol. 43


