扩展功能
文章信息
- 李浩渤, 杜勇, 陈勇, 李文静
- LI Haobo, DU Yong, CHEN Yong, LI Wenjing
- Notch 1对舌麟状细胞癌Tca8113细胞表皮生长因子受体表达的影响及其意义
- Influence of Notch 1 in expression of EGFR in tongue squamous cell carcinoma Tca8113 cells
- 吉林大学学报(医学版), 2017, 43(04): 715-719
- Journal of Jilin University (Medicine Edition), 2017, 43(04): 715-719
- 10.13481/j.1671-587x.20170410
-
文章历史
- 收稿日期: 2016-10-12
2. 河北省沧州市中心医院口腔科, 河北 沧州 061014
2. Department of Stomatology, Cangzhou Central Hospital, Hebei Province, Cangzhou 061014, China
口腔癌严重危害着人类健康,而舌鳞状细胞癌(舌鳞癌)是常见的口腔癌之一[1]。对舌鳞癌进行分子病理机制研究有助于寻找新型高效的治疗方法以提高其治愈率和生存率。近年来,Notch信号通道成为肿瘤靶向治疗研究的热点。Notch 1是Notch信号途径的受体之一,其与许多肿瘤的发生及病程的发展有紧密联系[2-3],但Notch 1在舌鳞癌组织中的表达及其作用至今仍未明确。人体病理生理的多个信号通路都与Notch信号通路产生交叉,有学者[4-6]发现:该通路与EGFr/Ras/ MAPK通路等有密切联系,通路间的相互作用可能参与调节细胞生命的整个过程。表皮生长因子受体(epidermal growth factor receptor,EGFR)信号通路参与细胞周期进程的调控,细胞增殖、黏附、迁移和分化的调节及损伤修复等。研究[7-8]显示:EGFR在舌鳞癌中为过表达状态,导致肿瘤生长加快、细胞增殖加快,并使患者对放化疗敏感性降低,最后会导致疾病预后不良并增加复发可能。Notch 1与EGFR信号通路参与多种癌症的发生发展,其在舌麟癌发生发展过程中的作用机制目前仍不清楚。本研究采用pRAMIC-I RES2-EGFP质粒转染舌麟癌Tca8113细胞,研究Notch 1在舌麟癌中的作用及其对EGFR表达的影响。
1 材料与方法 1.1 质粒、细胞、主要试剂和仪器pRAMIC-IRES2-EGFP目的质粒和pIRES2-EGFP对照质粒(美国Addgene公司)。Tca8113细胞(人舌麟癌细胞株,中科院上海细胞生物研究所)。RPMI1640培养基、Trypsin/EDTA胰蛋白酶和青/链霉素溶液(美国Gibico公司),胎牛血清(FBS,美国Hyclone公司),RT-PCR试剂盒(美国Promega公司),山羊抗人Noteh l多克隆抗体、鼠抗人EGFR单克隆抗体和鼠抗人beta-actin单克隆抗体(美国Santa Cruz公司)。低温离心机(德国Braun公司),PCR仪(美国TeehneTouehgene公司),细胞培养箱(美国FormaScienfific公司),酶标仪(美国Sigma公司)。
1.2 Tca8113细胞的培养将Tca8113细胞株用RPMI1640培养基(含10% FBS)在恒温培养箱(37℃、5%CO2)中进行常规培养及传代,当细胞处于对数生长期时即可用于实验。
1.3 细胞分组和转染细胞按不同的处理方法分为目的质粒pRAMIC-IRES2-EGFP转染组、对照质粒pIRES2-EGFP转染组和非转染组,转染实验前1d将细胞以1×105个/孔接种于6孔板上进行常规培养,待细胞密度为80%~90%时进行转染实验,采用脂质体lipofectamincTM 2000进行体外质粒转染,按试剂盒说明书操作。
1.4 RT-PCR法检测Notch 1和EGFR mRNA表达水平按试剂盒说明书提取细胞总RNA,逆转录获得cDNA,采用RT-PCR试剂盒进行PCR。PCR扩增的反应程序:94℃~96℃预加热,使模板DNA充分变性,然后进入扩增循环;94℃保持30s使模板变性,然后将温度降到复性温度(50℃~60℃),保持30 s,使引物与模板充分退火,72℃保持30s,使引物在模板上延伸,合成DNA,重复40次。采用7500 System Software系统进行分析,计算各组内参基因和目的基因的Ct值,相对定量采用2-△△Ct分析方法。GAPDH、Notch 1和EGFR基因引物委托上海生工生物工程有限公司进行设计并合成。GAPDH基因引物序列:5′-ggtgaaggtcggtgtgaacg-3′和3′-ctcgctcctggaagatggtg-5′,Notch 1基因引物序列:5′-acagcctcaacgggtacaag-3′和3′-ggcagacacaggagaagctc-5′,EGFR基因引物序列:5′-gcatgaccacggaggatagt-3′和3′-accactggaatcaagcatcc-5′。
1.5 Western blotting法检测Notch 1和EGFR蛋白表达水平取各组细胞,弃去培养液后采用PBS洗涤,共3次,采用BCA试剂盒按说明书提取细胞蛋白并进行定量。取适量裂解产物,以1:4比例加入样品和缓冲液,进行SDS-PAGE电泳,电泳完成后进行蛋白硝酸纤维素膜转移,采用5%脱脂牛奶在室温条件下封闭1 h,加入相应一抗(1:1000)4 ℃孵育4h,TBST洗涤3次,每次10 min;加入相应二抗(1:2000) 于室温下结合1.5 h,TBST洗涤3次,每次10 min; 按照ECL试剂盒说明书进行显色,成像扫描分析系统曝光拍照并保存图像。以灰度值计算蛋白表达水平。
1.6 免疫细胞化学法制备细胞爬片,各组Tca8113细胞接种前在6孔板底铺以灭菌处理的盖玻片,细胞进行常规培养,于转染48h后吸去培养液,PBS轻洗涤2次。将细胞爬片从6孔板内取出,放置于灭菌平皿中,采用4℃冰冻过的丙酮固定5min,1×PBS液轻洗,每次5 min,共3次。采用S-P法按免疫细胞化学试剂盒操作步骤进行检测。Notch 1一抗浓度为1:100, EGFR一抗浓度为1:150。以PBS代替一抗作为阴性对照。光学显微镜下观察,爬片背景清晰且细胞膜及细胞质有棕黄色颗粒状染色为阳性,反之结果为阴性。
1.7 MTT法检测Tca8113细胞存活率取处于对数生长期的Tca8113细胞,制备细胞悬液后以每孔约5×103个密度接种于96孔板。于接种后12、24、36和48 h加入MTT溶液,继续培养4 h后吸去培养液,每孔加入DMSO 100 μL,振摇10 min,酶标仪检测吸光度(A)值,吸收波长为488 nm。细胞存活率=加药孔A值/空白对照组A值×100%。
1.8 统计学分析采用SPSS19.0统计软件进行统计学分析。各组细胞Notch 1 mRNA及蛋白、EGFR mRNA及蛋白表达水平和细胞存活率以x± s表示,组间比较采用方差分析。以P < 0.05表示差异有统计学意义。
2 结果 2.1 各组细胞Notch 1mRNA和EGFR mRNA表达水平与非转染组和对照质粒pIRES2-EGFP转染组比较,目的质粒pRAMIC-IRES2-EGFP转染组Notch1 mRNA表达水平明显升高(P < 0.05),EGFR mRNA表达水平明显降低(P < 0.05);与非转染组比较,对照质粒pIRES2-EGFP转染组Notch 1 mRNA和EGFRmRNA表达水平差异均无统计学意义(P>0.05)。见表 1。
| (n=6, x± s) | ||
| Group | Notch 1 mRNA | EGFR mRNA |
| Non-transfection | 0.243±0.017 | 1.011±0.034 |
| pIRES2-EGFP | 0.218±0.022 | 0.968±0.041 |
| pRAMIC-IRES2-EGFP | 0.976±0.030*△ | 0.087±0.019*△ |
| *P < 0.05 compared with non-transfection group; △P < 0.05 compared with pIRES2-EGFP group. | ||
与非转染组和对照质粒pIRES2-EGFP转染组比较,目的质粒pRAMIC-IRES2-EGFP转染组Notch1蛋白表达水平明显升高(P < 0.05),EGFR蛋白表达水平明显降低(P < 0.05);与非转染组比较,对照质粒pIRES2-EGFP转染组Notch 1蛋白和EGFR蛋白表达水平差异均无统计学意义(P>0.05)。见图 1和表 2。免疫细胞化学法检测结果见图 2(插页三)。
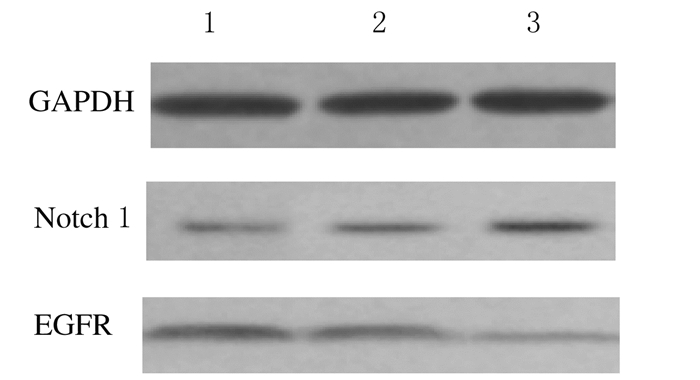
|
| Lane 1:Non-transfection group; Lane 2:pIRES2-EGFP group; Lane 3:pRAMIC-IRES2-EGFP group. 图 1 各组Tca8113细胞中Notch 1蛋白和EGFR蛋白表达电泳图 Figure 1 Expressionlevel of Notch 1 and EGFR proteins in Tca8113 cells in varous group |
|
|
| (n=6, x± s) | ||
| Group | Notch 1 protein | EGFR protein |
| Non-transfection | 0.183±0.021 | 0.818±0.041 |
| pIRES2-EGFP | 0.179±0.024 | 0.792±0.031 |
| pRAMIC-IRES2-EGFP | 0.989±0.032*△ | 0.101±0.017*△ |
| *P < 0.05 compared with non-transfection group; △P < 0.05 compared with pIRES2-EGFP group. | ||
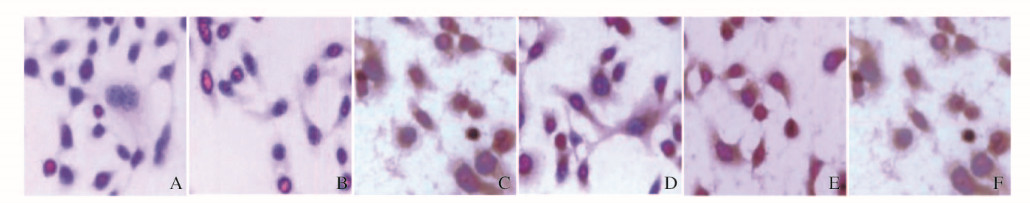
|
| A-C:Notch 1 protein; D-F:EGFR protein; A, D:Non-transfection group; B, E: pIRES2-EGFP group; C, F: pRAMIC-IRES2-EGFP group. 图 2 免疫细胞化学法检测各组Tcas8113细胞中Notch 1蛋白和EGFR蛋白表达(×400) Figure 2 Expressions of Notch 1 protein and EGFR protein in Tca8113 cells detected by immunocytochemistry(×400) |
|
|
与非转染组和对照质粒pIRES2-EGFP转染组比较,36和48h时目的质粒pRAMIC-IRES2-EGFP转染组Tca8113细胞增殖活性受到明显抑制,细胞存活率明显降低(P < 0.05或P < 0.01);与非转染组比较,对照质粒pIRES2-EGFP转染组细胞增殖活性无明显变化,细胞存活率差异无统计学意义(P>0.05)。见表 3。
| (n=6, x± s, η/%) | |||||
| Group | Survival rate | ||||
| (t/h) | 12 | 24 | 36 | 48 | |
| Non-transfection | 103.2±5.3 | 106.6±7.5 | 103.1±10.1 | 102.2±9.7 | |
| pIRES2-EGFP | 101.3±3.4 | 94.3±6.1 | 101.3±7.4 | 100.5±5.4 | |
| pRAMIC-IRES2-EGFP | 100.5±2.2 | 73.2±2.7 | 61.1±6.5*△ | 52.4±8.3**△△ | |
| *P < 0.05, ** P < 0.01 compared with non-transfection group;△P < 0.05, △△P < 0.01 compared with pIRES2-EGFP group | |||||
在细胞增殖、分化和凋亡等过程中,Notch信号通路起着十分重要的作用。在大多数组织中,受体Notch 1通常处于未分化状态或维持于干细胞状态,其表达下调可诱导细胞分化[9-10]。但在不同疾病中Notch 1的作用并非完全一致[11]。研究[12]显示:在皮肤表皮中Notch 1的作用主要是诱导角质细胞的成熟与分化,而在基底层中未见Notch 1点基因或蛋白表达,可能是由于Notch 1在基底层仍处于未分化或干细胞状态,以维持细胞和组织之间内在的动态平衡。研究[13]显示:口腔黏膜上皮的组织结构及生理功能与皮肤表皮有相似之处,Notch 1表达于口腔黏膜上皮角化层、部分颗粒层和棘层细胞,从角化细胞层开始阳性表达率和表达水平随着细胞层显示出层次性降低的情况。人体多种与病理生理有关的信号通路都可能存在Notch信号通路的调控,EGFR/Ras/MAPK、Shh(sonichedgehog)和Wnt/beta-catenin等信号通路与Notch信号通路有密切关系,细胞增殖、分化和凋亡过程都可能受这些通路间的相互效应影响[13-15]。Notch 1与EGFR信号通路在多种肿瘤的发生发展中相互作用,而由于肿瘤的组织来源和细胞类型的不同,其可能产生协同或拮抗的效应。研究[16-17]显示:舌鳞癌患者舌黏膜上发现高水平表达的EGFR,这可能是其下游信号途径持续激活的诱因,表明EGFR在癌细胞增殖过程中起促进作用。在不同临床分期舌鳞癌患者癌组织中,TNM分期越高, EGFR的表达水平也越高。
为了模拟靶基因调控与Notch配体诱导激活的Notch信号通路,通过基因转染技术将NICD编码基因导入靶细胞,达到组成性活化效应激活Notch下游信号是研究许多癌细胞生长凋亡的重要途径[18]。前列腺癌[19]和小细胞肺癌[20]等可通过这种方式来抑制癌细胞增殖并促进细胞凋亡。本研究结果显示:非转染组和对照质粒pIRES2-EGFP转染组的Tca8113细胞中有较低水平的Notch 1表达,EGFR mRNA和蛋白表达水平较高,通过体外瞬时转染重组质粒pRAMIC-IRES2-EGFP后,Notch 1的表达明显上调,而EGFRmRNA和蛋白表达水平明显降低,表明外源性导入过表达的NICD可起到诱导Notch 1信号通路的组成性活化效应;MTT结果显示:重组质粒pRAMIC-IRES2-EGFP转染组的Tca8113细胞增殖活性在转染72h后明显降低,推测Notch 1可能通过下调EGFR mRNA和蛋白表达水平而起到抑制舌麟癌细胞株Tcas113细胞增殖的作用。
本研究结果显示:在舌麟癌细胞株中Notch 1可能通过下调EGFR的表达而调节细胞增殖、分化和凋亡,两者之间可能存在负反馈调节作用。目前,Notch 1和EGFR信号通路在肿瘤治疗靶点研究中具有非常大的潜力,因此研究二者的调节通路及相互关系具有重要的价值和意义。
| [1] | Duan Y, Zhang S, Wang L, et al. Targeted silencing of CXCR4 inhibits epithelial-mesenchymal transition in oral squamous cell carcinoma[J]. Oncol Lett, 2016, 12(3): 2055–2061. |
| [2] | Mao L. NOTCH mutations:multiple faces in human malignancies[J]. Cancer Prev Res (Phila), 2015, 8(4): 259–261. DOI:10.1158/1940-6207.CAPR-15-0063 |
| [3] | Kangsamaksin T, Tattersall IW, Kitajewski J. Notch functions in developmental and tumour angiogenesis by diverse mechanisms[J]. Biochem Soc Trans, 2014, 42(6): 1563–1568. DOI:10.1042/BST20140233 |
| [4] | Kim HJ, Kim MJ, Kim A. The role of Notch1 signaling in anaplastic thyroid carcinoma[J]. Cancer Res Treat, 2017, 49(2): 509–517. DOI:10.4143/crt.2016.214 |
| [5] | Nusser-Stein S, Beyer A, Rimann I, et al. Cell-cycle regulation of NOTCH signaling during C. elegans vulval development[J]. Mol Syst Biol, 2012, 8: 618. DOI:10.1038/msb.2012.51 |
| [6] | Pellegrino MW, Farooqui S, Fr hli E, et al. LIN-39 and the EGFR/RAS/MAPK pathway regulate C. elegans vulval morphogenesis via the VAB-23 zinc finger protein[J]. Development, 2011, 138(21): 4649–4660. DOI:10.1242/dev.071951 |
| [7] | Jouan-Hureaux V, Boura C, Merlin JL, et al. Modulation of endothelial cell network formation in vitro by molecular signaling of head and neck squamous cell carcinoma (HNSCC) exposed to cetuximab[J]. Microvasc Res, 2012, 83(2): 131–137. DOI:10.1016/j.mvr.2011.07.008 |
| [8] | Ryott M, Wangsa D, Heselmeyer-Haddad K, et al. EGFR protein overexpression and gene copy number increases in oral tongue squamous cell carcinoma[J]. Eur J Cancer, 2009, 45(9): 1700–1708. DOI:10.1016/j.ejca.2009.02.027 |
| [9] | Wu G, Wilson G, George J, et al. Modulation of Notch signaling as a therapeutic approach for liver cancer[J]. Curr Gene Ther, 2015, 15(2): 171–181. DOI:10.2174/1566523214666141224100319 |
| [10] | Miele L. Notch signaling[J]. Clin Cancer Res, 2006, 12(4): 1074–1079. DOI:10.1158/1078-0432.CCR-05-2570 |
| [11] | Klinakis A, Lobry C, Abdel-Wahab O, et al. A novel tumour-suppressor function for the Notch pathway in myeloid leukaemia[J]. Nature, 2011, 473(7346): 230–233. DOI:10.1038/nature09999 |
| [12] | Huang WH, Liao WR, Sun RX. Astragalus polysaccharide induces the apoptosis of human hepatocellular carcinoma cells by decreasing the expression of Notch 1[J]. Int J Mol Med, 2016, 38(2): 551–557. |
| [13] | Deng Y, Greenwald I. Determinants in the LIN-12/Notch intracellular domain that govern its activity and stability during caenorhabditis elegans vulval development[J]. G3:Genes Genomes Genetics, 2016, 19: 116. |
| [14] | Hansson EM, Lendahl U, Chapman G, et al. Notch signaling in development and disease[J]. Semin Cancer Biol, 2004, 14(5): 320–328. DOI:10.1016/j.semcancer.2004.04.011 |
| [15] | Masuda S, Kumano K, Shimizu K, et al. Notch l oncoprotein antagonizes TGF-beta/Smad-mediated cell growth suppression via sequestration of coactivator p300[J]. Cancer Sci, 2005, 96(5): 274–282. DOI:10.1111/cas.2005.96.issue-5 |
| [16] | Gusenbauer S, Zanucco E, Knyazev P, et al. Erk2 but not Erk1 regulates crosstalk between Met and EGFR in squamous cell carcinoma cell lines[J]. Mol Cancer, 2015, 14(1): 54. DOI:10.1186/s12943-015-0319-z |
| [17] | Shao Y, Sha XY, Bai YX, et al. Effect of A disintegrin and metalloproteinase 10 gene silencing on the proliferation, invasion and migration of the human tongue squamous cell carcinoma cell line TCA8113[J]. Mol Med Rep, 2015, 11(1): 212–218. |
| [18] | 黄红杰. Notch 1在人舌麟癌中的作用及其与表皮生长因子受体信号的交互作用[D]. 杭州: 浙江大学, 2009. http://d.wanfangdata.com.cn/Thesis/Y1452748 |
| [19] | Ye QF, Zhang YC, Peng XQ, et al. siRNA-mediated silencing of Notch-1 enhances docetaxel induced mitotic arrest and apoptosis in prostate cancer cells[J]. Asian Pac J Cancer Prev, 2012, 13(6): 2485–2889. DOI:10.7314/APJCP.2012.13.6.2485 |
| [20] | Wang X, Song N, Zhang Y, et al. Coexpression of c-Met and Notch-1 correlates with poor prognosis in resected non-small-cell lung cancer[J]. Tumour Biol, 2015, 36(9): 7053–7059. DOI:10.1007/s13277-015-3404-4 |
 2017, Vol. 43
2017, Vol. 43


