扩展功能
文章信息
- 霍云龙, 王春玉, 赵越
- HUO Yunlong, WANG Chunyu, ZHAO Yue
- Mu2蛋白亚细胞结构定位及其相互作用蛋白的筛选
- Subcellular localization of Mu2 protein and screening of its interacting proteins
- 吉林大学学报(医学版), 2016, 42(06): 1054-1058
- Journal of Jilin University (Medicine Edition), 2016, 42(06): 1054-1058
- 10.13481/j.1671-587x.20160603
-
文章历史
- 收稿日期: 2016-04-21
2. 中国医科大学基础医学院染色质生物学研究室 教育部医学细胞生物学重点实验室 卫生部细胞生物学重点实验室, 辽宁 沈阳 110122
2. Department of Chromatin Biology, School of Basic Medical Sciences, Key Laboratory of Cell Biology, Ministry of Public Health, Key Laboratory of Medical Cell Biology, Ministry of Education, China Medical University, Shenyang 110122, China
果蝇是生物学研究中的重要模型。果蝇S2细胞系来源于黑腹果蝇晚期胚胎细胞,是适用于重组蛋白表达的一种常用工具细胞系。Mu2最初是于雌性果蝇生殖细胞系中发现的影响DNA双链断裂过程的基因[1-2]。Mu2基因突变造成卵细胞中DNA双链断裂处的修复率降低[3],其在体细胞中还具有细胞周期检查点功能[4]。本研究对Mu2相互作用的蛋白进行筛选以明确Mu2蛋白的功能。本研究以Mu2为诱饵,分离并鉴定了5个可能的Mu2相互作用蛋白,提示Mu2可能参与DNA损伤之外的新功能,为进一步研究Mu2蛋白的新功能奠定实验基础。
1 材料与方法 1.1 细胞和质粒果蝇S2细胞由中国医科大学盛京医院病理科保存。FLAG-Mu2表达质粒的构建:将Mu2 cDNA序列连接到N端带有FLAG标签的果蝇表达系统(购于美国Invitrogen公司);Mu2 cDNA序列(Clone ID LD44171,购于美国OPEN biosystems公司);构建的FLAG-Mu2质粒序列经测序验证正确[5]。
1.2 主要试剂和仪器Schneider’ s果蝇培养基购于美国Gibco公司,优级胎牛血清购于天津市灏洋生物制品科技有限责任公司,Lipofectamine2000脂质体转染试剂和核染料TOPRO-3购于美国Life Technologies公司,FLAG-M2琼脂糖珠购于美国Sigma公司,鼠源单克隆FLAG抗体购于上海睿星生物技术有限公司,cy-3标记驴抗鼠二抗购于美国Jackson Immuno Research公司,HRP标记羊抗鼠二抗购于北京中杉金桥生物技术有限公司,核浆分离试剂盒及ECL发光底物购于美国Pierce公司,其他生化试剂为国产分析纯级别。Mini-PROTEAN Tetra蛋白电泳系统购于美国Bio-Rad公司,细胞超净工作台购于苏州安泰空气技术有限公司,共聚焦激光扫描显微镜购于日本Olympus公司。
1.3 细胞培养S2细胞用含10%胎牛血清的Schneider’s果蝇培养基培养,添加100 U·mL-1青霉素和100 U·mL-1链霉素,于25℃无CO2的培养箱中培养,待细胞生长状态良好时铺板进行转染。
1.4 细胞转染转染工作液按转染说明书配置后,将其逐滴加入单层S2细胞中,轻柔摇晃混匀置于培养箱中孵育。6 h后弃去培养基,加入含血清培养基继续培养,48 h后进行后续实验。
1.5 共聚焦激光扫描显微镜下观察FLAG-Mu2蛋白在细胞中的定位细胞弃去培养基,加入4%多聚甲醛,室温固定15 min;PBS洗涤后,0.1%TritonX-100孵育10 min,进行透膜;加入驴血清室温封闭1 h;抗FLAG单克隆抗体(10 mg·L-1)室温孵育1 h;PBS洗涤后,Cy3-驴抗鼠二抗(1 mg·L-1)室温孵育1h;PBS洗涤后,TOPRO3室温孵育15 min;PBS洗涤后,采用60%甘油封片。采用共聚焦激光扫描显微镜观察Cy3标记的FLAG-Mu2蛋白和TOPRO染色的细胞核。
1.6 蛋白的提取弃去培养基并用PBS清洗后,在培养皿中加入预冷的含蛋白酶抑制剂的TNE裂解液,用细胞刮将细胞缓慢挂下,并收集至离心管中;超声破碎细胞后,4℃、12 000 g离心15 min;取上清至新离心管中,作为细胞总蛋白。
根据NE-PER核浆分离试剂盒说明书进行浆蛋白和核蛋白的提取。胰酶消化细胞后,500 g离心5 min收集细胞;PBS洗涤后,加入预冷的细胞浆提取试剂Ⅰ,剧烈涡旋15 s后,冰上静置10 min;加入预冷的细胞浆提取试剂Ⅱ后,剧烈涡旋5 s,之后冰上静置1 min;4℃、16 000 g离心5 min,迅速转移上清至新管中,作为浆蛋白;在原管离心后的沉淀中加入细胞核提取试剂,剧烈涡旋15 s后冰上静置10 min,重复4次;4℃、16 000 g离心10 min后,取上清至新离心管中,作为核蛋白。
1.7 免疫沉淀将FLAG-Mu2表达质粒转染至S2细胞后,收集细胞后均匀分配,分别进行总蛋白和核浆蛋白提取。向总蛋白、浆蛋白和核蛋白提取液中加入30 μL FLAG-M2琼脂糖珠,于4℃层析冷柜中旋转孵育3 h后,采用PBS洗涤3次,弃净上清后,加入与琼脂糖珠等体积的电泳上样缓冲液,进行蛋白质印记实验。
1.8 Western blotting法检测S2细胞核提取物中FLAG-Mu2蛋白的表达情况取10μg蛋白进行SDS-PAGE分离;4℃冷柜中以200 mA恒流3 h将蛋白转移至PVDF膜上;5%脱脂奶粉室温封闭1 h;FLAG单克隆抗体(1 mg·L-1)室温孵育2 h;TBST洗膜后,HRP标记的羊抗鼠二抗(1:5 000稀释)室温孵育1 h;TBST洗膜后,进行ECL发光检测。
1.9 FLAG-Mu2蛋白复合物的纯化和鉴定将FLAG-Mu2表达质粒稳定转染至S2细胞后进行核蛋白提取。在核提取液中加入FLAG M2琼脂糖珠,4℃冷柜中过夜孵育。孵育之后的FLAG琼脂糖珠经用含0.2 g·L-1FLAG多肽的溶液进行洗脱,经10%~60%甘油梯度离心后,再用Superose 6 10/300 GL凝胶过滤柱分离,之后行SDS-PAGE电泳,最终将凝胶进行硝酸银染色。并将染色条带所在处凝胶切下进行蛋白质质谱分析。
2 结果2.1共聚焦激光扫描显微镜下观察FLAG-Mu2蛋白在S2细胞中的定位
在546 nm的激发光下,大部分细胞中可以观察到红色荧光,说明转染效率较高。红色荧光指示的FLAG-Mu2蛋白阳性表达率达50%,其中FLAG-Mu2蛋白强表达率约为30%。FLAG-Mu2蛋白主要呈核着色,与蓝色荧光显示的细胞核重叠,说明FLAG-Mu2蛋白定位于细胞核中。见图 1(插页一)。

|
| A: Nucleus stained by TOPTO3 (blue); B: FLAG-mu2 stained by anti-FLAG antibody (red); C: Merged image. 图 1 共聚焦荧光显微镜下观察FLAG-mu2蛋白在S2细胞中的定位 Figure 1 Localization of FLAG-mu2 protein in S2 cells observed by confocal fluorescence microscope |
|
|
在转染FLAG-Mu2表达质粒的细胞蛋白中约140 000位置处检测到外源蛋白的表达,而对照细胞相同位置未见蛋白条带。FLAG-Mu2在细胞核提取物中的表达量较大,在细胞浆提取物中的表达量较小,说明FLAG-Mu2蛋白主要存在于S2细胞核提取物中。见图 2。
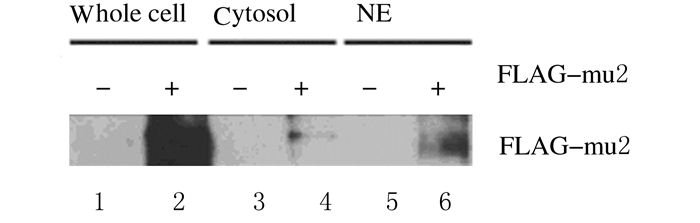
|
| Lane 1, 3, 5: Cells tranfected with control vector; Lane 2, 4, 6: Cells tranfected with FLAG-Mu2; Lane 1, 2: Whole cell lysates; Lane 3, 4: Cytosol extractions; Lane 5, 6: Nuclear extractions. 图 2 Western blotting法检测S2细胞核提取物中FLAG-Mu2蛋白表达 Figure 2 Expressions of FLAG-Mu2 protein in nuclear extractions of S2 cells detected by Western blotting method |
|
|
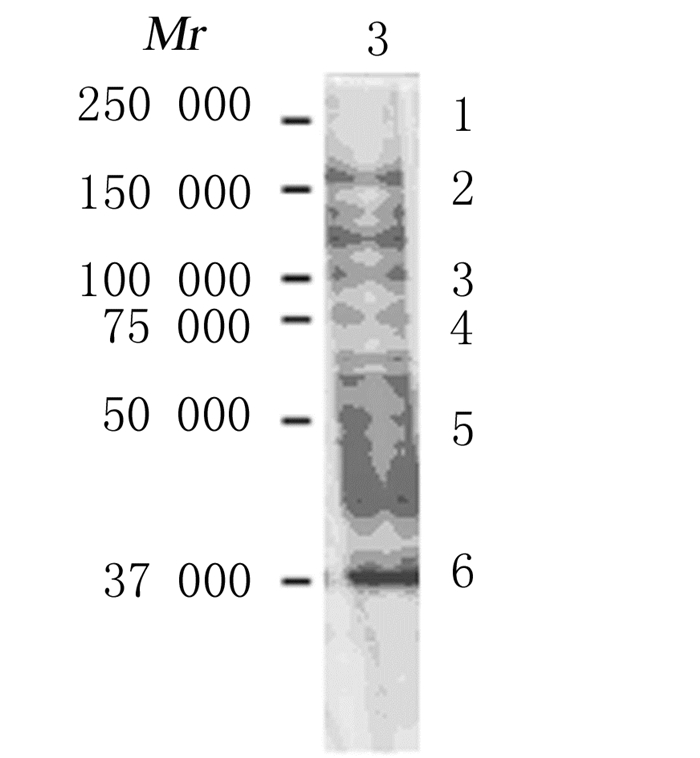
SDS-PAGE后对凝胶进行银染及Western blotting检测,以FLAG抗体进行检测显示FLAG-Mu2蛋白主要存在于第3泳道的洗脱液组分中,其蛋白复合物相对分子质量约为300 000。见图 3。

|
| Lane 1-13:Fractionated purifications; Upper: Silver staining method; Bottom: Western blotting method. 图 3 免疫沉淀纯化FLAG-Mu2蛋白复合物的电泳图 Figure 3 Electrophoregram of purified FLAG-Mu2 protein complex by immuno-precipitation |
|
|
将第3泳道中的主要条带切下后进行质谱测序。作为诱饵蛋白的FLAG-Mu2被成功沉淀(条带3),初步鉴定其蛋白复合物的其他可能组分包括pif1A (PFRAIRE-interacting factor 1A)、CG31782、mip120(Myb-interacting protein 120)、CG31690和G protein-sa60A。见图 4。
|
| Lane1:PFTAIRE-interacting factor 1A, pif1A;Lane 2:CG31782;Lane 3:MU-2(bait); Lane 4:Myb-interacting protein 120, mip120;Lane 5:CG31690;Lane 6:G protein-sa60A. 图 4 FLAG-Mu2蛋白复合物成分初步鉴定的电泳图 Figure 4 Electrophoregram of identification of components of FLAG-Mu2 protein complex |
|
|
果蝇S2细胞是一个高效的蛋白表达系统,具有表达量高并且产物蛋白构像接近天然状态的优点,且培养要求低并且容易操作,因此适用于蛋白质的功能研究。蛋白所行使的生物学功能与其在细胞中的定位密切相关,根据蛋白的亚细胞定位可以合理推测基因的功能。Mu2蛋白由1 353个氨基酸组成,相对分子质量为139 000, 其N末端具有FHA结构域和HLH结构域,C末端具有2个串联的BRCT结构域,中间和靠C末端处有3个核定位信号,提示了该蛋白的核定位及具有与DNA结合的能力[6]。BRCT结构域是一个磷酸化的蛋白结合结构域,许多参与DNA损伤修复和细胞周期调控的蛋白均具有该结构域[7]。迄今为止,关于Mu2蛋白的生物学功能的研究也主要集中在其DNA损伤修复功能。在卵细胞中,Mu2蛋白突变造成DNA双链断裂处的修复率降低[3];在体细胞中,Mu2蛋白还具有细胞周期检查点功能[4]。本研究结果显示:在果蝇S2细胞中Mu2蛋白主要定位于细胞核,这与相关研究[3-4]结果相符。
根据Mu2蛋白的氨基酸序列及其结合蛋白的特点,提示其与人的DNA损伤检查点介质1(mediator of DNA damage checkpoint protein 1, MDC1)同源[3]。MDC1是参与哺乳动物细胞中DNA双链断裂修复的重要因子[8],其介导许多磷酸化依赖的蛋白相互作用[9-10]。基于其在DNA损伤修复过程中的重要作用,MDC1影响肿瘤细胞对放化疗的敏感性[11-12]。同时,MDC1还具有转录调控功能,其与雄激素受体(androgen receptor,AR)相互作用,作为AR的辅调节因子通过影响组蛋白乙酰化酶GCN5的招募而促进AR介导的转录,在前列腺癌中参与抑制肿瘤的作用[5];MDC1还能与雌激素受体(estrogen receptor,ER)相互作用,上调ER的转录活性,在乳腺癌中具有抑制肿瘤细胞生长的功能[13]。此外,MDC1还在精子发生过程中起作用[14]。MDC1的多功能性提示Mu2蛋白在果蝇中也可能具有DNA损伤修复外的新的功能。因此,筛选Mu2蛋白的相互作用蛋白提示Mu2蛋白中存在未发现的其他功能,并同时为其同源蛋白MDC1的进一步研究提供了实验依据。
通过纯化FLAG-Mu2蛋白复合物并进行质谱分析,本课题组发现了Mu2蛋白复合物中5种可能的蛋白组分。其中,pif1A是具有hZIP结构域的转录因子[15];CG31782含有C2H2型锌指结构域,预测其具有转录因子活性;mip120是Myb复合物的组分之一,于特定染色质位置上依赖其他因子在DNA复制过程中起双重作用[16];CG31690具有N-乙酰葡糖胺转移酶活性,含TRP结构域,该结构域在蛋白-蛋白相互作用中起重要作用[17];G protein-sa60A参与G蛋白偶联受体信号通路[18]。尽管这些蛋白与Mu2的相互作用需要进一步验证,但其参与不同信号通路、拥有不同的生物学功能,提示除参与DNA损伤修复的作用外,Mu2的生物学功能仍有待深入研究。
本研究成功地进行了FLAG-Mu2表达载体的转染和表达,观察了Mu2蛋白在S2细胞中的亚细胞结构定位,并筛选到5种可能与Mu2形成复合体的蛋白,为进一步研究Mu2蛋白的功能奠定了实验基础。
| [1] | Mason JM, Strobel E, Green MM. Mu-2:mutator gene in Drosophila that potentiates the induction of terminal deficiencies[J]. PNAS, 1984, 81(19): 6090–6094. DOI:10.1073/pnas.81.19.6090 |
| [2] | Mason JM, Champion LE, Hook G. Germ-line effects of a mutator, Mu2, in Drosophila melanogaster[J]. Genetics, 1997, 146(4): 1381–1397. |
| [3] | Dronamraju R, Mason JM. Recognition of double strand breaks by a mutator protein (MU2) in Drosophila melanogaster[J]. PLoS Genet, 2009, 5(5): e1000473. DOI:10.1371/journal.pgen.1000473 |
| [4] | Kurzhals RL, Titen SW, Xie HB, et al. Chk2 and p53 are haploinsufficient with dependent and independent functions to eliminate cells after telomere loss[J]. PLoS Genet, 2011, 7(6): e1002103. DOI:10.1371/journal.pgen.1002103 |
| [5] | Wang C, Sun H, Zou R, et al. MDC1 functionally identified as an androgen receptor co-activator participates in suppression of prostate cancer[J]. Nucleic Acids Res, 2015, 43(10): 4893–4908. DOI:10.1093/nar/gkv394 |
| [6] | Kasravi A, Walter MF, Brand S, et al. Molecular cloning and tissue-specific expression of the mutator2 gene (Mu2) in Drosophila melanogaster[J]. Genetics, 1999, 152(3): 1025–1035. |
| [7] | Dronamraju R, Mason JM. MU2 and HP1a regulate the recognition of double strand breaks in Drosophila melanogaster[J]. PLoS One, 2011, 6(9): e25439. DOI:10.1371/journal.pone.0025439 |
| [8] | Rassoolzadeh H, Coucoravas C, Farnebo M. The proximity ligation assay reveals that at DNA double-strand breaks WRAP53beta associates with gammaH2AX and controls interactions between RNF8 and MDC1[J]. Nucleus, 2015, 6(5): 417–424. DOI:10.1080/19491034.2015.1106675 |
| [9] | Jungmichel S, Stucki M. MDC1:The art of keeping things in focus[J]. Chromosoma, 2010, 119(4): 337–349. DOI:10.1007/s00412-010-0266-9 |
| [10] | Luo S, Ye K. Dimerization, but not phosphothreonine binding, is conserved between the forkhead-associated domains of Drosophila MU2 and human MDC1[J]. FEBS Lett, 2012, 586(4): 344–349. DOI:10.1016/j.febslet.2012.01.023 |
| [11] | Zeng Q, Wang Z, Liu C, et al. Knockdown of NFBD1/MDC1 enhances chemosensitivity to cisplatin or 5-fluorouracil in nasopharyngeal carcinoma CNE1 cells[J]. Mol Cell Biochem, 2016, 418(1-2): 137–146. DOI:10.1007/s11010-016-2739-5 |
| [12] | Cirauqui B, Margeli M, Quiroga V, et al. DNA repair pathways to regulate response to chemoradiotherapy in patients with locally advanced head and neck cancer[J]. Tumour Biol, 2016, 37(10): 13435–13443. DOI:10.1007/s13277-016-5149-0 |
| [13] | Zou R, Zhong X, Wang C, et al. MDC1 enhances estrogen receptor-mediated transactivation and contributes to breast cancer suppression[J]. Int J Biol Sci, 2015, 11(9): 992–1005. DOI:10.7150/ijbs.10918 |
| [14] | Xiao Y, Pollack D, Andrusier M, et al. Identification of cell-specific targets of sumoylation during mouse spermatogenesis[J]. Reproduction, 2016, 151(2): 149–166. DOI:10.1530/REP-15-0239 |
| [15] | Reinke AW, Baek J, Ashenberg O, et al. Networks of bZIP protein-protein interactions diversified over a billion years of evolution[J]. Science, 2013, 340(6133): 730–734. DOI:10.1126/science.1233465 |
| [16] | Sim CK, Perry S, Tharadra SK, et al. Epigenetic regulation of olfactory receptor gene expression by the Myb-MuvB/dREAM complex[J]. Genes Develop, 2012, 26(22): 2483–2498. DOI:10.1101/gad.201665.112 |
| [17] | Hoskins RA, Carlson JW, Kennedy C, et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin[J]. Science, 2007, 316(5831): 1625–1628. DOI:10.1126/science.1139816 |
| [18] | Wolfgang WJ, Hoskote A, Roberts IJ, et al. Genetic analysis of the Drosophila Gs (alpha) gene[J]. Genetics, 2001, 158(3): 1189–1201. |
 2016, Vol. 42
2016, Vol. 42


