扩展功能
文章信息
- 杨麒巍, 杜珍武, 于杉, 高素洁, 赵冠杰, 张琳, 卢佳, 任明, 张桂珍
- YANG Qiwei, DU Zhenwu, YU Shan, GAO Sujie, ZHAO Guanjie, ZHANG Lin, LU Jia, REN Ming, ZHANG Guizhen
- 孕妇血浆DNA中内参基因拷贝数稳定性的评价
- Evaluation on stability of copy number of reference gene in plasma DNA of pregnant woman
- 吉林大学学报(医学版), 2016, 42(03): 523-529
- Journal of Jilin University (Medicine Edition), 2016, 42(03): 523-529
- 10.13481/j.1671-587x.20160320
-
文章历史
- 收稿日期: 2015-09-29
Lo等[1] 1997年首次证明:孕妇血浆中存在有少量的游离胎儿DNA(cell-free fetal DNA,cffDNA),因此许多研究者将目光集中于应用cffDNA进行无创性产前检测(non-invasive prenatal testing,NIPT)。目前研究cffDNA的方法中,实时荧光定量PCR法(Real-time fluoresence quantitative PCR,qPCR)是最为基础、准确并且常用的方法之一。但是,qPCR法的准确性也受到起始样品量、模板质量和PCR效率等诸多内外因素的影响[2],因此需要使用内参基因进行结果的标准化。近年来越来越多的研究[3, 4, 5, 6]表明:目前的研究中经常使用的内参基因有β-肌动蛋白(ACTB)和甘油醛-3-磷酸脱氢酶(GAPDH)等,在不同的研究中其表达水平有明显的差异。在选择内参用于cffDNA分析之前对于其拷贝数稳定性进行比较和评价,从而选择一种更为稳定的内参基因,可以有效地增加结果的可靠性。目前针对孕妇血浆游离DNA样本中内参基因拷贝数稳定性的评价尚无报道。本研究首次探讨了在cffDNA研究中经常使用的6种内参基因分别在孕妇血浆DNA、非孕妇血浆DNA和孕妇血浆DNA中长度大于300 bp的DNA片段(视为母体来源的DNA)和孕妇血浆DNA中长度小于300 bp的DNA片段(视为胎儿来源的DNA)中的拷贝数稳定性。这6种内参基因分别为β-珠蛋白(HBB)、端粒酶(TERT)、GAPDH、白蛋白(ALB)、β-肌动蛋白(ACTB)和T细胞受体γ(TRG),内参基因选自已经发表的利用qPCR法研究cffDNA的文献[7, 8]。采用geNorm、NormFinder和BestKeeper 3种统计学分析程序计算内参基因的拷贝数稳定性。本研究旨在为应用孕妇血浆游离DNA进行无创性产前检测等定量研究中如何选择最为稳定的内参基因提供实验依据。
1 材料与方法 1.1 主要试剂和仪器SYBR Premix Ex Taq (2×)、DNA Marker DL1000和10× Loading Buffer(日本Takara公司),QIAamp DNA mini kit(德国QIAGEN公司),绿如蓝DNA染料(北京天恩泽公司),DNA引物合成(上海生工生物工程有限公司),实验所用水为电阻率18 MΩocm的去离子水。LightCycle 480实时荧光定量PCR扩增仪(美国Roche公司),Microfuge 22R低温高速离心机(美国Beckman Coulter公司),Synergy HT酶标仪(美国BioTek公司),TProfessional Thermocycler型 PCR仪(德国Biometra公司),DYY-Ⅲ7型电泳仪(北京六一仪器厂),GIS-2008凝胶成像分析系统(上海培清科技有限公司)。
1.2 研究对象的一般资料孕妇组:2012年2—5月在吉林大学中日联谊医院妇产科门诊进行产前检查的健康孕妇18名,年龄20~35岁,孕龄(12.87±1.25)周。非孕妇组:2012年2—5月在吉林大学中日联谊医院进行健康体检的健康未怀孕女性18名,年龄18~40岁。研究对象分为孕妇与非孕妇整体组 、孕妇组、非孕妇组、母体与胎儿整体组、母源DNA组和胎源DNA组。
1.3 血浆DNA的提取和分离所有样本按照QIAamp DNA mini Kit操作说明提取血浆DNA,提取操作在采血后4 h内完成。
取10 μL孕妇组研究对象的血浆DNA,分别与1 μL 10× Loading Buffer混匀,加样于1%琼脂糖凝胶,以绿如蓝为DNA染料,0.5× TBE电泳缓冲液,调整电压为10 V·cm-1进行电泳。电泳结束后在紫外光下以DL500 DNA marker为标志,在300 bp处将每条泳道切割为2部分,使用AxyPrep DNA凝胶回收试剂盒分别回收DNA,将孕妇血浆DNA分为>300 bp(视为母体来源的DNA)和≤300 bp(视为胎儿来源的DNA)2部分。
1.4 qPCR法检测各组内参基因的Ct值采用LightCycle 480实时荧光定量PCR扩增仪进行qPCR检测。6种内参基因引物序列见表 1。反应条件:95℃预变性10 min;95℃变性15 s,58℃退火15 s,72℃延伸30 s,重复40个循环;72℃延伸3 min。将DNA样品进行2倍梯度稀释并扩增,采用Ct值绘制趋势线,根据公式E=2-1/slope计算引物扩增效率。每个样品同时进行3复孔,取5 μL qPCR扩增产物进行电泳观察。
| Symbol | Gene | Primer sequence (5′-3′) | Produet size (bp) | qPCR efficiency |
| HBB | beta-globin | F, GTGCACCTGACTCCTGAGGAGAR,CCTTGATACCAACCTGCCCAG | 101 | 2.58 |
| TERT | telomerase | F,GGTGAACCTCGTAAGTTTATGCAAR,GGCACACGTGGCTTTTCG | 97 | 2.00 |
| GAPDH | glyceraldehyde-3-phosphate dehydrogenase | F,GGACTGAGGCTCCCACCTTTR,GCATGGACTGTGGTCTGCAA | 157 | 1.72 |
| ALB | albumin | F,TGAAACATACGTTCCCAAAGAGTTTR,CTCTCCTTCTCAGAAAGTGTGCATAT | 80 | 1.79 |
| ACTB | beta-actin | F,CCTGTACGCCAACACAGTGCR,ATACTCCTGCTTGCTGATCC | 211 | 2.08 |
| TRG | T cell receptor gamma | F,AGGGTTGTGTTGGAATCAGGR,CGTCGACAACAAGTGTTGTTCCAC | 160 | 1.82 |
将样本Ct值按要求分别输入geNorm、NormFinder和BestKeeper 3种程序中计算内参基因的拷贝数稳定性。geNorm程序计算基因稳定值(average expression stability,M)和配对变异参数(pairwise variations,V),M值代表一个内参基因与其他内参基因比较的配对变异性,每次比较后排除M值最高的内参基因,其余内参基因重新比较,直到仅有2组内参基因为止。M值越低代表该基因稳定性越高。将M=1.5作为分界线,高于该值的内参基因被视为不稳定的内参基因。V(n/n+1)用来确定为了达到一定的稳定性,推荐联合使用内参基因的最少数量。以V(n/n+1)=0.15为分界线,低于该值代表n个内参基因组合已经可以提供较高的稳定性。
NormFinder基于ANOVA模型计算内参基因的含量稳定性,其值越低代表含量稳定性越高。同时还能计算内参基因的组内和组间稳定性。
BestKeeper通过计算每个候选基因的标准差(standard deviation,SD)和相关系数(coefficient of correlation,R)分析基因的稳定性。SD是每一个内参基因的标准差,相关系数是通过Pearson’s配对相关性分析法计算每个候选基因与所有Ct值的几何平均数之间的相关系数。当SD>1.0时,该基因被认为是不稳定的,其余的内参基因根据R值排序,R值越高代表稳定性越高。
1.6 统计学分析采用SPSS17.0统计软件进行数据分析。样本Ct值以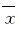 ± s
表示,2组间样本Ct值比较采用t检验,多组间样本Ct值比较采用方差分析。以α=0.05为检验水准。
± s
表示,2组间样本Ct值比较采用t检验,多组间样本Ct值比较采用方差分析。以α=0.05为检验水准。
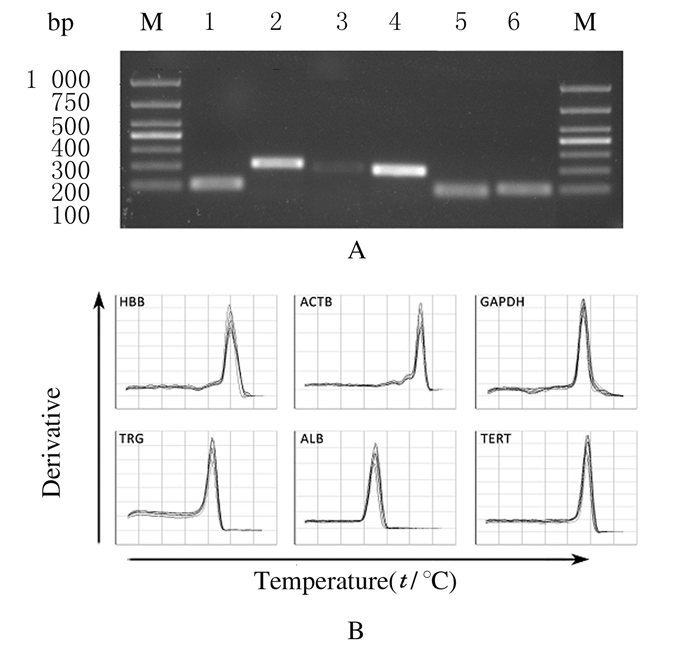
qPCR产物电泳结果显示:所有PCR产物条带清晰、条带位置与设计长度相符,无杂带、无引物二聚体,见图 1A。6种内参基因扩增产物熔解曲线示:所有PCR产物均呈单峰,证明所有PCR产物均为特异性扩增,见图 1B。
|
| 图 1 PCR产物电泳图(A)和熔解曲线图(B) Fig.1 Electrophoregram (A) and derivative curve images (B) of PCR products M:DL 1000DNA marker;Lane 1:HBB;Line 2:ACTB;Lane 3:GAPDH;Lane 4:TRG;Lane 5:ALB;Lane 6:TERT. |
采用Ct值进行候选内参基因扩增特点的描述,各组内参基因Ct值见表 2。不同内参基因Ct值组间比较差异无统计学意义(P>0.05)。各组中ACTB有最低的Ct值,HBB次之,这一结果表明ACTB和HBB在血浆DNA中拷贝数较高。
(n=18,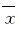 ± s) ± s) |
||||||
| Group | Ct value | |||||
| ACTB | HBB | TERT | ALB | TRG | GAPDH | |
| Pregnant+non-pregnant | 26.80±1.70 | 28.74±1.95 | 28.97±1.24 | 30.49±1.94 | 30.87±3.04 | 33.23±3.10 |
| Pregnant | 25.99±0.99 | 28.54±2.19 | 28.64±1.16 | 30.50±2.37 | 29.82±1.43 | 32.66±3.21 |
| Non-pregnant | 28.02±1.86 | 29.04±1.64 | 29.46±1.25 | 30.48±1.19 | 32.45±4.13 | 34.09±2.92 |
| Maternal+fetal | 32.80±0.41 | 34.82±0.92 | 38.42±1.06 | 36.34±1.08 | 36.25±2.00 | 37.78±1.08 |
| Maternal | 32.82±0.49 | 34.97±0.84 | 38.74±1.32 | 36.04±1.21 | 37.19±2.63 | 38.44±0.78 |
| Fetal | 32.78±0.40 | 34.67±1.09 | 38.10±0.79 | 36.64±0.08 | 35.32±0.35 | 37.11±0.79 |
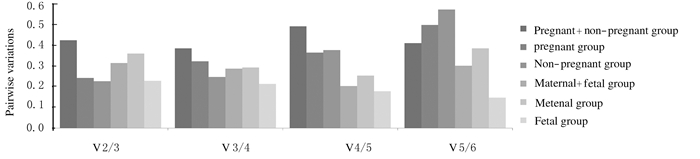
根据计算所得M值将内参基因按照拷贝数稳定性由高到低进行排序(表 3),在孕妇与非孕妇整体组和孕妇组中ACTB和TERT的拷贝数稳定性最高,GAPDH的拷贝数稳定性最低;在非孕妇组HBB和ALB的拷贝数稳定性最高,TRG的拷贝数稳定性最低;在母体与胎儿整体组和胎源DNA组GAPDH和HBB的拷贝数稳定性最高,TRG及TERT的拷贝数稳定性最低;在母源DNA组ALB和TERT的拷贝数稳定性最高,TRG的拷贝数稳定性最低。在确定内参基因的最佳数量时,几乎所有组的Pairwise Variations值均大于0.15,表明没有可推荐的联合使用内参基因的最佳数量。见图 2。
|
| 图 2 各组内参基因的最佳数量 Fig.2 Optimal number of reference genes in various groups |
| Group | Rank of ACTB | Rank of HBB | Rank of TERT | ||||||
| geNorm | NormFinder | BestKeeper | geNorm | NormFinder | BestKeeper | geNorm | NormFinder | BestKeeper | |
| Pregnant+non-pregnant | 1 | 2 | 2 | 3 | 3 | 6 | 1 | 1 | 1 |
| Pregnant | 1 | 1 | 1 | 4 | 5 | 5 | 1 | 2 | 3 |
| Non-pregnant | 4 | 4 | 5 | 1 | 1 | 3 | 3 | 3 | 4 |
| Maternal+fetal | 3 | 3 | 6 | 1 | 1 | 1 | 4 | 2 | 4 |
| Maternal | 5 | 5 | 3 | 3 | 1 | 1 | 1 | 4 | 5 |
| Fetal | 3 | 1 | 4 | 1 | 5 | 1 | 6 | 6 | 3 |
| Group | Rank of ALB | Rank of TRG | Rank of GAPDH | ||||||
| geNorm | NormFinder | BestKeeper | geNorm | NormFinder | BestKeeper | geNorm | NormFinder | BestKeeper | |
| Pregnant and Non-pregnant | 4 | 5 | 4 | 5 | 6 | 3 | 6 | 4 | 5 |
| Pregnant | 5 | 4 | 2 | 3 | 3 | 4 | 6 | 6 | 6 |
| Non-pregnant | 1 | 2 | 1 | 6 | 6 | 6 | 5 | 5 | 2 |
| Maternal+fetal | 5 | 5 | 5 | 6 | 6 | 2 | 1 | 4 | 3 |
| Maternal | 1 | 2 | 4 | 6 | 6 | 2 | 4 | 3 | 6 |
| Fetal | 4 | 2 | 6 | 5 | 4 | 5 | 1 | 3 | 2 |
根据计算所得稳定值将内参基因按照拷贝数稳定性由高到低进行排序(表 3),在孕妇与非孕妇整体组、孕妇组、非孕妇组和母体与胎儿整体组拷贝数稳定性最高的内参基因分别为TERT、ACTB、HBB及HBB,经NormFinder软件分析所得出的拷贝数稳定性最高和最低的基因,与geNorm软件的分析结果几乎相同;在母源DNA组HBB的拷贝数稳定性最高,在胎源DNA组ACTB的拷贝数稳定性最高。
2.5 BestKeeper软件分析各组内参基因的拷贝数稳定性根据计算所得R值将内参基因按照拷贝数稳定性由高到低进行排序(表 3),在孕妇与非孕妇整体组、孕妇组、非孕妇组、母体与胎儿整体组、母源DNA组和胎源DNA组拷贝数稳定性最高的基因分别为TERT、ACTB、ALB、HBB、HBB及HBB。经BestKeeper软件分析,各组拷贝数稳定性最高的内参基因,与geNorm软件分析结果几乎相同。SD计算结果显示:各组拷贝数最为稳定的内参基因所对应的SD值均小于1.0,证明该结果可以采用。
2.6 3种软件结果综合分析各组内参基因的拷贝数稳定性综合分析3种程序的统计结果,本文作者认为:孕妇与非孕妇整体组、孕妇组、非孕妇组、母体与胎儿整体组、母源DNA组和胎源DNA组拷贝数稳定性最高的基因分别为TERT、ACTB、ALB、HBB、HBB和HBB。采用方差分析进行上述组别之间稳定性最高的内参基因的Ct值的两两比较,结果显示:母体与胎儿整体组、母源DNA组、母源DNA组和胎源DNA组的Ct值比较差异均有统计学意义(F=114.84,P < 0.05),其余各组之间Ct值比较差异均无统计学意义(P>0.05)。
3 讨 论cffDNA的发现使其成为NIPT的重点研究方向。在健康的孕妇体中,从怀孕第7周起即可以检测到cffDNA[9],其含量随着孕程逐渐增加,3个月后达到平台期,在孕晚期继续增加,在产后2 h内迅速从母体中清除。目前的研究[10]认为:孕妇血浆中的胎儿DNA主要来源于凋亡的胎盘绒毛膜细胞,是小片段的DNA分子穿过胎盘屏障进入到母体血液循环中所致。因此,cffDNA具有一些不同于母体来源的DNA的特点,即cffDNA分子多小于300 bp,而母体来源的DNA分子多大于300 bp[11]。基于该特点,研究者可以通过电泳等方法将cffDNA从大量的母体DNA背景中特异性分离出来。这些特点使得cffDNA成为NIPT最理想的检材。目前应用于研究cffDNA的方法有很多,如甲基化免疫沉淀法(MeDiP)、数字化PCR法(digital PCR)和大规模平行测序法(MPS)等,其中qPCR是研究cffDNA最为基础、灵敏、准确并且广泛应用的实验方法,由于qPCR实验成本低、操作简单,已经成功地应用于诸如性别鉴定、β-地中海贫血、RhD血型鉴定和非整倍体疾病等多种先天性疾病的产前诊断。尽管qPCR的应用已经极为广泛,但是在其应用过程中仍然面临着诸多问题,其中最重要的是如何能够尽量避免或减小加样误差、反应体系中的抑制物成分对反应效率的影响、起始模板量的差异等系统误差引起的分析结果不准确[12]。为了解决该问题,研究者使用内参基因进行qPCR结果的校正和标准化,从而减小系统误差。内参基因被定义为在各种生物样品中广泛存在并且稳定表达的基因,在DNA研究中经常用作确定DNA的存在和定量分析各组样品中的DNA拷贝数[13]。然而,目前的研究尚未发现有任何一种内参基因在所有组织中表达量均保持稳定。对于血浆cffDNA的qPCR研究,理想的内参基因不应该受到母体怀孕孕程、应激反应、外界刺激或其他生理病理因素的影响,其在血浆中的拷贝数在不同身体状态下以及不同个体之间应该保持稳定[14]。然而近期的研究[15]结果表明:广泛使用的内参基因的表达量在不同研究中有很大的差异。例如,有研究[16]显示:B2M、ACTB 和SDHA的表达量在癫痫患者的脑组织中有明显差异,经常被用做代表细胞数量的单拷贝内参基因HBB也被证实不是最为可靠的内参基因[13]。采用qPCR进行分析之前应对内参基因的表达稳定性进行评价,从而确定可靠的内参基因,尤其是当cffDNA含量极少,并且非整倍体疾病中DNA拷贝数差异不是很明显的情况下,这是一个不可或缺的步骤。
本研究首次评价了6种常用的内参基因在孕妇血浆DNA、非孕妇女性血浆DNA和孕妇血浆中不同长度的DNA片段之间的拷贝数稳定性。本研究采用孕中期(即12~24孕周)的孕妇血浆DNA进行分析,因为在这一时期内孕妇血浆DNA含量最为稳定。6种内参基因分别为HBB、TERT、GAPDH、ALB、ACTB和TRG,本研究采用qPCR法检测其在所有样品中的拷贝数,数据分析采用geNorm、NromFinder、BestKeeper 3种统计软件。针对6种内参基因的拷贝数稳定性排序,3种软件所得出的结果略有不同,这种差异可能是由于3种软件使用的是不同的统计学计算方法所造成的[17]。综合3种软件的统计结果,本文作者认为:在孕妇与非孕妇整体组、孕妇组、非孕妇组、母体与胎儿整体组、母源DNA组和胎源DNA组中,拷贝数稳定性最高的基因分别为TERT、ACTB、ALB、HBB、HBB及HBB。其中母体与胎儿整体组和母源DNA组、母源DNA组和胎源DNA组内参基因的Ct值比较差异有统计学意义,这种差异可能来自于同一基因在不同样本中或不同基因在同一样本中的表达量不尽相同,但这种差异并不影响同一基因在同一样本中的表达稳定性。如果实验中计划联合使用多个内参基因进行qPCR结果的标准化校正,则可以根据geNorm结果中的V值确定最佳的内参基因联合使用数量,本研究结果显示:几乎所有V值均大于0.15这一分界值,即无合适推荐的内参基因联合使用数量。有学者[18]认为:在实验条件允许的情况下,可以联合使用3种内参基因进行qPCR结果的校正。
血浆中DNA的含量极低,并且主要来源于细胞的凋亡过程。这也可能是影响血浆DNA扩增效率的因素之一。原因可能为:首先,在血浆DNA的扩增过程中,扩增的目的片段应该尽量短,因为目的片段越长,其模板越有可能在细胞凋亡过程中被降解,导致有效的模板量降低;其次,实验无法保证通过血浆DNA每次都能够成功地扩增目的基因。越来越多的研究正致力于将血浆DNA应用于临床诊断,血浆DNA的应用对于NIPT有极为重要的影响。尽管如此,目前在应用qPCR法进行的针对血浆DNA的研究中,所有的内参基因都是根据经验选择的,其在血浆中的拷贝数是否稳定并未经过系统评价和比较。本研究证实了在孕中期孕妇血浆DNA中拷贝数最为稳定的内参基因,本研究结果为利用孕妇血浆DNA进行后续实验提供了理论依据。
| [1] | Lo YM,Corbetta N,Chamberlain PF,et al.Presence of fetal DNA in maternal plasma and serum[J].Lancet,1997,350(9076):485-487. |
| [2] | Zhong Q,Zhang Q,Wang Z,et al.Expression profiling and validation of potential reference genes during Paralichthys olivaceus embryogenesis[J].Mar Biotechnol,2008,10(3):310-318. |
| [3] | Ali H,Du Z,Li X,et al.Identification of suitable reference genes for gene expression studies using quantitative polymerase chain reaction in lung cancer in vitro[J].Mol Med Rep,2015.11(5):3767-3773. |
| [4] | Li X,Yang Q,Bai J,et al.Identification of appropriate reference genes for human mesenchymal stem cell analysis by quantitative real-time PCR[J].Biotechnol Lett,2015,37(1):67-73. |
| [5] | Ma H,Yang Q,Li D,et al.Validation of suitable reference genes for quantitative polymerase chain reaction analysis in rabbit bone marrow mesenchymal stem cell differentiation[J].Mol Med Rep,2015,12(2):2961-2968. |
| [6] | Yang Q,Ali HA,Yu S,et al.Evaluation and validation of the suitable control genes for quantitative PCR studies in plasma DNA for noninvasive prenatal diagnosis[J].Int J Mol Med,2014,34(6):1681-1687. |
| [7] | Qin XY,Li GX,Qin YZ,et al.Quantitative assessment of hematopoietic chimerism by quantitative real-time polymerase chain reaction of sequence polymorphism systems after hematopoietic stem cell transplantation[J].Chin Med J,2011,124(15):2301-2308. |
| [8] | Picchiassi E,Coata G,Fanetti A,et al.The best approach for early prediction of fetal gender by using free fetal DNA from maternal plasma[J].Prenat Diagn,2008,28(6):525-530. |
| [9] | Galbiati S,Smid M,Gambini D,et al.Fetal DNA detection in maternal plasma throughout gestation[J].Hum Genet,2005,117(2-3):243-248. |
| [10] | Alberry M,Maddocks D,Jones M,et al.Free fetal DNA in maternal plasma in anembryonic pregnancies:confirmation that the origin is the trophoblast[J].Prenat Diagn,2007,27(5):415-418. |
| [11] | Chan KC,Zhang J,Hui AB,et al.Size distributions of maternal and fetal DNA in maternal plasma[J].Clin Chem,2004,50(1):88-92. |
| [12] | Bustin SA,Benes V,Nolan T,et al.Quantitative real-time RT-PCR-a perspective[J].J Mol Endocrinol,2005,34(3):597-601. |
| [13] | Steinau M,Rajeevan MS,Unger ER.DNA and RNA references for qRT-PCR assays in exfoliated cervical cells[J].J Mol Diagn,2006,8(1):113-118. |
| [14] | Peters IR,Peeters D,Helps CR,et al.Development and application of multiple internal reference (housekeeper) gene assays for accurate normalisation of canine gene expression studies[J].Vet Immunol Immunopathol,2007,117(1/2):55-66. |
| [15] | Li X,Yang Q,Bai J,et al.Identification of optimal reference genes for quantitative PCR studies on human mesenchymal stem cells[J].Mol Med Rep,2015,11(2):1304-1311. |
| [16] | Wierschke S,Gigout S,Horn P,et al.Evaluating reference genes to normalize gene expression in human epileptogenic brain tissues[J].Biochem Biophys Res Commun,2010,403(3/4):385-390. |
| [17] | Chang E,Shi S,Liu J,et al.Selection of reference genes for quantitative gene expression studies in platycladus orientalis (Cupressaceae) using real-time PCR[J].PLoS One,2012,7(3):e33278. |
| [18] | Liman M,Wenji W,Conghui L,et al.Selection of reference genes for reverse transcription quantitative real-time PCR normalization in black rockfish (Sebastes schlegeli)[J].Mar Genomics,2013,11(1):67-73. |
 2016, Vol. 42
2016, Vol. 42


