扩展功能
文章信息
- 糟航, 王旭, 李薇
- ZAO Hang, WANG XU, LI WEI
- CDA基因单核苷酸多态性对中晚期非小细胞肺癌接受吉西他滨化疗患者预后的影响
- Influence of single nucleotide polymorphisms of CDA in prognosis of advanced non-small cell lung cancer patients treated with gemcitabine
- 吉林大学学报(医学版), 2016, 42(02): 316-320
- Journal of Jilin University (Medicine Edition), 2016, 42(02): 316-320
- 10.13481/j.1671-587x.20160224
-
文章历史
- 收稿日期: 2015-11-06
2. 新疆医科大学研究生学院, 新疆乌鲁木齐 830011
2. Graduated School, Xinjiang Medical University, Urumqi 830011, China
肺癌是最常见的恶性肿瘤之一,在世界卫生组织2012年最新的调查数据中显示:每年约有840万人死于肿瘤,其中有160万人死于肺癌,居肿瘤相关死亡原因的首位[1]。约80%的病例是非小细胞肺癌(non-small cell lung cancer,NSCLC),2/3的患者确诊时即为晚期,故目前化疗仍然是主要治疗手段[2]。吉西他滨联合铂类方案是晚期NSCLC化疗最常用的标准方案之一[3],有研究[3, 4]显示:相较于铂类联合紫杉醇等其他第三代化疗药,铂类联合吉西他滨化疗后患者1年生存获益可提高4.2%,但晚期NSCLC患者化疗后的1年生存率仍然不超过40%。研究[5]显示:NSCLC的化疗已经达到了平台期,以后关于NSCLC化疗方案的临床研究应该包括分子预后指标。
单核苷酸多态性(single nucleotide polymorphisms,SNPs)是指在染色体序列上由于单个核苷酸位置上存在转换、颠换、插入和缺失等变异而引起的DNA序列多态性,且至少在一个人群中这种变异的发生频率在1%以上[6]。研究[7, 8]发现:某些功能基因的SNPs对化疗的疗效及预后具有一定影响,但尚无统一定论。吉西他滨主要依赖胞苷脱氨酶催化失活,但其SNPs的作用仍然有争议。本研究分析了CDA基因79A>CSNPs与接受吉西他滨化疗的中晚期NSCLC患者临床预后的关系,以期寻找可能预测NSCLC化疗预后的指标,从而为根据患者遗传特点进行个体化治疗提供理论依据。
1 资料与方法 1.1 研究对象选择2011年3月1日—2014年7月1日于吉林大学第一医院住院化疗的93例肺癌患者,所有患者均为病理学确诊的不能手术治疗的中晚期NSCLC初治患者,化疗前功能状态KPS评分均>60分,血常规、 肝肾功能在正常范围内,心电图正常。随访截止日期为2015年3月15日。
1.2 化疗方案所有患者均接受吉西他滨联合顺铂的一线化疗方案,具体方案为:吉西他滨1000mg·m-2,d1,8;顺铂25 mg·m-2,d2~4;3~4周重复1次。所有患者均接受4~`6个周期化疗。
1.3 临床观察及评价以总生存时间(overall survival,OS)和无进展生存期(progression free survival,PFS)作为最终评价指标。OS指确诊日至患者末次随访或死亡(死于肿瘤或并发症)的时间。PFS指从确诊日至患者疾病出现进展的时间。患者疗效评价根据RECIST1.1标准进行评估[9]。
1.4 标本采集及CDA基因SNPs的检测初诊时采集患者抗凝全血3~5 mL,将其分装到1.5 mL的EP管中置于-20℃冰箱中进行保存,应用Promega公司全血DNA提取试剂盒说明提取外周血DNA,应用Sequenom Massarray genotype系统进行CDA基因79A>C SNPs的检测。
1.5 统计学分析应用SPSS 17.0软件进行统计学分析。采用χ2检验进行基因型分布与临床病理特征之间的比较,采用Fisher’ s确切概率法进行检验。采用Kaplan-meier法分析生存情况,Log-rank法行单因素检验,COX回归法行多因素分析。以P<0.05为差异有统计学意义。
2 结果 2.1 CDA基因79A>C基因型分布在93例NSCLC患者中,所有的基因型均被检出。64例携带A/A基因型,4例携带C/C基因型,25例携带A/C基因型。其中A等位基因出现的频率为83.2%,C等位基因出现的频率为16.8%。见表 1。
| Clinical characteristic | N(η/%) | Genotype[n(η/%)] | χ 2 | P | |
| AA | AC+CC | ||||
| Gender | |||||
| MaleFemale | 78(83.9) 15(16.1) |
54(69.3) 10(66.7) |
24(30.8) 5(33.3) |
0.039 | 0.845 |
| Age(year) | |||||
| <60≥60 | 51(54.8) 42(45.2) |
37(72.5) 27(64.3) |
14(27.5) 15(35.7) |
0.733 | 0.501 |
| BMI(kg·m -2) | |||||
| <18 | 2(2.2) | 1(50.0) | 1(50.0) | ||
| 18-24 | 59(63.4) | 43(72.9) | 16(27.1) | 1.379 | 0.358 |
| >24 | 32(34.4) | 20(62.5) | 12(37.5) | ||
| Smoking state | |||||
| Never-smokingEver-or current smokers | 30(32.3) 63(67.7) |
23(76.7) 41(65.1) |
7(23.3) 22(34.9) |
1.272 | 0.340 |
| ECOG score | |||||
| 0 | 13(14.0) | 8(61.5) | 5(38.5) | ||
| 1 | 79(84.9) | 55(69.6) | 24(30.4) | 0.798 | 0.683 |
| 2 | 1(1.1) | 1(100.0) | 0(0.0) | ||
| Histopathological type | |||||
| Squamous cell cancer | 46(49.4) | 34(73.9) | 5(26.1) | ||
| Adenocarcinoma | 41(44.1) | 24(58.5) | 24(41.5) | 5.295 | 0.073 |
| Qthers | 6(6.5) | 6(100.0) | 0(0.0) | ||
| Clinical stage | |||||
| ⅢA | 15(16.2) | 10(66.7) | 5(33.3) | ||
| ⅢB | 27(29.0) | 19(70.4) | 8(29.6) | 0.064 | 0.969 |
| Ⅳ | 51(54.8) | 35(68.6) | 16(31.4) | ||
| Second-line treament | |||||
| ChemotherapyTargeted therapy | 50(71.6) 12(19.4) |
37(74.0) 7(58.3) |
13(26.0) 5(41.7) |
1.153 | 0.305 |
| AC+CC: Carrying at least one allele C;campared with wild -type AA. | |||||
93例患者全部完成随访,随访截止日期为2015年3月15日。其中死亡63例,存活30例。CDA基因79A>C SNPs与NSCLC患者临床及肿瘤病理特征均未见明显相关性。患者总体中位PFS为10.1个月(95%CI,9.2~11.0个月),中位OS为21.8个月(95%CI,19.5~24.0个月)。 携带野生型基因型(A/A)患者的中位PFS为9.3个月,携带突变型基因型(A/C+C/C)患者中位PFS为11.0个月,携带突变型基因型患者的中位PFS有延长趋势(P=0.061)。而携带野生型基因型A/A患者的中位OS为21.6个月,携带突变型基因型A/C+C/C患者中位 OS为22.1个月,二者中位OS比较差异无统计学意义(P=0.513)。CDA基因79A>C不同基因型患者生存曲线见图 1。
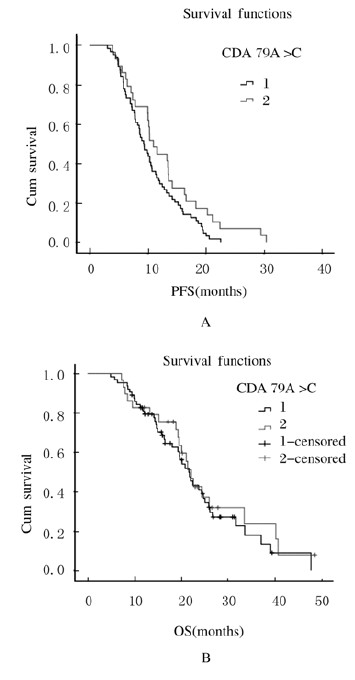
|
| 1 represented AA genotype;2 represented AC+CC genotypes. 图1 CDA基因79A>C不同基因型患者PFS和OS曲线 Fig.1 Curves of PFS and OS of patients with different genotypes of CDA gene 79A>C |
经COX比例风险模型对性别、年龄、体质量指数(body mass index,BMI)、美国东部肿瘤协作组(Eastern Cooperative Oncology Group,ECOG)评分、吸烟状态、病理类型、分期和二线治疗是否包含靶向治疗等因素进行校正发现:与携带野生型基因型的个体比较,携带突变型等位基因的个体死亡风险比值有所降低,但差异无统计学意义(HR,0.836;95%CI,0.488~1.431;P=0.514)。而相对于野生型个体,突变等位基因的携带者疾病进展风险却明显降低(HR,0.647;95%CI,0.409~1.024;P=0.063),但不能作为影响疾病进展的独立因素。见表 2和3。
| CDA genotype | Medeian PFS(months)(95%CI) | χ 2 | P | HR | P |
| A/A | 9.3(7.9-10.7) | 1 | |||
| A/A+A/C | 11.0(8.7-13.3) | 3.509 | 0.061 | 0.641(0.409-1.024) | 0.063 |
| CDA genotype | Medeian OS(months)(95%CI) | χ 2 | P | HR | P |
| A/A | 21.6(18.7-24.6) | 1 | |||
| A/A+A/C | 22.1(19.8-24.5) | 0.428 | 0.513 | 0.836(0.488-1.431) | 0.514 |
在化疗方案与给药剂量相同的情况下,不同个体由于遗传背景的差异,对化疗药物的反应存在很大的差异。近年来通过检测SNPs遗传标记,根据个体的遗传特性或癌组织的遗传标记制定化疗方案,是目前面临的一项重要挑战。Marsh 等[10]的研究指出:由于SNPs属于遗传学标记,所以其分布于全身所有组织细胞,在肿瘤组织与正常组织中检测结果无差异。对于晚期未行肿瘤手术切除的患者,如能检测血液学标记,则对预测药物疗效和判 断预后非常有益。CDA基因位于人体染色体 1p36.2-p35,是一种在失活吉西他滨作用机制中发挥重要作用的关键酶,酶活性受损的患者可能发生严重的毒性反应,而过表达又可能会降低吉西他滨的抗肿瘤效果。众所周知,CDA是趋于多态性的酶,Teng等[11]在人类中性粒细胞电泳观察分析时发现存在3种CDA基因表型,并提出存在纯合子和杂合子2种等位基因的观点。随后有2个研究小组明确了CDA突变基因的2个cDNA编码序列,其以1个非保守的氨基酸取代了在27位点的氨基酸序列,CDA1位点携带谷氨酸残基(Q),CDA2位点携带赖氨酸残基(K)[12, 13]。有研究[14]证实:CDA基因 79A>C SNPs导致了编码区内K27Q的改变。研究[15]报道:K27Q的多态性会轻度降低CDA的活性,但被取代的第27位氨基酸序列所编码的蛋白质区域接近于四聚体的保守区,而这些区域并不直接参与酶的催化过程。
在中国,CDA 基因79A>C SNP的发生率在癌症患者和健康人群中分别是12.0%和12.3%,这与非裔美国人群中发生率[16]基本一致。本研究中CDA 基因79A>C 突变率为16.8%,研究结果显示:CDA基因 79A>C SNPs对患者PFS有一定的延长作用,但未发现与OS明显相关。在最近的一项以中国人群为研究对象的研究[17]中发现:CDA基因79A>C SNPs与预后无关,这与之前日本学者[7]进行的一项对于256例接受吉西他滨化疗的患者CDA SNPs的研究结论相似。目前关于CDA基因79A>C SNPs预测吉西他滨化疗效果的报道也存在争议,而大部分的研究来自欧美人群。
Gilbert等[14]指出:CDA基因79A>C SNPs可导致吉西他滨脱氨酶活性显著降低,这一结论在Tibaldi等[8]的研究中也得到了证实,该研究结果显示:CDA基因79A>C SNPs中A/A基因型的患者具有更好的化疗获益率、更长的PFS以及OS,可作为预测化疗效果和毒性反应的独立指标。而Kirch等[18]则报道了与之截然相反的结果。Maring等[19]就NSCLC患者CDA基因79A>C 所导致的非同义K27Q突变对吉西他滨药代动力学的影响进行研究,未发现CDA基因K27Q对药物下阴影面积、清除率及血浆半衰期有任何影响。其他的一些体外研究结果也并未显示CDA活性与吉西他滨的敏感性之间存在关联,仅在转染细胞中显示出CDA基因79A>C SNPs可导致较高的CDA活性和较低的吉西他滨的敏感性。
总之,本研究结果显示:CDA基因79A>C SNPs与接受吉西他滨化疗的NSCLC患者的预后无关。也许不同研究得出的不同结果与样本大小和在本研究中评估的人种有关。本次调查结果的一个解释可能是由于吉西他滨代谢受多种因素的影响:CDA基因转录、CDA的酶活性以及参与吉西他滨代谢其他的一些辅助酶影响因素等。目前对于从血液样本中和癌组织中直接获得的基因型SNP是否一致仍然存在争议。SNPs代表的遗传变异存在于身体的所有细胞,但基因型代表一个静态值可能无法应对不同的环境变化,在化疗的环境 中可能不能反映肿瘤的DNA的变化,如杂合性缺失。因此,需要进行更多的研究来建立更加有效和可靠的预测指标为晚期NSCLC患者化疗提供依据。
| [1] | Ferlay J,Soerjomataram I,Dikshit R,et al.Cancer incidence and mortality worldwide:sources,methods and major patterns in GLOBOCAN 2012[J].Int J Cancer,2015,136(5):E359-386. |
| [2] | Jemal A,Siegel R,Ward E,et al.Cancer statistics,2007[J].CA Cancer J Clin,2007,57(1):43-66. |
| [3] | Le Chevalier T,Scagliotti G,Natale R,et al.Efficacy ofgemcitabine plus platinum chemotherapy compared with otherplatinum containing regimens in advanced non-small-cell lung cancer:ameta-analysis of survival outcomes[J].Lung Cancer,2005,47(1):69-80. |
| [4] | Azzoli CG,Baker S Jr,Temin S,et al.American Society of Clinical Oncology Clinical Practice Guideline update on chemotherapy for stage IV non-small-cell lung cancer[J].J Clin Oncol,2010,13(3):171-189. |
| [5] | Schiller JH,Harrington D,Belani CP,et a1.Comparisonof four chemotherapy regimens for advanced non-small cell lung cancer[J].N Engl J Med,2002,346(2):92-98. |
| [6] | Taylor JG,Choi EH,Foster CB,et al.Using genetic variation to study humandisease[J].Trends Mol Med,2001,7(11):507-512. |
| [7] | Sugiyama E,Kaniwa N,Kim SR,et al.Pharmacokinetics of gemcitabinein Japanese cancer patients:the impact of a cytidine deaminase polymorphism[J].J Clin Oncol,2007,25(1):32-42. |
| [8] | Tibaldi C,Giovannetti,Vasile E,et al.Correlation of CDA,ERCC1,and XPD polymorphisms with response and survivalin gemcitabine/cisplatin treated advanced non-small cell lung cancer patients[J].Clin Cancer Res,2008,14(6):1797-1803 |
| [9] | Eisenhauer EA,Therasse P,Bogaerts,et al.New response evaluation criteria in solid tumours:revised RECIST guideline (version 1.1)[J].Eur J Cancer,2009,45(2):228-247. |
| [10] | Marsh S,Mallon MA,Goodfellow P,et al.Concordance of pharmacogenetic markers in germline and colorectal tumor DNA[J].Pharmacogenomics,2005,6(8):873-877. |
| [11] | Teng YS,Anderson JE,Giblett ER. Cytidine deaminase:a new genetic polymorphism demonstrated in human granulocytes[J].Am J Hum Genet,1975,27(4):492-497. |
| [12] | Kühn K,Bertling WM,Emmrich F.Cloning of a functional cDNA for human cytidine deaminase (CDD) and its use as a marker of monocyte/macrophage differentiation[J].Biochem Biophys Res Commun,1993,190(1):1-7. |
| [13] | Laliberté J,Momparler RL.Human cytidine deaminase:purification of enzyme,cloning,and expression of its complementary DNA[J].Cancer Res,1994,54(20):5401-5407. |
| [14] | Gilbert JA,Salavaggione OE,Ji Y,et al.Gemcita-bine pharmacogenomics:cytidine deaminase and deoxycytidylate deaminase gene resequencing and functional genomics[J].Clin Cancer Res,2006,12(6):1794-1803. |
| [15] | Vincenzetti S,Mariani PL,Cammertoni N,et al.Isoenzymatic forms of human cytidine deaminase[J].Protein Eng Des Sel,2004,17(12):871-877. |
| [16] | Xu J,Zhou Y,Zhang J,et al.High incidence of severe neutropenia after gemcitabine-based chemotherapy in Chinese cancer patients with CDA 79A>C mutation[J].Clin Chim Acta,2012,413(15/16):1284-1287. |
| [17] | Zhou M,Ding YJ,Feng Y,et al.Association of xeroderma pigmentosum group D(Asp312Asn,Lys751Gln) and cytidine deaminase (Lys27Gln,Ala70Thr) polymorphisms with outcome in Chinese non-small cell lung cancer patients treated with cisplatin-gemcitabine[J].Genet Mol Res,2014,13(2):3310-3318. |
| [18] | Kirch HC,SchroderJ,Hoppe H,et al.Recombinant gene products of two natural variants of the humancytidine deaminase gene conferdifferent deamination rates of cytarabine in vitro[J].Exp Hematol,1998,26(5):421-425. |
| [19] | Maring JG, Wachters FM,Slijfer M,et al.Pharmacokinetics of gemcitabine in non-small-cell lung cancer patients:impact of the 79A>C cytidine deaminase polymorphism[J].Eur J Clin Pharmacol,2010,66(6):611-617. |
 2016, Vol. 42
2016, Vol. 42


