扩展功能
文章信息
- 乔莎, 黄映辉, 王世宝, 孙延霞, 卢振霞
- QIAO Sha, HUANG Yinghui, WANG Shibao, SUN Yanxia, LU Zhenxia
- 不同培养方法对乳腺癌MDA-MB-231细胞DNA甲基化状态的影响
- Influence of different culture methods in DNA methylation status of breast cancer MDA-MB-231 cells
- 吉林大学学报(医学版), 2016, 42(02): 271-276
- Journal of Jilin University (Medicine Edition), 2016, 42(02): 271-276
- 10.13481/j.1671-587x.20160216
-
文章历史
- 收稿日期: 2015-11-11
2. 北京工业大学生命科学与生物工程学院肿瘤研究所, 北京 100124
2. Cancer Institute, College of Life Sciences and Bioengineering, Beijing Polytechnic University, Beijing 100124, China
肿瘤发生是多因素诱导并在体内的复杂环境逐步演化而成的过程。二维(two-Dimensional,2D)培养是肿瘤研究较成熟且主要的技术,简单、方便,细胞的生存力好,但缺少完整的组织微环境。小鼠原位移植(mouse orthotopic transplantion,简称Ti)模型虽有肿瘤细胞生长介质,但模型的构建容易受动物、细胞和外界环境等因素影响,较难完成,且实验成本高不易实施。三维(three-dimensional,3D)模型是一种新的实验技术和方法,模拟体内复杂的生长环境,如细胞与基质相互作用、细胞之间黏附,模拟肿瘤组织复杂的信号转导通路;与内皮细胞共培养时模拟新生血管形成;细胞成团生长时模拟细胞团中心缺氧、坏死等。3D模型类似于Ti模型,模拟细胞复杂的生长环境且节约成本,又可像2D模型方便构建,但与肿瘤细胞体内生长环境仍有差距。越来越多的学者[1, 2]采用此技术探索肿瘤形态学、增殖分化、基因表达、侵袭和转移、药物代谢等生物学行为。研究[3]发现:肿瘤发生、浸润、转移与表观遗传学密切相关。表观遗传学是指细胞不依赖DNA序列改变而发生的可遗传性变化,其机制主要包括4个方面:DNA甲基化、组蛋白修饰、染色体重塑和非编码RNAs[3]。DNA甲基化是基因表达重要调节因素之一,其在肿瘤发生发展中的作用也是研究的主题。有研究[4, 5]显示:细胞可通过改变甲基化状态调控基因的表达导致肿瘤生成、恶性转变。但DNA甲基化状态的改变是否受细胞培养方法的影响尚不清楚。目前国内外有关这方面的报道较少。本研究旨在探讨乳腺癌MDA-MB-231细胞经2D、3D和Ti模型培养后全基因组DNA甲基化水平的差异,为3D培养模型相比于其他模型的利弊研究奠定理论基础。
1 材料与方法 1.1 细胞、主要试剂和仪器人乳腺癌MDA-MB-231细胞系(吉林大学中日联谊医院科学研究中心提供)。DMEM培养基(美国Invitrogen公司),胎牛血清(美国Biological Industries公司),Matrigel胶(美国BD公司),胰酶粉(美国Sigma公司)。BX51生物显微镜和IX51倒置荧光显微镜(日本Olympus公司),5810R冷冻离心机和细胞计数仪(美国Invitrogen公司),371型二氧化碳培养箱(美国Forma公司)。
1.2 细胞培养与收集2D细胞培养模型:采用完全培养基(90%DMEM、10%FBS)将MDA-MB-231细胞置于37℃、5%CO2细胞培养箱中培养,取对数生长期的细胞用0.25%胰酶(含1%EDTA)消化、离心,收集细胞,并迅速放入液氮中冻存。3D细胞培养模型:将1×103个处于对数生长期的MDA-MB-231细胞(2D模型)与不含FBS的DMEM培养基按1∶1制成细胞悬液,取10μL均匀铺于预冷的6孔板中Matrigel胶面上,使细胞成单个分布,置于37℃、5%CO2细胞培养箱中培养20min,使细胞固定于胶面上,再轻轻沿着孔壁加入1500μL完全培养基。每隔1d更换完全培养基。7d后抽弃上层培养液,用预冷的细胞回收液溶解Matrigel胶,后用剪去头部的100μL枪头吹打至无絮状物,4℃、3000r·min-1离心5 min,吸弃上清,PBS重悬转至1.5 mL EP管中,4℃、 6000 r·min-1离心3min收集细胞,并迅速放入液氮中冻存。Ti模型:取对数生长期的MDA-MB-231细胞(2D模型)用无血清培养液稀释成1×106~1×107mL-1细胞悬液,切开裸鼠胸壁皮肤,暴露乳房脂肪垫,注入0.2 mL(含1×107个细胞)细胞悬液,缝合切口。正常喂养。14 d后将小鼠皮下形成的肿瘤组织取出,切成3 mm×3 mm×3 mm的小块后,迅速放入液氮中冻存。
1.3 DNA的提取及甲基化芯片全基因组检测采用QIAamp DNA Mini Kit试剂盒(上海欧易生物医学科技有限公司)对上述收集的细胞及肿瘤组织团块进行DNA提取,然后采用DNA甲基化450K芯片对不同培养环境培养后的MDA-MB-231细胞进行全基因组检测。由该公司按照以下几个步骤完成:①用EZ DNA甲基化试剂盒对提取的基因组DNA进行亚硫酸氢盐转化处理;②制备亚硫酸氢盐转换的DNA模板及碱变性-全基因组扩增(MSA4);③对扩增后的DNA样品进行酶切片段化处理,并对其用沉淀液PM1及异丙醇进行沉淀,对沉淀后的DNA用RA1重悬液进行重悬;④将重悬的DNA样本分散于BeadChip芯片上,用Illumina杂交炉进行杂交处理、荧光染色和芯片扫描。
1.4 甲基化差异计算采用Genomestudio软件(2011.1版本,美国Illumina公司)对芯片扫描的数据进行标准化处理后得出标准化信号值及检出基因情况(检出标准:P < 0.05)。①Illumina甲基化芯片针对每1个CpG位点设计双色荧光探针检测甲基化和非甲基化的等位基因。SignalA为未甲基化探针信号值,SignalB为甲基化探针信号值,根据公式计算CpG位点的β值,评估该位点的甲基化水平。②Delta_Beta=βref-βcond,评估该位点甲基化差异程度;DiffScore=10×sgn(βref-βcond)×log10(P),评估甲基化状态。其中βref为对照组的β值,βcond为实验组的β值,P为差异显著水平。P取0.05,DiffScore=±13。设对照组的Delta_Beta为1,Delta_Beta为正数表示实验组基因甲基化增加,负数则相反。DiffScore取正值代表实验组甲基化程度升高,负值则相反。β=Max(signalB.0)/[Max(signalA.0)+Max(signalB.0)+100]。
1.5 统计学分析①差异甲基化基因筛选:采用Genomestudio软件(2011.1版本,美国Illumina公司)进行统计分析,用于比较的2组设有三重复及以上采用t检验,不满足上述条件的则采用Illumina Custom Model。差异甲基化基因:实验组样本P<0.05,Diffscore值小于-13或大于13;且Delta_ Beta大于0.17或小于-0.17。②差异甲基化基因富集分析:采用DAVID在线分析工具(DAVID v6.7)对筛选出的差异甲基化基因根据基因本体论(Gene Ontology,GO)和京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)进行功能描述和Pathway分析,模型间每个差异甲基化基因在GO条目、细胞通路中返回的值以P表示。以P≤0.05表示显著富集。
2 结果 2.1 3种培养方法下MDA-MB-231细胞生长方式不同于传统的2D培养,3D模型下MDA-MB-231细胞呈团块状生长,Ti模型培养的MDA-MB-231细胞则在胸壁下形成肿瘤肿块。见图 1(插页四)。
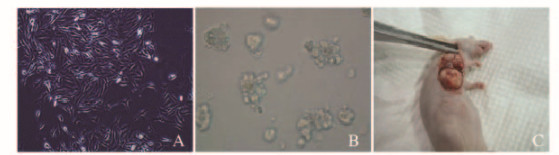
|
| A:2D cell culture model; B: 3D cell culture model; C: Mouse orthotopic transplantation model 图1 3种培养方法下MDA-MB-231细胞生长方式 Fig.1 Growth of MDA-MB-231 cells under different culture methods |
3D与2D模型的差异甲基化基因个数较3D与Ti模型少,3D与Ti模型和Ti与2D模型数目相似。3D与2D模型、Ti与2D模型差异基因的甲基化程度以降低为主,3D与Ti模型则以升高为主。见表 1。
| (x± s ) | |||
| Methylation gene | Differential screening | Lower degree of methylation | Higher degree of methylation |
| 3D vs 2D | 480 | 267 | 213 |
| 3D vs Ti | 86448 | 13342 | 73106 |
| Ti vs 2D | 90005 | 73539 | 16466 |
GO分析包括三大板块:生物学过程、细胞成分和分子功能。每个GO板块中,3D与2D模型的MDA-MB-231细胞差异甲基化基因富集个数明显较3D与Ti模型少;3D与Ti模型富集的差异甲基化基因个数相比于Ti与2D模型略少,结果见表 2。3D与Ti模型和Ti与2D模型的差异甲基化基因主要富集在与细胞生长黏附分化、信号转导、膜相关组成以及肽酶活性、序列特异性DNA结合、Ca2+结合等条目中。3D与2D模型、3D与Ti模型和Ti与2D模型的差异甲基化基因均在多细胞生物体发育、细胞分化条目上有富集。见图 2。富集的差异甲基化基因主要有RhO蛋白家族、CYFIP1、VEGFR1、HIC1、E-钙黏蛋白(E-cadherin)和整合素等。
| GO analysis | Biological process(BP) | Cellular component(CC) | Molecular function(MF) | |||
| Population Totala | List Totalb | Population Totala | List Totalb | Population Totala | List Totalb | |
| 3D vs 2D | 14200 | 172 | 16399 | 195 | 14962 | 175 |
| 3D vs Ti | 14200 | 9950 | 16399 | 11464 | 14962 | 10463 |
| Ti vs 2D | 14200 | 10153 | 16399 | 11707 | 14962 | 10661 |
| aThe number of genes available on the entire microarray and having at least one GO BP/CC/ MF annotation; b The number of differential methylation genes having at least one GO BP/CC/MF annotation,P≤0.05. | ||||||

|
| *BP;▲CC;●MF. 图2 不同模型间差异甲基化基因在GO部分条目中的富集情况 Fig.2 Enrichment of differential methylation genes in GO partial terms between different models |
3D与2D模型的差异甲基化基因富集的信号通路均比3D与Ti模型和Ti与2D模型少(表 3),主要是MAPK信号通路、细胞黏附分子和肌动蛋白骨架调节通路。相比于3D与2D模型,3D与Ti模型和Ti与2D模型富集的信号通路相似,除了在上述通路有富集外,在致肿瘤相关通路、细胞因子与其受体相互作用通路、Ca2+信号通路和Wnt信号通路也有一定量的富集。见图 3。
| Pathway analysis | 3D vs 2D | 3D vs Ti | Ti vs 2D |
| Population Totala | 5981 | 5981 | 5981 |
| List Totalb | 70 | 4161 | 4227 |
| a The number of genes available on the entire microarray and having at least one KEGG annotation;b The number of differential methylation genes having at least one KEGG annotation. | |||

|
| 图3 不同模型间差异甲基化基因富集的部分信号通路 Fig.3 Partial signaling pathway of differential methylation gene enrichment between different models |
目前肿瘤生物学行为研究多在传统的2D模型中进行,但其不能真实、充分反映肿瘤细胞生长环境对肿瘤生物学行为的影响。而肿瘤细胞生长环境对肿瘤发生发展至关重要,尤其是细胞间相互作用和细胞外基质(extracellular matrix,ECM)重塑[6]。Ti模型是通过动物构建肿瘤生长环境,较为复杂,易受多种因素影响。3D模型具备了ECM,在2D模型基础上形成与人体内相似的细胞间连接和细胞与基质相互作用的肿瘤微环境,是一种新的细胞培养方法,是2D培养和动物模型的桥接。3种模型均被应用于肿瘤研究。表观遗传学是近年来肿瘤研究较热门的一门学科,DNA甲基化是其机制之一。研究[7, 8]表明:DNA甲基化常发生在CpG岛中5′CpG3′的胞嘧啶残基上,与编码基因表达、基因组稳定性有关。异常甲基化状态影响细胞功能,导致肿瘤发生、浸润和转移。肿瘤DNA甲基化的研究多采用上述实验方法,而比较3种细胞培养模型对肿瘤细胞DNA甲基化影响的研究少有报道。本实验通过高通量甲基化芯片检测技术总体上分析不同模型营造的生长环境对人类乳腺癌MDA-MB-231细胞全基因组甲基化的影响。
本研究结果显示:不同模型间细胞生长方式不同,2D模型中MDA-MB-231细胞呈平面生长,3D和Ti模型的细胞形成连接,长成不同大小的团块,说明细胞在不同模型中的生长方式受其所在的培养环境影响,并因此可能表现的生物学行为不同。Bissell等[9]发现:阻断3D ECM和2D培养的恶性T4-2细胞生长因子受体信号通路中的关键受体后,3D ECM的细胞恶性表型逆转;同时下调β1整合素和表皮生长因子受体(epidermal growth factor receptor,EGFR)蛋白水平,后者在2D中并未变化,提示3D和2D模型营造的不同环境对肿瘤细胞生物学行为产生不同的影响;ECM可能使T4-2细胞形成微通路,连接了生长因子信号通路与β1整合素和EGFR表达。Benton等[10]发现:在2D和含Ⅰ型胶原蛋白的3D模型中呈单层分布的MCF-10A和MCF-7细胞则在含层黏连蛋白1的3D模型中形成了类似正常的腺泡结构或肿瘤团;高度恶性的MDA-MB-231细胞在含层黏连蛋白1的3D模型出现细胞连接而在2D和含Ⅰ型胶原蛋白的3D模型中未见,说明3D模型中微环境介质与肿瘤细胞生物学行为也有密切关系。
本研究结果显示:MDA-MB-231细胞基因组甲基化状态在3种模型间有差异,说明生长环境可能影响基因组甲基化状态。模型间差异甲基化基因涉及细胞生长分化、骨架蛋白合成及调节、细胞黏附和MAPK信号通路等方面。CYFIP1、RhO蛋白家族、VEGFR1、HIC1和E-钙黏蛋白等基因参与上述过程,且甲基化状态在不同模型间有差异,提示这些基因的异常甲基化可能与细胞在不同模型间的不同生物学行为有关。研究[11, 12]表明:Rho蛋白家族参与细胞形态改变、细胞与基质黏附及细胞骨架重组,调节肿瘤细胞侵袭和转移。研究[13]发现:超过30%人类肿瘤有Ras基因发生突变,但Rho蛋白家族作为Ras超家族,尚未见有关其基因突变的报道,且Rho信号通路下调与包括癌症在内的许多疾病发生发展有关,说明异常Rho蛋白信号可能与基因甲基化状态改变有关,并参与肿瘤发生过程。E-钙黏蛋白是1种肿瘤转移抑制基因,其表达的减弱或消失与肿瘤形成、浸润和转移密切相关。Benton等[10]发现:层黏连蛋白1能够降低MDA-MB-231细胞中E-钙黏蛋白启动子甲基化和DNA甲基化转移酶1(DNA methyltransferase-1,DNMT1)的表达,使在2D和含Ⅰ型胶原蛋白的3D模型中不表达E-钙黏蛋白的细胞出现再表达,提示ECM能通过改变启动子甲基化的状态影响细胞黏附分子的表达。DesRochers等[5]发现:不表达E-钙黏蛋白的鳞状上皮细胞Inv-1a有更强侵袭能力,浸润后的部分Inv-1a细胞再表达E-钙黏蛋白与E-钙黏蛋白启动子甲基化状态、DNMT1表达有关,提示E-钙黏蛋白的表观遗传学调控参与细胞侵袭力改变、肿瘤浸润和转移灶的形成。Teoh-Fitzgerald等[4]发现:细胞外超氧化物歧化酶(extracellular superoxide dismutase,EcSOD)在2D和3D模型培养的人乳腺上皮细胞(human mammary epithelial cell,HMEC)及肿瘤组织中表达水平与CpG甲基化改变有关,且与肿瘤发生、转移和临床分期有关。以上研究均表明:细胞生长环境影响基因组甲基化状态,且改变细胞生物学行为,证实了表观遗传学在肿瘤细胞发生、浸润和转移中作用。
本研究结果显示:3D与Ti模型和Ti与2D模型的差异甲基化基因在跨膜运输过程、Ca2+结合、细胞因子及受体作用通路中均有差异显著性富集,而3D与2D模型无差异,提示2D和3D模型不能模拟真实的上述细胞过程,与真实的肿瘤生长环境有一定差距,不能完全替代动物模型。但3D模型不受动物影响,可以通过ECM及细胞共培养研究细胞形态学、细胞与基质的相互作用和细胞极性等[1]。分析生物学行为及细胞生长分化、骨架蛋白合成及调节、细胞黏附等方面在3D和2D培养方法的差异发现:3D较2D模型在此类研究方面有一定的优势。
综上所述,细胞生长环境能改变DNA甲基化的状态,且影响细胞生长和相关信号通路;甲基化状态改变可能与乳腺肿瘤的发生、浸润和转移有关。3D模型是1种新的实验方法,构建类似肿瘤生长环境,桥接2D和动物模型,对肿瘤生物学行为研究有重要意义,但不能完全替代动物模型。
| [1] | Ravi M,Paramesh V,Kaviya SR,et al. 3D cell culture systems:advantages and applications[J]. J Cell Physiol,2015,230(1):16-26. |
| [2] | Lovitt CJ,Shelper TB,Avery VM.Evaluation of chemotherapeutics in a three-dimensional breast cancer model[J].J Cancer Res Clin Oncol,2015,141(5):951-959. |
| [3] | Dawson MA,Kouzarides T.Cancer epigenetics:from mechanism to therapy[J].Cell,2012,150(1):12-27. |
| [4] | Teoh-Fitzgerald ML, Fitzgerald MP,Zhong W,et al.Epigenetic reprogramming governs EcSOD expression during human mammary epithelial cell differentiation,tumorigenesis and metastasis[J]. Oncogene,2014,33(3):358-368. |
| [5] | DesRochers TM,Shamis Y,Alt-Holland A,et al. The 3D tissue microenvironment modulates DNA methylation and E-cadherin expression in squamous cell carcinoma[J]. Epigenetics,2012,7(1):34-46. |
| [6] | Cheung KJ,Ewald AJ.Illuminating breast cancer invasion:diverse roles for cell-cell interactions[J]. Curr Opin Cell Biol,2014,30C:99-111. |
| [7] | You JS,Jones PA.Cancer genetics and epigenetics:two sides of the same coin[J].Cancer Cell,2012,22(1):9-20. |
| [8] | Goel A,Boland CR.Epigenetics of colorectal cancer[J].Gastroenterology,2012,143(6):1442-1460. |
| [9] | Bissell MJ,Kenny PA,Radisky DC.Microenvironmental regulators of tissue structure and function also regulate tumor induction and progression:the role of extracellular matrix and its degrading enzymes[J].Cold Spring Harb Symp Quant Biol,2005,70:343-356. |
| [10] | Benton G,Crooke E,George J.Laminin-1 induces E-cadherin expression in 3-dimensional cultured breast cancer cells by inhibiting DNA methyltransferase 1 and reversing promoter methylation status[J]. FASEB J,2009,23(11):3884-3895. |
| [11] | Baranwal S,Alahari SK.Rho GTPase effector functions in tumor cell invasion and metastasis[J]. Curr Drug Targets,2011,12(8):1194-1201. |
| [12] | Luis-Ravelo D,Anton I,Zandueta C,et al. RHOB influences lung adenocarcinoma metastasis and resistance in a host-sensitive manner[J]. Mol Oncol,2014,8(2):196-206. |
| [13] | Ellenbroek SI,Collard JG.Rho GTPases:functions and association with cancer[J].Clin Exp Metastasis,2007,24(8):657-672. |
 2016, Vol. 42
2016, Vol. 42


