扩展功能
文章信息
- 魏旭静, 李林, 张红真, 王景, 徐静
- WEI Xujing, LI Lin, ZHANG Hongzhen, WANG Jing, XU Jing
- LncRNA CCAT1通过TGF-β/Smad信号通路对子宫内膜癌细胞增殖、侵袭和迁移的影响
- Effects of LncRNA CCAT1 on proliferation, invasion and migration of endometrial cancer cells through TGF-β1/smad signaling pathway
- 吉林大学学报(医学版), 2020, 46(05): 1016-1022
- Journal of Jilin University (Medicine Edition), 2020, 46(05): 1016-1022
- 10.13481/j.1671-587x.20200519
-
文章历史
- 收稿日期: 2019-09-23
子宫内膜癌是女性常见的恶性肿瘤,其发病机制复杂,探讨其发生发展机制对其诊治及改善其预后具有重要价值。近年来,子宫内膜癌细胞增殖、侵袭迁移及其分子机制方面的研究取得了巨大进步,长链非编码核糖核酸(long non coding RNA,LncRNA)在胚胎发育、基因表达和肿瘤发生等过程中发挥重要作用,多种LncRNA参与子宫内膜癌的发生发展过程[1]。近年来研究[2]发现:LncRNA CCAT1在多种恶性肿瘤的增殖和侵袭迁移过程中发挥重要作用,在子宫内膜癌中的作用也受到研究者关注,YU等[3]研究发现:LncRNA CCAT1可促进子宫内膜癌细胞的增殖和迁移。但其通过何种信号通路在子宫内膜癌发生发展中发挥作用尚不清楚。转化生长因子β(transforming growth factor-β,TGF-β)/Smad信号通路参与子宫内膜癌的增殖及侵袭迁移过程,SAHOO等[4]研究显示:抑制细胞外基质介导的TGF-β信号转导可抑制子宫内膜癌的转移。本实验通过观察沉默CCAT1对子宫内膜癌细胞增殖、侵袭和迁移及TGF-β/Smad信号通路的影响,探讨其在子宫内膜癌中的可能作用机制。
1 材料与方法 1.1 细胞、主要试剂和仪器人子宫内膜癌Ishiwaka细胞株和正常人子宫内膜基质细胞T-HESC(中国科学院上海细胞库);Lipofectamine 2000试剂盒(美国Invitrogen公司),CCAT1模拟物siRNA(CCAT1-siRNA)和阴性对照模拟物siRNA(广州博锐生物科技公司),ECL化学发光试剂盒、逆转录(RT)试剂盒、Trizol试剂、聚合酶链反应(PCR)试剂盒、LY364947(TGF-β/ Smad抑制剂)和结晶紫(美国Sigma公司),CCK8试剂、胰蛋白酶和DMEM培养基(美国BPB公司),兔抗人增殖细胞核抗原(PCNA)、E-钙黏蛋白(E-cadherin)、波形蛋白(vimentin)、锌指转录因子(snail)、凋亡抑制蛋白(Twist)、白细胞抑制因子2/3(Smad2/3)、磷酸化Smad 2/3(p-Smad2/3)、转化生长因子β1(TGF-β1)和兔抗人TGF-β1单克隆抗体(美国Santa Cruz公司);MultiskanTM FC酶标仪、Applied Biosystems PCR仪和NERLTM流式细胞仪(美国赛默飞世尔科技公司),Transwell小室(美国TaKaRa公司)等。
1.2 细胞培养Ishiwaka细胞和T-HESC细胞置于DMEM(含双抗和10%胎牛血清)培养,细胞贴壁后每2 d换液1次,细胞生长至达80%以上融合时进行细胞传代培养。
1.3 实时荧光定量PCR(RT-PCR)法检测Ishiwaka细胞和T-HESC细胞中CCAT1 mRNA表达水平取Ishiwaka和T-HESC细胞,加入Trizol裂解液裂解细胞,提取细胞总RNA,将RNA逆转录为cDNA,PCR法检测细胞中CCAT1 mRNA表达水平;CCAT1上游引物:5'-CCATTCCATTCAT-TTCTCTTTCCTA-3',下游引物:5'-GGCGTA-GGCGATTGGGGATCG-3'。PCR反应条件:95℃、30 s;95℃、5 s,58℃、30 s,72℃、30 s,共42个循环。以U6为内参照。每组设7个复孔。以2-∆∆Ct法计算细胞中CCAT1 mRNA表达水平。
1.4 细胞分组和转染取对数生长期的Ishiwaka细胞分为空白对照组、阴性对照组、CCAT1-siRNA组和CCAT1-siRNA+LY364947组,将各组对数生长期的Ishiwaka细胞接种到6孔细胞培养板中,每孔2×105个细胞,培养至细胞达90%以上融合时进行转染,阴性对照组转染阴性对照siRNA,CCAT1-siRNA组转染CCAT1-siRNA,CCAT1-siRNA + LY364947组转染CCAT1-siRNA同时加入LY364947(3 µL)[5],空白对照组不转染。转染步骤严格参照Lipofectamine 2000试剂盒说明书进行。转染24 h后采用RT-PCR法检测细胞中CCAT1 mRNA表达水平,方法同“1.2”。
1.5 CCK8法检测Ishiwaka细胞增殖能力取各组生长良好的Ishiwaka细胞接种到96孔细胞培养板中,每孔5×103个细胞,培养24、48和72 h时每孔分别加入10 µL的CCK8试剂,继续培养2 h,每组设7个复孔。酶标仪检测450 nm波长处各孔吸光度(A)值,以A值表示细胞增殖能力。
1.6 Transwell小室实验检测各组侵袭细胞数和迁移细胞数取各组生长状态良好的Ishiwaka细胞用双无培养基悬浮,将含血清的正常培养基加入24孔细胞培养板中,将24孔细胞培养板放入Transwell小室,调整各组细胞个数,使200 µL培养液中含5×104个细胞,将各组细胞放入Transwell小室培养24 h,然后取出小室,甲醇固定30 min,棉签擦去内室细胞,用结晶紫染色30 min,纤维镜下观察迁移细胞数。侵袭实验在实验前用基质胶包被小室,其余步骤同迁移实验。
1.7 Western blotting法检测Ishiwaka细胞中PCNA、E-cadherin、vimentin、snail、Twist、Smad2/3、p-Smad2/3和TGF-β1蛋白表达水平取各组转染24 h细胞加入蛋白裂解液提取细胞总蛋白,BCA法检测细胞蛋白浓度。取100 µg蛋白电泳,经转膜、5%脱脂奶粉封闭,加入一抗:兔抗人PCNA、E-cadherin、vimentin、snail、Twist、Smad2/3、p-Smad2/3和TGF-β1单克隆抗体,稀释比例1:200,过夜孵育。加入二抗(1:5 000)孵育2 h,以β-actin为内参,化学发光法显色,每组设7个复孔,凝胶电泳成像仪采集图像,Quantity One软件分析条带灰度值。目标蛋白表达水平=目标蛋白条带灰度值/β-actin条带灰度值。
1.8 统计学分析采用SPSS 20.0统计软件进行统计学分析。不同细胞中CCAT1 mRNA表达水平、各组细胞增殖能力、迁移细胞数、侵袭细胞数以及细胞中PCNA、E-cadherin、vimentin、snail、Twist、Smad2/3、p-Smad2/3和TGF-β1蛋白表达水平经检验均符合正态分布,以x±s表示,多组间样本均数比较采用单因素方差分析,组间两两比较采用LSD-t检验。以P < 0.05为差异有统计学意义。
2 结果 2.1 不同细胞中CCAT1mRNA表达水平Ishiwaka细胞中CCAT1 mRNA表达水平(1.94±0.17)明显高于T-HESC细胞(1.00±0.09)(t =12.929,P < 0.01)。
2.2 各组Ishiwaka细胞中CCAT1 mRNA表达水平与空白对照组(1.00±0.08)和阴性对照组(0.99±0.10)比较,CCAT1-siRNA组和CCAT1-siRNA + LY364947组Ishiwaka细胞中CCAT1 mRNA表达水平(0.52±0.06和0.49±0.07)明显降低(P < 0.01),空白对照组与阴性对照组及CCAT1-siRNA组与CCAT1-siRNA + LY364947组Ishiwaka细胞中CCAT1表达水平比较差异无统计学意义(P>0.05)。
2.3 各组Ishiwaka细胞增殖能力培养24 h,各组Ishiwaka细胞增殖能力比较差异均无统计学意义(P>0.05);培养48和72 h,与空白对照组和阴性对照组比较,CCAT1-siRNA组和CCAT1-siRNA+ LY364947组Ishiwaka细胞增殖能力明显降低(P < 0.05),与CCAT1-siRNA组比较,CCAT1-siRNA+LY364947组Ishiwaka细胞增殖能力明显降低(P < 0.05),空白对照组与阴性对照组Ishiwaka细胞比较差异无统计学意义(P>0.05)。见表 1。
| (n=7, x±s) | |||||||||||||||||||||||||||||
| Group | A value | ||||||||||||||||||||||||||||
| (t/h) 24 | 48 | 72 | |||||||||||||||||||||||||||
| Blank control | 0.28±0.07 | 0.55±0.08 | 0.86±0.12 | ||||||||||||||||||||||||||
| Negative control | 0.27±0.09 | 0.57±0.09 | 0.85±0.13 | ||||||||||||||||||||||||||
| CCAT1-siRNA | 0.23±0.06 | 0.42±0.06*△ | 0.60±0.10*△ | ||||||||||||||||||||||||||
| CCAT1-siRNA+LY364947 | 0.22±0.05 | 0.31±0.05*△# | 0.46±0.08*△# | ||||||||||||||||||||||||||
| F | 1.271 | 20.06 | 22.595 | ||||||||||||||||||||||||||
| P | 0.31 | < 0.01 | < 0.01 | ||||||||||||||||||||||||||
| *P < 0.05 compared with blank control group; △P < 0.05 compared with negative control group; # P < 0.05 compared with CCAT1-siRNA group. | |||||||||||||||||||||||||||||
与空白对照组和阴性对照组比较,CCAT1-siRNA组和CCAT1-siRNA + LY364947组Ishiwaka细胞中侵袭细胞数和迁移细胞数明显降低(P < 0.05);与CCAT1-siRNA组比较,CCAT1-siRNA + LY364947组Ishiwaka细胞侵袭细胞数和迁移细胞数明显降低(P < 0.05),空白对照组与阴性对照组Ishiwaka细胞中侵袭细胞数和迁移细胞数比较差异无统计学意义(P>0.05)。见表 2、图 1(插页五)和图 2(插页五)。
| (n=7, x±s) | |||||||||||||||||||||||||||||
| Group | Number of invasion cells | Number of migration cells | |||||||||||||||||||||||||||
| Blank control | 50.24±4.16 | 46.15±3.97 | |||||||||||||||||||||||||||
| Negative control | 51.43±3.98 | 47.02±3.73 | |||||||||||||||||||||||||||
| CCAT1-siRNA | 36.48±3.52*△ | 24.16±3.52*△ | |||||||||||||||||||||||||||
| CCAT1-siRNA + LY364947 | 21.43±3.26*△# | 18.26±3.42*△# | |||||||||||||||||||||||||||
| F | 98.494 | 114.873 | |||||||||||||||||||||||||||
| P | < 0.01 | < 0.01 | |||||||||||||||||||||||||||
| *P < 0.05 compared with blank control group; △P < 0.05 compared with negative control group; # P < 0.05 compared with CCAT1-siRNA group. | |||||||||||||||||||||||||||||
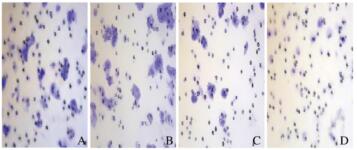
|
| A: Blank control group; B: Negative control group;C:CCATl-siRNA group; D:CCAT1-siRNA十LY36494 group. 图 1 Transwell 小室法测定各组 Ishiwaka 细胞侵袭能力(结晶紫,×200) Fig. 1 Invasive abilities of Ishiwaka cells in var1ous groups determined by Transwell chamber(Crystal violet, ×200) |
|
|
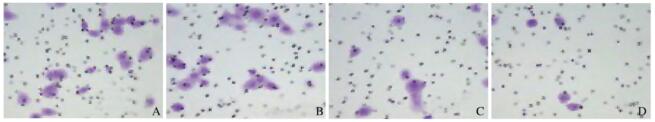
|
| A: Blank control group; B: Negative control group;C:CCATl-siRNA group; D:CCAT1-siRNA十LY36494 group. 图 2 Transwell 小室测定各组 Ishiwaka 细胞迁移能力 (结晶紫,×200) Fig. 2 Migration capacities of Ishiwaka cells in various groups determined by Transwell chamber (Crystal violet, ×200) |
|
|
与空白对照组和阴性对照组比较,CCAT1-siRNA组和CCAT1-siRNA + LY364947组Ishiwaka细胞中PCNA、vimentin、snail和Twist蛋白表达水平均明显降低(P < 0.05),E-cadherin蛋白表达水平明显升高(P < 0.05);与CCAT1-siRNA组比较,CCAT1-siRNA + LY364947组Ishiwaka细胞E-cadherin蛋白表达水平明显升高(P < 0.05),其他蛋白表达水平均明显降低(P < 0.05),但空白对照组与阴性对照组Ishiwaka细胞中上述蛋白表达水平比较差异均无统计学意义(P>0.05)。见表 3和图 3。
| (n=7,x±s) | |||||||||||||||||||||||||||||
| Group | PCNA | E-cadherin | vimentin | snail | Twist | ||||||||||||||||||||||||
| Blank control | 0.83±0.17 | 0.18±0.07 | 0.58±0.11 | 0.85±0.12 | 0.51±0.08 | ||||||||||||||||||||||||
| Negative control | 0.79±0.15 | 0.21±0.08 | 0.56±0.09 | 0.86±0.14 | 0.49±0.07 | ||||||||||||||||||||||||
| CCAT1-siRNA | 0.26±0.05*△ | 0.56±0.11*△ | 0.27±0.03*△ | 0.64±0.08*△ | 0.33±0.05*△ | ||||||||||||||||||||||||
| CCAT1-siRNA+LY364947 | 0.12±0.03*△# | 0.74±0.14*△# | 0.18±0.02*△# | 0.32±0.07*△# | 0.24±0.04*△# | ||||||||||||||||||||||||
| F | 67.275 | 48.55 | 53.515 | 39.533 | 30.591 | ||||||||||||||||||||||||
| P | < 0.01 | < 0.01 | < 0.01 | < 0.01 | < 0.01 | ||||||||||||||||||||||||
| *P < 0.05 compared with blank control group; △P < 0.05 compared with negative control group; # P < 0.05 compared with CCAT1-siRNA group. | |||||||||||||||||||||||||||||

|
| Lane 1:Blank control group; Lane 2:Negative control group; Lane 3:CCAT1-siRNA group; Lane 4:CCAT1-siRNA + LY364947 group. 图 3 Western blotting法检测Ishiwaka细胞中PCNA、E-cadherin、vimentin、snail和Twist蛋白表达电泳图 Fig. 3 Expression levels of PCNA, E-cadherin, vimentin, snail and Twist proteins in Ishiwaka cells in virous groups determined with Western blotting method |
|
|
与空白对照组和阴性对照组比较,CCAT1-siRNA组和CCAT1-siRNA + LY364947组Ishiwaka细胞中p-Smad2/3和TGF-β1蛋白表达水平明显降低(P < 0.05);与CCAT1-siRNA组比较,CCAT1-siRNA + LY364947组Ishiwaka细胞中p-Smad2/3和TGF-β1蛋白表达水平明显降低(P < 0.05);空白对照组与阴性对照组Ishiwaka细胞中p-Smad2/3和TGF-β1蛋白表达水平比较差异无统计学意义(P> 0.05)。见图 4和表 4。

|
| Lane 1:Blank control group; Lane 2:Negative control group; Lane 3:CCAT1-siRNA group; Lane 4:CCAT1-siRNA + LY364947 group. 图 4 Electrophoregram of expressions of Smad2/3, p-Smad2/3 and TGF-β1 proteins in Ishiwaka cells in various groups determined by Western blotting method |
|
|
| (n=7, x±s) | |||||||||||||||||||||||||||||
| Group | Smad2/3 | p-Smad2/3 | TGF-β1 | ||||||||||||||||||||||||||
| Blank control | 0.82±0.13 | 0.71±0.12 | 0.64±0.12 | ||||||||||||||||||||||||||
| Negative control | 0.84±0.12 | 0.73±0.13 | 0.61±0.11 | ||||||||||||||||||||||||||
| CCAT1-siRNA | 0.85±0.14 | 0.37±0.05*△ | 0.25±0.03*△ | ||||||||||||||||||||||||||
| CCAT1-siRNA+LY364947 | 0.87±0.15 | 0.26±0.04*△# | 0.18±0.02*△# | ||||||||||||||||||||||||||
| F | 0.165 | 44.894 | 57.41 | ||||||||||||||||||||||||||
| P | 0.92 | < 0.01 | < 0.01 | ||||||||||||||||||||||||||
| *P < 0.05 compared with blank control group; △P < 0.05 compared with negative control group; # P < 0.05 compared with CCAT1-siRNA group. | |||||||||||||||||||||||||||||
LncRNA是非编码RNA家族成员,长度大于200 nt,不具备蛋白质编码功能,在染色质修饰、表观遗传和转录调控等多方面发挥重要调控作用,可促进蛋白基因表达,也可抑制基因表达[6]。近年来研究[7-8]显示:多种LncRNA与恶性肿瘤的发生发展关系密切,在恶性肿瘤的发生发展中发挥抑癌或促癌作用,与靶蛋白组织的复杂的调控网络在细胞增殖、分化、凋亡和侵袭迁移中发挥重要的调控作用。LncRNA CCAT1位于c-Myc的一个增强子区,在多种恶性肿瘤中异常表达[9],与多种恶性肿瘤的预后关系密切,如CCAT1可靶向miR-219a调节宫颈癌HeLa细胞生长、侵袭和迁移[10];激活CCAT1可通过调节SPRY4和HOXB13在食管鳞状细胞癌中的表达影响食管鳞状细胞癌细胞增殖和迁移[11];LncRNA CCAT1可被c-Myc激活促进胰腺癌细胞增殖和迁移[12]。潘洪丽等[13]对子宫内膜癌细胞中LncRNA CCAT1表达研究发现:子宫内膜癌组织中LncRNA CCAT1呈高水平表达,沉默子宫内膜癌细胞中LncRNA CCAT1水平可抑制子宫内膜癌细胞的侵袭和迁移。本文作者对子宫内膜癌Ishiwaka细胞中LncRNA CCAT1水平研究发现:Ishiwaka细胞中LncRNA CCAT1 mRNA表达水平升高,沉默Ishiwaka细胞LncRNA CCAT1表达可抑制子宫内膜癌细胞的增殖、侵袭和迁移能力。但LncRNA CCAT1在子宫内膜癌中的作用机制尚不清楚。
多种信号通路参与子宫内膜癌的发生发展过程,其中TGF-β/Smad信号通路为子宫内膜癌细胞增殖、侵袭和迁移过程中的重要通路之一。TGF-β/Smad信号通路在肿瘤的发生发展中发挥抑制和促进肿瘤进展的双重作用,在恶性肿瘤的起始阶段,TGF-β/Smad信号通路可抑制恶性肿瘤细胞增殖、促进肿瘤细胞凋亡,从而抑制恶性肿瘤的发展;在恶性肿瘤的晚期,TGF-β/Smad信号通路可促进肿瘤细胞增殖、抑制肿瘤细胞凋亡,从而促进恶性肿瘤的进展和转移过程[14]。TGF-β/Smad信号通路在恶性肿瘤的发生发展中,通过调节上皮间质转化(EMT)导致恶性肿瘤细胞免疫逃避、细胞转移和诱导血管生成等,促进恶性肿瘤的进展[15-16]。
LncRNA CCAT1可通过多种信号通路参与恶性肿瘤的发生发展,如LncRNA CCAT1通过激活丝裂原活化蛋白激酶(mitogen-activated protein kinases,MAPK)信号通路影响胃癌细胞的增殖、凋亡、侵袭和迁移过程[17];LncRNA CCAT1在恶性肿瘤中的作用与CCAT1可影响恶性肿瘤细胞EMT有关[18];LncRNA CCAT1通过miR-490-3p上调TGF-β受体1从而促进TGF-β1诱导的卵巢癌细胞EMT[19]。LncRNA CCAT1可通过TGF-β诱导细胞EMT,EMT在细胞的侵袭迁移中发挥重要作用[20],因此推测LncRNA CCAT1在子宫内膜癌细胞增殖、侵袭和迁移中的作用可能与TGF-β/Smad信号通路及EMT有关。本研究结果显示:沉默CCAT1及加入TGF-β/Smad信号抑制剂均可降低细胞中PCNA、E-cadherin、vimentin、snail、Twist、p-Smad2/3和TGF-β1蛋白表达水平,细胞中E-cadherin蛋白表达水平升高。PCNA在细胞核中合成,检测细胞中PCNA蛋白水平可用于评价细胞的增殖状态[21]。E-cadherin为重要的维持上皮细胞表型的细胞间黏附分子,E-cadherin表达缺失或者表达降低是EMT的重要标志[22]。Vimentin为间质细胞的标志物,其水平升高也是EMT的标志物[23]。snail和Twist为TGF-β/Smad信号通路的上游转录因子,二者水平升高可促进TGF-β/Smad信号通路激活[24-25]。p-Smad2/3和TGF-β1蛋白表达水平可反映TGF-β/Smad信号通路状况,p-Smad2/3和TGF-β1蛋白表达水平升高表明TGF-β/Smad信号通路激活;反之,p-Smad2/3和TGF-β1蛋白水平降低表明TGF-β/Smad信号通路受到抑制。因此,本研究中沉默CCAT1及加入TGF-β/Smad信号抑制剂均可抑制子宫内膜癌细胞增殖、侵袭和迁移,降低细胞中PCNA、E-cadherin、vimentin、snail、Twist、p-Smad2/3和TGF-β1蛋白表达水平,细胞中E-cadherin蛋白表达水平升高,表明沉默CCAT1抑制子宫内膜癌细胞增殖、侵袭和迁移的机制可能与抑制TGF-β/Smad信号通路激活、从而抑制子宫内膜癌细胞EMT有关。
综上所述,沉默LncRNA CCAT1对子宫内膜癌细胞的增殖、侵袭和迁移具有抑制作用,其机制可能与沉默LncRNA CCAT1可抑制子宫内膜癌细胞TGF-β/Smad信号通路活化从而抑制EMT有关。
| [1] |
LI Z, YU Z, MENG X, et al. Long noncoding RNA GAS5 impairs the proliferation and invasion of endometrial carcinoma induced by high glucose via targeting miR-222-3p/p27[J]. Am J Transl Res, 2019, 11(4): 2413-2421. |
| [2] |
ZHANG Z, XIE H, LIANG D, et al. Long non-coding RNA CCAT1 as a diagnostic and prognostic molecular marker in various cancers:a meta-analysis[J]. Oncotarget, 2018, 9(34): 23695-23703. DOI:10.18632/oncotarget.24923 |
| [3] |
YU J, JIANG L, GAO Y, et al. LncRNA CCAT1 negatively regulates miR-181a-5p to promote endometrial carcinoma cell proliferation and migration[J]. Exp Ther Med, 2019, 17(5): 4259-4266. |
| [4] |
SAHOO S S, QUAH M Y, NIELSEN S, et al. Inhibition of extracellular matrix mediated TGF-β signalling suppresses endometrial cancer metastasis[J]. Oncotarget, 2017, 8(42): 71400-71417. DOI:10.18632/oncotarget.18069 |
| [5] |
毛晓丹, 谭宇思, 李玉红, 等. 转化生长因子β1作用下绒毛膜癌JEG-3细胞c-myc mRNA表达[J]. 临床和实验医学杂志, 2014, 13(3): 168-171. |
| [6] |
LIU Y, WANG L L, CHEN S, et al. LncRNA ABHD11-AS1 promotes the development of endometrial carcinoma by targeting cyclin D1[J]. J Cell Mol Med, 2018, 22(8): 3955-3964. DOI:10.1111/jcmm.13675 |
| [7] |
SUN M Y, ZHU J Y, ZHANG C Y, et al. Autophagy regulated by lncRNA HOTAIR contributes to the cisplatin-induced resistance in endometrial cancer cells[J]. Biotechnol Lett, 2017, 39(10): 1477-1484. DOI:10.1007/s10529-017-2392-4 |
| [8] |
XIE P, CAO H, LI Y, et al. Knockdown of lncRNA CCAT2 inhibits endometrial cancer cells growth and metastasis via sponging miR-216b[J]. Cancer Biomark, 2017, 21(1): 123-133. DOI:10.3233/CBM-170388 |
| [9] |
LU L, QI H, LUO F, et al. Feedback circuitry via let-7c between lncRNA CCAT1 and c-Myc is involved in cigarette smoke extract-induced malignant transformation of HBE cells[J]. Oncotarget, 2017, 8(12): 19285-19297. DOI:10.18632/oncotarget.15195 |
| [10] |
王圣坦, 朱根海, 洪澜, 等. 长链非编码CCAT1靶向miR-219a对宫颈癌Hela细胞生长、侵袭和迁移的调节作用[J]. 安徽医科大学学报, 2018, 53(9): 1348-1353. |
| [11] |
ZHANG E, HAN L, YIN D, et al. H3K27 acetylation activated-long non-coding RNA CCAT1 affects cell proliferation and migration by regulating SPRY4 and HOXB13 expression in esophageal squamous cell carcinoma[J]. Nucleic Acids Res, 2017, 45(6): 3086-3101. DOI:10.1093/nar/gkw1247 |
| [12] |
YU Q, ZHOU X, XIA Q, et al. Long non-coding RNA CCAT1 that can be activated by c-Myc promotes pancreatic cancer cell proliferation and migration[J]. Am J Transl Res, 2016, 8(12): 5444-5454. |
| [13] |
潘洪丽, 何宝玉, 韦东, 等. 长链非编码RNA CCAT1对子宫内膜癌细胞迁移侵袭及上皮-间质转化的影响[J]. 实用医学杂志, 2017, 33(4): 598-602. |
| [14] |
GARCIA-RENDUELES A R, RODRIGUES J S, GARCIA-RENDUELES M E, et al. Rewiring of the apoptotic TGF-β-SMAD/NFκB pathway through an oncogenic function of p27 in human papillary thyroid cancer[J]. Oncogene, 2017, 36(5): 652-666. DOI:10.1038/onc.2016.233 |
| [15] |
COLAK S, TEN DIJKE P. Targeting TGF-β signaling in cancer[J]. Trends Cancer, 2017, 3(1): 56-71. |
| [16] |
Syed V. TGF-β Signaling in Cancer[J]. J Cell Biochem, 2016, 117(6): 1279-1287. DOI:10.1002/jcb.25496 |
| [17] |
余南荣, 曾祥, 徐厚巍, 等. LncRNA CCAT通过调控MYC蛋白的表达激活MAPK信号通路影响胃癌细胞的生物学行为[J]. 中国免疫学杂志, 2018, 34(9): 1298-1303. |
| [18] |
LIN H P, CHENG W, YAN H H, et al. Overexpression of the long noncoding RNA CCAT1 promotes metastasis via epithelial to mesenchymal transition in lung adenocarcinoma[J]. Oncol Lett, 2018, 1809-1814. |
| [19] |
MU Y, LI N, CUI Y L. The lncRNA CCAT1 upregulates TGFβR1 via sponging miR-490-3p to promote TGFβ1-induced EMT of ovarian cancer cells[J]. Cancer Cell Int, 2018, 18: 145. DOI:10.1186/s12935-018-0604-1 |
| [20] |
WANG X K, LAI Q H, HE J, et al. LncRNA SNHG6 promotes proliferation, invasion and migration in colorectal cancer cells by activating TGF-β/Smad signaling pathway via targeting UPF1 and inducing EMT via regulation of ZEB1[J]. Int J Med Sci, 2019, 16(1): 51-59. DOI:10.7150/ijms.27359 |
| [21] |
KOWALSKA E, BARTNICKI F, FUJISAWA R, et al. Inhibition of DNA replication by an anti-PCNA aptamer/PCNA complex[J]. Nucleic Acids Res, 2018, 46(1): 25-41. DOI:10.1093/nar/gkx1184 |
| [22] |
MENDONSA A M, NA T Y, GUMBINER B M. E-cadherin in contact inhibition and cancer[J]. Oncogene, 2018, 37(35): 4769-4780. DOI:10.1038/s41388-018-0304-2 |
| [23] |
ZHAO L, ZHANG P, SU X J, et al. The ubiquitin ligase TRIM56 inhibits ovarian cancer progression by targeting vimentin[J]. J Cell Physiol, 2018, 233(3): 2420-2425. DOI:10.1002/jcp.26114 |
| [24] |
WANG G H, MA W, LI Y, et al. Prognostic value of Twist, Snail and E-cadherin expression in pathological N0 non-small-cell lung cancer:a retrospective cohort study[J]. Eur J Cardiothorac Surg, 2018, 54(2): 237-245. DOI:10.1093/ejcts/ezy022 |
| [25] |
MA J, WANG L, LI J, et al. Swainsonine inhibits invasion and the EMT process in esophageal carcinoma cells by targeting Twist1[J]. Oncol Res, 2018, 26(8): 1207-1213. DOI:10.3727/096504017X15046134836575 |
 2020, Vol. 46
2020, Vol. 46


