扩展功能
文章信息
- 王治, 洪莉, 李素廷, 曾婉玲
- WANG Zhi, HONG Li, LI Suting, ZENG Wanling
- 基于GEO数据库生物信息学方法分析子宫内膜癌相关基因和候选通路
- Analysis on endometrial cancer-related genes and candidate pathways based on GEO database bioinformatics methods
- 吉林大学学报(医学版), 2020, 46(04): 804-809
- Journal of Jilin University (Medicine Edition), 2020, 46(04): 804-809
- 10.13481/j.1671-587x.20200422
-
文章历史
- 收稿日期: 2019-10-30
子宫内膜癌(endometrial carcinoma, EC)是妇科常见的生殖系统恶性肿瘤之一[1],占女性生殖系统恶性肿瘤20%~30%,好发于围绝经期及绝经后女性。研究[2]显示:EC每年发病率约为0.2/10000~1/10000,且近年来其发病率逐渐升高,并呈现年轻化趋势。EC发病原因和机制尚不明确,其早期诊断及治疗方面尽管已有较大进展,但仍有相当一部分的病例发展至晚期才被确诊。因此,研究EC发病原因和疾病发展相关机制及干预靶标,有助于进一步改善EC的诊断、治疗和预后。随着基因芯片技术的发展以及生物信息学相关工具的完善,目前基因表达汇编(Gene Expression Omnibus,GEO)数据库[3]提供了大量的基因表达谱相关数据,对其进行数据分析可以快速发现肿瘤组织中的差异表达基因,并可通过进一步分析寻找到影响疾病发生发展的分子靶标。在肺癌[4]、胃癌[5]、乳腺癌[6]、神经母细胞瘤[7]、肠癌[8]和卵巢癌[9]等多种肿瘤中,对GEO数据库进行分析已经在多种肿瘤机制研究中得到了广泛应用,该分析方法已经被证实切实有效。通过生物信息学技术筛选EC患者组织的差异表达基因(differentially expressed genes, DEGs)来发现潜在标志物,对EC的诊断和治疗方案的确定具有重要意义。本研究通过对GEO数据库中EC组织的基因表达数据集GSE3678和GSE17025进行联合分析,筛选出具有高可信度的DEGs和重要的相关信号通路,构建了DEGs的蛋白质-蛋白质互作网络(protein-protein interaction network,PPI),并筛选出了关键基因作为分子靶标,以期通过DEGs和PPI的分析结果为EC的研究提供新思路。
1 资料与方法 1.1 基因芯片数据集信息通过GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载高通量测序数据集GSE63678和GSE17025, 2组基因芯片数据类型均为Expression profiling by array,种属均为Homo sapiens;GSE63678数据集注释平台为GPL571,包括5例正常组织样本(GSM1555092~GSM1555096)和7例EC样本(GSM1555085~GSM1555091);GSE17025数据集注释平台为GPL570,包括12例正常组织样本(GSM425927~GSM425938)和79例EC样本(GSM425837~GSM425915)。
1.2 DEGs的提取和分析GEO2R(http://www.ncbi.nlm.nih.gov/geo/geo2r/)可用于分析GEO数据集中不同数据组的差异表达水平。在基因表达数据集中,以正常组织样本为对照组,以EC组织为实验组,使用GEO2R在线分析工具分别分析并筛选出GSE63678和GSE17025的DEGs,筛选标准为P<0.05并且|log(FC)|>2,使用R软件ggplot2和pheatmap程序包分析DEGs并进行可视化,分别生成火山图与热图。最后用FunRich软件生成venn图筛选出在GSE63678和GSE17025中同时上调或下调的DEGs用于进一步分析。
1.3 基因本体论(gene ontology,GO)富集分析和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes, KEGG)通路分析对大规模基因进行GO分析已经成为生物信息学分析的常用手段,可从细胞组分、生物过程和分子功能等多方面对DEGs进行全面注释。KEGG数据库已被广泛用于通路富集分析,可得到EC发展过程中的关键通路。使用R语言clusterProfiler包对DEGs分别进行GO富集分析和KEGG信号通路分析,并对结果进行可视化。
1.4 PPI网络构建和分析String在线数据库(http://string.embl.de/)可用于检索分子间相互作用以及预测蛋白质互作关系。使用String数据库对上述所得的206个DEGs进行分析,构建PPI网络并使用Cytoscape软件进行可视化,使用Cytohubba插件对PPI网络上所有DEGs进行评分,以其中最高相关度的前10个DEGs作为EC发病相关的关键基因,即hub基因。最后使用Cytoscape软件MCODE插件对DEGs进行聚类功能模块构建,关键模块筛选标准为MCODE评分>4,并对关键模块所包含基因进行GO富集分析和KEGG通路富集分析。模块筛选标准:Degree Cutoff = 2,K-core = 2,Maximum Depth= 100,Node Score Cutoff = 0.2。
2 结果 2.1 DEGs的提取对GSE63678数据集进行处理分析后,共鉴定出459个DEGs,其中包括242个上调基因(52.7%)和217个下调基因(47.3%)(图 1A,见插页七);GSE17025数据集分析,与对照组比较,EC组共有1508个DEGs,有797个基因发生了上调(52.9%),711个基因发生了下调(47.1%)(图 1B,见插页七)。2组芯片数据集的差异基因表达热图分别见图 1C和1D(插页七)。将筛选出来的差异表达基因进行Venny分析,结果显示:在数据集GSE3678和GSE17025中同时发生上调的基因有100个,同时发生下调的基因有106个(图 1E,见插页七)。
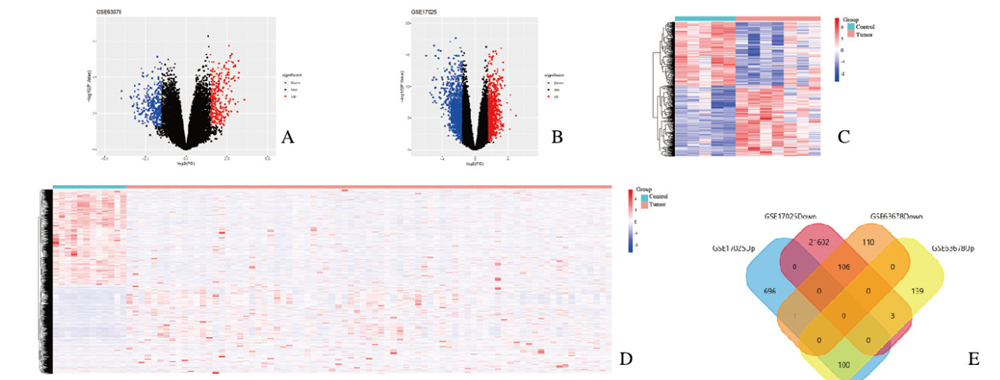
|
| A:Volcano map of GSE63678; B:Volcano map of GSE17025; C:Heat map of GSE63678; D: Heat map of GSE17025; E: Venn analysis of GSE63678 and GSE17025. 图 1 GSE63678和GSE17025数据集中DEGs的筛选 Fig. 1 Screening of DEGs in GSE63678 and GSE17025 data sets |
|
|
对共同上调或下调的DEGs进行GO富集分析,GO注释主要分为细胞组成、生物过程和分子功能3个部分。DEGs主要富集在有丝分裂染色体分离、核分裂和细胞器分裂等生物学过程。KEGG信号通路分析结果显示:差异基因主要富集于细胞周期、miRNA、p53信号通路和2型糖尿病等信号通路过程。见图 2(插页七)。
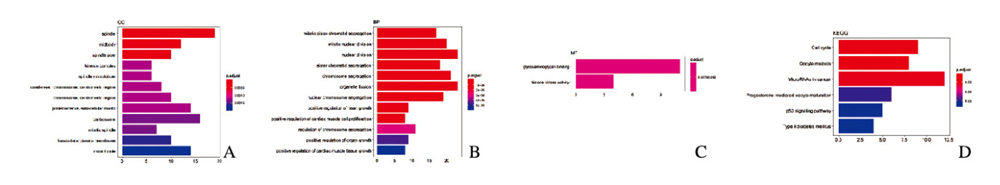
|
| A: Analysis of cell composition; B: Analysis of biological process; C: Analysis of molecular function; D: Analysis of KEGG signal pathway. 图 2 DEGs的GO富集分析和KEGG信号通路分析 Fig. 2 GO enrichment analysis and KEGG signal pathway analysis of DEGs |
|
|
基于String数据库在线分析DEGs的PPI关系,纳入PPI标准为综合得分>0.4,共得到165个PPI节点和1131个蛋白互作关系,在所有DEGs中占比80.1%。使用Cytoscape软件对PPI网络进行可视化及分析(图 3A)。使用CentiScape插件对PPI节点进行评估,选择具有最高度连通性的10个节点DEG作为EC的关键基因(图 3B),可能在EC发生发展中起到关键作用的基因有细胞分裂周期基因20(cell division cycle gene 20, CDC20)、极光激酶A(aurora kinase A, AURKA)、细胞周期蛋白B1(Cyclin B1, CCNB1)、泛素E3连接酶(denticleless E3 ubiquitin protein ligase, DTL)、中心体相关蛋白55(centrosomal protein 55, CEP55)、细胞周期蛋白依赖性激酶1(Cyclin-dependent kinase 1, CDK1)、驱动蛋白家族成员11(kinesin family member 11, KIF11)、母系胚胎亮氨酸拉链激酶(maternal embryonic leucine zipper kinase, MELK)、细胞周期蛋白B2(Cyclin B2, CCNB2)和苯并咪唑出芽抑制解除同源物蛋白1 (budding uninhibited by benzimidazoles 1, BUB1)。见表 1。

|
| A:PPI network, including 165 nodes and 1131 interaction of proteins; B:Interaction of 10 hub genes analyzed by CentiScape plugin. 图 3 DEGs的PPI分析 Fig. 3 PPI analysis of DEGs |
|
|
| Gene | Betweenness unDir | Bridging unDir | Degree unDir | EigenVector unDir | MCODE_Score | Radiality unDir | Stress unDir |
| CDC20 | 104.22 | 1.93 | 43 | 0.16 | 37.95 | 6.80 | 730 |
| AURKA | 1 300.43 | 11.53 | 45 | 0.16 | 37.95 | 6.91 | 3 602 |
| CCNB1 | 148.04 | 3.08 | 43 | 0.16 | 37.95 | 6.82 | 1 302 |
| DTL | 4.35 | 0.10 | 42 | 0.16 | 37.95 | 6.79 | 160 |
| CEP55 | 4.35 | 0.10 | 42 | 0.16 | 37.95 | 6.79 | 160 |
| CDK1 | 826.79 | 14.26 | 44 | 0.16 | 37.95 | 6.86 | 2 754 |
| KIF11 | 4.35 | 0.10 | 42 | 0.16 | 37.95 | 6.79 | 160 |
| MELK | 4.35 | 0.10 | 42 | 0.16 | 37.95 | 6.79 | 160 |
| CCNB2 | 148.04 | 3.08 | 43 | 0.16 | 37.95 | 6.82 | 1 302 |
| BUB1 | 4.35 | 0.10 | 42 | 0.16 | 37.95 | 6.79 | 160 |
使用MCODE插件对PPI进行关键模块的筛选,共筛选出3个重要的子模块,筛选标准为K-core=2,Node Score Cutoff=0.2,Degree Cutoff=2,Maximum Depth= 100。A模块MCODE得分为42.048,由43个节点和883个相互作用关系构成(图 4A);B模块MCODE得分为4.25,由9个节点和17个相互作用关系构成(图 4B);C模块MCODE得分为4.0,由4个节点和6个相互作用关系构成(图 4C)。
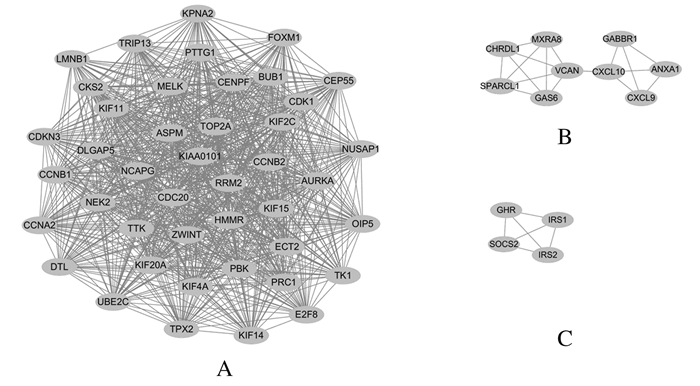
|
| A:Interaction of submodule A; B:Interaction of submodule B; C:Interaction of submodule C. 图 4 MCODE分析DEGs关键子模块 Fig. 4 MCODE analysis on key submodule of DEGs |
|
|
对PPI中3个重要子模块所包含的56个DEGs进行GO富集分析和KEGG信号通路分析。GO富集分析结果显示:DEGs主要在细胞核分裂和细胞周期调节等生物过程中发挥重要作用。KEGG信号通路分析结果显示:DEGs主要与细胞周期、卵母细胞分裂、p53、细胞衰老、2型糖尿病和叉头转录因子O(FoxO)等信号通路有密切关联。见图 5(插页七)。

|
| A: Analysis of cell composition; B: Analysis of biological process; C: Analysis of molecular function; D: Analysis of KEGG signal pathway. 图 5 重要子模块中DEGs的GO富集分析和KEGG信号通路分析 Fig. 5 GO enrichment analysis and KEGG signal pathway analysis of DEGs in important submodules |
|
|
EC是临床常见的妇科肿瘤之一,在全球妇科肿瘤中发病率位列第4位,在过去数十年间其发病率和死亡率逐年升高。EC典型的临床表现为阴道出血,肥胖、高血压和糖尿病是EC的“三联征”。目前EC治疗以手术为主,同时给予化疗或内分泌治疗,但其治疗效果却不甚理想,存在手术不彻底、化疗药物毒性大和易产生耐药性等问题[10-11]。发现较早的Ⅰ期EC患者综合治疗后5年生存率可达85%以上,中晚期患者生存率则明显降低,Ⅱ期EC患者5年生存率约为70%,Ⅲ期患者则仅为50%[12]。早期发现和治疗是提高EC患者生存率的最佳途径。随着对EC发生发展机制的不断探索,已有多项研究[13-15]证实:多基因组学改变和信号转导通路的异常在EC发病中起重要的调控作用,多基因多通路的复杂连锁反应过程也是肿瘤发生机制研究的难点和重点。
随着生物信息学技术及二代测序技术的飞速发展,通过基因芯片数据研究EC发病机制成为了可能,也是研究EC的重要方向。本研究通过生物信息学技术对GEO数据库中2个EC相关数据集GSE63678和GSE17025进行联合分析,共鉴定出了共同发生上调的差异基因100个,同时发生下调的差异基因106个;GO富集分析表明:差异基因主要富集于有丝分裂染色体分离、核分裂、细胞器分裂等生物学过程;KEGG信号通路分析表明:差异基因主要富集于细胞周期、miRNA、p53信号通路和2型糖尿病等信号通路过程。对差异基因构建的PPI网络进行进一步分析后,CDC20、AURKA、CCNB1、DTL、CEP55、CDK1、KIF11、MELK、CCNB2和BUB1被筛选为关键基因,其在EC发生发展过程中可能发挥重要作用。本文作者在PPI网络中提取出了3个重要的子模块,其所包含基因依然富集于细胞核分裂和细胞周期调节等生物过程,KEGG通路富集分析则表示这些基因主要与细胞周期、卵母细胞分裂、p53信号通路、细胞衰老、Ⅱ型糖尿病和FoxO信号通路等有关联。
CDC20在许多种肿瘤中具有高表达,对肿瘤的发生起促进作用,抑制CDC20活性可加速细胞凋亡、调节细胞分裂周期[16-17],因此CDC20可作为抗肿瘤治疗的有效靶点。AURKA是一个周期性蛋白,通过参与中心体的复制分离和成熟在细胞周期中起着重要作用,其高表达可使多种抑癌蛋白失活同时激活多种致癌基因的表达[18]。CCNB1和CCNB2均属于细胞周期蛋白(Cyclin)家族成员,研究[3, 19-23]表明:CCNB1基因在乳腺癌、卵巢癌和直肠癌等多种肿瘤组织中同样高表达,在结直肠癌和肺癌组织中CCNB2高表达能加快细胞周期进程、促进细胞异常增殖。未来有望通过靶向CCNB1和CCNB2基因的治疗方法来抑制肿瘤细胞增殖。DTL介导细胞泛素化修饰及降解,与多种肿瘤的发生发展有密切关联,研究[24]表明:DTL通过介导雌激素促进EC细胞侵袭迁移和上皮-间质转换,其机制可能为EC诊断及治疗提供新思路。CEP55是中心体相关蛋白家族成员,在中心体依赖相关的细胞功能如细胞周期进展、胞质分裂中心体复制等过程中发挥重要作用[25]。CDK1可与细胞周期蛋白B(CyclinB,CCNB)组成复合物即有丝分裂促进因子(mitosis-promoting factor,MPF),促使细胞周期从G2期进入M期,是哺乳动物细胞增殖所必需的基因,CDK1活性失调会导致染色体破裂和细胞死亡[26]。KIF11是一种经典的分子马达,在染色体的分离和双极纺锤体的组装过程中发挥重要作用,与细胞增殖有密切关联[27]。MELK是一种周期依赖性激酶,研究[28-29]显示:MELK在多种肿瘤细胞中高表达,并与肿瘤侵袭和转移有密切关联。BUB1是纺锤体的重要组成部分,其主要功能是监控并保证有丝分裂过程的进行[30]。
高度有序的细胞周期活动是细胞维持正常代谢与增殖过程的保证,当细胞周期[31]调控发生异常,例如周期相关蛋白、复合蛋白或细胞周期监测点等细胞周期调控网络发生异常时,会导致细胞增殖失控,含有异常受损DNA的细胞无法凋亡,最终导致细胞无限增殖和肿瘤形成[32]。本研究基于微阵列数据集,通过生物信息学技术分析了EC发生发展相关的易感基因和调控网络,富集分析和PPI网络分析表明:细胞周期相关的基因和通路在EC发生发展过程中起重要作用,PPI网络中筛选出的关键基因绝大多数与细胞周期调控有关联,由此可见,细胞周期失调是EC发病的主要机制。本研究结果为EC相关的发病机制、诊断、治疗和预后判断等研究提供了重要依据。结合进一步实验验证,以上参与EC发病的关键分子有望成为具有临床价值的EC生物标志物和治疗靶标。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
STEWART C J, DOHERTY D A, HAVLAT M, et al. Transtubal spread of endometrial carcinoma:correlation of intra-luminal tumour cells with tumour grade, peritoneal fluid cytology, and extra-uterine metastasis[J]. Pathology, 2013, 45(4): 382-387. |
| [3] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO:archive for functional genomics data sets:update[J]. Nucleic Acids Res, 2013, 41(Database issue): D991-D995. |
| [4] |
LI Y, GU J, XU F K, et al. Transcriptomic and functional network features of lung squamous cell carcinoma through integrative analysis of GEO and TCGA data[J]. Sci Rep, 2018, 8(1): 15834. |
| [5] |
ZHANG X Q, ZHANG W F, JIANG Y Y, et al. Identification of functional lncRNAs in gastric cancer by integrative analysis of GEO and TCGA data[J]. J Cell Biochem, 2019, 120(10): 17898-17911. DOI:10.1002/jcb.29058 |
| [6] |
LIU P F, JIANG W H, ZHOU S Y, et al. Combined analysis of ChIP sequencing and gene expression dataset in breast cancer[J]. Pathol Oncol Res, 2017, 23(2): 361-368. DOI:10.1007/s12253-016-0116-z |
| [7] |
MELAIU O, MINA M, CHIERICI M, et al. PD-L1 is a therapeutic target of the bromodomain inhibitor JQ1 and, combined with HLA class I, a promising prognostic biomarker in neuroblastoma[J]. Clin Cancer Res, 2017, 23(15): 4462-4472. DOI:10.1158/1078-0432.CCR-16-2601 |
| [8] |
RUAN G T, GONG Y Z, LIAO X W, et al. Diagnostic and prognostic values of CXC motif chemokine ligand 3 in patients with colon cancer[J]. Oncol Rep, 2019, 42(5): 1996-2008. |
| [9] |
MEN C D, LIU Q N, REN Q. A prognostic 11 genes expression model for ovarian cancer[J]. J Cell Biochem, 2018, 119(2): 1971-1978. DOI:10.1002/jcb.26358 |
| [10] |
PAUTIER P, POMMERET F. Systemic therapy for advanced endometrial cancer[J]. Bull Cancer, 2017, 104(12): 1046-1053. DOI:10.1016/j.bulcan.2017.10.007 |
| [11] |
ETHIER J L, DESAUTELS D N, AMIR E, et al. Is hormonal therapy effective in advanced endometrial cancer? A systematic review and meta-analysis[J]. Gynecol Oncol, 2017, 147(1): 158-166. |
| [12] |
CONWAY J L, LUKOVIC J, FERGUSON S E, et al. Clinical outcomes of surgically unresectable endometrial cancers[J]. Am J Clin Oncol, 2019, 42(10): 777-782. DOI:10.1097/COC.0000000000000600 |
| [13] |
LIU J H, NIE S P, LIANG J Y, et al. Competing endogenous RNA network of endometrial carcinoma:a comprehensive analysis[J]. J Cell Biochem, 2019, 120(9): 15648-15660. DOI:10.1002/jcb.28831 |
| [14] |
RAFFONE A, TRAVAGLINO A, MASCOLO M, et al. TCGA molecular groups of endometrial cancer:Pooled data about prognosis[J]. Gynecol Oncol, 2019, 155(2): 374-383. |
| [15] |
WANG Y W, HUANG T, SUN X, et al. Identification of a potential prognostic lncRNA-miRNA-mRNA signature in endometrial cancer based on the competing endogenous RNA network[J]. J Cell Biochem, 2019, 120(11): 18845-18853. DOI:10.1002/jcb.29200 |
| [16] |
HOLLAND A J, CLEVELAND D W. Boveri revisited:chromosomal instability, aneuploidy and tumorigenesis[J]. Nat Rev Mol Cell Biol, 2009, 10(7): 478-487. DOI:10.1038/nrm2718 |
| [17] |
IZAWA D, PINES J. The mitotic checkpoint complex binds a second CDC20 to inhibit active APC/C[J]. Nature, 2015, 517(7536): 631-634. DOI:10.1038/nature13911 |
| [18] |
DAR A A, GOFF L W, MAJID S, et al. Aurora kinase inhibitors——rising stars in cancer therapeutics?[J]. Mol Cancer Ther, 2010, 9(2): 268-278. |
| [19] |
李莉, 熊国平, 颜琳, 等. CCNB1在卵巢癌中的表达及意义[J]. 现代妇产科进展, 2016, 25(11): 818-820, 824. |
| [20] |
方益锋. Chk1介导的CCNB1高表达在结直肠癌中的作用及机制研究[D].杭州: 浙江大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10335-1016283002.htm
|
| [21] |
SCHNITTGER A, DE VEYLDER L. The dual face of cyclin B1[J]. Trends Plant Sci, 2018, 23(6): 475-478. |
| [22] |
王杰, 陈大兴, 郭宁, 等. CCNB2过表达对非小细胞肺癌不良预后的预测效果研究[J]. 中国现代医学杂志, 2017, 27(17): 45-49. DOI:10.3969/j.issn.1005-8982.2017.17.009 |
| [23] |
辛萱, 陈军, 严钢莉, 等. CCNB2在结直肠癌中的表达及临床意义[J]. 现代生物医学进展, 2014, 14(26): 5127-5131. |
| [24] |
徐华丽, 杨孝明, 杜娜, 等. UBE3C在雌激素促进子宫内膜癌细胞迁移及侵袭中的作用机制研究[J]. 现代妇产科进展, 2018, 27(5): 334-338, 342. |
| [25] |
ZHAO W M, SEKI A, FANG G W. Cep55, a microtubule-bundling protein, associates with centralspindlin to control the midbody integrity and cell abscission during cytokinesis[J]. Mol Biol Cell, 2006, 17(9): 3881-3896. DOI:10.1091/mbc.e06-01-0015 |
| [26] |
ASGHAR U, WITKIEWICZ A K, TURNER N C, et al. The history and future of targeting cyclin-dependent kinases in cancer therapy[J]. Nat Rev Drug Discov, 2015, 14(2): 130-146. |
| [27] |
VALE R D, MILLIGAN R A. The way things move:looking under the hood of molecular motor proteins[J]. Science, 2000, 288(5463): 88-95. DOI:10.1126/science.288.5463.88 |
| [28] |
NAKANO I, PAUCAR A A, BAJPAI R, et al. Maternal embryonic leucine zipper kinase (MELK) regulates multipotent neural progenitor proliferation[J]. J Cell Biol, 2005, 170(3): 413-427. DOI:10.1083/jcb.200412115 |
| [29] |
BEULLENS M, VANCAUWENBERGH S, MORRICE N, et al. Substrate specificity and activity regulation of protein kinase MELK[J]. J Biol Chem, 2005, 280(48): 40003-40011. DOI:10.1074/jbc.M507274200 |
| [30] |
GRABSCH H, TAKENO S, PARSONS W J, et al. Overexpression of the mitotic checkpoint genes BUB1, BUBR1, and BUB3 in gastric cancer:association with tumour cell proliferation[J]. J Pathol, 2003, 200(1): 16-22. |
| [31] |
ZHANG L, WAN Y C, JIANG Y, et al. Upregulation HOXA10 homeobox gene in endometrial cancer:role in cell cycle regulation[J]. Med Oncol, 2014, 31(7): 52. DOI:10.1007/s12032-014-0052-2 |
| [32] |
DE FELICE F, MARCHETTI C, TOMBOLINI V, et al. Immune check-point in endometrial cancer[J]. Int J Clin Oncol, 2019, 24(8): 910-916. |
 2020, Vol. 46
2020, Vol. 46


