扩展功能
文章信息
- 商浩, 李春森, 李锋, 刘春霞
- SHANG Hao, LI Chunsen, LI Feng, LIU Chunxia
- microRNA-9-5p靶向MEF2C对腺泡状横纹肌肉瘤细胞生物学行为的影响
- Effect of microRNA-9-5p targeting MEF2C on biological behaviors of alveolar rhabdomyosarcoma cells
- 吉林大学学报(医学版), 2020, 46(03): 482-491
- Journal of Jilin University (Medicine Edition), 2020, 46(03): 482-491
- 10.13481/j.1671-587x.20200309
-
文章历史
- 收稿日期: 2019-07-17
2. 浙江省立同德医院司法鉴定所, 浙江 杭州 310012;
3. 首都医科大学附属北京朝阳医院病理科, 北京 100020
2. Judical Appraisal Institute, Tongde Hospital, Zhejiang Province, Hangzhou 310012, China;
3. Department of Pathology, Beijing Chaoyang Hospital, Capital Medical University, Beijing 100020, China
横纹肌肉瘤是儿童和青少年中最常见的软组织肉瘤,好发于头部和颈部[1]。根据临床特点和细胞形态等,世界卫生组织(WHO)将横纹肌肉瘤分为腺泡状横纹肌肉瘤(alveolar rhabdomyosarcoma, ARMS)、胚胎性横纹肌肉瘤、多形性横纹肌肉瘤和梭形细胞/硬化性横纹肌肉瘤[2]。ARMS常存在t(2;13)(q35;q14)或t(1;13)(q36,q14)染色体易位,形成融合基因PAX3-FOXO1或PAX7-FOXO1,其分化程度低,且预后差。尽管放射疗法和化疗方案等在ARMS中广泛应用,但ARMS患儿5年生存率仍然较低。当ARMS出现转移时,患儿5年生存率常低于20%[3-4]。因此有必要揭示ARMS的分子机制,以帮助鉴定新的治疗靶点和分子诊断标志物。
小分子核糖核酸(microRNAs,miRNAs)是长度约为22个核苷酸的非编码RNA,通过与靶基因的3′-UTR结合诱导mRNA降解或抑制mRNA翻译控制蛋白质编码基因的表达[5-6]。miRNA已被证实是多种肿瘤的关键调节因子,作为肿瘤癌基因或抑癌基因参与细胞增殖、侵袭、迁移、凋亡和分化等。研究[7-9]表明:microRNA-9-5p(miR-9-5p)在胃癌、前列腺癌和非小细胞肺癌等多种人类恶性肿瘤组织中呈异常表达,参与肿瘤发生发展。然而miR-9-5p在ARMS中的表达及其作用机制尚未见报道。本研究探讨miR-9-5p在ARMS组织中的表达及其作用机制,旨在为研究ARMS发病机制及治疗提供依据。
1 材料与方法 1.1 细胞、质粒、主要试剂和仪器ARMS RH30细胞购自上海复翔生物技术有限公司,ARMS PLA802细胞购自上海斯信生物科技有限公司,正常骨骼肌HSKMC细胞购自北纳生物技术有限公司,人胚肾293T细胞由石河子大学医学院新疆地方病和民族疾病重点实验室提供。肌细胞增强因子2C(myocyte enhancer 2C,MEF2C)过表达质粒和空载体购自上海吉凯基因化学技术有限公司。MEF2C siRNA及其阴性对照、miR-9-5p模拟物及其阴性对照和miR-9-5p抑制剂及其阴性对照购自上海吉玛制药技术有限公司,含有miR-9-5p结合位点的野生型荧光素酶质粒(MEF2C-3′-UTR-WT)、含有miR-9-5p结合位点突变的突变型荧光素酶质粒(MEF2C-3′-UTR-MUT)、空载体荧光素酶质粒(MEF2C-3′-UTR-NC)和海肾荧光素酶质粒购自上海吉凯基因化学技术有限公司,anti-MEF2C和anti-β-actin购自美国Abcam公司。Lipofectamine 2000转染试剂购自美国Life Technologies公司,miR-9-5p和U6引物购自北京天根生化科技有限公司,MEF2C和β-actin引物购自生工生物工程(上海)技术股份有限公司,miRNeasy Mini Kit、miRNeasy FFPE Kit、miScript ⅡRT Kit和SYBR Green PCR Kit购自德国Qiagen公司,CCK-8试剂购自日本Dojindo公司,Annexin Ⅴ-APC/PI凋亡检测试剂盒购自江苏凯基生物技术股份有限公司,Transwell小室购自美国Costar公司,Matrigel基质胶购自美国BD Biosciences公司。双荧光素酶报告基因检测系统购自美国Promega公司,倒置显微镜购自日本Olympus公司,7500实时荧光定量PCR仪购自美国Applied Biosystems公司,电泳仪、转膜仪、凝胶成像系统和全自动酶标仪购自美国Bio-Rad公司,PAS型流式细胞仪购自德国Partec公司。
1.2 组织标本收集2010-2015年石河子大学医学院第一附属医院病理科经甲醛固定石蜡包埋的ARMS组织标本和正常骨骼肌组织标本男女各5例,标本所属研究对象年龄为7个月~66岁。所有研究对象临床资料完整且详细,且术前均未进行放化疗,参照WHO软组织肿瘤分类明确诊断结果,经病理学专家证实病理诊断。本研究方案获石河子大学伦理委员会批准,在手术前获患者及其家属知情同意,并签署知情同意书。
1.3 细胞培养和转染处于对数生长期的ARMS RH30细胞、ARMS PLA802细胞、正常骨骼肌HSKMC细胞和人胚肾293T细胞置于含10%胎牛血清(FBS)和1%双抗(青-链霉素)的DMEM培养基中,于37℃、5% CO2条件下培养。根据实验需求,使用Lipofectamine 2000按照说明书进行转染。
1.4 qRT-PCR法检测不同组织和细胞中miR-9-5p和MEF2C mRNA表达水平以miR-9-5p和MEF2C为目的基因,以U6和β-actin作为内参基因。采用miRNeasy Mini Kit从4种细胞中提取总RNA,采用miRNeasy FFPE Kit从不同组织样品中提取总RNA,采用miScript Ⅱ RT Kit将RNA样品逆转录成cDNA,采用miScript SYBR Green PCR Kit在实时荧光定量PCR仪中进行扩增(反应条件:95℃预变性15 min,94℃、15 s,55℃、30 s,70℃、30 s,共40个循环)。分析实验结果的有效性,导出实验数据,采用2-△△CT法计算不同组织和细胞中miR-9-5p和MEF2C mRNA表达水平。后续实验采用miR-9-5p表达水平最高且MEF2C mRNA表达水平最低的细胞,将细胞分为miR-NC组(加入miR-9-5p模拟物)和miR-9-5p抑制剂组(加入miR-9-5p抑制剂)。
1.5 各组细胞增殖率和细胞凋亡率检测以每孔5×103个细胞的密度接种于含有100 μL 10% FBS-DMEM培养基的96孔细胞培养板(每组设5个复孔)中,待细胞贴壁0、24、48和72 h后,向每孔加入10 μL CCK-8试剂,置于细胞培养箱孵育1.5 h,酶标仪检测在450 nm处吸光度(A)值,以A值代表各组细胞增殖率。每个样本收集2.5×105个细胞,以预冷的PBS缓冲液洗涤2次,按照Annexin Ⅴ-APC/PI凋亡检测试剂盒说明书采用流式细胞术检测各组细胞凋亡率。
1.6 各组细胞侵袭数和迁移数检测采用Transwell小室进行细胞侵袭和迁移能力的检测。细胞侵袭能力,以0.2 mL DMEM培养基重悬的4×105个细胞接种于以Matrigel处理的上室中,0.6 mL 20% FBS-DMEM培养基加入下室中,置于细胞培养箱中48 h,取出上室,对侵袭细胞固定、染色,随机选取5个视野拍照,计数侵袭细胞数。细胞迁移能力检测与细胞侵袭能力检测步骤基本相同,不同之处仅为无需在上室中加入Matrigel以及细胞加入后只需培养24 h,随机选取5个视野拍照,计数迁移细胞数。
1.7 双荧光素酶报告基因检测293T细胞中荧光素酶活性采用数据库(TargetScan 7.2[10]、miRDB 6.0[11-12]和mirwalk 3.0[13])预测miR-9-5p和MEF2C mRNA 3′-UTR区的结合位点。0.1μL MEF2C-3′-UTR-WT/MEF2C-3′-UTR-MUT/MEF2C-3′-UTR-NC报告质粒、0.4 μL miR-9-5p/NC和0.02 μL海肾荧光素酶质粒共转染293T细胞,置于细胞培养箱中48 h,采用双荧光素酶报告基因检测系统检测293T细胞中荧光素酶活性。
1.8 Western blotting法检测RH30细胞中MEF2C蛋白表达水平取10 μL各蛋白样本加入10% SDS-PAGE凝胶行电泳分离,电转目的蛋白MEF2C至PVDF膜,5%脱脂奶粉封闭2 h,一抗(anti-MEF2C,1:1 000;anti-β-actin,1:2 000) 4℃孵育过夜,洗膜,对应二抗(1:10 000)室温孵育2 h,洗膜,ECL化学发光试剂盒显色,曝光,显影,定影,采用Quantity One软件分析结果。
1.9 转染MEF2C过表达质粒或MEF2CsiRNA后RH30细胞中MEF2CmRNA、细胞增殖率、细胞凋亡率、细胞侵袭数和细胞迁移数检测RH30细胞转染MEF2C过表达质粒(计为MEF2C)及其对照质粒(计为EV)、RH30细胞转染siMEF2C过表达质粒(计为siMEF2C)及其对照质粒(计为si-NC),检测RH30细胞中MEF2CmRNA表达水平、细胞增殖率、细胞凋亡率、细胞侵袭数和细胞迁移数。同时进行miR-9-5p抑制剂与MEF2CsiRNA共转染,分为miR-NC+si-NC组、miR-9-5p-inhibitor+si-NC组、miR-NC+siMEF2C组和miR-9-5p-inhibitor+siMEF2C组。检测各组细胞增殖率、细胞凋亡率、细胞侵袭数和细胞迁移数。
1.10 统计学分析采用SPSS 19.0统计软件进行统计学分析。各种组织和细胞中miR-9-5p和MEF2C mRNA的表达水平,各组细胞增殖率和凋亡率,各组细胞侵袭数和细胞迁移数,293T细胞中荧光素酶活性,RH30细胞中MEF2C蛋白表达水平均以x±s表示,组间两两比较采用LSD法,多组间样本均数比较采用单因素方差分析。以P<0.05为差异有统计学意义。
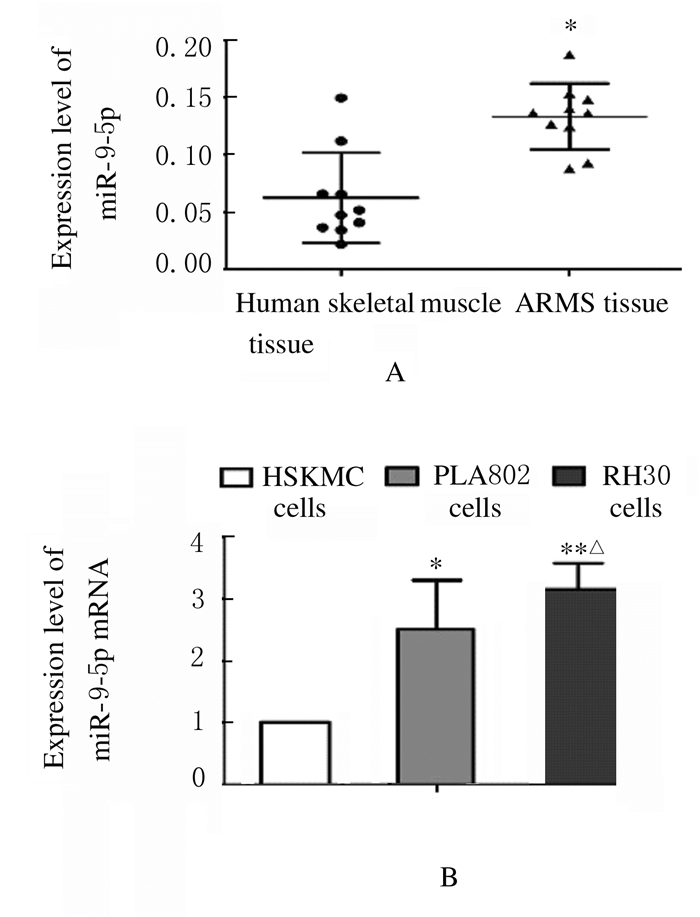
2 结果 2.1 不同组织和细胞中miR-9-5p表达水平ARMS组织中miR-9-5p表达水平高于人正常骨骼肌组织(P < 0.01),见图 1A。RH30细胞和PLA802细胞中miR-9-5p表达水平高于HSKMC细胞(P < 0.05),且RH30细胞中miR-9-5p表达水平高于PLA802细胞(P < 0.05),见图 1B。因此后续实验采用RH30细胞进行。
|
| A: Scatter plot of expression level of miR-9-5p in human skeletal muscle and ARMS tissues; *P < 0.01 vs human skeletal muscle tissue; B: Histogram of expression levels of miR-9-5p in HSKMC, PLA802 and RH30 cells, *P < 0.05, * * P < 0.01 vs HSKMC cells; △P < 0.05 vs PLA802 cells. 图 1 不同组织和细胞中miR-9-5p表达水平 Fig. 1 Expression levels of miR-9-5p in different kinds of tissues and cells |
|
|
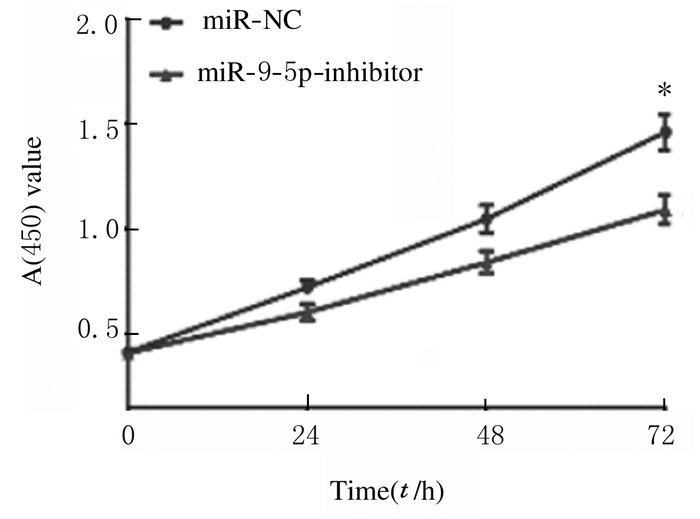
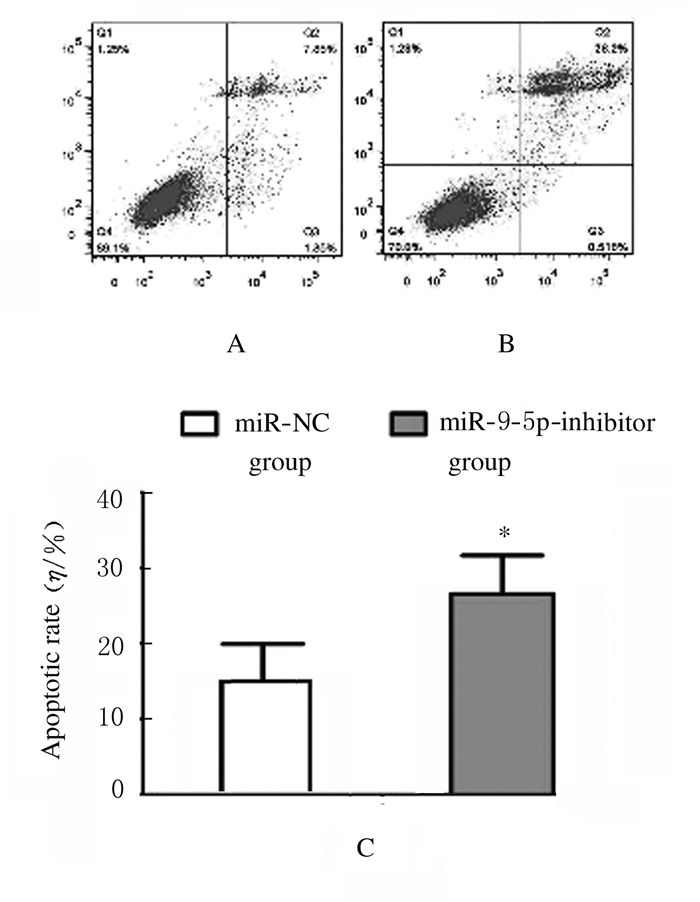
与RH30细胞中转染miR-9-5p抑制剂比较,RH30细胞中miR-9-5p表达水平明显降低。CCK-8法检测结果显示:与miR-NC组比较,miR-9-5p抑制剂组RH30细胞增殖率降低(P < 0.05),即干扰miR-9-5p抑制了RH30细胞增殖,见图 2。流式细胞术检测结果显示:与miR-NC组比较,miR-9-5p抑制剂组RH30细胞凋亡率升高(P < 0.05),即干扰miR-9-5p促进了RH30细胞凋亡。见图 3。
|
| *P < 0.05 vs miR-NC group. 图 2 不同时间点2组RH30细胞增殖率 Fig. 2 Proliferation rates of RH30 cells in two groups at different time points |
|
|
|
| A, B: Flow cytometry diagram; A:miR-NC group; B:miR-9-5p inhibitor group; C:Histogram(*P < 0.05 vs miR-NC group). 图 3 2组RH30细胞凋亡率 Fig. 3 Apoptotic rates of RH30 cells in various groups |
|
|
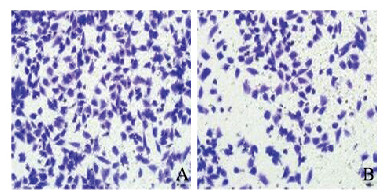
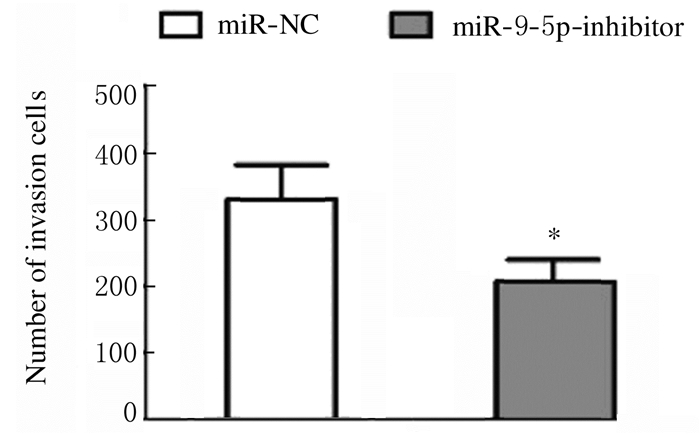
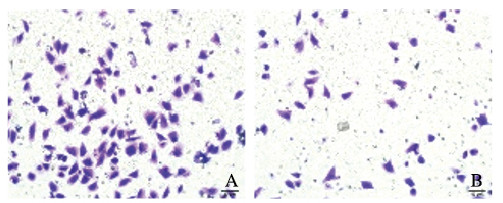
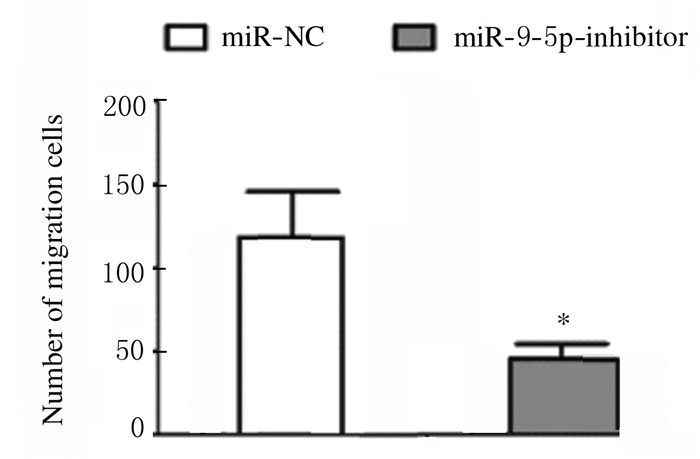
Transwell小室法检测结果显示:与miR-NC组比较,miR-9-5p抑制剂组的RH30细胞侵袭数降低(P < 0.05),见图 4(插页四)和图 5,细胞迁移数降低(P < 0.05),见图 6(插页四)和图 7,即干扰miR-9-5p抑制RH30细胞的侵袭和迁移。
|
| A: miR-NC group; B:miR 9-5p inhibitor group. 图 4 2组RH30细胞侵袭形态表现(HE, ×200) Fig. 4 Invasion morphology of RH30 cells in two groups(HE, ×200) |
|
|
|
| *P < 0.05 vs miR-NC group. 图 5 2组RH30细胞侵袭数 Fig. 5 Number of invasion RH30 cells in two groups |
|
|
|
| A: miR-NC group; B: miR-9-5p inhibitor group. 图 6 2组RH30细胞迁移形态表现(HE, ×200) Fig. 6 Migration morphology of RH30 cells in two groups (HE, ×200) |
|
|
|
| *P < 0.05 vs miR-NC group. 图 7 2组RH30细胞迁移数 Fig. 7 Number of migration RH30 cells in two groups |
|
|
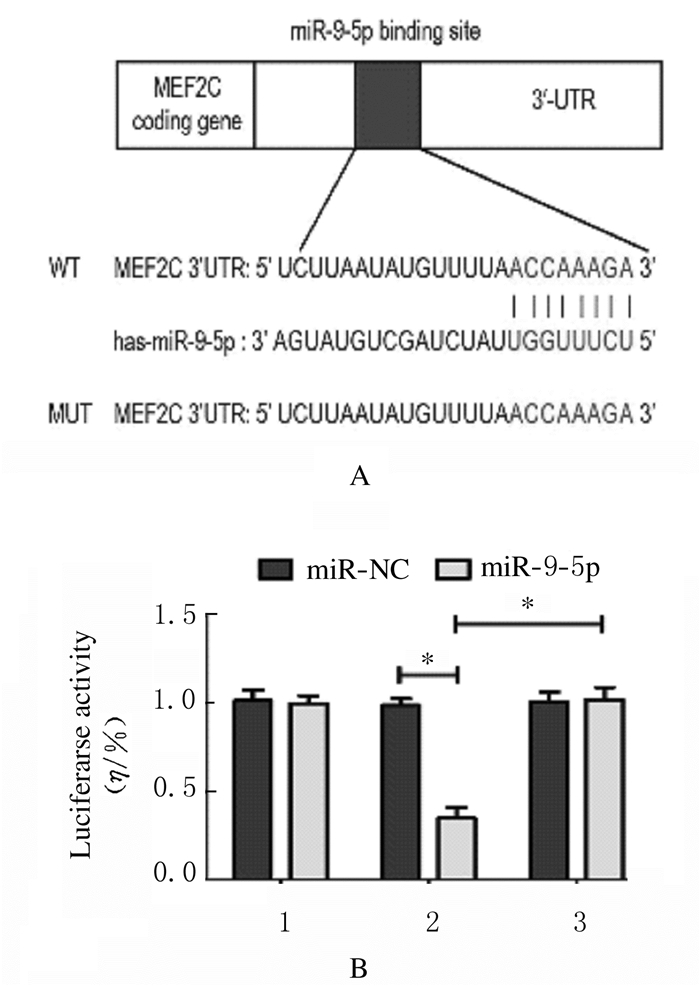
miR-9-5p与MEF2C 3′-UTR区预测的结合位点如图 8A所示。与miR-NC组比较,加入miR-9-5p后, 共转染MEF2C-3′-UTR-WT的293T细胞中荧光素酶活性降低(P < 0.01),而共转染MEF2C-3′-UTR-MUT和MEF2C-3′-UTR-NC的293T细胞中的荧光素酶活性无改变,见图 8B。
|
| 1:MEF2C-3′-UTR-NC; 2:MEF2C-3′-UTR-WT; 3:MEF2C-3′-UTR-MUT.*P < 0.01. 图 8 miR-9-5p与MEF2C-3′-UTR区预测的结合位点(A)和各组293T细胞荧光素酶活性(B) Fig. 8 Predicted binding sites of mir-9-5p and mef2c-3′- UTR region (A) and luciferase activities in 293T cells in various groups (B) |
|
|
与miR-NC组比较,miR-9-5p抑制剂组RH30细胞中MEF2C mRNA表达水平升高(t= 8.64,P < 0.05),见图 9A;与miR-NC组比较,miR-9-5p抑制剂组RH30细胞中MEF2C蛋白表达水平升高(t= 27.85,P < 0.05),见图 9B。
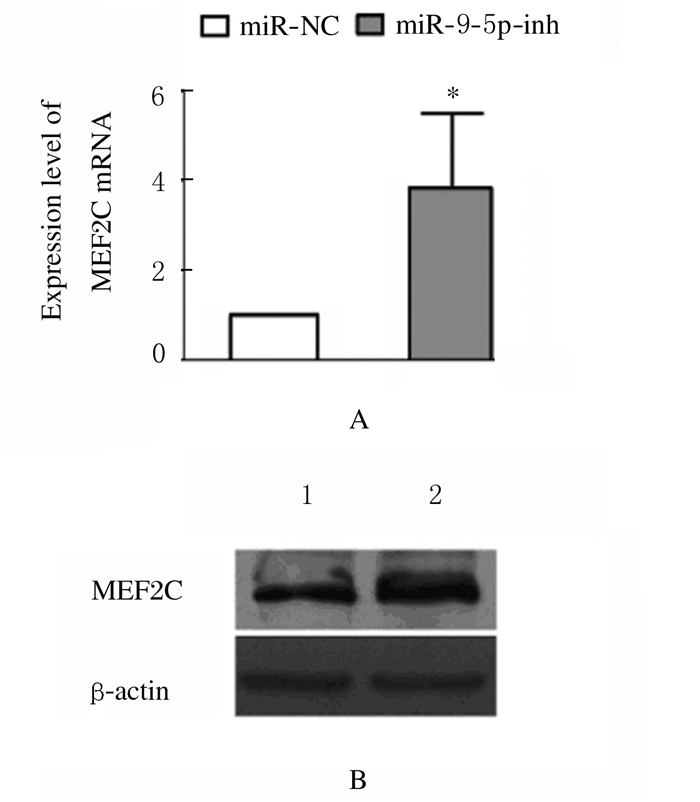
|
| A: Histogram of expression of MEF2C mRNA(*P < 0.05 vs miR-NC group); B:Electrophoregram of expressions of MEF2C protein (Lane 1:miR-NC group; Lane 2:miR-9-5p inhibitor group). 图 9 2组RH30细胞中MEF2C mRNA和蛋白表达情况 Fig. 9 Expressions of MEF2C mRNA and protein in RH30 cells in two groups |
|
|
ARMS组织中MEF2C mRNA表达水平低于正常骨骼肌组织(t=12.64,P < 0.01),见图 10A,且与ARMS组织中miR-9-5p表达水平呈负相关关系(r=-0.542,P < 0.05),见图 10B。RH30细胞和PLA802细胞中MEF2C mRNA表达水平低于HSKMC细胞(P < 0.01),见图 10C。
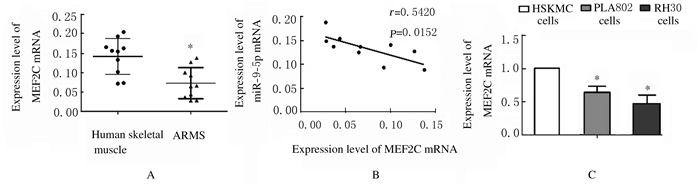
|
| A:Expression levels of MEF2C mRNA in human skeletal muscle and ARMS tissues(*P < 0.01 vs human skeletal muscle); B:Correlation between miR-9-5p and MEF2C mRNA expressions in ARMS tissue; C:Expression levels of MEF2C mRNA in HSKMC, PLA802 and RH30 cells(*P < 0.01 vs HSKMC cells). 图 10 不同组织和细胞中MEF2CmRNA表达水平 Fig. 10 Expression levels of MEF2C mRNA in different tissues and cells |
|
|
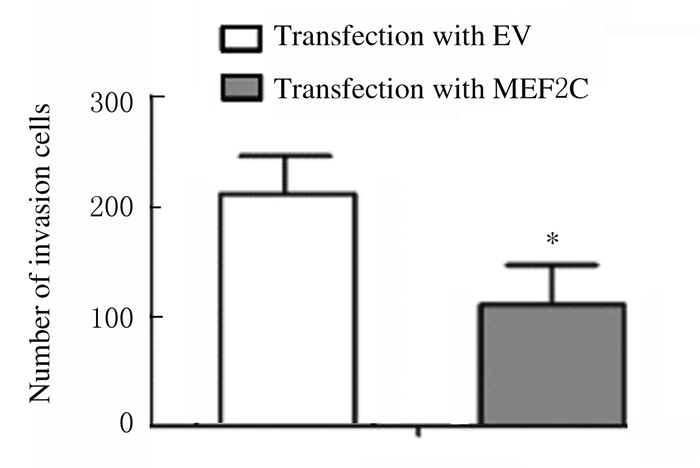
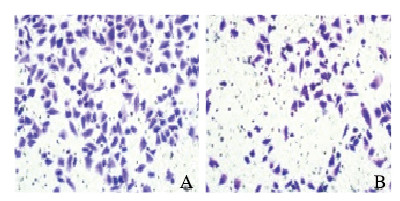
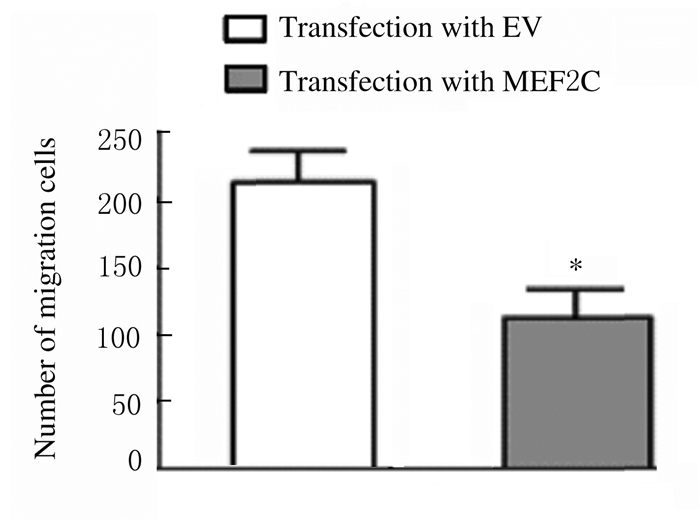
与转染对照质粒(EV)比较,转染MEF2C过表达质粒后RH30细胞中MEF2C mRNA表达水平升高(P < 0.01),见图 11。CCK-8法检测结果显示:与转染对照质粒(EV)比较,转染MEF2C过表达质粒后RH30细胞增殖率降低(P < 0.01),见图 12。流式细胞术检测结果显示:与转染对照质粒(EV)比较,转染MEF2C过表达质粒后RH30细胞凋亡率升高(P < 0.05),见图 13。Transwell小室检测结果显示:与转染对照质粒比较,转染MEF2C过表达质粒后RH30细胞侵袭数降低(P < 0.05),见图 14(插页四)和图 15,细胞迁移数降低(P < 0.01),见图 16(插页五)和图 17。
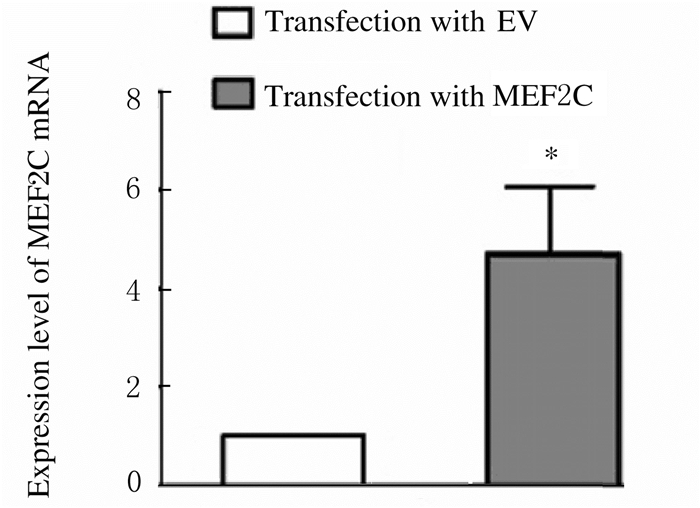
|
| *P < 0.01 vs transfection with EV. 图 11 转染MEF2C过表达质粒后RH30细胞中MEF2C mRNA表达水平 Fig. 11 Expression levels of MEF2C mRNA in RH30 cells after transfection with MEF2 over-expression plasmid |
|
|
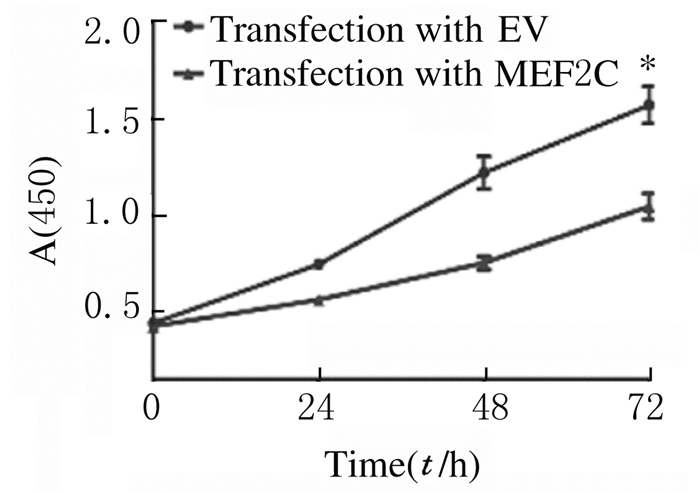
|
| *P < 0.01 vs transfection with EV. 图 12 转染MEF2C过表达质粒后RH30细胞增殖率 Fig. 12 Proliferation rates of RH30 cells after transfection with MEF2C over-expression plasmid |
|
|

|
| A, B: Flow cytometry diagram; A:Transfection with EV; B:Transfection with MEF2C;C:Histogram(*P < 0.05 vs transfection with EV). 图 13 转染MEF2C过表达质粒后RH30细胞凋亡率 Fig. 13 Apoptotic rates of RH30 cells after transfection with MEF2C over-expression plasmid |
|
|

|
| A: Transfection with EV; B: Transfection with MEF2C. 图 14 转染MEF2C过表达质粒后RH30细胞侵袭形态表现(HE, ×200) Fig. 14 Invasion morphology of RH30 cells after transfection with MEF2C over-expression plasmid(HE, ×200). |
|
|
|
| *P < 0.05 vs transfection with EV. 图 15 转染MEF2C过表达质粒后RH30细胞侵袭数 Fig. 15 Number of invasion RH30 cells after transfection with MEF2C over-expression plasmid |
|
|
|
| A: Transfection with EV; B: Transfection with MEF2C-siNC. 图 16 转染MEF2C过表达质粒后RH30细胞迁移形态表现(HE, ×200) Fig. 16 Migrate of RH30 cells after transfection with MEF2C over-expression plasmid(HE, ×200) |
|
|
|
| *P < 0.01 vs transfection with EV. 图 17 转染MEF2C过表达质粒后RH30细胞迁移数 Fig. 17 Number of migration RH30 cells after transfection with MEF2C over-expression plasmid |
|
|
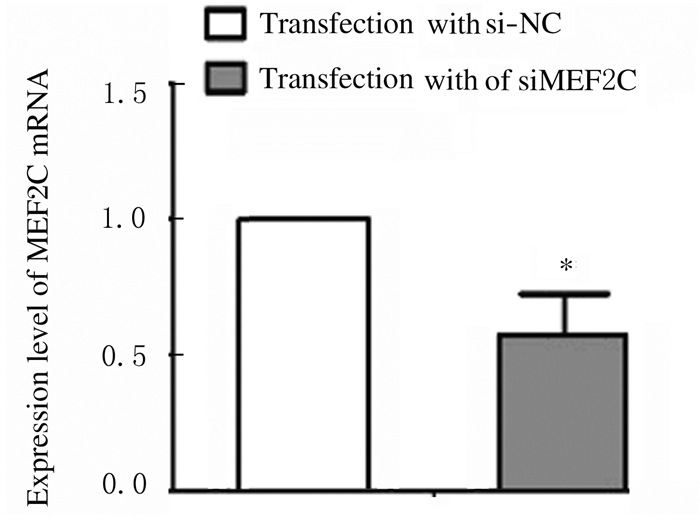
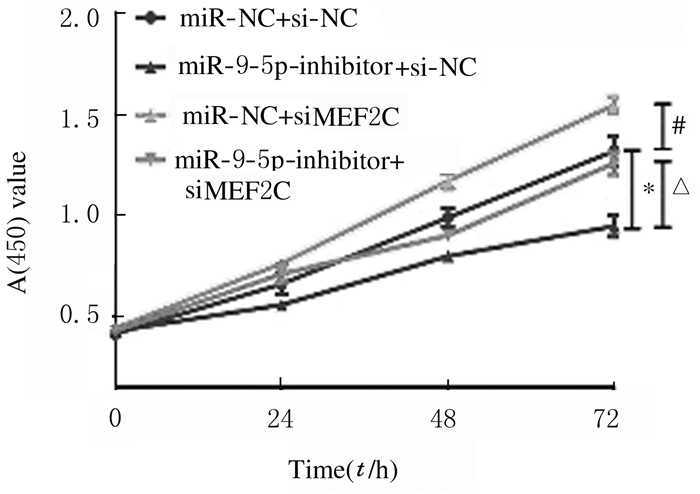
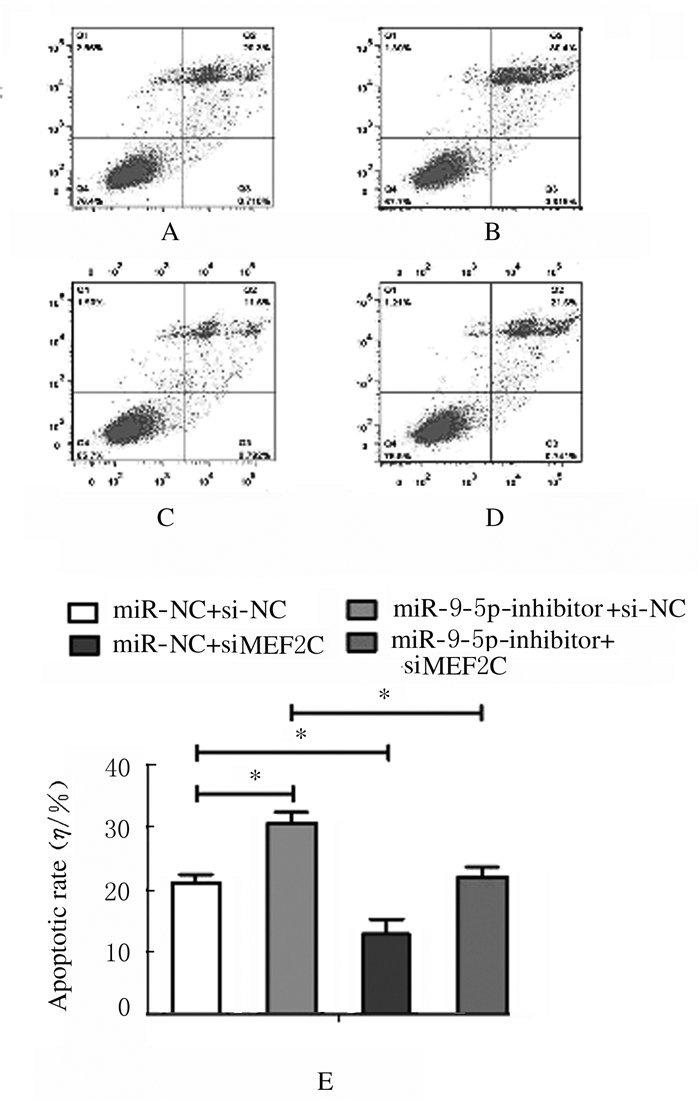
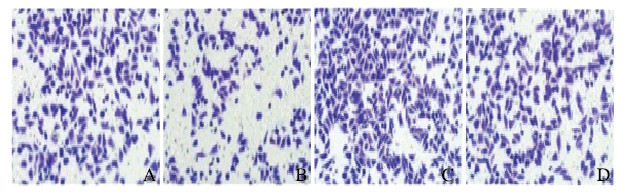
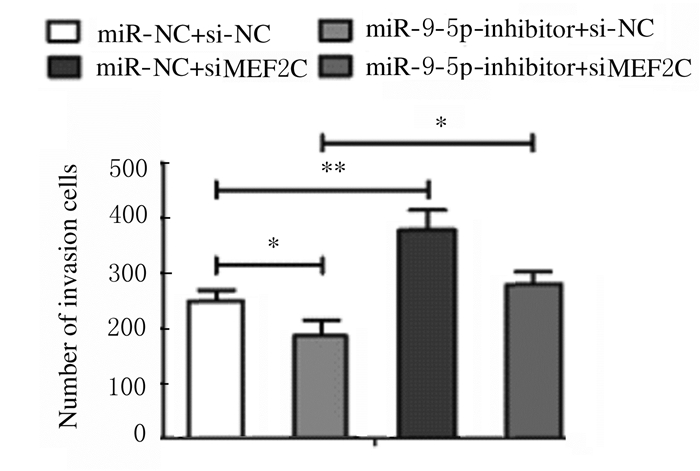
与转染si-NC比较,转染MEF2C siRNA后,RH30细胞中MEF2C mRNA表达水平降低(P < 0.01),见图 18。CCK-8法检测结果显示:与转染si-NC比较,转染MEF2C siRNA后RH30细胞增殖率升高(P < 0.05),即干扰MEF2C逆转了miR-9-5p抑制剂对细胞增殖的抑制作用,见图 19。流式细胞术检测结果显示:与转染si-NC比较,转染MEF2C siRNA后RH30细胞凋亡率降低(P < 0.01),即干扰MEF2C减弱了miR-9-5p抑制剂对细胞凋亡的促进作用,见图 20。Transwell小室检测结果显示:与转染si-NC比较,转染MEF2C siRNA后RH30细胞侵袭数升高(P < 0.01),见图 21(插页五)和图 22,细胞迁移数升高(P < 0.01),见图 23(插页五)和图 24,即干扰MEF2C逆转了miR-9-5抑制剂对细胞侵袭和迁移的抑制作用。
|
| *P < 0.01 vs transfection with si-NC. 图 18 转染MEF2C siRNA后RH30细胞中MEF2C mRNA表达水平 Fig. 18 Expression levels of MEF2C mRNA in RH30 cells after transfection with MEF2C siRNA |
|
|
|
| *P < 0.01 vs miR-9-5p-inhibitor+si-NC group; △P < 0.01 vs miR-9-5p-inhibitor+siMEF2C group; #P < 0.05 vs miR-NC+siMEF2C group. 图 19 转染MEF2C siRNA质粒后各组RH30细胞增殖率 Fig. 19 Proliferation rates of RH30 cells in various groups after transfection with MEF2C siRNA |
|
|
|
| A-D: Flow cytometry diagram; A:miR-NC+si-NC group; B:miR-9-5P-inhibitor+si-NC group; C:miR-NC+siMEF2C group; D:miR-9-5P-inhibitor+siMEF2C group; E:Histogram.*P < 0.01. 图 20 转染MEF2CsiRNA后各组RH30细胞凋亡率 Fig. 20 Apoptotic rates of RH30 cells in various groups after transfection with MEF2C siRNA |
|
|
|
| A:miR-NC+ si-NC group; B: miR-9-5 p-inhibitor+si-NC group; C: miR-NC+siMEF2C group; D: miR-9-5p-inhibitor + siMEF2C group. 图 21 转染MEF2C siRNA后RH30细胞侵袭形态表现(HE, ×200) Fig. 21 Invasion morphology of RH30 cells after transfection with MEF2C siRNA(HE, ×200) |
|
|
|
| *P < 0.05, * *P < 0.01. 图 22 转染MEF2CsiRNA后各组RH30细胞侵袭数 Fig. 22 Number of invasion RH30 cells in various groups after transfection with MEF2C siRNA |
|
|

|
| A: miR-NC+ si-NC group; B: miR-9-5p-inhibitor + si-NC group; C: miR-NC + siMEF2C group; D: miR-9-5p-inhibitor + siMEF2C group. 图 23 转染MEF2CsiRNA后RH30细胞迁移形态表现(HE, ×200) Fig. 23 Migrate morphology of RH30 cells after transfection with MEF2C siRNA(HE, ×200) |
|
|
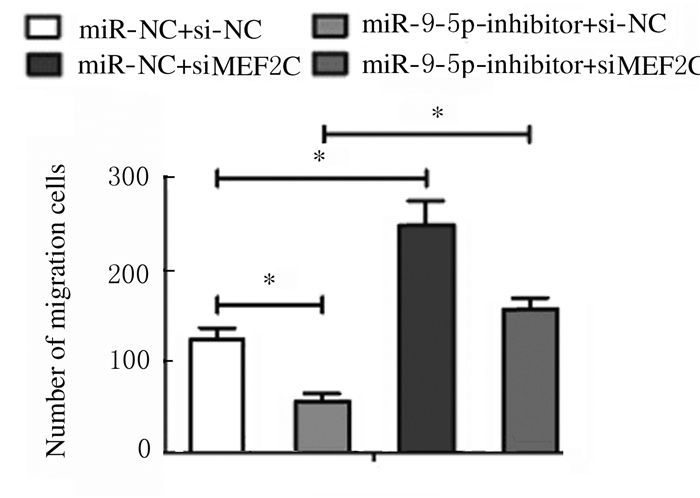
|
| *P < 0.01. 图 24 转染MEF2C siRNA后各组RH30细胞迁移数 Fig. 24 Number of migration RH30 cells in various groups after transfection with MEF2C siRNA |
|
|
miR-9源于染色体1q22(miR9-1)、5q14.3(miR9-2)和15q26.1(miR9-3),最终成为功能成熟的miR-9-5p和miR-9-3p。其中,miR-9-5p在多种癌症中存在异常表达,作为癌基因或抑癌基因参与肿瘤进展。在乳腺癌和胆管癌中miR-9-5p被认为是一种致癌miRNA[14-15],但在胃癌、卵巢癌和结肠癌中被认为是抑癌miRNA[16-18]。由于miR-9-5p参与多种肿瘤的发生发展,且具有两面性。因此,miR-9-5p在ARMS中发挥何作用成为研究重点。本研究结果显示:miR-9-5p在ARMS组织和细胞中高表达,且干扰miR-9-5p抑制细胞的增殖、侵袭、迁移以及抗凋亡能力,表明miR-9-5p在ARMS中可能起致癌基因的作用。进一步研究显示:miR-9-5p可直接靶向MEF2C mRNA的3′-UTR诱导mRNA降解调控MEF2C表达。
MEF2C是MEF2家族成员之一,最初在骨骼肌细胞中被发现,在肌细胞分化中扮演重要角色[19]。同时,MEF2C与心肌、神经嵴和软骨细胞分化发育等过程有关[20]。近年来研究[21-23]显示:MEF2C在多种肿瘤组织中存在异常表达,参与肝细胞癌、T细胞淋巴瘤和白血病等肿瘤进展。ARMS是起源于横纹肌细胞或向横纹肌细胞分化的间叶细胞的一种恶性肿瘤。因此,本文作者推测:MEF2C在ARMS中可能也发挥一定作用,MEF2C在ARMS中低表达,过表达MEF2C可抑制细胞的增殖、侵袭、迁移以及抗凋亡能力,且干扰MEF2C可部分逆转抑制miR-9-5p对ARMS细胞的抑制作用,表明MEF2C在ARMS中可能起抑癌基因的作用。
miRNA也可通过靶向mRNA的3′-UTR抑制其翻译来调节蛋白表达,如miR-874抑制GEFT蛋白表达而不影响其mRNA表达,表明miR-874通过靶向GEFT mRNA的3′-UTR导致其翻译抑制[24]。砷通过调节miR-31表达抑制SATB2翻译,使SATB2蛋白表达降低而不影响其mRNA表达[25]。这种现象引起了本文作者对miRNA抑制蛋白合成机制的关注。在植物中,miRNA通常与3′-UTR区或mRNA编码区的靶序列完全互补配对降解mRNA;在动物中,miRNA通常与靶mRNA的3′-UTR碱基不完全互补配对通过抑制翻译或引起mRNA降解来调节靶基因的表达[26]。
综上所述,miR-9-5p可通过直接靶向MEF2C促进ARMS细胞的增殖、侵袭、迁移和抗凋亡能力,miR-9-5p在ARMS中可能扮演癌基因的角色。
| [1] |
PONTES F S, DEOLIVEIRA J I, DESOUZA L L, et al. Clinicopathological analysis of head and neck rhabdomyosarcoma:A series of 10 cases and literature review[J]. Med Oral Patol Oral Cir Bucal, 2018, 23(2): e188-e197. |
| [2] |
ZAMBO I, VESELY K. WHO classification of tumours of soft tissue and bone 2013:the main changes compared to the 3rd edition[J]. Cesk Patol, 2014, 50(2): 64-70. |
| [3] |
CLEARY M M, MANSOOR A, SETTELMEYER T P, et al. NFκB signaling in alveolar rhabdomyosarcoma[J]. Dis Model Mech, 2017, 10(9): 1109-1115. DOI:10.1242/dmm.030882 |
| [4] |
BOMPAS E, CAMPION L, ITALIANO A, et al. Outcome of 449 adult patients with rhabdomyosarcoma:an observational ambispective nationwide study[J]. Cancer Med, 2018, 7(8): 4023-4035. DOI:10.1002/cam4.1374 |
| [5] |
GUO H L, INGOLIA N T, WEISSMAN J S, et al. Mammalian microRNAs predominantly act to decrease target mRNA levels[J]. Nature, 2010, 466(7308): 835-840. DOI:10.1038/nature09267 |
| [6] |
FABIAN M R, SONENBERG N, FILIPOWICZ W. Regulation of mRNA translation and stability by microRNAs[J]. Annu Rev Biochem, 2010, 79(1): 351-379. DOI:10.1146/annurev-biochem-060308-103103 |
| [7] |
FAN Y Y, SHI Y, LIN Z H, et al. miR-9-5p Suppresses malignant biological behaviors of human gastric cancer cells by negative regulation of TNFAIP8L3[J]. Dig Dis Sci, 2019, 64(10): 2823-2829. DOI:10.1007/s10620-019-05626-2 |
| [8] |
CHEN L, HU W F, LI G H, et al. Inhibition of miR-9-5p suppresses prostate cancer progress by targeting StarD13[J]. Cell Mol Biol Lett, 2019, 24(3): 20. |
| [9] |
LI G, WU F, YANG H, et al. MiR-9-5p promotes cell growth and metastasis in non-small cell lung cancer through the repression of TGFBR2[J]. Biomed Pharmacother, 2017, 96(12): 1170-1178. |
| [10] |
AGARWAL V, BELL G W, NAM J W, et al. Predicting effective microRNA target sites in mammalian mRNAs[J]. ELife, 2015, 4(8). DOI:10.7554/eLife.05005 |
| [11] |
LIU W J, WANG X W. Prediction of functional microRNA targets by integrative modeling of microRNA binding and target expression data[J]. Genome Biol, 2019, 20(1): 18. |
| [12] |
WONG N, WANG X W. MiRDB:an online resource for microRNA target prediction and functional annotations[J]. Nucleic Acids Res, 2015, 43(Databaseissue): D146-D152. |
| [13] |
STICHT C, DETORRE C, PARVEEN A, et al. MiRWalk:an online resource for prediction of microRNA binding sites[J]. PLoS One, 2018, 13(10): e0206239. DOI:10.1371/journal.pone.0206239 |
| [14] |
LIU D Z, CHANG B, LI X D, et al. MicroRNA-9 promotes the proliferation, migration, and invasion of breast cancer cells via down-regulating FOXO1[J]. Clin Transl Oncol, 2017, 19(9): 1133-1140. DOI:10.1007/s12094-017-1650-1 |
| [15] |
SHIGEHARA K, YOKOMURO S, ISHIBASHI O, et al. Real-time PCR-based analysis of the human bile microRNAome identifies miR-9 as a potential diagnostic biomarker for biliary tract cancer[J]. PLoS One, 2011, 6(8): e23584. DOI:10.1371/journal.pone.0023584 |
| [16] |
ZHENG L D, QI T, YANG D H, et al. MicroRNA-9 suppresses the proliferation, invasion and metastasis of gastric cancer cells through targeting cyclin D1 and Ets1[J]. PLoS One, 2013, 8(1): e55719. DOI:10.1371/journal.pone.0055719 |
| [17] |
TANG H S, YAO L Q, TAO X, et al. MiR-9 functions as a tumor suppressor in ovarian serous carcinoma by targeting TLN1[J]. Int J Mol Med, 2013, 32(2): 381-388. DOI:10.3892/ijmm.2013.1400 |
| [18] |
CEKAITE L, RANTALA J K, BRUUN J, et al. MiR-9, -31, and -182 deregulation promote proliferation and tumor cell survival in colon cancer[J]. Neoplasia, 2012, 14(9): 868-879. DOI:10.1593/neo.121094 |
| [19] |
GOSSETT L A, KELVIN D J, STERNBERG E A, et al. A new myocyte-specific enhancer-binding factor that recognizes a conserved element associated with multiple muscle-specific genes[J]. Mol Cell Biol, 1989, 9(11): 5022-5033. DOI:10.1128/MCB.9.11.5022 |
| [20] |
POTTHOFF M J, OLSON E N. MEF2:a central regulator of diverse developmental programs[J]. Development, 2007, 134(23): 4131-4140. DOI:10.1242/dev.008367 |
| [21] |
BAI X L, ZHANG Q, YE L Y, et al. Myocyte enhancer factor 2C regulation of hepatocellular carcinoma via vascular endothelial growth factor and Wnt/β-catenin signaling[J]. Oncogene, 2015, 34(31): 4089-4097. DOI:10.1038/onc.2014.337 |
| [22] |
HOMMINGA I, PIETERS R, LANGERAK A W, et al. Integrated transcript and genome analyses reveal NKX2-1 and MEF2C as potential oncogenes in T cell acute lymphoblastic leukemia[J]. Cancer Cell, 2011, 19(4): 484-497. DOI:10.1016/j.ccr.2011.02.008 |
| [23] |
FABER J, KRIVTSOV A V, STUBBS M C, et al. HOXA9 is required for survival in human MLL-rearranged acute leukemias[J]. Blood, 2009, 113(11): 2375-2385. DOI:10.1182/blood-2007-09-113597 |
| [24] |
SHANG H, LIU Y, LI Z Z, et al. MicroRNA-874 functions as a tumor suppressor in rhabdomyosarcoma by directly targeting GEFT[J]. Am J Cancer Res, 2019, 9(4): 668-681. |
| [25] |
CHEN Q Y, LI J Q, SUN H, et al. Role of miR-31 and SATB2 in arsenic-induced malignant BEAS-2B cell transformation[J]. Mol Carcinog, 2018, 57(8): 968-977. DOI:10.1002/mc.22817 |
| [26] |
PILLAI R S, BHATTACHARYYA S N, FILIPOWICZ W. Repression of protein synthesis by miRNAs:how many mechanisms?[J]. Trends Cell Biol, 2007, 17(3): 118-126. DOI:10.1016/j.tcb.2006.12.007 |
 2020, Vol. 46
2020, Vol. 46


