扩展功能
文章信息
- 徐凤英, 刘儒, 马丽, 曲一帆, 肖伟利, 王玉珍, 谢基明
- XU Fengying, LIU Ru, MA Li, QU Yifan, XIAO Weili, WANG Yuzhen, XIE Jiming
- 乳腺癌MDA-MB-231细胞中Rab7a基因的网络分析
- Network analysis on Rab7a gene in breast cancer MDA-MB-231 cells
- 吉林大学学报(医学版), 2020, 46(03): 464-469
- Journal of Jilin University (Medicine Edition), 2020, 46(03): 464-469
- 10.13481/j.1671-587x.20200306
-
文章历史
- 收稿日期: 2019-06-04
2. 内蒙古自治区人民医院检验科, 内蒙古 呼和浩特 010010;
3. 内蒙古农业大学生命科学学院, 内蒙古 呼和浩特 010018
2. Department of Clinical Laboratory, People's Hospital, Inner Mongolia Autonomons Region, Hohhot 010010, China;
3. College of Life Science, Inner Mongolia Agricultural University, Hohhot 010018, China
Rab7a基因和Rab7b基因是Rab7基因家族的2个同源亚型,具有较高的序列相似性[1]。与负责从核内体向高尔基体转运的Rab7b不同[2],Rab7a位于晚期核内体,控制向内吞降解室的转运[3]。Rab7a是溶酶体、吞噬体和自噬体成熟所必需的,并已被证明可调节细胞的存活和凋亡。最近报道[4]表明:Rab7a在癌症中也发挥了重要作用。波形丝蛋白是反映上皮间质转化的一个指标[5],在癌细胞中由Rab7a调控[6]。伏立诺和辛伐他汀可通过抑制Rab7协同抑制三阴性乳腺癌(tripe-negative breast cancer, TNBC)的发展[7]。然而目前尚未见关于Rab7a相关联基因如何参与TNBC发生发展的报道。本研究利用慢病毒载体敲除TNBC MDA-MB-231细胞中Rab7a基因,通过与阴性对照组比较,对差异基因进行分析,推断Rab7a及其关联基因的相互作用以及其可能参与的重要病理过程。
1 材料与方法 1.1 细胞、病毒、主要试剂和仪器人乳腺癌ZR-75-30、T-47D、HCC-1937、MDA-MB-231和MCF-7细胞购自美国模式菌种收集中心(American Type Culture Collection, ATCC)。慢病毒lv-Rab7a-sgRNA和阴性对照CON251购自上海吉凯基因化学技术有限公司。DMEM培养基购自美国Invitrogen公司,Ausbian胎牛血清(FBS)购自上海威正翔禹生物科技有限公司,BCA试剂盒和RIPA裂解液均购自上海碧云天生物有限公司。anti-Rab7a和anti-GAPDH均购自美国Santa Cruz公司,TRIzol试剂盒购自上海普飞公司,M-MLV反转录试剂盒、dNTP和Rnase Inhibitor均购自北京Promega公司,SYBR Master Mixture购自日本TaKaRa公司,人基因表达谱芯片(货号:901838)、安捷伦RNA 6000 Nano试剂盒、3′IVT PLUS基因芯片和基因芯片杂交洗涤染色试剂盒均购自美国Affymetrix公司。
1.2 细胞培养人乳腺癌ZR-75-30、T-47D和HCC-1937细胞置于RPMI1640培养基中培养,乳腺癌MCF-7细胞采用DMEM培养基培养,MDA-MB-231细胞采用L-15培养基培养。培养基中血清含量均为10%。MDA-MB-231细胞隔天换液1次,当细胞生长至80%~90%融合时,采用含EDTA的PBS平衡盐溶液漂洗,再采用0.25%胰蛋白酶消化传代,均在37℃、5%CO2培养箱中培养。
1.3 Western blotting法检测乳腺癌ZR-75-30、MCF-7、T-47D、MDA-MB-231和HCC-1937细胞中Rab7a蛋白表达水平收集对数生长期目的细胞,提取总蛋白,采用BCA试剂盒测定蛋白水平。取50 μg蛋白样品按比例加入上样缓冲液,沸水浴5 min,冷却离心后进行十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE),采用湿转法120 V、90 min将蛋白质电转至聚偏二氟乙烯(PVDF)膜上,然后采用磷酸盐缓冲液和吐温20 (TBST)溶解的5%脱脂奶粉室温封闭4 h,阻断与抗体的非特异性结合。膜与一抗(1:1 000) 4℃孵育过夜,次日采用TBST洗涤3次(每次10 min),室温孵育辣根过氧化物酶标记的二抗(1:10 000) 2 h,TBST洗涤3次(每次10 min)后检测PVDF膜上Rab7a蛋白表达水平。
1.4 慢病毒载体介导的Rab7a基因敲除将处于对数生长期的MDA-MB-231细胞经胰酶消化,完全培养基制成细胞密度为(3~5)×104mL-1的细胞悬液,取2 mL接种于6孔板,培养24 h后,采用感染复数(MOI)为10的慢病毒感染MDA-MB-231细胞以敲除Rab7a基因,培养12 h后采用完全培养液代替病毒感染液继续培养。慢病毒载体上的绿色荧光蛋白(green fluorescent protein, GFP)表达率为90%时,收集细胞进行后续Rab7a基因敲除效率的验证。Rab7a基因敲除组根据敲除的4种不同Rab7a基因序列分为KD1组、KD2组、KD3组和KD4组,并设置阴性对照组。各组Rab7a基因敲除序列见表 1。
| Group | Sequence (5′-3′) |
| Negtive control | TTCTCCGAACGTGTCACGT |
| KD1 | ACGAATTTCCTGAACCTAT |
| KD2 | AACAAGATTGACCTCGAAA |
| KD3 | CACAATAGGAGCTGACTTT |
| KD4 | CATTTGTTGTGTTGGGAAA |
采用TRIzol试剂盒对病毒感染的各组乳腺癌MDA-MB-231细胞进行裂解和总RNA提取。取2 μg总RNA和2 μL反转录引物,根据M-MLV试剂盒说明进行反转录合成cDNA,采用qPCR法扩增目的基因和内参基因。Rab7a基因敲除效率为Rab7a mRNA表达水平降低百分比,Rab7a mRNA表达水平以每组测得的目的基因Rab7a和内参基因GAPDH的CT值并采用2-ΔΔCt法进行计算。ΔCt=目的基因Ct值-内参基因Ct值,ΔΔCt=阴性对照组ΔCt平均值-各样品ΔCt值。Rab7a基因引物序列见表 2。
| Gene | Sequence (5′-3′) |
| Rab7a sence | GTCGGGAAGACATCACTCA |
| Rab7a antisence | CTAGCCTGTCATCCACCAT |
| GAPDH sence | TGACTTCAACAGCGACACCCA |
| GAPDH antisence | CACCCTGTTGCTGTAGCCAAA |
采用TRIzol法抽提阴性对照组和KD2组样品总RNA,抽提所得总RNA经NanoDrop 2000和Agilent Bioanalyzer 2100质检,合格样本进入芯片实验。芯片实验的步骤主要包括样品的制备、芯片杂交、芯片洗染、芯片扫描和结果分析。
1.7 Rab7a基因敲除后一体化通路分析(IPA)IPA是一个一体化在线整合分析软件(www.ingenuity.com)[8],用于基因、microRNA数据和小规模实验数据的分析。IPA建立了可视化的实验系统,用于理解基因、蛋白质、化学物质和药物等属性,同时可呈现分子之间相互作用的关系网。IPA使用网络生成算法,按照本课题组预设的网络大小将分子之间的网络图分割成多个网络,并给每一个网络进行打分。Score基于P值计算,反映了网络中的数据集分子出现在该网络中为随机过程的概率。Score打分是基于超几何分布,通过右尾的Fisher精确检验获得显著性水平的负对数值。所有的网络使用Score值进行排序。Score值大于2代表该功能被显著激活,Score值小于-2代表该功能被显著抑制。
1.8 统计学分析采用SPSS 20.0统计软件进行统计学分析。Rab7a基因敲除效率以x±s表示,多组间样本均数比较采用完全随机设计的单因素方差分析,组间两两比较采用SNK-q检验。以P < 0.05为差异有统计学意义。
2 结果 2.1 各组乳腺癌细胞中Rab7a蛋白表达以表达Rab7a的293T细胞作为阳性对照,收集对数生长期乳腺癌ZR-75-30、MCF-7、T-47D、MDA-MB-231和HCC-1937细胞总蛋白,Western blotting法分析结果显示:与阳性对照组比较,乳腺癌ZR-75-30、T-47D、MDA-MB-231和HCC-1937细胞中均可见Rab7a蛋白表达。见图 1。
|
| Lane 1: Positive control group(293T cells); Lane 2: ZR-75-30 cells; Lane 3: MCF-7 cells; Lane 4: T-47D cells; Lane 5: MDA-MB-231 cells; Lane 6: HCC-1937 cells. 图 1 各组乳腺癌细胞中Rab7a蛋白表达电泳图 Fig. 1 Electrophoregram of expressions of Rab7a protein in breast cancer cells in various groups |
|
|
MDA-MB-231细胞感染慢病毒96 h后,阴性对照组和KD1、KD2、KD3及KD4组细胞生长状态良好,病毒感染率达到90%以上。见图 2(插页三)。

|
| A, F: Negative control group; B, G: KD1 group; C, H: KD2 group; D, I:KD3 group; E, J: KD4 group; A-E: Bright field; F-J: Green fluorescence field. 图 2 各组慢病毒感染MDA-MB-231细胞中荧光表达(×100) Fig. 2 Fluorescence expression in MDA-MB-231 cells in various groups infected by lentivirus(×100) |
|
|
与阴性对照组比较,KD1组MDA-MB-231细胞中Rab7a mRNA表达水平下调了76.2%,即Rab7a基因敲除效率为76.2%;KD2组MDA-MB-231细胞中Rab7a mRNA表达水平下调了87.9%,KD3组MDA-MB-231细胞中Rab7a mRNA表达水平下调了86.2%,KD4组MDA-MB-231细胞中Rab7a mRNA表达水平下调了58.2%;与其他各组比较,KD2组细胞的靶序列具有最高的敲除效率(P < 0.001),即KD2组细胞的靶序列是敲除效率最高的靶点。见图 3。

|
| *P < 0.01 vs negative control group. 图 3 各组MDA-MB-231细胞中Rab7a mRNA表达水平 Fig. 3 Expression levels of Rab7a mRNA in MDA-MB-231 cells in various groups |
|
|
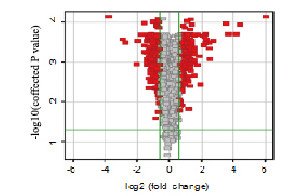
对Rab7a敲除效率最高的KD2组与阴性对照组进行基因芯片检测结果显示:与阴性对照组比较,KD2组检测的上调基因数(262个)增多(P < 0.01),下调基因数(372个)增多(P < 0.01)。火山图展示了与阴性对照组和KD2组差异基因分布情况。横坐标代表经过以2为底对数转换的差异倍数,纵坐标代表经过以10为底对数转换的矫正显著性水平;红色表示差异倍数大于1.5、显著性水平小于0.05的所有探针。横坐标0左侧代表下调的基因数,右侧代表上调的基因数。见图 4(插页三)。
|
| Red: KD2 group; Grey: Negative control group. 图 4 阴性对照组和KD2组差异基因分布 Fig. 4 Distribution of differential genes in negative control group and KD2 group |
|
|
对这634个差异基因进行疾病与功能的IPA,疾病与功能柱状图展示差异基因在疾病与功能分类中的富集情况。所有疾病与功能使用-Log(P value)值由高到底排序(即按照P值由小到大排序)。与阴性对照组比较,KD2组中与Rab7a相关联的基因主要与癌症、机体损伤和异常及细胞存活等有密切关联。见图 5。
|
| 1:Cancer; 2:Organismal injuries and abomalities; 3:Cell death and survival; 4:Cell growth; 5:Protein synthesis; 6:Cell-to-cell signaling and interaction; 7:Cellular movement; 8:Cellular development; 9:Tumor morphology; 10:Organismal survival; 11:Gastrointestinal disease; 12:Hematological system development and function; 13:Immune cell trafficking; 14:Hematological disease; 15:Immunological disease. 图 5 差异基因相关疾病与功能关系的直条图 Fig. 5 Histogram of disease and function associated with different genes |
|
|
根据指定基因VEGFA、IRS1、RPS6KB1、EIF4E和PRKAA1,添加目的基因Rab7a,通过IPA分析平台绘制基因关系网络图。在网络图中,Rab7a基因敲除导致eIF4F和IRS1表达下调以及激酶RPS6KB1表达下调;激酶PRKAA1和IKBKE表达上调及VEGFA和转录因子ATF2表达上调。见图 6。

|
| 图 6 指定基因的IPA网络图 Fig. 6 IPA network diagram of specific genes |
|
|
乳腺癌已成为全球女性发病率最高的恶性肿瘤[9],且死亡率持续升高[10], 其中TNBC占浸润性乳腺癌的10%~20%,严重影响女性健康与生活质量[11]。由于TNBC不表达雌激素受体(estrogenreceptor,ER)、孕激素受体(progesteronereceptor,PR)和人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2),因此内分泌治疗及抗HER2分子靶向治疗对TNBC患者无效[12]。目前有效的全身治疗方法是具有细胞毒性的化疗,这种治疗方式不良反应大且耐药性高[13]。因此,探索TNBC发生变化的分子机制可能有助于深入了解这种疾病的治疗策略[14]。
本课题组前期的研究[4]已经证实:Rab7a在乳腺癌中发挥致癌基因的作用, 促进乳腺癌的生长和侵袭。本研究中采用重组的4种不同靶序列的慢病毒敲除MDA-MB-231细胞的Rab7a基因,并成功获得持久基因沉默、稳定低表达Rab7a的MDA-MB-231细胞。通过对不同靶点敲除效率的验证,筛选出Rab7a敲除效率最高的KD2组进行基因芯片分析,结果显示:与Rab7a敲除相关联的基因有262个上调基因,372个下调基因。对差异基因进行IPA分析可见Rab7a及其相关联基因与癌症、细胞存活、机体损伤和异常、炎症、免疫反应等有密切联系,提示Rab7a及相关基因参与多种病理过程[15-18]。在指定的基因列表中,本研究通过添加目的基因Rab7a进行IPA分析,在基因网络分析图中,Rab7a与指定各基因相互作用并发生变化。真核起始因子(eIFs)在肿瘤发生发展起重要作用,mTOR/eIF4F轴有助于乳腺癌的维持和进展[19]。本研究中,eIF4F被Rab7a敲除后表达下调,提示Rab7a可能通过调节eIF4F而促进乳腺癌发生。此外,Rab7a沉默还导致RPS6KB1表达降低,有研究[20]显示:RPS6KB1在乳腺癌组织中发生了改变。本研究结果显示:Rab7a耗竭增强了PRKAA1的表达。目前PRKAA1在癌症发展中的作用尚不明确,PRKAA1和RPS6KB1在乳腺癌发展中的作用需要进一步研究。
综上所述,可将TNBC细胞中Rab7a作为一个靶基因,对其相关联基因进行IPA分析,全面预测Rab7a可能参与的疾病进程,并将Rab7a与其相关基因进行相互关联分析,为将来更深层次的研究奠定基础。
| [1] |
BOCK J B, MATERN H T, PEDEN A A, et al. A genomic perspective on membrane compartment organization[J]. Nature, 2001, 409(6822): 839-841. DOI:10.1038/35057024 |
| [2] |
JAGER S, BUCCI C, TANIDA I, et al. Role for Rab7 in maturation of late autophagic vacuoles[J]. J Cell Sci, 2004, 117(Pt 20): 4837-4848. |
| [3] |
BUCCI C, THOMSEN P, NICOZIANI P, et al. Rab7:a key to lysosome biogenesis[J]. Mol Biol Cell, 2000, 11(2): 467-480. DOI:10.1091/mbc.11.2.467 |
| [4] |
XIE JM, YAN Y, LIU F, et al. Knockdown of Rab7a suppresses the proliferation, migration, and xenograft tumor growth of breast cancer cells[J]. Biosci Rep, 2019, 39(2): BSR20180480. DOI:10.1042/BSR20180480 |
| [5] |
COGLI L, PROGIDA C, BRAMATO R, et al. Vimentin phosphorylation and assembly are regulated by the small GTPase Rab7a[J]. Biochim Biophys Acta, 2013, 1833(6): 1283-1293. DOI:10.1016/j.bbamcr.2013.02.024 |
| [6] |
MARGIOTTA A, PROGIDA C, BAKKE O, et al. Rab7a regulates cell migration through Rac1 and vimentin[J]. Biochim Biophys Acta Mol Cell Res, 2017, 1864(2): 367-381. DOI:10.1016/j.bbamcr.2016.11.020 |
| [7] |
KOU X, YANG Y, JIANG X, et al. Vorinostat and simvastatin have synergistic effects on triple-negative breast cancer cells via abrogating Rab7 prenylation[J]. Eur J Pharmacol, 2017, 813: 161-171. DOI:10.1016/j.ejphar.2017.08.022 |
| [8] |
CALVANO S E, XIAO W, RICHARDS D R, et al. A network-based analysis of systemic inflammation in humans[J]. Nature, 2005, 437(7061): 1032-1037. DOI:10.1038/nature03985 |
| [9] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [10] |
ZHENG Y, ZHANG M. Disparities of breast cancer burden between China and western countries and its implication[J]. Chin J Surg, 2015, 53(12): 905-909. |
| [11] |
XU S, THOMPSON W, ANCOLI-ISRAEL S, et al. Cognition, quality-of-life, and symptom clusters in breast cancer:Using Bayesian networks to elucidate complex relationships[J]. Psychooncology, 2018, 27(3): 802-809. DOI:10.1002/pon.4571 |
| [12] |
HARBECK N, GNANT M. Breast cancer[J]. Lancet, 2017, 389(10074): 1134-1150. DOI:10.1016/S0140-6736(16)31891-8 |
| [13] |
WEIN L, LOI S. Mechanisms of resistance of chemotherapy in early-stage triple negative breast cancer (TNBC)[J]. Breast, 2017, 34(Suppl 1): S27-S30. |
| [14] |
JHAN J R, ANDRECHEK E R. Triple-negative breast cancer and the potential for targeted therapy[J]. Pharmacogenomics, 2017, 18(17): 1595-1609. DOI:10.2217/pgs-2017-0117 |
| [15] |
ZHAO T, DING X, YAN C, et al. Endothelial Rab7 GTPase mediates tumor growth and metastasis in lysosomal acid lipase-deficient mice[J]. J Biol Chem, 2017, 292(47): 19198-19208. DOI:10.1074/jbc.M116.773093 |
| [16] |
WANG Q, LI H, ZHOU K, et al. Rab7 controls innate immunity by regulating phagocytosis and antimicrobial peptide expression in Chinese mitten crab[J]. Fish Shellfish Immunol, 2019, 95: 259-267. DOI:10.1016/j.fsi.2019.10.037 |
| [17] |
MOHAPATRA G, GAUR P, MUJAGOND P, et al. A SUMOylation-dependent switch of RAB7 governs intracellular life and pathogenesis of Salmonella typhimurium[J]. J Cell Sci, 2019, 132(1): jcs222612. DOI:10.1242/jcs.222612 |
| [18] |
BARBIE T U, RITTER J L, ZHU Z, et al. Phosphorylation of RAB7 by TBK1/IKKepsilon regulates innate immune signaling in triple negative breast cancer[J]. Cancer Res, 2020, 80(1): 44-56. DOI:10.1158/0008-5472.CAN-19-1310 |
| [19] |
NASR Z, ROBERT F, PORCO J A J R, et al. eIF4F suppression in breast cancer affects maintenance and progression[J]. Oncogene, 2013, 32(7): 861-871. DOI:10.1038/onc.2012.105 |
| [20] |
ADEM C, SODERBERG C L, HAFNER K, et al. ERBB2, TBX2, RPS6KB1, and MYC alterations in breast tissues of BRCA1 and BRCA2 mutation carriers[J]. Genes Chromosomes Cancer, 2004, 41(1): 1-11. DOI:10.1002/gcc.20057 |
 2020, Vol. 46
2020, Vol. 46


