扩展功能
文章信息
- 冉楠, 马明星, 庞志强, 王泽雨, 刘月, 郑瑞鹏, 卢俊英, 张超, 陈光, 章宏, 王放
- RAN Nan, MA Mingxing, PANG Zhiqiang, WANG Zeyu, LIU Yue, ZHENG Ruipeng, LU Junying, ZHANG Chao, CHEN Guang, ZHANG Hong, WANG Fang
- 血栓形成易感基因芯片的研制及效果评价
- Development and evaluation of thrombosis susceptibility gene chip
- 吉林大学学报(医学版), 2020, 46(01): 182-187
- Journal of Jilin University (Medicine Edition), 2020, 46(01): 182-187
- 10.13481/j.1671-587x.20200132
-
文章历史
- 收稿日期: 2019-02-20
2. 吉林寰基生物科技有限公司, 吉林 长春 130021;
3. 吉林大学基础医学院生理学系, 吉林 长春 130021;
4. 吉林大学第一医院血管外科, 吉林 长春 130021
2. Jilin Huanji Biotechnology Co., Ltd., Changchun 130021, China;
3. Department of Physiology, School of Basic Medical Sciences, Jilin University, Changchun 130021, China;
4. Department of Vascular Surgery, First Hospital, Jilin University, Changchun 130021, China
血栓性疾病已经成为全球死亡率最高的疾病[1]。近年来,我国血栓性疾病的发病率、致残率、死亡率和复发率呈逐年增高的趋势[2]。血栓性疾病受到多种因素的影响,其中遗传性因素在其发生发展中起着重要的作用[3]。目前血栓性疾病的诊断主要依据影像学和凝血指标的检查,缺乏灵敏度及特异度[4]。基因芯片技术作为最具发展潜力的生物高新技术体系,具有快速、高效、敏感、经济、平行化和自动化等特点[5-6]。近年来,基因芯片技术被广泛应用到生命科学的众多领域中,例如基因表达检测[7-9]、核酸突变检测、基因组多态性分析[10-14]、基因文库作图[15]和杂交测序[16]等。目前基因芯片在血栓性疾病检测方面的应用甚少。本研究拟通过探讨血栓形成易感基因芯片的设计、研制及初步临床验证,建立一种快速且高通量检测血栓形成易感基因突变的方法, 以期为易患血栓者提供一定的生活指导,为血栓性疾病的早期诊断、预防和个体化治疗奠定基础。
1 材料与方法 1.1 血栓形成易感基因及其多态性位点的确定根据GenBank上已发表的血栓形成易感基因序列,初步选择了4种凝血-纤溶系统相关因子的7种单核苷酸多态性(SNP)位点基因改变作为检测目标:纤维蛋白原β链-455G/A突变(rs1800790)、纤维蛋白原β链-148位C/T突变(rs1800787)、糖蛋白Ⅵ 13254位A/G突变(rs1613662)、凝血因子Ⅸ-580位A/G突变(rs6048)、凝血因子VLeidenG/A突变(rs6025)、凝血酶原20210位G/A突变(rs1799963)和亚甲基四氢叶酸还原酶677位C/T突变(rs1801133)[17-19]。
1.2 主要试剂和仪器QIAGEN RT-PCR Kit和核酸纯化试剂盒购自德国QIAGEN公司,Cy3荧光染料购自美国Amersham Pharmacia Biotech公司,十二烷基磺酸钠(SDS)和氯化钠柠檬酸钠清洗缓冲液(SSC)购自北京索莱宝科技有限公司。生物芯片点样仪(美国Genomic Solution公司),生物芯片扫描仪(美国Packard Bioscience公司,ABI 7300型荧光定量PCR仪(美国Applied Biosystems公司),紫外交联仪(美国Spectrolinker公司),芯片杂交仪(美国General Electric公司)。
1.3 芯片探针和引物的合成首先从GenBank或文献中搜索选定目的基因的多态性位点,根据直接杂交检测SNP的方法,通过ArrayDesginer软件设计检测探针,将检测探针送美国塞莱拉基因技术公司进行合成,探针的5′末端氨基修饰。探针序列见表 1。
| Probe name | Probe sequence | |
| Wild type (5′-3′) | Mutant type(5′-3′) | |
| rs1801133 | CTGCGGGAGCCGATTTCA | CTGCGGGAGTCGATTTCA |
| rs6025 | CTGGACAGGCGAGGAATACAG | CTGGACAGGCAAGGAATACAG |
| rs6048 | CCGTGCTGAGACTGTTTTTCC | CCGTGCTGAGGCTGTTTTTCC |
| rs1613662 | CTGCTACCGAGGAAGGTGG | CTGCTACCGGGGAAGGTGG |
| rs1799963 | GACTCTCAGCGAGCCTCAATG | GACTCTCAGCAAGCCTCAATG |
| rs1800790 | TTTAATGGCCCCTTTTGA | TTTAATAGGCCCCTTTTGA |
| rs1800787 | CAGAAATGATTATGTTGATATG | CAGAAATGATTACGTTGATATG |
根据GenBank搜索的序列结果对基因组DNA进行引物设计,针对每个基因的SNP检测位点设计2~3对检测引物,下游引物采用Cy3荧光标记。引物合成后,以人类基因组为模板进行PCR扩增,扩增产物进行琼脂糖凝胶电泳检测,找到能够高效扩增待测片段的引物组合。引物序列见表 2。
| Primer name | Sequence(5′-3′) | Primer length(bp) | Tm(θ/℃) |
| rs1801133 | CTGTGCTGTGCTGTTGGAAG (F) GACGGTGCGGTGAGAGTG (R) |
20 18 | 55.4 |
| rs6025 | TCTTTCAGGCAGGAACAACAC (F) ACACATCCAATACCAACAGACC (R) |
21 22 | 54.6 |
| rs6048 | TTCTCAGAAGTGACAAGGATGG (F) CCAACAACCCGAGTGAAGTC (R) |
22 20 | 53.8 |
| rs1613662 | GAGAAGTCTAGGCAGAGAGGAG (F) ACAACAGTCCAGTGATGTAAGC (R) |
22 22 | 54.9 |
| rs1799963 | TGTTTCTAAAACTATGGTTCCC (F) GAGAGCTGCCCATGAATAG (R) |
22 19 | 50.1 |
| rs1800780 | AAAAGGGTCTTTCTGATGTGTATTTTTCA (F) CCATAAATGCTTATGTTTTCTGACAATGA (R) |
29 29 | 54.3 |
| rs1800787 | TCGGAGCTTGTGTAGTTTCCACAT(F) GGTTCATTGTTGGCTGAACCAT(R) |
24 23 | 52.3 |
从GenBank或文献中搜索各检测位点及其邻近片段的基因序列,各检测位点基因序列分别设计突变型和正常型,送美国塞莱拉基因技术公司进行合成。
1.4.2 芯片片基的醛基化修饰芯片片基的醛基化修饰按之前研究中的方式进行[20],将清洗过的玻片放入含10 mmol·L-1氨基丙基三乙氧基硅烷(3-amin opropyltrieth oxysilane,APES)的无水乙醇溶液浸泡30 min,经过一系列处理后晾干,暗盒保存备用。
1.4.3 设计方案根据设计的探针建立6×12芯片探针布局,包括5个部分:HC为杂交对照点探针,NC为阴性参考点探针,BC为空白点,IC为提取对照点探针,rs编号为相应待测基因。W表示该基因的正常基因型检测探针,M代表该基因的突变基因型检测探针,每个探针至少有3个重复位点。芯片探针布局见表 3。
| HC | BC | rs6025-M | rs6025-W | BC | rs1613662-M | rs1613662-W | BC | rs1799963-M | rs1799963-W | BC | NC |
| HC | BC | rs6025-M | rs6025-W | BC | rs1613662-M | rs1613662-W | BC | rs1799963-M | rs1799963-W | BC | NC |
| HC | BC | rs6025-M | rs6025-W | BC | rs1613662-M | rs1613662-W | BC | rs1799963-M | rs1799963-W | BC | NC |
| IC | BC | rs6048-M | rs6048-W | rs18000790-M | rs18000790-W | rs1801133-M | rs1801133-W | rs1800787-M | rs1800787-W | BC | HC |
| IC | BC | rs6048-M | rs6048-W | rs18000790-M | rs18000790-W | rs1801133-M | rs1801133-W | rs1800787-M | rs1800787-W | BC | HC |
| IC | BC | rs6048-M | rs6048-W | rs18000790-M | rs18000790-W | rs1801133-M | rs1801133-W | rs1800787-M | rs1800787-W | BC | HC |
将合成探针和点样仪专用Glycerol Buffer按一定比例混合点样于醛基化修饰玻片上,每个点的直径为100~130 μm。通过紫外交联后使探针固定于片基上。
1.4.5 芯片杂交、洗脱、扫描和结果判读取75 μL经42℃预热的杂交缓冲液和25 μL参考品扩增-标记PCR产物(PCR扩增体系:参考品DNA5 μL, PCR扩增试剂混合物12.5 μL,PCR扩增引物7.5 μL)充分混匀后滴加在芯片矩阵位置,42℃杂交60 min。按流程将芯片洗脱后自然晾干,置于激光扫描仪中使用532 nm波长激光扫描,获得扫描数据及图谱。结果判读由血栓形成易感基因检测芯片判读系统进行,荧光信号强度的范围为0~350 AU。0 AU表示未检测到荧光信号,说明检测的基因组DNA中不包含相应位点的基因型。大于0 AU表示检测到荧光信号,说明检测的基因组DNA中包含相应位点的基因型。
1.5 血栓形成易感基因芯片的效果评价 1.5.1 基因芯片特异度和灵敏度测定采用靶序列经过测序验证的选定位点突变的人类基因组DNA为模板,对芯片设计的血栓形成易感基因检测探针特异性进行评价。灵敏度测定用紫外吸收法测定标准基因组DNA浓度,然后将标准DNA进行逐级稀释,按照基因芯片检测方法进行检测,记录检测信号最弱的浓度即可计算出芯片的检测灵敏度(最低靶基因浓度)。
1.5.2 临床样本检测试验研究对象:2017年1月—2018年5月,于寰基生物芯片产业有限公司的吉林、河南和云南3个地区的医学检验所共收集有不明原因血栓性疾病家族史的血栓患者24例,男女比例约为1.67:1,年龄36~45岁,其中吉林省9例,河南省8例,云南省7例;同时在3个地区体检中心共招募健康受试者150人,男女比例约为1.5:1,年龄36~55岁,其中吉林省60人,河南省50人,云南省40人。血栓患者纳入标准:经临床超声等检查确诊为动静脉血栓者;有血栓性疾病家族史;无临床可查证的导致血栓形成的诱因;自愿签署知情同意书入组本研究。健康受试者纳入标准:无慢性疾病,目前未患有系统性疾病;血液生化、血常规、便常规+潜血、尿常规、心电图、胸透、乙肝两对半及腹部B超等检查指标未发现具有临床意义的异常;签署知情同意书。血栓患者与健康受试者排除标准:恶性肿瘤患者;重要脏器(心、肝、肾和脑)有原发性疾病者;妊娠期或哺乳期妇女;有精神病病史者;治疗依从性差者。
为评价该芯片对血栓形成易感基因突变的检出率,取上述有不明原因血栓性疾病家族史的血栓患者和健康受试者口腔上皮细胞标本[21-22],按照1.4.5中的操作步骤进行基因芯片检测。
1.6 统计学分析采用SPSS20.0统计软件进行统计学分析。健康受试者和血栓患者血栓形成易感基因突变检出率以百分率(%)表示,组间比较采用χ2检验。以P < 0.05为差异有统计学意义。
2 结果 2.1 基因芯片杂交结果当HC和IC的3个重复位点2个以上有绿色荧光信号,且NC和BC的3个重复位点2个以上无绿色荧光信号时,芯片检测结果为有效结果。根据表 3中的芯片探针布局进行基因位点检测,3个重复位点中2个重复位点rs-W荧光值/rs-M荧光值比值≥2.0判定为正常基因型,2个以上重复位点rs-W荧光值/rs-M荧光值比值为0.5~1.5判定为杂合型,2个以上重复位点rs-W荧光值/rs-M荧光值比值≤0.5则判定为突变基因型。芯片杂交结果见图 1(封三)。

|
| A: Hybridization result of mutant gene; B: Hybridization result of normal gene; C: Hybridization result of negative reference. 图 1 以阳性参考品和阴性参考品为模板的基因芯片杂交结果 Fig. 1 Gene chip hybridization results with positive reference samples and negative reference samples as templates |
|
|
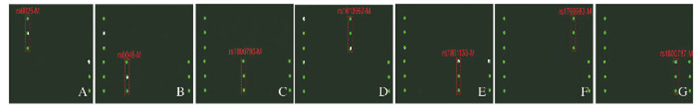
特异度检测结果:特定位点突变的人类基因组DNA在基因芯片上与目标探针出现特异性的杂交信号,杂交斑点清晰,与非目的探针几乎无杂交信号(图 2,见封三)。灵敏度检测结果:该基因芯片的灵敏度为50~100 mg·L-1。
|
| A: Special mutation for rs6025; B: Special mutation for rs6048; C: Special mutation for rs1800790; D: Special mutation for rs1613662; E: Special mutation for rs1801133; F: Special mutation for rs1799963; G: Special mutation for rs11800787. 图 2 7种血栓形成易感基因特异性突变位点基因芯片检测结果 Fig. 2 Gene ship special test of seven specific mutation sites of thrombogenic susceptibility genes |
|
|
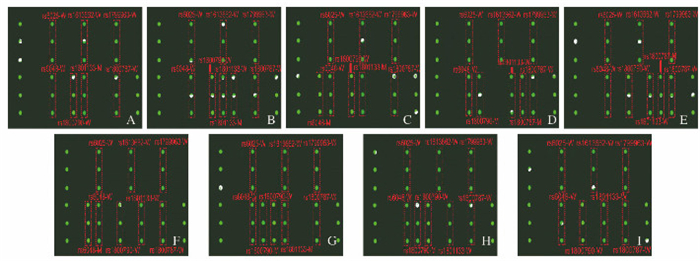
在健康受试者150人中共发现血栓形成易感基因突变者8人,血栓形成易感基因芯片检测的突变率为5.3%(8/150)(图 3,见封三);在24例有不明原因血栓性疾病家族史的血栓患者中检测到20例血栓形成易感基因突变者,血栓形成易感基因芯片检测的突变率达83.3%(20/24)。2组受试者血栓形成易感基因突变率比较差异有统计学意义(χ2=4.14,P < 0.05),有不明原因血栓性疾病家族史的血栓患者血栓形成易感基因突变检出率明显高于健康受试者。
|
| A: rs1801133 mutation (Homozygosity); B: rs1801133 mutation (Heterozygosity); C: Combined mutation of rs1801133 (Homozygosity) and rs6048 (Heterozygosity); D: Combined mutation of rs1800790 (Homozygosity) and rs1800787 (Heterozygosity); E: rs1800787 mutation (Heterozygosity); F: rs6048 mutation (Heterozygosity); G: Combined mutation of rs1801133 (Homozygosity) and rs1800790 (Heterozygosity); H: rs1800790 mutation (Heterozygosity); I: Gene ship picture of normal people 图 3 8例血栓形成易感基因突变检测者基因芯片杂交结果 Fig. 3 Gene chip hybridization results of 8 cases of thrombosis susceptibility gene mutation detectors |
|
|
血栓性疾病是复杂的遗传-环境多因素疾病,疾病负担呈逐年增长的趋势[23]。遗传因素在血栓性疾病的发生和发展中起着重要的作用。血栓性疾病的诊治重点在于早期诊断和靶向治疗,而目前血栓风险预防从预警监控到预防措施都存在明显的缺陷,还习惯于通过血压、血脂、血糖、肥胖、吸烟史、年龄和凝血参数等指标预测血栓风险。一般来说上述指标与血栓发生具有间接的关系,对实际预警血栓意义非常有限。单纯依据这些指标并不能准确反映血栓或出血的风险,也正是由于目前血栓预防措施的严重缺陷才导致血栓性疾病危害严重[24]。
基因芯片技术已经广泛应用于生命科学的各个领域,在疾病的早期诊断及个体化治疗方面起着重要的作用, 然而其在血栓性疾病方面的研究和应用较少。程克斌[25]用基因芯片技术研究了肺栓塞患者及正常人基因表达谱的差异,并选择其中11条相关基因进行荧光定量PCR验证,结果显示:基因芯片筛选基因表达异常是可靠的,但此研究仅适用于肺栓塞检测。本研究以血栓形成易感基因为研究对象,制备检测速度快、灵敏度高和特异性强的基因芯片,用于血栓性疾病的早期诊断及风险评估,使高危人群获益于临床前预防,并为血栓性疾病提供靶向治疗位点。
PCR检测技术对体系痕量核酸分子进行扩增,灵敏度高,但存在不可避免的缺陷,如产物可能有非特异性扩增等[26]。本研究中血栓形成易感基因芯片采用荧光标记引物进行RT-PCR扩增,对扩增产物进行标记,将PCR技术和分子杂交技术相结合,增加了血栓性疾病早期诊断的灵敏度和特异度。与常规PCR检测技术比较,基因芯片具有高通量检测、平行对照、避免假阴性、成本大幅度降低和快速方便等特点,对血栓性疾病的早期诊断、风险评估和疾病进程预测具有重要意义。芯片制备过程中,用于探针固定的基片种类有很多种,醛基化修饰的载玻片不仅荧光本底低,而且可以与氨基修饰的探针进行缩合反应,不需要点样后的其他处理,操作简单,提高了杂交效率和检测灵敏度。
用靶序列经过测序验证的人类基因组DNA为模板对血栓形成易感基因芯片的特异性进行检测,在芯片上出现特异性杂交信号。该基因芯片的灵敏度检测结果显示:标准基因组DNA浓度为50~100 mg·L-1,可以在芯片上显示杂交信号,而PCR检测的灵敏度为25 mg·L-1,基因芯片的灵敏度略低于PCR检测灵敏度,但特异性明显增加,假阴性率明显降低。本研究应用该基因芯片对来自吉林、河南和云南3个地区有不明原因血栓性疾病家族史的血栓患者和健康受试者进行检测,结果显示:该基因芯片对不明原因血栓性疾病家族史的血栓患者血栓形成易感基因突变检出率明显高于健康受试者。由于血栓性疾病是一个由遗传、环境和生活习惯等多因素影响的复杂疾病,因此易感基因检测无法全面反映血栓形成风险的全部问题,但是可以对人们的日常生活给予指导,有一定早期诊断价值,可降低血栓发生的风险。
本研究设计的血栓形成易感基因芯片在对血栓性疾病患者的早期诊断、临床前预防和个体化治疗方存在明显的优越性,但不足之处在于该基因芯片的检测位点未覆盖所有血栓形成易感基因,需在后续的研究中进一步完善。
| [1] |
朱楚洪. 组织工程化血管研究状况与展望[J]. 解剖学杂志, 2013, 36(1): 1-3. |
| [2] |
JIANG GH, WANG D Z, LI W, et al. Coronary heart disease mortality in China:age, gender and urban-rural gaps during epidemiological transition[J]. Rev Panam Salud Publica, 2012, 31(4): 317-324. DOI:10.1590/S1020-49892012000400008 |
| [3] |
TANG L, HU Y. Ethnic diversity in the genetics of venous thromboembolism[J]. Thromb Haemost, 2015, 114(5): 901-909. |
| [4] |
吴俊. 出血与血栓性疾病实验室诊断方法探讨[J]. 中华临床实验室管理电子杂志, 2018, 6(3): 185-186. DOI:10.3877/cma.j.issn.2095-5820.2018.03.011 |
| [5] |
ZHU T, GAO Y F, ChEN Y X, et al. Genome-scale analysis identifies GJB2 and ERO1LB as prognosis markers in patients with pancreatic cancer[J]. Oncotarget, 2017, 8(13): 21281-21289. |
| [6] |
TING C, JUN A, SHUN Z, et al. Detection of the common resistance genes in Gram-negative bacteria using gene chip technology[J]. Indian J Med Microbiol, 2013, 31(2): 142-147. |
| [7] |
SCHENA M, SHALON D, DAVIS R W, et al. Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J]. Science, 1995, 270(5235): 467-470. DOI:10.1126/science.270.5235.467 |
| [8] |
CENTLOW M, WINGREN C, BORREBAECK C, et al. Differential gene expression analysis of placentas with increased vascular resistance and pre-eclampsia using whole-genome microarrays[J]. J Pregnancy, 2011, 2011: 472354. |
| [9] |
SHALON D, SMITH S J, BROWN P O. A DNA microarray system for analyzing complex DNA samples using two-color fluorescent probe hybridization[J]. Genome Res, 1996, 6(7): 639-645. DOI:10.1101/gr.6.7.639 |
| [10] |
KASHUBA V, DMITRIEV A A, KRASNOV G S, et al. NotI microarrays:novel epigenetic markers for early detection and prognosis of high grade serous ovarian cancer[J]. Int J Mol Sci, 2012, 13(10): 13352-13377. |
| [11] |
VAN EIJK P, TENG Y M, BENNET M R, et al. Integrated microarray-based tools for detection of genomic DNA damage and repair mechanisms[J]. Methods Mol Biol, 2018, 1672: 77-99. |
| [12] |
MIRAGHAZADEH A, ZHANG P, HARDING C, et al. The use of SNP hybridisation arrays and cytogenetics to characterise deletions of chromosome 4B in hexaploid wheat (Triticum aestivum L)[J]. Theor Appl Genet, 2016, 129(11): 2151-2160. DOI:10.1007/s00122-016-2763-6 |
| [13] |
ZHOU F, LIN Y, LUO Q, et al. The clinical application of gene chips combined with CT examination in the diagnosis of large vestibular aqueduct syndrome patients[J]. J Clin Otorhinolaryngol Head Neck Surg, 2013, 27(19): 1073-1075. |
| [14] |
沈国松, 张甦, 何平亚. 基因芯片技术在复发性流产遗传学诊断中的应用[J]. 中华妇产科杂志, 2013, 48(7): 515-518. DOI:10.3760/cma.j.issn.0529-567x.2013.07.009 |
| [15] |
SAPOLSKY R J, LIPSHUTZ R J. Mapping genomic library clones using oligonucleotide arrays[J]. Genomics, 1996, 33(3): 445-456. DOI:10.1006/geno.1996.0219 |
| [16] |
ELEFTHERIOS P D. Sequencing with microarray technology-a powerful new tool for molecular diagnostics[J]. Clin Chem, 2000, 46(10): 1523-1525. DOI:10.1093/clinchem/46.10.1523 |
| [17] |
KLOVAITE J, NORDESTGAARD B G, TYBJAERG-HANSEN A, et al. Elevated fibrinogen levels are associated with risk of pulmonary embolism, but not with deep venous thrombosis[J]. Am J Respir Crit Care Med, 2013, 187(3): 286-293. DOI:10.1164/rccm.201207-1232OC |
| [18] |
KUMARI B, SRIVASTAVA S, CHATTERJEE T, et al. Study of associated genetic variants in indian subjects reveals the basis of ethnicity related differences in susceptibility to venous thromboembolism[J]. Thrombosis, 2014, 2014: 182762. |
| [19] |
JIANG J, LIU K, ZOU J J, et al. Associations between polymorphisms in coagulation-related genes and venous thromboembolism[J]. Medicine (Baltimore), 2017, 96(13): e6537. DOI:10.1097/MD.0000000000006537 |
| [20] |
韩雪清, 林祥梅, 侯义, 等. 禽流感病毒分型基因芯片的研制[J]. 微生物学报, 2008, 48(9): 1241-1249. DOI:10.3321/j.issn:0001-6209.2008.09.018 |
| [21] |
CLAIRE M, ISABELLE S, JACQUELINE C, et al. Collection of human genomic DNA from buccal cells for genetics studies:comparison between cytobrush, mouthwash, and treated Card[J]. J Biomed Biotechnol, 2005, 2005(3): 291-296. DOI:10.1155/JBB.2005.291 |
| [22] |
KVCHLER E, TANNURE P N, FALAGAN-LOTSCH P, et al. Buccal cells DNA extraction to obtain high quality human genomic DNA suitable for polymorphism genotyping by PCR-RFLP and Real-Time PCR[J]. J Appl Oral Sci, 2012, 20(4): 467-471. DOI:10.1590/S1678-77572012000400013 |
| [23] |
WENDELBOE A M, RASKOB G E. Global burden of thrombosis[J]. Circ Res, 2016, 118(9): 1340-1347. DOI:10.1161/CIRCRESAHA.115.306841 |
| [24] |
TANTRY U S, BONELLO L, ARADI D, et al. Consensus and update on the definition of on-treatment platelet reactivity to adenosine diphosphate associated with ischemia and bleeding[J]. J Am Coll Cardiol, 2013, 62(24): 2261-2273. DOI:10.1016/j.jacc.2013.07.101 |
| [25] |
程克斌.基因芯片检测肺栓塞-深静脉血栓形成相关基因群的研究[D].上海: 同济大学, 2006. http://cdmd.cnki.com.cn/article/cdmd-10247-2007226344.htm
|
| [26] |
刘姗, 刘美丽, 刘丹, 等. 人CREG启动子报告基因质粒的构建及转录活性分析[J]. 解放军医学杂志, 2019, 44(7): 585-592. |
 2020, Vol. 46
2020, Vol. 46


