扩展功能
文章信息
- 谭琦, 任立群, 张祎冰, 王亚帝, 谷泽慧, 黄鹏, 陈素贤
- TAN Qi, REN Liqun, ZHANG Yibing, WANG Yadi, GU Zehui, HUANG Peng, CHEN Suxian
- 生物信息学在三阴性乳腺癌miRNA生物标志物筛选中的应用
- Application of bioinformatics in screening of miRNA biomarkers in triple-negative breast cancer
- 吉林大学学报(医学版), 2019, 45(05): 1098-1105
- Journal of Jilin University (Medicine Edition), 2019, 45(05): 1098-1105
- 10.13481/j.1671-587x.20190522
-
文章历史
- 收稿日期: 2019-02-20
2. 锦州医科大学附属第三医院病理科, 辽宁 锦州 121001;
3. 吉林大学药学院实验药理与毒理学教研室, 吉林 长春 130021
2. Department of Pathology, Affiliated Third Hospital, Jinzhou Medical University, Jinzhou 121001, China;
3. Department of Experimental Pharmacology and Toxicology, School of Pharmacy, Jilin University, Changchun 130021, China
乳腺癌是目前世界范围内最为常见的女性恶性肿瘤,位于导致女性死亡的肿瘤性疾病第2位[1]。随着社会和经济的发展以及人民生活水平的不断提高,乳腺癌的发病率逐年上升。三阴性乳腺癌(triple-negative breast cancer, TNBC)是指雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)和人类表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)均为阴性的乳腺癌,是乳腺癌中侵袭性最强的一种亚型,占全部乳腺癌的15%~20%。由于缺乏对应的治疗靶点使得TNBC目前尚无有效的治疗方法[2]。因此,筛选有效的药物干预靶点,可为研究TNBC的发生发展提供新的理论依据,是治疗TNBC的重要策略。微小RNA(microRNA, miRNA)是一类高度保守进化且长度约为22nt的内源性单链非编码RNA[3],其通过与靶向mRNA分子的3′端互补配对,从而影响靶向mRNA翻译水平的变化,进而参与细胞生长分化和细胞凋亡等生理过程[4]。目前关于miRNA在肿瘤中作用的研究逐年增多,而miR-21-5p作为miRNA的一种,其在肿瘤的发生发展过程中起着重要作用。研究[5]显示:在非小细胞肺癌中miR-21-5p的表达水平增加,并通过调节SMAD7基因促进癌症的发展。在前列腺癌患者尿液中,miR-21-5p表达水平明显增加,成为一种区分前列腺病例中良性和侵袭性肿瘤的诊断分子[6]。miR-21-5p在多种癌症中持续高表达,促进癌症的发生发展,但在TNBC中的参与机制尚未得到证实。本研究通过对医学癌症数据库(The Cancer Genome Atlas, TCGA)与基因表达(Gene Expression Omnibus,GEO)数据库中miRNAs在TNBC中的相关研究数据进行整理,并对miR-21-5p进行靶基因预测及生物信息学分析,阐明miR-21-5p在TNBC发生发展中的主要作用机制和发挥生物学功能的主要途径,为TNBC的相关靶向治疗提供初步理论依据。
1 材料与方法 1.1 TCGA数据库数据筛选TCGA数据库(http://www.cancergenome.nih.gov/)是美国国家癌症研究所(National Cancer Institute, NCI)和美国国立人类基因组研究所(National Human Genome Research Institute,NHGRI)共同监督的一个项目,TCGA数据库收录的乳腺癌病例共有1099例,其中针对miRNA测序相关的临床研究共202例,为了研究乳腺癌组织与癌旁组织中miRNAs的差异表达,对该研究数据进行下载整理。共得到1161项患者组织中miRNAs表达的研究数据,其中包括1071例乳腺癌组织和90例癌旁组织的研究数据。使用R语言对数据进行分析处理,得到乳腺癌组织与癌旁组织中差异表达miRNAs的结果,界定logFC>1且P<0.05的miRNAs为差异性表达miRNAs。
1.2 GEO数据库数据筛选GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)是由美国国立生物技术信息中心创建并维护的基因表达数据库。以“TNBC”和“triple negative breast cancer”为关键词进行搜索,之后界定物种为人类以及非编码RNA分析数据。共确定了20个数据集。纳入标准:①每个数据集包括TNBC样本组(TNBC组织和TNBC细胞系)和对照样本组(健康乳腺组织、相邻的非癌组织、经药物治疗后组织、正常乳腺细胞系和良性乳腺增生组织);②候选miRNAs的表达样本在2组中均存在;③每组至少包含2个样本。排除标准:①重复数据集,是其他数据集的一部分;②仅有长链非编码RNA(lncRNAs)或环状RNA(CircRNAs),无候选miRNAs表达数据;③数据集缺乏正常对照组样本。另外在GSE108286中,对TNBC细胞系的26种细胞株中miRNAs的表达进行了研究,虽本研究无对照样本组(即无正常乳腺细胞系)对比,但是分析了TNBC细胞系的26种细胞株miRNAs的表达,与本研究内容相符故对该研究进行纳入。最后共纳入GSE108286和GSE70754。
1.3 候选miRNAs的生物信息学分析使用Targetscan 7.2(http://www.targetscan.org/vert_72/)和miRDB(http://mirdb.org/)数据库对候选miRNAs进行靶基因预测,将数据库所得目标靶基因取交集用于进一步研究。使用注释、可视化和整合发现的数据库(Database forAnnotation, Visualization and Interrogated Discovery, DAVID)在线分析工具(https://david.ncifcrf.gov/)对预测的靶基因进行基因本体(gene ontology, GO)功能注释与京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路富集分析。通过STRING数据库(https://string-db.org/)分析其蛋白质-蛋白质相互作用(PPI),并使用Cytoscape构建可视化网络图。选择相互作用分数>0.4确定靶基因相互作用关系。
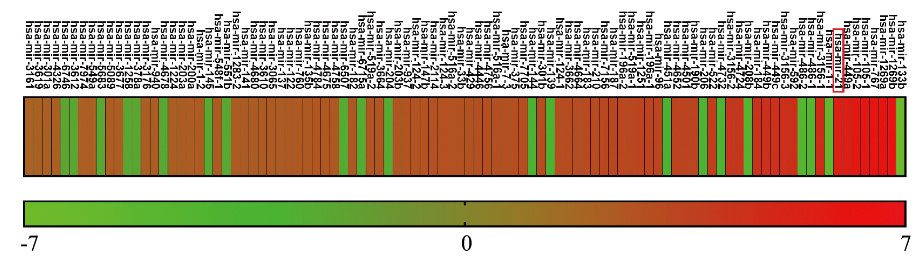
2 结果 2.1 基于TCGA分析检测乳腺癌组织和癌旁组织中miR-21-5p的表达对miRNA测序相关的202例乳腺癌病例中的1161项miRNA表达的研究数据使用R语言进行处理,并对结果整理后,乳腺癌组织与癌旁组织比较共存在335个miRNA表达具有明显改变(logFC>1且P<0.05),与癌旁组织比较,乳腺癌组织上调234个miRNA,下调101个miRNA。将限定范围调整到logFC>2共获得100个差异表达miRNA,其中上调78个miRNA,下调22个miRNA,miR-21-5p表达明显上调(logFC=5.557, P<0.01)。见图 1(插页六)和表 1。
|
| 图 1 乳腺癌组织和癌旁组织中miRNAs差异表达图 Fig. 1 Differential expression diagram of miRNAs in breast cancer tissue and adjacent tissue |
|
|
| miRNA_ID | logFC | P |
| hsa-mir-133b | -6.581 85 | 1.03E-172 |
| hsa-mir-1269b | 6.180 37 | 2.19E-14 |
| hsa-mir-1269a | 6.051 87 | 1.21E-17 |
| hsa-mir-767 | 5.982 52 | 2.31E-18 |
| hsa-mir-105-1 | 5.970 14 | 1.62E-18 |
| hsa-mir-105-2 | 5.873 04 | 4.68E-17 |
| hsa-mir-449a | 5.621 05 | 3.96E-23 |
| hsa-mir-21 | 5.556 87 | 3.74E-109 |
| hsa-mir-1-1 | -5.555 04 | 2.26E-225 |
| hsa-mir-3156-1 | 4.712 38 | 9.55E-16 |
| hsa-mir-486-1 | -4.685 07 | 9.05E-299 |
| hsa-mir-486-2 | -4.682 69 | 2.17E-297 |
| hsa-mir-592 | 4.563 74 | 1.12E-72 |
| hsa-mir-3156-3 | 4.398 96 | 4.33E-15 |
| hsa-mir-449c | 4.370 93 | 7.78E-17 |
| E standed for the power of 10. | ||
GSE108286中,分析了2055个miRNA在MDA-MB-231、BT549和BT20等26种TNBC细胞株中的表达,之后对数据进行筛选, 以miRNA在TNBC单株细胞中相对表达水平>70 000作为界定标准,共选出25个miRNA。之后对25个miRNA在26种细胞株中的表达进行整理,结果显示:miR-92a-3p、miR-182-5p、let-7f-5p、let-7a-5p、miR-22-5p、miR-21-5p、miR-191-5p和miR-30a-5p在TNBC细胞系中表达明显(在26种细胞株中表达超过20种)。见表 2。GSE70754中,分析8例患者的32个组织样本和27例患者的108个血清样本,分析临床数据中TNBC患者通过新辅助化疗(NAC)后血清中miRNAs表达情况。共发现18个miRNA与TNBC化疗预后相关,与NAC前比较,miR-200b、miR-141、miR-200a和miR-21-5p表达水平差异有统计学意义(logFC>5且P<0.01)。见表 3。与癌旁组织比较,乳腺癌组织中miR-21-5p表达水平明显升高,且TNBC患者经过化疗后血清中miR-21-5p表达水平明显下调(logFC=-5.07,P<0.01)。
| miRNA_ID | Number of expression |
| hsa-miR-92a-3p | 26 |
| hsa-miR-182-5p | 25 |
| hsa-let-7f-5p | 25 |
| hsa-let-7a-5p | 25 |
| hsa-miR-22-3p | 24 |
| hsa-miR-21-5p | 24 |
| hsa-miR-191-5p | 24 |
| hsa-miR-30a-5p | 21 |
| hsa-miR-423-5p | 20 |
| hsa-miR-26a-5p | 20 |
| hsa-miR-181a-5p | 20 |
| miRNA_ID | logFC | P |
| hsa-miR-200b | -5.476 | 3.15E-09 |
| hsa-miR-141 | -5.344 | 1.56E-10 |
| hsa-miR-200a | -5.332 | 4.12E-09 |
| hsa-miR-21 | -5.070 | 4.50E-09 |
| hsa-miR-203 | -4.830 | 1.46E-10 |
| hsa-miR-183 | 4.269 | 6.76E-08 |
| hsa-miR-148a | -4.105 | 1.86E-09 |
| hsa-miR-29c | 3.563 | 1.07E-08 |
| hsa-miR-192 | -3.322 | 6.29E-08 |
| hsa-miR-30e | -3.218 | 7.76E-08 |
| E standed for the power of 10. | ||
分别采用Targetscan和miRDB数据库预测miR-21-5p靶基因,得到362和469个结果,将结果取交集后共得到186个靶基因。
2.4 miR-21-5p在多物种间保守型分析hsa-miR-21-5p基因序列号为MIMAT0000076。通过miRBase对不同物种的miR-21-5p序列进行保守性分析:miR-21-5p的成熟序列5′-UAGCUUAUCAGACUGAUGUUGA-3′在人类、小鼠、大鼠和猕猴等多种物种间均具有很高的保守性。见表 4。
| Accession | miRNA_ID | Sequence |
| MIMAT0000076 | hsa-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0014527 | tgu-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0000530 | mmu-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0000790 | rno-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0002165 | ssc-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0002320 | mml-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0003528 | bta-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGACU-3′ |
| MIMAT0003774 | gga-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0004091 | mdo-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0004417 | cgr-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
| MIMAT0007160 | oan-miR-21-5p | 5′-UAGCUUAUCAGACUGAUGUUGA-3′ |
miR-21-5p在生物学功能(biological process)中主要参与RNA聚合酶Ⅱ启动子的转录、血管重构和细胞分化等功能(P<0.05)。在细胞组成(cellular component)方面参与细胞核、细胞质和蛋白质复合物等构成(P<0.05)。在分子功能(molecular function)中与转录因子活性、蛋白质结合和RNA聚合酶Ⅱ核心启动子近端区序列特异性DNA结合等作用相关(P<0.05)。KEGG通路富集分析结果:miR-21-5p靶基因主要富集在促分裂原活化蛋白激酶(MAPK)、转化生长因子β(TGF-β)、肿瘤坏死因子(TNF)和胰岛素抵抗等信号通路中(P<0.05)。见表 5。将186个靶基因导入STRING数据库,发现STAT3、SMAD7和PIK3R1在基因网络中起到中枢调节基因作用。见图 2。
| Category | Term | P | Gene |
| Biological process | |||
| GO:0000122 | Negative regulation of transcription from RNApolymerase Ⅱ promoter | 4.9E-8 | MEF2C, SATB1, FASLG, SMAD7 |
| GO:0045944 | Positive regulation of transcription from RNApolymerase Ⅱ promoter | 6.8E-5 | PALG1, MEF2C, THRB, SMAD7 |
| GO:0006351 | Transcription, DNA-templated | 6.1E-4 | PLAG1, MEF2C, FASLG, SMAD7 |
| GO:0001974 | Blood vessel remodeling | 6.8E-4 | MEF2C, BMRP2, JAG1, RBPJ |
| GO:0060021 | Palate development | 7.7E-4 | MEF2C, OSR1, ALX4, ALX1 |
| GO:0051056 | Regulation of small GTPase mediated signaltransduction | 8.1E-4 | TAGAP, TIAM1, ARHGAP24, ARHGEF12 |
| GO:2000737 | Negative regulation of stem cell differentiation | 1.0E-3 | ZFP36L2, JAG1, STAT3 |
| Cellular component | |||
| GO:0005654 | Nucleoplasm | 1.5E-3 | MEF2C, SATB1, THRB, SMAD7 |
| GO:0005634 | Nucleus | 1.5E-3 | MEF2C, ELF2, THRB, CPEB3 |
| GO:0005911 | Cell-cell junction | 1.2E-2 | TIAM1, KRIT1, BMPR2, PIK3R1 |
| GO:0005829 | Cytosol | 1.5E-2 | MEF2C, BMPR2, PIK3R1, VCL |
| GO:0005667 | Transcription factor complex | 1.8E-2 | SMAD7, RBPJ, ALX4, PITX2 |
| GO:0043234 | Protein complex | 2.0E-2 | MEF2C, SASH1, DCAF7, SMAD7 |
| GO:0005737 | Cytoplasm | 2.7E-2 | MEF2C, RALGPS2, ELF2, CPEB3 |
| Molecular function | |||
| GO:0005515 | Protein binding | 2.0E-5 | MEF2C, ELF2, THRB, CPEB3 |
| GO:0003700 | Transcription factor activity, sequence-specific DNAbinding | 5.0E-5 | PLAG1, MEF2C, ELF2, THRB |
| GO:0005085 | Guanyl-nucleotide exchangefactor activity | 4.4E-4 | RALGPS2, TAGAP, TIAM1, RASGRP1 |
| GO:0001077 | Transcriptional activator activity, RNA polymeraseⅡcore promoter proximal region sequence- specificbinding | 1.7E-3 | PLAG1, MEF2C, RBPJ, NFIA |
| GO:0000978 | RNA polymerase Ⅱ core promoter proximal regionsequence-specific DNA binding | 3.1E-3 | PLAG1, MEF2C, GATAD2B, RBPJ |
| GO:0031490 | Chromatin DNA binding | 3.8E-3 | SUZ12, THRB, STAT3, PITX2 |
| GO:0046872 | Metal ion binding | 2.6E-2 | SLC8A3, PLAG1, KLF6, SMAD7 |
| KEGG pathway | |||
| hsa04010 | MAPK signaling pathway | 4.7E-6 | MEF2C, NTF3, DUSP8, FASLG |
| hsa04060 | Cytokine-cytokine receptorinteraction | 6.4E-4 | IL6R, IL12A, CCL1, FASLG |
| hsa04062 | Chemokine signaling pathway | 2.4E-2 | PIK3R1, STAT3, FASLG, IL12A |
| hsa04931 | Insulin resistance | 1.6E-2 | PPP1R3A, PIK3R1, STAT3, PPP1R38 |
| hsa04668 | TNF signaling pathway | 1.5E-2 | MAP2K3, PIK3R1, CCL20, JAG1 |
| hsa04350 | TGF-β signaling pathway | 9.9E-4 | SMAD7, PITX2, BMPR2 |
| E standed for the power of 10. | |||

|
| Note: The node size represented the degree of target gene interaction. 图 2 miR-21-5p靶基因的蛋白质-蛋白质相互作用网络 Fig. 2 Protein-protein interaction network of miR-21-5p target genes |
|
|
使用DAVID数据库预测miR-21-5p在MAPK通路中关键靶基因, 分别为FASLG(Fas ligand)、MEF2C(myocyte enhancer factor 2C)、DUSP8(dual specificity phosphatase 8)、MAP2K3(mitogen-activated protein kinase kinase 3)、NTF3(neurotrophin 3)、MAP3K1(mitogen-activated protein kinase kinase kinase 1)和RASGRP1(RAS guanyi releasing protein 1)。将MAPK通路中关键靶基因通过Targetscan数据库与miR-21-5p的相互关系进行查询,对比靶基因与miR-21-5p之间的权重信誉分数, 结果显示:所选靶基因中FASLG、NTF3和DUSP8与miR-21-5p权重分数较高。见表 6。在靶基因中对miR-21-5p进行分析,发现FASLG、NTF3、MAP2K3、DUSP8与miR-21-5p权重分数较高。见表 7。
| Representative transcript | Target gene | Weighted context score |
| ENST00000340030.3 | FASLG | -0.64 |
| ENST00000423158.3 | NTF3 | -0.47 |
| ENST00000397374.3 | DUSP8 | -0.42 |
| ENST00000342679.4 | MAP2K3 | -0.30 |
| ENST00000399503.3 | MAP3K1 | -0.29 |
| ENST00000559830.1 | RASGRP1 | -0.12 |
| ENST00000340208.5 | MEF2C | -0.10 |
| Gene | Position of gene | Context score precentile | Weighted context score |
| FASLG | 853-860 | 99 | -0.46 |
| NTF3 | 133-139 | 99 | -0.47 |
| DUSP8 | 231-237 | 80 | -0.16 |
| MAP2K3 | 77-84 | 96 | -0.30 |
| MAP3K1 | 1432-1439 | 96 | -0.27 |
| RASGRP1 | 1058-1065 | 99 | -0.12 |
| MEF2C | 192-198 | 76 | -0.09 |
相对于其他类型的乳腺癌,TNBC具有复发率高、易转移、生存期短和预后差等特点。因其不表达激素受体和HER2,无法从内分泌治疗和HER2靶向治疗中获益。鉴于TNBC呈现的高度异质性,对这种类型乳腺癌的细化研究、设计出针对癌细胞的靶向治疗药物、与现行的常规化疗药物联合或序贯应用,是提高总体治愈率的重要环节[7]。目前虽然已经发现了TNBC的一些潜在的分子靶点[8],包括MAPK/细胞外信号调节激酶(ERK)信号通路、磷酸肌醇3-激酶/AKT/哺乳动物靶标雷帕霉素(PI3K/AKT/mTOR)通路等,然而其研究结果并不理想。RNA的分子变异与TNBC预后及治疗策略有密切关联。尤其是对miRNA相关靶基因调控的分子生物学研究,为提高TNBC诊断的精确性和治疗的靶向性开辟了新思路。
miRNAs是一类具有调控基因表达作用的分子,通过转录后识别其需要靶向调节的mRNA, 影响mRNA的稳定性和抑制mRNA的翻译进而对基因表达、蛋白编码基因和逆行转录后进行调控[3]。研究[9]表明:miRNA在TNBC的发生发展过程中起重要作用,可成为TNBC的诊断和预后相关因子。BHARDWAJ等[10]发现:miR-29c表达水平的下降在TNBC的早期发展中起着关键作用,表明miRNA不仅可以作为癌症诊断以及预后的观察指标,还可以应用到临床疾病基因靶向治疗中,为TNBC的临床治疗提供了新的研究思路。
对TCGA数据库中乳腺癌患者临床miRNAs表达数据进行下载,分析整理后显示:与TNBC癌旁组织比较,TNBC组织中miR-133b表达水平明显下调,miR-105和miR-21-5p表达水平明显上调,提示上述miRNA可能参与TNBC的发生发展。对GEO数据集的分析显示:在NAC期间miR-21-5p血清水平降低,并且较高的血清水平与不良预后有关,提示miR-21-5p可以作为三阴性乳腺癌化疗预后的生物学标志物。研究[11]显示:let-7miRNA通过调节JAK-STAT3/c-Myc途径控制TNBC细胞的转移,miR-21-5p调节肿瘤抑制蛋白TIMP3和PDCD4在TNBC中的表达进而参与TNBC的发生发展。另有研究[12]显示:在TNBC中miR-21-5p表达过高与上皮间充质转化(epithelial-mesenchymal transition, EMT)、不良预后及5年生存率较短有密切关联,提示miR-21-5p可能与TNBC的发生发展以及不良预后密切相关。
为了进一步探讨miR-21-5p的生物学功能,本研究通过Targetscan7.2软件预测miR-21-5p的靶基因,之后通过生物信息学进一步分析发现:miR-21-5p主要通过MAPK通路实现对TNBC发生发展的调控。在MAPK通路中MAP2K3、DUSP8和FASLG起主要作用。而靶基因的生物学功能主要集中在转录、细胞分化、信号转导、细胞凋亡、PI3P生物合成、蛋白连接和泛素蛋白转移酶活性等过程。研究[13]显示:在TNBC中DUSP8作为miR-122的靶基因与放射治疗的预后相关,而DUSP8作为一种磷酸酶在MAPK通路中去磷酸化抑制ERK、JNK和P38的表达以影响TNBC的发生发展,揭示了miR-21-5p参与TNBC发病的机制。
本研究对miR-21-5p的作用机制进行研究显示:某些信号转导通路涉及miR-21-5p的生物学效应。在食管鳞癌中,miR-21-5p靶向细胞黏附分子2(cell adhesion molecule 2,CADM2)调节AKT信号通路对其细胞凋亡起抑制作用[14]、通过调节组织金属蛋白酶抑制因子3(TIMP metallopeptidase inhibitor 3,TIMP3)信号通路抑制血管生成[15]。为了验证这项结论,在Targetscan 7.2软件中查询CADM2和TIMP3与miR-21-5p种子区域的配对类型分别为7mer-A1和8mer, 其与miR-21-5p非种子区域3′端存在不同长度的互补特性。因此CADM2和TIMP3的3′UTR与miR-21-5p种子区域的完全配对可以增强miR-21-5p对其2个基因的沉默作用。另外在多种癌症中miR-21-5p也被确定为重要的血清标记物。miR-21-5p可以作为年轻胃癌患者复发的预测因子[16],在透明细胞肾癌中也发现miR-21-5p通过靶向SATB1与预后不良有密切关联[17],miR-21-5p也是结肠癌的预后因子[18]。因此miR-21-5p可能会成为一种癌症患者预后和复发情况的监测标准和判断指标。
本实验的局限性:由于GEO数据库对于TNBC的miRNA表达数据集收录数量太少,并且本研究利用公共数据库挖掘数据和在线预测工具进行生物信息学分析,可能会导致分析结果存在较大的异质性。
综上所述,与正常乳腺组织和癌旁组织比较,TNBC组织样本中miR-21-5p的表达水平明显升高,而TNBC患者经化疗后miR-21-5p的表达水平较化疗前明显下降,表明miR-21-5p可以成为判断TNBC预后效果的指标。此外,生物信息学分析揭示了miR-21-5p通过调控其靶基因DUSP8、FASLG和MAP2K3等影响TNBC的发生发展。除以上分析研究之外,还需要同时建立、扩展TNBC患者的临床标本和随访数据库,验证miR-21-5p、DUSP8、FASLG和MAP2K3与TNBC的疾病预测及临床预后的关系。通过对miRNAs调控基因表达及相关信号转导通路的研究,对于阐明TNBC发病的确切分子机制,寻找与疾病诊断、治疗及预后相关的潜在作用靶点,具有重要的临床意义。
| [1] | HARBECK N, GNANT M. Breast cancer[J]. Lancet, 2017, 389(10074): 1134–1150. DOI:10.1016/S0140-6736(16)31891-8 |
| [2] | DENKERT C, LIEDTKE C, TUTT A, et al. Molecular alterations in triple-negative breast cancer-the road to new treatment strategies[J]. Lancet, 2017, 389(10087): 2430–2442. DOI:10.1016/S0140-6736(16)32454-0 |
| [3] | 何江洋, 李奉喜, 黄垂国, 等. 胰腺癌特异性差异表达lncRNA相关的ceRNA调控网络的构建[J]. 郑州大学学报:医学版, 2018, 53(4): 457–462. |
| [4] | FABIAN M R, SONENBERG N, FILIPOWICZ W. Regulation of mRNA translation and stability by microRNAs[J]. Annu Rev Biochem, 2010, 79: 351–379. DOI:10.1146/annurev-biochem-060308-103103 |
| [5] | LI X D, WU X F. MiR-21-5p promotes the progression of non-small-cell lung cancer by regulating the expression of SMAD7[J]. Onco Targets Ther, 2018, 11: 8445–8454. DOI:10.2147/OTT.S172393 |
| [6] | GHORBANMEHR N, GHARBI S, KORSCHING E, et al. miR-21-5p, miR-141-3p, and miR-205-5p levels in urine-promising biomarkers for the identification of prostate and bladder cancer[J]. Prostate, 2019, 79(1): 88–95. DOI:10.1002/pros.23714 |
| [7] | 何建林, 梅振宇, 盛勇, 等. 三阴性乳腺癌新辅助治疗研究进展[J]. 中国肿瘤外科杂志, 2018, 10(5): 330–332. DOI:10.3969/j.issn.1674-4136.2018.05.015 |
| [8] | HE Q, XUE S Y, TAN Y Q, et al. Dual inhibition of Akt and ERK signaling induces cell senescence in triple-negative breast cancer[J]. Cancer Lett, 2019, 448: 94–104. DOI:10.1016/j.canlet.2019.02.004 |
| [9] | FAN C, LIU N. Identification of dysregulated microRNAs associated with diagnosis and prognosis in triple-negative breast cancer:an in silico study[J]. Oncol Rep, 2019, 41(6): 3313–3324. |
| [10] | BHARDWAJ A, SINGH H, RAJAPAKSHE K, et al. Regulation of miRNA-29c and its downstream pathways in preneoplastic progression of triple-negative breast cancer[J]. Oncotarget, 2017, 8(12): 19645–19660. |
| [11] | MALLA R R, KUMARI S, GAVARA M M, et al. A perspective on the diagnostics, prognostics, and therapeutics of microRNAs of triple-negative breast cancer[J]. Biophys Rev, 2019, 11(2): 227–234. DOI:10.1007/s12551-019-00503-8 |
| [12] | PIASECKA D, BRAUN M, KORDEK R, et al. MicroRNAs in regulation of triple-negative breast cancer progression[J]. J Cancer Res Clin Oncol, 2018, 144(8): 1401–1411. DOI:10.1007/s00432-018-2689-2 |
| [13] | PEREZ-AÑORVE I X, GONZALEZ-DE LA ROSA C H, SOTO-REYES E, et al. New insights into radioresistance in breast cancer identify a dual function of miR-122 as a tumor suppressor and oncomiR[J]. Mol Oncol, 2019, 13(5): 1249–1267. |
| [14] | LI X H, CHEN D, LI M F, et al. The CADM2/Akt pathway is involved in the inhibitory effect of miR-21-5p downregulation on proliferation and apoptosis in esophageal squamous cell carcinoma cells[J]. Chem Biol Interact, 2018, 288: 76–82. DOI:10.1016/j.cbi.2018.04.021 |
| [15] | DAI J W, LIN Y Y, DUAN Y F, et al. Andrographolide inhibits angiogenesis by inhibiting the Mir-21-5p/TIMP3 signaling pathway[J]. Int J Biol Sci, 2017, 13(5): 660–668. DOI:10.7150/ijbs.19194 |
| [16] | PARK S K, PARK Y S, AHN J Y, et al. MiR 21-5p as a predictor of recurrence in young gastric cancer patients[J]. J Gastroenterol Hepatol, 2016, 31(8): 1429–1435. DOI:10.1111/jgh.13300 |
| [17] | KOWALCZYK A E, KRAZINSKI B E, GODLEWSKI J, et al. SATB1 is down-regulated in clear cell renal cell carcinoma and correlates with miR-21-5p overexpression and poor prognosis[J]. Cancer Genomics Proteomics, 2016, 13(3): 209–217. |
| [18] | YU W H, ZHU K X, WANG Y L, et al. Overexpression of miR-21-5p promotes proliferation and invasion of colon adenocarcinoma cells through targeting CHL1[J]. Mol Med, 2018, 24(1): 36. |
 2019, Vol. 45
2019, Vol. 45


