扩展功能
文章信息
- 曲添, 张红, 张志民, 杜奥博, 邵思奇, 杨瑶瑶, 刘璐
- QU Tian, ZHANG Hong, ZHANG Zhimin, DU Aobo, SHAO Siqi, YANG Yaoyao, LIU Lu
- 变形链球菌耐氟菌株eriCF基因差异性表达及其意义
- Differential expression of eriCF gene in fluoride-resistant strains of Streptococcus mutans and its significance
- 吉林大学学报(医学版), 2019, 45(03): 551-557
- Journal of Jilin University (Medicine Edition), 2019, 45(03): 551-557
- 10.13481/j.1671-587x.20190314
-
文章历史
- 收稿日期: 2018-11-28
龋病是最常见的口腔疾病之一,其主要致龋菌是变形链球菌(Streptococcus mutans,S.mutans)。氟化物既可以保护牙齿硬组织,促进牙齿再矿化,又能够抑制S.mutans等口腔致病菌的生长和代谢活动[1-2],因此被广泛用于龋病的预防和治疗[3]。但是氟化物长期广泛的使用可导致口内耐氟链球菌的产生,其中最常见的是S.mutans耐氟菌株。耐氟菌株的出现不仅降低了氟化物的抗龋作用,也干扰了口腔内微生态系统的正常运行,有研究者[4]称致病菌菌株之间的差异与患者患龋率有密切关系,严重影响患者的口腔健康。目前研究[5]显示一些细菌体内有其他离子毒力缓释系统,却无氟毒力缓释系统。因此,通过对细菌氟抗性机制的研究,人们提出了氟化物核糖开关的概念。2012年,BAKER等发现了与氟化物核糖开关相关的一种氟转运蛋白:通透的氯离子通道蛋白(EriCF)和微小的细胞膜蛋白(CrcB),并认为这2种蛋白的表达与氟化物核糖开关以及樟脑酚的抗性作用有关。有研究[6-7]显示:在S.mutans中不存在氟化物核糖开关,而存在EriCF;EriCF是由2个串联的同源基因编码产生,在GenBank中基因标记为SMU_1289C和SMU_1290C。有学者[8]在对编码基因进行过表达时发现在不同的氟化物作用下,目的基因存在差异性表达,从而证明了S.mutans氟化物转运蛋白与细菌的氟抗性有关,但是S.mutans耐氟菌株UA159-FR中关于氟抗性相关基因的研究尚未见报道。本课题组经过前期研究BLAST比对发现:S.mutans耐氟菌株UA159-FR中存在2个串联的同源基因eriCF1和eriCF2,在GenBank中基因标记为SMUFR_1127和SMUFR_1128[9],序列同源性分析发现S.mutans耐氟菌株UA159-FR菌株中的2个基因与eriCF基因的同源性为52%。有研究[6]显示丁香假单胞菌中的eriCF基因可表达氟化物通道蛋白。而STOCKBRIDGGE等[10]发现:通过引入其他细菌的氟抗性相关基因eriCF,可以使敲除crcB基因的大肠杆菌突变体恢复氟抗性能力。
基于以上研究结果,本研究通过克隆表达S.mutans耐氟菌株UA159-FR中eriCF1和eriCF2基因,构建重组大肠杆菌,并通过检测绘制重组菌在不同氟化钠浓度下的生长曲线,研究S.mutans耐氟菌株UA159-FR中eriCF1和eriCF2基因的差异性表达与大肠杆菌氟抗性的关系,以期为研究口腔链球菌及其耐氟菌株的氟抗性机制和维持口腔微生态环境的稳定性提供指导。
1 材料与方法 1.1 菌种、主要试剂和仪器S.mutans耐氟菌株UA159-FR为吉林大学口腔医院实验室保存菌株;大肠埃希菌感受态细胞TransT1、E.coli和BL21,克隆载体pEASY-Blunt Zero,表达载体Peasy-Blunt E2和PCR扩增高保真酶均购自北京全式金生物公司。细菌DNA少量提取试剂盒和质粒提取试剂盒购自天根生物公司,细菌蛋白提取试剂盒购自江苏凯基生物公司,BCA法蛋白定量测定试剂盒购自南京建成生物工程研究所。
1.2 菌株分组和给药菌株分3组,eriCF1组(eriCF1+Peasy-Blunt E2)、eriCF2组(eriCF2+Peasy-Blunt E2)和KZ组(Peasy-Blunt E2)。3组菌株均以含克隆载体的大肠杆菌为模板,分别将表达载体(eriCF1+Peasy-Blunt E2、eriCF2+Peasy-Blunt E2和Peasy-Blunt E2)转化至大肠埃希菌感受态细胞BL21中,经过冰浴30 min、热激90 s、冰浴5 min后,于无氨苄霉素的LB培养液(37℃、200 r·min-1)中培养1 h,再转入含氨苄霉素的BL固体培养基(37℃、200 r·min-1)中过夜培养,挑单菌落增加菌体浓度。
1.3 基因克隆和重组大肠杆菌的构建 1.3.1 引物设计根据GenBank提供的S.mutans耐氟菌株UA159-FR中eriCF1与eriCF2(登录号: CP007016.1)的基因序列,eriCF1基因长度为1221 bp,内侧引物扩增产物长度为1 341 bp,外侧引物扩增产物长度为1 546 bp。2个目的基因内侧引物设计时要求引物起始端以ATG开头或者ATG前方的碱基数目是3的倍数,避免开放阅读框架发生移码突变。各基因引物由库美生物公司设计并合成。见表 1。
| Gene | Primer | Sequence(5′-3′) |
| eriCF1 | Outer forward primer | CGTTTTCGTGCGTCCATTACA |
| Outer reverse primer | TGGCTTTGAAAACATTCCTTACT | |
| Inner forward primer | CTAATCTCTAATTTTTTGAAGCG | |
| Inner reverse primer | ATGATTGAAAAAAAGATTAAAGAAG | |
| eriCF2 | Outer forward primer | CCATCCCCTTACTGCTTTTAG |
| Outer reverse primer | GAGGCTATGGACTTACAAGAAAT | |
| Inner forward primer | TTAAGCAGTAACCTTTTGTGCAC | |
| Inner reverse primer | ATGGGGATTAAGATCAAGTCTTTT |
提取S.mutans UA159-FR的全基因组作为模板,通过巢式PCR扩增目的基因。eriCF1和eriCF2的外侧引物扩增反应条件相同:①95℃预变性1 min;②35个克隆循环:95℃变性20 s,55℃退火20 s,72℃延伸2.5 min;③72℃延伸10 min。利用琼脂糖凝胶电泳对PCR扩增产物进行检测,并保存PCR产物作为内侧引物扩增的反应体系模板。内侧引物反应除模板与引物发生改变,其他反应物及反应条件同外侧引物扩增条件相同。
目的基因扩增后,将ericF1和ericF2内侧PCR产物分别与pEASY-Blunt Zero克隆载体混合,于25℃恒温箱中反应20 min;再将产物分别转入Trans1-T1感受态细胞中,并设置阳性对照组(含有空载质粒的感受态细胞)和阴性对照组(用双蒸水代替克隆载体的感受态细胞),经过42℃热激45 s,转置冰上2 min后,首先转入37℃、200 r·min-1条件下的无氨苄霉素的LB液体培养基培养1 h,37℃、200 r·min-1含氨苄霉素的LB固体培养基中培养过夜。12 h后,挑选单菌落至无氨苄霉素的LB液体培养基中增加菌液浓度,通过琼脂糖凝胶电泳检测PCR扩增后的目的基因eriCF1与eriCF2的蛋白(EriCF1和EriCF2)表达水平,克隆成功后,将含目的基因的克隆载体送至库美生物公司测序检测。
1.3.3 eriCF1和eriCF2表达载体的构建以含克隆载体的大肠杆菌为模板,将扩增后的eriCF1和eriCF2分别与表达载体Peasy-Blunt E2连接,构建eriCF1+Peasy-Blunt E2、eriCF2+Peasy-Blunt E2和Peasy-Blunt E23组表达载体,并分别转化至大肠埃希菌感受态细胞BL21中,经过冰浴30 min,热激90 s,冰浴5 min后,置于37℃、200 r·min-1的无氨苄霉素LB培养液中培养1 h,再转入37℃、200 r·min-1环境下含氨苄霉素的LB固体培养基中过夜培养,挑单菌落增加菌体浓度,进行目的基因检测鉴定。
1.3.4 SDS-PAGE法检测各组EriCF1与EriCF2蛋白表达水平将eriCF1组、eriCF2组和KZ组菌液分别接种至4 mL含氨苄霉素的LB培养基中,于37℃、200 r·min-1振荡培养菌液至600 nm处吸光度(A)值为0.5~0.8 [8],再加入4 μL IPTG继续振荡培养8 h, 之后取4 mL菌液离心收集沉淀,采用PBS洗涤混匀2~3次,向EP管中加入Lysis工作液混悬。采用BCA法、SDS-PAGE电泳法、考马斯亮蓝染色法和凝胶成像等方法检测目的蛋白表达水平。
1.4 3组重组大肠杆菌菌株生长曲线的测定① 菌株条件:将3组重组大肠杆菌分别置于含氨苄霉素的LB液体培养基中培养至菌液浓度A(600)值约为0.600;②测定时间点:选取培养0、1、2、3、4、6、8、10、12、14、16、18和20 h共13个时间点;③氟化钠浓度:采用低氟浓度(1.5和2.0 g·L-1),高氟浓度(2.5和3.0 g·L-1)共4个氟化钠浓度[11];④测定方式:在室温≥15℃条件下,向管内分别加入0.8 mL含氨苄霉素的LB液体培养基、0.1 mL菌液和0.1 mL预配的10倍浓度氟化钠,对3组重组大肠杆菌按照计划时间点分别取菌液测定A(600)值,每组实验重复3次,记录分析数据,绘制细菌生长曲线。
1.5 统计学分析采用SPSS 17.0统计软件进行统计学分析。3组重组大肠杆菌的A值以x±s表示,多组间样本均数比较采用单因素方差分析。以P<0.05为差异有统计学意义。
2 结果 2.1 eriCF1和eriCF2基因克隆及表达载体的构建通过巢式PCR和外侧引物扩增,目的基因eriCF1预期片段长度为1 546 bp,eriCF2预期片段长度为1 828 bp;经琼脂糖凝胶电泳检测以及库美生物公司测序检验的实验结果一致,见图 1。通过内侧引物扩增,目的基因eriCF1, 预期片段长度为1 341 bp,eriCF2预期片段长度为1331 bp;经库美生物公司测序检验的重组克隆质粒测序结果与预期相符,提示目的基因克隆成功。将目的基因分别与表达载体Peasy-Blunt E2连接,构建重组表达载体, 经琼脂糖凝胶电泳检测结果显示表达载体连接成功,见图 2。
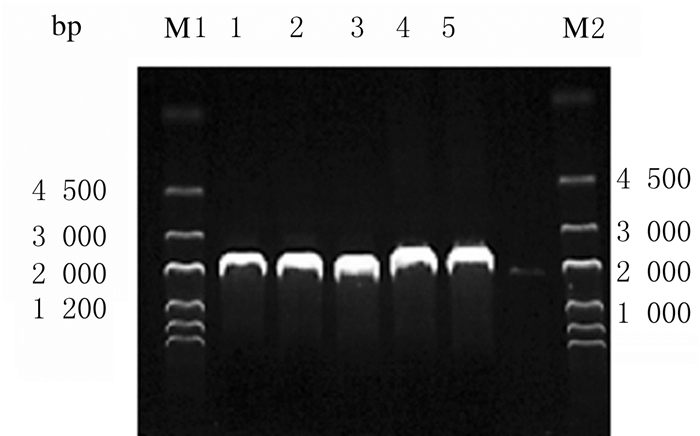
|
| M1, M2:DL4500 DNA marker; Lane 1-3: eriCF1 amplification product; Lane 4-5: eriCF2 amplification product. 图 1 eriCF1和eriCF2外侧基因片段表达电泳图 Fig. 1 Electrophoregram of expressions of lateral gene fragmentsof eriCF1 and eriCF2 |
|
|
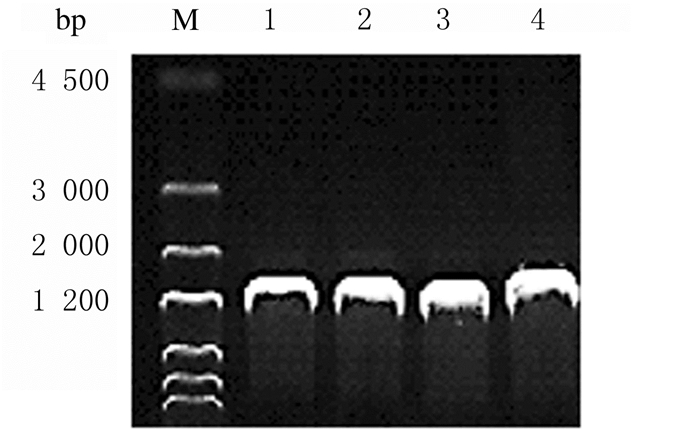
|
| M:DL4500 DNA marker; Lane 1-3: eriCF1; Lane 4: eriCF2. 图 2 重组表达载体表达电泳图 Fig. 2 Electrophoregram of expressions of recombinant expression vectors |
|
|
EriCF1蛋白相对分子质量约为31 500,EriCF2蛋白相对分子质量约为31 200,本实验通过IPTG诱导和SDS-PAGE蛋白分析结果显示:EriCF1和EriCF2蛋白相对分子质量为25 000~35 000(图 3),与现有研究结果一致,即本实验目的蛋白EriCF1和EriCF2诱导表达成功。
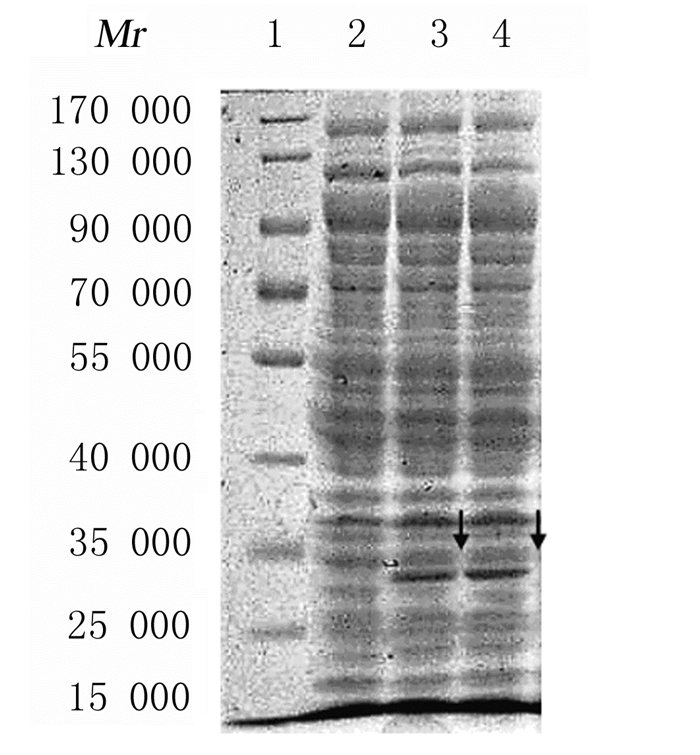
|
| Lane 1:DL10000DNA marker; Lane 2:Blank control group; Lane 3:EriCF1 protein; Lane 4:EriCF2 protein; ↓:Target protein. 图 3 EriCF1和EriCF2蛋白表达电泳图 Fig. 3 Electrophoregram of expressions of EriCF1 and EriCF2 proteins |
|
|
以时间点为横坐标,A(600)值为纵坐标,绘制4种氟化钠浓度下3组大肠杆菌的生长曲线,空白对照组菌液浓度为0,提示实验过程中无其他细菌影响。见图 4。
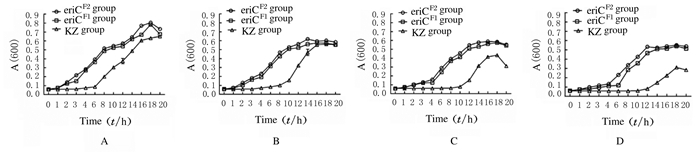
|
| A:1.5 g·L-1 sodium fluoride; B:2.0 g·L-1 sodium fluoride; C:2.5 g·L-1 sodium fluoride; D:3.0 g·L-1 sodium fluoride. 图 4 不同浓度氟化钠作用下3组大肠杆菌的生长曲线 Fig. 4 Growth curves of E.coli after treated with different concentrations of sodium fluoride in three groups |
|
|
3组菌株均大致呈现出迟缓期、对数期、稳定期和衰退期4个时期的生长变化过程[12],且随着氟化钠浓度的增加,3组菌株生长速度均逐渐减慢,菌液浓度也逐渐降低,提示氟化钠对重组大肠杆菌的生长有抑制作用;1.5 g·L-1氟化钠培养的KZ组菌液稳定期A(600)值比空白对照组降低了63.2%,eriCF1组菌液A(600)值降低了23.8%,eriCF2组菌液A(600)值降低了24.1%,提示氟化钠浓度越高,对重组大肠杆菌的生长抑制作用越强,其中氟化钠浓度对KZ组菌株的抑制作用最为明显;而与KZ组受抑制的程度比较,eriCF1组和eriCF2组菌株受抑制程度相对降低,且在高氟浓度下,受抑制的差异性越明显。
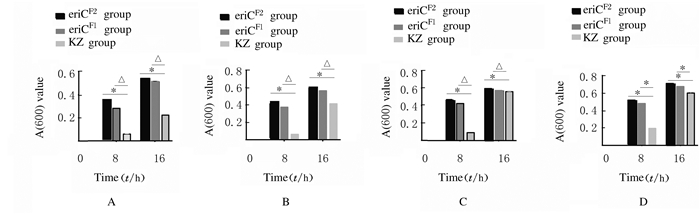
2.4 8和16 h时3组重组菌株A(600)值在对数期(8 h)和稳定期(16 h)时间点,在4个氟化钠浓度条件下,eriCF1组和eriCF2组菌液A(600)值均高于KZ组(P<0.01);且eriCF2组重组菌株的菌液A(600)值均高于eriCF1组(P<0.01)。见表 2。
| (n=3, x±s) | |||||||||
| Group | A(600)(8 h) | A(600)(16 h) | |||||||
| (sodium 3.0 g·L-1 fluoride) | 2.5 g·L-1 | 2.0 g·L-1 | 1.5 g·L-1 | (sodium 3.0 g·L-1 fluoride) | 2.5 g·L-1 | 2.0 g·L-1 | 1.5 g·L-1 | ||
| eriCF2 | 0.334±0.001 | 0.394±0.002 | 0.463±0.002 | 0.510±0.001 | 0.539±0.002 | 0.592±0.002 | 0.599±0.002 | 0.711±0.002 | |
| eriCF1 | 0.281±0.002* | 0.375±0.001* | 0.425±0.001* | 0.481±0.002* | 0.513±0.001* | 0.561±0.003* | 0.576±0.002* | 0.674±0.001* | |
| KZ | 0.059±0.001*△ | 0.062±0.001*△ | 0.093±0.002*△ | 0.195±0.001*△ | 0.222±0.001*△ | 0.412±0.001*△ | 0.560±0.001*△ | 0.600±0.001*△ | |
| F | 27 226.333 | 52 138.500 | 41 428.000 | 58 474.500 | 46 461.500 | 8 085.903 | 442.304 | 303.384 | |
| P | <0.01 | <0.01 | <0.01 | <0.01 | <0.01 | <0.01 | <0.01 | <0.01 | |
| *P < 0.01 compared with eriCF2 group;△P < 0.01 compared with eriCF1 group. | |||||||||
3组重组菌株在8和16 h菌液A(600)值比较:eriCF2组>eriCF1组>KZ组(P<0.01),提示eriCF2组菌株在不同氟环境下的生存能力最强,eriCF1组次之,KZ组最弱;eriCF1和eriCF2基因的导入均提高了大肠杆菌的氟抗性能力。见图 5。
|
| A-D:3.0, 2.5, 2.0, and 1.5 g·L -1 sodium fluoride.*P < 0.01 vs eriCF2 group; △P < 0.01 vs eriCF1 group. 图 5 不同浓度氟化钠条件下培养8和16 h时各组大肠杆菌A(600)值 Fig. 5 A(600) valuesofE.coliat 8 and 16 h after culture under different concentrations of sodium fluoride |
|
|
目前,关于S.mutans耐氟菌株中eriCF1和eriCF2基因及其作用的报道较少,但有研究学者[13]发现:许多原核生物中均含有CLC型氯离子选择性通道蛋白,而且在S.mutans中存在通透的氯离子通道,对氟化物具有高度的选择特异性。经过BLAST同源性分析[14]证实:大肠杆菌衍生的EriC是CLC型氯离子选择性通道蛋白的同源物,在部分真核和原核生物中作为氟离子转运蛋白,可以增强生物体自身的氟抗性能力。S.mutans中EriC属于CLC型氯离子通道蛋白的1个亚型,可以转运F-与H+,进而改变细菌细胞内外的pH值,减弱细菌细胞内的氟化物毒性与酸性环境所带来的生存压力[15]。上述研究提示:大肠杆菌对于氟化物具有一定的抵抗能力。LIAO等[8]在对S.mutans耐氟菌株的研究中发现:在无氟环境中培养S.mutans亲代菌株和耐氟菌株,氟抗性相关基因出现高表达,说明细菌的氟抗性并非由氟化物诱导引起,而是基因的内源性表达导致。研究[13-16]显示:在细菌中eriCF基因并不常见, 但在S.mutans的基因组中却存在2个同源基因,而且这2个目的基因(eriCF1和eriCF2)是以串联形式存在。LIAO等[17-18]发现:2个不同的S.mutans耐氟菌株中氟转运蛋白启动子mutp活性相似,进而提出EriCF1和EriCF2蛋白可能是细菌获得氟抗性能力的普遍途径。基于以上理论,本研究成功克隆S.mutans耐氟菌株中2个编码通透氯离子通道蛋白的eriCF1和eriCF2基因, 进而证实了基因内源性表达这一研究理论, 为S.mutans耐氟菌株的耐氟机制提供了研究基础;通过IPTG诱导可以实现表达EriCF1和EriCF2融合蛋白,这为研究S.mutans氟转运相关蛋白的结构、氟转运机制及防龋疫苗的研制奠定了理论基础[19]。
BAKER等[6]提出:同一个生物体内eriCF基因和crcB基因不会同时存在。大肠杆菌可以通过crcB基因获得氟抗性能力,当crcB基因缺失时,会导致突变株对氟离子高度敏感。因此,MURATA等[13]将S.mutans中2个编码氯离子选择性通道蛋白的基因转入到大肠杆菌crcB基因敲除株中,证明了eriCF和crcB基因在氟抗性方面具有互补作用。MEN等[20]在此基础上发现:虽然2种基因均与细菌耐氟性有关,但口腔链球菌中eriCF基因的氟抗性能力强于crcB基因。然而,当来自同一链球菌属内不同的eriC和crcB基因共存于同一口腔链球菌体内时,却未出现菌株氟抗性能力增强的叠加效应。因此,为了进一步研究S.mutans耐氟菌株中eriCF1和eriCF2的氟抗性作用,本研究检测了3组重组大肠杆菌的氟抗性能力。本研究结果显示:在大肠杆菌crcB基因存在的情况下,不论在高氟环境下(2.5和3.0g·L-1)还是低氟环境下(1.5和2.0 g·L-1),eriCF1组和eriCF2组菌株菌液终浓度及生长速度均高于KZ组,且eriCF2组菌液终浓度高于eriCF1组,提示S.mutans耐氟菌株中eriCF1和eriCF2基因的导入均提高了大肠杆菌的氟抗性能力, 这与S.mutans中通透的氯离子通道作用相同。本文作者认为:EriCF1和EriCF2蛋白可能通过逆转细菌体内的氟离子浓度,从而减弱氟化物的毒性作用[21], 或者是与大肠杆菌内部产酸和耐酸机制的改变有关[22],CAI等[23]提出:当细菌较长时间生存在低pH值环境中时,生物膜中所含有的氟化物可诱导细菌产生氟抗性能力。本研究结果显示:与原代大肠杆菌BL21比较,eriCF1和eriCF2的导入不仅能增强细菌的氟抗性能力,而且eriCF2的氟抗性能力较eriCF1强,提示来自不同菌属的氟抗性相关蛋白可以起到叠加效应,增强原代菌株的氟抗性能力,这与MEN等[20]的研究结论不同。本文作者认为出现叠加效应的原因,可能为不同菌属之间目的基因产物的相互作用[24],但具体机制仍有待于后续的研究和验证。
综上所述, S.mutans耐氟菌株中eriCF1和eriCF2基因均与细菌的氟抗性能力有关, 而且来自不同菌属的氟抗性相关蛋白可以出现氟抗性叠加效应。本研究结果对于探索氯离子选择性通道蛋白与氟制剂联合应用来抑制S.mutans的生长具有重要的指导意义,利用氟抗性相关蛋白机制促进氟化物通过细菌的细胞膜,有选择性地降低细菌的氟抗性,提高氟化物的抗菌作用,不仅会改善人类的口腔健康状况,对人体的全身健康状况也具有现实意义。
| [1] | CATE J M. Fluorides in caries prevention and control:empiricism or science[J]. Caries Res, 2004, 38(3): 254–257. DOI:10.1159/000077763 |
| [2] | SUBRAMANIAM P, NADAN N. Effect of xylitol, sodium fluoride and triclosan containing mouth rinse on Streptococcus mutans[J]. Contemp Clin Dent, 2011, 2(4): 287–290. DOI:10.4103/0976-237X.91790 |
| [3] | BUZALAF M A, PESSAN J P, HONÓRIO H M, et al. Mechanisms of action of fluoride for caries control[J]. Monogr Oral Sci, 2011, 22: 97–114. DOI:10.1159/000325151 |
| [4] | OISHI Y, WATANABE K, KUMADA H, et al. Purification and characterization of a novel secondary fimbrial protein from Porphyromonas gulae[J]. J Oral Microbiol, 2012, 4(1): 1–10. |
| [5] | WEINBERG Z, WANG J X, BOGUE J, et al. Comparative genomics reveals 104 candidate structured RNAs from bacteria, archaea, and their metagenomes[J]. Genome Biol, 2010, 11(3): R31. DOI:10.1186/gb-2010-11-3-r31 |
| [6] | BAKER J L, SUDARSANN, WEINBERGZ, et al. Widespread genetic switches and toxicity resistance proteins for fluoride[J]. Science, 2012, 335(6065): 233–235. DOI:10.1126/science.1215063 |
| [7] | BREAKER R R. New insight on the response of bacteria to fluoride[J]. Caries Res, 2012, 46(1): 78–81. DOI:10.1159/000336397 |
| [8] | LIAO Y, CHEN J, BRANDT B W, et al. Identification and functional analysis of genome mutations in a fluoride-resistant Streptococcus mutans strain[J]. PLoS One, 2015, 10(4): e0122630. DOI:10.1371/journal.pone.0122630 |
| [9] | 卢春英.变形链球菌耐氟菌株全基因组测序[D].长春: 吉林大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10183-1014296052.htm |
| [10] | STOCKBRIDGE R B, LIM H H, OTTEN R, et al. Fluoride resistance and transport by riboswitch-controlled CLC antiporters[J]. Proc Natl Acad Sci USA, 2012, 109(38): 15289–15294. DOI:10.1073/pnas.1210896109 |
| [11] | 罗晓庆, 孙济宇, 王慧, 等. 铜绿假单胞菌外膜蛋白OprH原核表达载体的构建与表达[J]. 中国烧伤创疡杂志, 2014(3): 195–198. DOI:10.3969/j.issn.1001-0726.2014.03.006 |
| [12] | 王升智, 高波, 周智慧, 等. 重组大肠杆菌DH5α生长曲线的测定[J]. 黑龙江畜牧兽医, 2010(11): 96–98. |
| [13] | MURATA T, HANADA N. Contribution of chloride channel permease to fluoride resistance in Streptococcus mutans[J]. FEMS Microbiol Lett, 2016, 363(11): fnw101. DOI:10.1093/femsle/fnw101 |
| [14] | LI SS, SMITH K D, DAVIS J H, et al. Eukaryotic resistance to fluoride toxicity mediated by a widespread family of fluoride export proteins[J]. Proc Natl Acad Sci U S A, 2013, 110(47): 19018–19023. DOI:10.1073/pnas.1310439110 |
| [15] | ZHU LK, ZHANG ZM, LIANG JP. Fatty-acid profiles and expression of the fabM gene in a fluoride-resistant strain of Streptococcus mutans[J]. Arch Oral Biol, 2012, 57(1): 10–14. DOI:10.1016/j.archoralbio.2011.06.011 |
| [16] | ACCARDI A, PICOLLO A. CLC channels and transporters:Proteins with borderline personalities[J]. Biochim Biophys Acta, 2010, 1798(8): 1457–1464. DOI:10.1016/j.bbamem.2010.02.022 |
| [17] | LIAO Y, YANG JM, BRANDT B W, et al. Genetic loci associated with fluoride resistance in Streptococcus mutans[J]. Front Microbiol, 2018, 9: 3093. DOI:10.3389/fmicb.2018.03093 |
| [18] | LIAO Y, BRANDT B W, ZHANG M, et al. A single nucleotide change in the promoter mutp enhances fluoride resistance of Streptococcus mutans[J]. AntimicrobAgents Chemother, 2016, 60(12): 7509–7512. |
| [19] | 超博, 张红, 李文月, 等. 变形链球菌耐氟菌株耐酸相关基因ropA的克隆及蛋白表达[J]. 微生物学杂志, 2017, 37(4): 23–27. DOI:10.3969/j.issn.1005-7021.2017.04.004 |
| [20] | MEN X C, SHIBATA Y, TAKESHITA T, et al. Identification of anion channels responsible for fluoride resistance in oral streptococci[J]. PLoS One, 2016, 11(11): e0165900. DOI:10.1371/journal.pone.0165900 |
| [21] | JI C H, STOCKBRIDGE R B, MILLER C. Bacterial fluoride resistance, Fluc channels, and the weak acid accumulation effect[J]. J Gen Physiol, 2014, 144(3): 257–261. DOI:10.1085/jgp.201411243 |
| [22] | PICOLLO A, XU Y, JOHNER N, et al. Synergistic substrate binding determines the stoichiometry of transport of a prokaryotic H(+)/Cl(-) exchanger[J]. Nat Struct Mol Biol, 2012, 19(5): 525–531. DOI:10.1038/nsmb.2277 |
| [23] | CAI Y L, LIAO Y, BRANDT B W, et al. The fitness cost of fluoride resistance for different Streptococcus mutans strains in Biofilms[J]. Front Microbiol, 2017, 8: 1630. DOI:10.3389/fmicb.2017.01630 |
| [24] | LIAO Y, BRANDT B W, LI J Y, et al. Fluoride resistance in Streptococcus mutans:A mini review[J]. J Oral Microbiol, 2017, 9(1): 1344509. DOI:10.1080/20002297.2017.1344509 |
 2019, Vol. 45
2019, Vol. 45


