扩展功能
文章信息
- 穆天驰, 刘超, 富帅, 李启阳, 王馨梦, 肖建英
- MU Tianchi, LIU Chao, FU Shuai, LI Qiyang, WANG Xinmeng, XIAO Jianying
- 基础转录激活因子1蛋白在胃癌组织中的表达及其临床意义
- Expression of activator of basal transcription 1 in gastric cancer tissue and its clinical significance
- 吉林大学学报(医学版), 2019, 45(01): 94-99
- Journal of Jilin University (Medicine Edition), 2019, 45(01): 94-99
- 10.13481/j.1671-587x.20190118
-
文章历史
- 收稿日期: 2018-01-26
2. 锦州医科大学基础医学院发育生物学教研室, 辽宁 锦州 121001
2. Department of Developmental Biology, School of Basic Medical Sciences, Jinzhou Medical University, Jinzhou 121001, China
RNA聚合酶Ⅱ的转录调控是一个高度动态的细胞过程,需要一系列调节蛋白的协调相互作用。该过程的核心是TATA结合蛋白(TATA- binding protein, TBP),TBP是蛋白复合物转录因子ⅡD(transcription factor ⅡD, TFⅡD)的关键组成成分[1]。作为一种新的TBP结合蛋白,基础转录激活因子1(activator of basal transcription 1,ABT1)促进体外和哺乳动物细胞内RNA聚合酶(PolⅡ)介导的基础转录。ABT1在小鼠各组织中广泛表达,定位于细胞核和核仁,从酵母到哺乳动物细胞保守,提示ABT1在细胞中发挥基础性作用。酵母ABT1缺陷细胞无法生长,表明ABT1的重要性[2]。鼠ABT1是一种核蛋白,具有独特的酸性区域,在许多转录因子如融合表达转录因子VP16、酵母转录因子GAL4及GCN4和P53中作为转录激活域[3-8]。然而,ABT1在其他转录因子和DNA结合蛋白无已知的功能模体[9]。结肠腺瘤中TBP的表达增加,因此TBP表达增强可能是结肠癌发生发展过程中的早期事件[10]。然而,目前ABT1在实体瘤特别是胃癌组织中的表达状态尚未见报道。为了更好地理解ABT1的功能和作用机制,本研究通过免疫组织化学法,结合组织芯片技术,研究ABT1蛋白在胃癌发生发展、转移和预后中的潜在作用。
1 资料与方法 1.1 胃癌组织样本100例人胃腺癌组织芯片样本(hstma180su08)(直径1.5 mm,深度4.0μm)购自上海芯超生物科技有限公司的组织样本库(上海,http://www.superchip.com.cn/)。100例胃腺癌患者平均年龄为(64.45±6.03)岁(32~81岁),行原发性胃癌根治手术的时间为2000年7月—2007年4月,所有患者术前均未接受化疗或放疗,死于无关病因的患者和术后1个月内死亡的患者均未纳入本研究。所有病例的详细临床和病理资料,包括临床和病理TNM分期及总生存期(overall survival,OS)均可获得。另外80例胃腺癌患者癌旁组织芯片样本来自与其配对的胃癌组织。所有患者术后随访至2015年7月,均有详细和完整的临床病理资料。随访时间为8.2~9.0年。所有组织标本均进行组织学检查,采用美国癌症联合委员会(AJCC)分类(2010)/国际抗癌联盟(UICC)2002/TNM分类和分期系统。本组患者的临床病理参数见表 1。
| Clinicopathological parameter | n(η/%) |
| Gender | |
| Male | 64(64.00) |
| Female | 36(36.00) |
| Age(year) | |
| ≤65 | 50(51.02) |
| >65 | 48(48.98) |
| Tumor size (d/cm) | |
| ≤5 | 50(51.02) |
| >5 | 48(48.98) |
| Pathological grading | |
| Ⅱ | 15(15.00) |
| Ⅲ | 74(74.00) |
| Ⅳ | 11(11.00) |
| Primary tumor | |
| T1 | 8(8.08) |
| T2 | 11(11.11) |
| T3 | 66(66.67) |
| T4 | 14(14.14) |
| Regional lymph nodes | |
| N0 | 27(27.55) |
| N1 | 15(15.31) |
| N2 | 26(26.53) |
| N3 | 30(30.61) |
| Distant metastasis | |
| M0 | 91(91.92) |
| M1 | 8(8.08) |
| TNM | |
| TNM1 | 10(10.20) |
| TNM2 | 32(32.65) |
| TNM3 | 49(50.00) |
| TNM4 | 7(7.14) |
| Survival status | |
| Survive | 31(31.31) |
| Death | 68(68.69) |
采用标准免疫组织化学染色法分析ABT1蛋白表达。胃癌组织芯片在二甲苯、梯度酒精脱水,浸入EDTA抗原缓冲液和微波进行抗原修复,3%过氧化氢阻断内源性过氧化物酶活性和3%牛血清白蛋白培养封闭非特异性结合。兔多克隆anti-ABT1抗体(1:25, 美国Abcam公司)与组织芯片4℃过夜孵育。免疫过氧化物酶染色采用多用途HRP连接试剂盒(美国Dako公司), 采用病理切片扫描成像系统(美国Aperio公司)分析。由2名经验丰富的病理科医生对免疫组织化学染色结果进行独立评估。染色细胞的比例和染色程度作为半定量评价的标准。细胞染色百分比和染色强度评价均超过病理切片扫描成像系统的5个视野。阳性细胞的百分比评分分为0分(0%)、1分(1%~25%)、2分(26%~50%)、3分(51%~75%)和4分(76%~100%);染色强度分为阴性(0分)、弱(1分)、中度(2分)和强(3分)。每个样本的最终免疫反应评分是通过染色百分比和染色强度相乘来计算。最后的得分≤2分为低表达,而>2分为高表达[11-12]。
1.3 Kaplan-Meier生存曲线分析胃癌患者生存期生存期是指从手术日期到死亡的时间。Kaplan- Meier生存曲线分析使用在线资源(http://kmplot.com/),并按照Chun KH实验室的描述分析胃癌患者的生存期[13-14]
1.4 统计学分析采用Graphpad prism 5.0统计软件进行统计学分析。胃癌患者胃癌和癌旁组织中ABT1蛋白表达水平以x±s表示,组间比较采用配对Wilcoxon检验。ABT1蛋白表达水平与胃癌患者临床病理参数的相关性采用Pearson和Spearman’ s相关性分析。ABT1蛋白表达水平与胃癌患者生存期和预后的关系采用Kaplan-Meier生存分析法和Log-rank检验。以P < 0.05为差异有统计学意义。
2 结果 2.1 ABT1蛋白在胃癌组织和癌旁组织中的表达水平ABT1蛋白在胃癌组织和癌旁组织的细胞核和细胞质中均有表达(图 1,见插页三)。ABT1在胃癌组织中的表达水平(5.130±0.229)明显低于癌旁组织(5.938±0.262),差异有统计学意义(P=0.021)。随着胃癌病理分级的增加,ABT1蛋白在病理分级Ⅱ期、Ⅲ期和Ⅳ期胃癌组织中的表达水平(6.195±0.241、4.862±0.272和2.562±0.201)逐渐降低,组间比较差异有统计学意义(P < 0.01)。见图 2(插页三)。

|
| 图 1 ABT1蛋白在胃癌组织(A)和癌旁组织(B)中的表达(免疫组织化学,×400) Fig. 1 Expressions of ABT1 protein in gastric cancer tissue (A)and paracancerous tissue(B)(Immunohistochemistry, ×400) |
|
|

|
| A:Gastric cancer stage Ⅱ;B:Gastric cancer stage Ⅲ;C:Gastric cancer stage Ⅳ;D:Blank control with PBS instead of ABT1 antibody. 图 2 ABT1蛋白在不同病理分级胃癌组织中的表达(免疫组织化学,×400) Fig. 2 Expressions of ABT1 protein in gastric cancer tissue with different pathological grades(Immunohistochemistry, ×400) |
|
|
Spearman’ s相关分析显示:胃癌患者胃癌组织中ABT1蛋白表达水平与胃癌组织病理分级呈负相关关系(r=-0.224,P=0.026),即胃癌病理分级越高,ABT1蛋白表达水平越低,但胃癌组织中ABT1蛋白表达水平与胃癌患者性别、年龄、肿瘤大小、T分期、N分期、M分期和TNM分期无明显相关关系(P>0.05)。见表 2。
| Clinicopathological parameter | n | Expression of ABT1 protein | r | P | |
| Low | High | ||||
| Gender | -0.015 | 0.882 | |||
| Male | 64 | 8 | 56 | ||
| Female | 36 | 6 | 30 | ||
| Age(year) | 0.118 | 0.251 | |||
| ≤65 | 50 | 6 | 44 | ||
| >65 | 48 | 8 | 40 | ||
| Tumor size (d/cm) | -0.132 | 0.198 | |||
| ≤5 | 50 | 4 | 46 | ||
| >5 | 48 | 10 | 38 | ||
| Pathological grading | -0.224 | 0.026 | |||
| Ⅱ | 15 | 0 | 15 | ||
| Ⅲ | 74 | 9 | 65 | ||
| Ⅳ | 11 | 5 | 6 | ||
| Primary tumor | 0.048 | 0.636 | |||
| T1 | 8 | 1 | 7 | ||
| T2 | 11 | 0 | 11 | ||
| T3 | 66 | 10 | 56 | ||
| T4 | 14 | 2 | 12 | ||
| Regional lymph node | -0.025 | 0.807 | |||
| N0 | 27 | 4 | 23 | ||
| N1 | 15 | 1 | 14 | ||
| N2 | 26 | 3 | 23 | ||
| N3 | 30 | 5 | 25 | ||
| Distant metastasis | 0.066 | 0.519 | |||
| M0 | 91 | 13 | 78 | ||
| M1 | 8 | 1 | 7 | ||
| TNM staging | 0.007 | 0.946 | |||
| TNM1 | 10 | 0 | 10 | ||
| TNM2 | 32 | 6 | 26 | ||
| TNM3 | 49 | 7 | 42 | ||
| TNM4 | 7 | 1 | 6 | ||
首先,采用Kaplan-Meier生存曲线分析预测了876例患者(来源:Kaplan-Meier plotter网站)胃癌组织中ABT1蛋白表达水平与总生存期的关系(图 3A),Log-rank检验结果显示:ABT1低表达的胃癌患者预后相对较差,而ABT1高表达的胃癌患者生存期较长(HR=1.483,P < 0.01)。采用Kaplan-Meier生存曲线分析本研究中100例胃癌患者胃癌组织中ABT1蛋白表达水平与总生存期的关系(图 3B),Log-rank检验结果显示:ABT1高表达的胃癌患者预后较好,ABT1蛋白表达水平与患者生存期呈正相关关系(HR=2.411,P=0.027)。
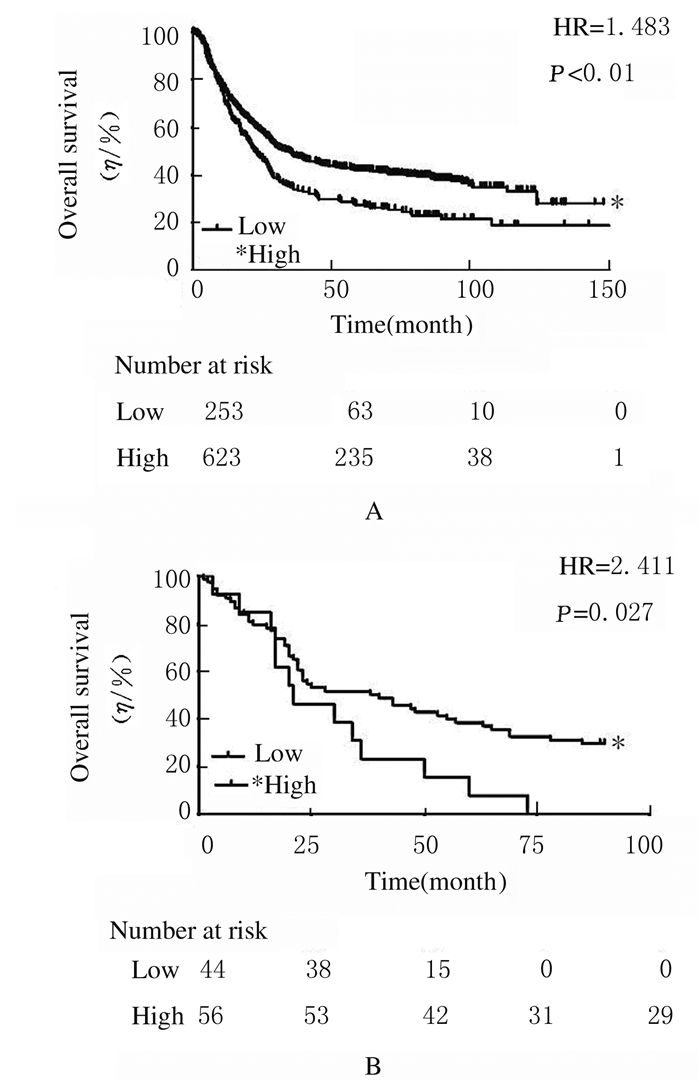
|
| A:Kaplan-Meier survival analysis and Log-rank statistical analysis of the relationship between the ABT1 protein expression and the overall survival in 876 patients with gastric cancer;B:Kaplan-Meier survival analysis and Log-rank statistical analysis of the relationship between the expression of ABT1 protein expression and the overall survival of 100 patients with gastric cancer. 图 3 ABT1蛋白表达水平与胃癌患者预后的相关性分析 Fig. 3 Correlation analysis between ABT1 protein expression levels and prognosis of gastric cancer patients |
|
|
胃癌是中国癌症相关死亡的主要原因之一。到目前为止,手术切除胃原发肿瘤仍是最有效的治疗方法。然而,只有不到一半的胃癌患者在早期被诊断,并接受外科治疗[15-19]。因此,生物标志物的不断发展将有助于预测胃癌患者的预后和对治疗的反应。众所周知,TBP可以直接结合到抑癌基因p53,而这种相互作用的生物学意义尚不明确[8, 20]。鉴于ABT1是一种新的TBP结合蛋白,因此分析ABT1在不同类型癌症中的表达特性有重要意义。
为了研究ABT1蛋白表达与胃癌发生发展、转移和预后的关系,本研究选择了100例临床信息丰富带有随访信息的胃癌病例,采用免疫组织化学技术和生物信息学及统计分析方法研究ABT1蛋白在胃癌组织中的表达和临床意义。本研究结果显示:ABT1蛋白在细胞核和细胞质中均有表达,且在胃癌组织中的表达水平明显低于癌旁组织。ABT1的主要生物学功能定位于细胞核中[2],但在胃癌细胞胞浆中也呈强阳性表达,因此ABT1在细胞质中表达定位可能在胃癌发生发展中发挥重要的生物学功能。本研究结果显示:胃癌组织中ABT1蛋白表达水平与胃癌患者的病理分级呈负相关关系,与Ⅱ期胃癌比较,在Ⅲ期和Ⅳ期胃癌组织中ABT1表达水平明显降低,提示ABT1可能在胃癌发生中起到抑制作用;细胞中ABT1表达水平与胃癌病理分级之间的反比关系,提示ABT1低表达参与肿瘤的进程,并可能通过未知的机制调节细胞增殖。因此,细胞中ABT1表达水平与胃癌患者病理分级之间的负相关关系可能提示ABT1在肿瘤进程中发挥抗肿瘤作用。然而,本研究尚未发现胃癌患者其他相关的临床或病理参数与ABT1表达有关联。
为了探讨ABT1是否为胃癌预后不良的预测因子,本文作者首先利用Kaplan-Meier plotter网站分析了876例胃癌患者ABT1表达与预后生存的关系。Kaplan Meier plotter能够用10 461种癌症样本来评估54675种基因对生存的影响,其中包括5143例乳腺癌、1816例卵巢癌、2437例肺癌和1065例胃癌患者,平均随访时间分别为69、40、49和33个月[21]。本研究结果表明:ABT1高表达胃癌患者的生存率高于ABT1低表达胃癌患者,即ABT1低表达与胃癌患者的不良预后呈正相关关系。在生物信息学分析的基础上,本文作者利用Kaplan Meier法分析了本研究中100例胃癌患者ABT1表达水平与预后因素之间的相关性,总生存率分析结果显示:ABT1高表达胃癌患者拥有更长总生存期,与网站的预测分析结果一致。
综上所述,ABT1的功能和调节作用尚未完全清楚,TBP结合蛋白的功能在胃癌中作为预后指标的作用也不明确,因此ABT1在胃癌中的异位表达可能具有不同的生物学作用和意义。本研究首次报道了ABT1在胃癌中可作为一个潜在的独立预后预测因子,但还应进行前瞻性临床试验进一步评估ABT1单独或联合其他标志物在不同类型癌症的预后评估或诊断中的价值。
| [1] | PEREIRA L A, KLEJMAN M P, TIMMERS H T. Roles for BTAF1 and Mot1p in dynamics of TATA-binding protein and regulation of RNA polymerase Ⅱ transcription[J]. Gene, 2003, 315: 1–13. DOI:10.1016/S0378-1119(03)00714-5 |
| [2] | ODA T, KAYUKAWA K, HAGIWARA H, et al. A novel TATA-binding protein-binding protein, ABT1, activates basal transcription and has a yeast homolog that is essential for growth[J]. Mol Cell Biol, 2000, 20(4): 1407–1418. DOI:10.1128/MCB.20.4.1407-1418.2000 |
| [3] | RAVARANI C H, CHALANCON G, BREKER M, et al. Affinity and competition for TBP are molecular determinants of gene expression noise[J]. Nat Commun, 2016, 7: 10417. DOI:10.1038/ncomms10417 |
| [4] | ARKOVA O, KUZNETSOV N, FEDOROVA O, et al. A real-time study of the interaction of TBP with a TATA box-containing duplex identical to an ancestral or minor allele of human gene LEP or TPI[J]. J Biomol Struct Dyn, 2016, 35(14): 3070–3081. |
| [5] | MAEDA R, TAMASHIRO H, TAKANO K, et al. TBP-like protein (TLP) disrupts the p53-MDM2 interaction and induces long-lasting p53 activation[J]. J Biol Chem, 2017, 292(8): 3201–3212. DOI:10.1074/jbc.M116.763318 |
| [6] | LOMVARDAS S, THANOS D. Nucleosome sliding via TBP DNA binding in vivo[J]. Cell, 2001, 106(6): 685–696. DOI:10.1016/S0092-8674(01)00490-1 |
| [7] | CHEN D, HINKLEY C S, HENRY RW, et al. TBP dynamics in living human cells:constitutive association of TBP with mitotic chromosomes[J]. Mol Biol Cell, 2002, 13(1): 276–284. DOI:10.1091/mbc.01-10-0523 |
| [8] | TORA L. A unified nomenclature for TATA box binding protein (TBP)-associated factors (TAFs) involved in RNA polymerase Ⅱ transcription[J]. Genes Dev, 2002, 16(6): 673–675. DOI:10.1101/gad.976402 |
| [9] | ODA T, FUKUDA A, HAGIWARA H, et al. ABT1-associated protein (ABTAP), a novel nuclear protein conserved from yeast to mammals, represses transcriptional activation by ABT1[J]. J Cell Biochem, 2004, 93(4): 788–806. DOI:10.1002/(ISSN)1097-4644 |
| [10] | JOHNSON S A S, LIN J J, WALKEY C J, et al. Elevated TATA-binding protein expression drives vascular endothelial growth factor expression in colon cancer[J]. Oncotarget, 2017, 8(30): 48832–48845. |
| [11] | YAN J, WEI Q, JIAN W, et al. IMP3 predicts invasion and prognosis in human lung adenocarcinoma[J]. Lung, 2016, 194(1): 137–146. DOI:10.1007/s00408-015-9829-0 |
| [12] | CHEN W, GUO W, LI M, et al. Upregulation of cleavage and polyadenylation specific factor 4 in lung adenocarcinoma and its critical role for cancer cell survival and proliferation[J]. PLoS ONE, 2013, 8(12): e82728. DOI:10.1371/journal.pone.0082728 |
| [13] | KIM H Y, CHO Y, KANG H, et al. Targeting the WEE1 kinase as a molecular targeted therapy for gastric cancer[J]. Oncotarget, 2016, 7(31): 49902–49916. |
| [14] | KIM S J, HWANG J A, RO J Y, et al. Galectin-7 is epigenetically-regulated tumor suppressor in gastric cancer[J]. Oncotarget, 2013, 4(9): 1461–1471. |
| [15] | GARATTINI S K, BASILE D, CATTANEO M, et al. Molecular classifications of gastric cancers:Novel insights and possible future applications[J]. World J Gastrointest Oncol, 2017, 9(5): 194–208. DOI:10.4251/wjgo.v9.i5.194 |
| [16] | WANG Z, LIU C. Lgr5-positive cells are cancer-stem-cell-like cells in gastric cancer[J]. Cell Physiol Biochem, 2015, 36(6): 2447–2455. DOI:10.1159/000430205 |
| [17] | LI Y, YANG X, WU Y, et al. B7-H3 promotes gastric cancer cell migration and invasion[J]. Oncotarget, 2017, 8(42): 71725–71735. |
| [18] | ZHOU Y, ZHANG G J, WANG J, et al. Current status of lymph node micrometastasis in gastric cancer[J]. Oncotarget, 2017, 8(31): 51963–51969. |
| [19] | EGELAND E V, FLATMARK K, NESLAND J M, et al. Expression and clinical significance of Wee1 in colorectal cancer[J]. Tumour Biol, 2016, 37(9): 12133–12140. DOI:10.1007/s13277-016-5081-3 |
| [20] | CRIGHTON D, WOIWODE A, ZHANG C, et al. p53 represses RNA polymerase Ⅲ transcription by targeting TBP and inhibiting promoter occupancy by TFⅢB[J]. EMBO J, 2003, 22(11): 2810–2820. DOI:10.1093/emboj/cdg265 |
| [21] | SZÁSZ A M, LÁNCZKY A, NAGY Á, et al. Cross-validation of survival associated biomarkers in gastric cancer using transcriptomic data of 1065 patients[J]. Oncotarget, 2016, 7(31): 49322–49333. |
 2019, Vol. 45
2019, Vol. 45


