扩展功能
文章信息
- 陈炳鹏, 任明, 李容杭, 韩青, 王金成
- CHEN Bingpeng, REN Ming, LI Ronghang, HAN Qing, WANG Jincheng
- 基因芯片技术检测NOB1对骨肉瘤细胞相关基因的调控作用
- Regulatory effect of NOB1 on osteosarcoma cell-related genes detected by gene microarray technique
- 吉林大学学报(医学版), 2018, 44(05): 929-934
- Journal of Jilin University (Medicine Edition), 2018, 44(05): 929-934
- 10.13481/j.1671-587x.20180508
-
文章历史
- 收稿日期: 2018-03-26
骨肉瘤是一种最常见的组织学原发性骨肿瘤,常发于青少年人群中。尽管近年来包括化疗和手术治疗在内的现代多学科治疗方法都在不断的发展,骨肉瘤患者的5年生存率仍然只是维持在60%~70%[1-2]。最近随着肿瘤相关信号通路和新型基因靶向治疗的研究进展,为预防和治疗骨肉瘤提供了新的策略和途径。目前与骨肉瘤发生发展相关的关键基因包括FAS、Ezrin、CXCR4、COX-2、HER2、以及NOTCH和HES1等[3-5]。但是仍缺乏一个能准确预测骨肉瘤进展、转移和对化疗反应性的标志性分子。NOB1蛋白是26S蛋白酶体的一个亚单位,在蛋白酶功能和RNA代谢过程中发挥重要作用。研究[6-10]显示:NOB1在多种肿瘤中异常表达且发挥重要功能。但是, NOB1在骨肉瘤中的功能及其分子机制尚不十分清楚。本研究应用慢病毒介导的短发夹RNA(short hourpin RNA, shRNA)干扰技术建立抑制NOB1 mRNA表达的Lv-shNOB1-U2OS骨肉瘤细胞株,利用mRNA表达谱芯片对Lv-shCon-U2OS组和Lv-shNOB1-U2OS组细胞分别行表达谱分析,筛选出可能受NOB1调控的基因,并对差异基因进行相关分析,为进一步研究NOB1在骨肉瘤中的功能奠定分子基础。
1 材料与方法 1.1 细胞株、主要试剂和仪器Lv-shNOB1-U2OS细胞株及其相应的对照细胞Lv-shCon-U2OS细胞株由本课题组建立。RNA LabChip® kits、RNA 6000 nano marker、表达谱芯片配套试剂盒、Gene Expression Hybridization Kit和Gene Expression Wash Buffer Kit均购自美国Agilent Technologies公司,RPMI 1640培养基购自美国Gibco公司, 胰蛋白酶和胎牛血清购自美国Invitrogen公司。NanoDrop ND-2000和载物片洗缸为美国Thermo公司产品,Agilent Bioanalyzer 2100生物分析仪、杂交炉和Agilent Scanner G2505C为美国Agilent Technologies公司产品。Feature Extraction软件version 10.7.1.1和Genespring软件version 12.5为美国Agilen Technologies公司产品。
1.2 定量PCR法检测人NOB1基因的干扰效率提取感染重组病毒Lv-shNOB1-U2OS细胞、感染空病毒Lv-shCon-U2OS细胞、未感染病毒的Saos-2细胞和U2OS细胞的总RNA,以总RNA为模板,β-actin为内参照,用定量PCR法检测细胞中NOB1 mRNA表达变化。Real-time PCR引物采用Primer Premier 5.0设计,送上海生工公司合成。引物序列如下:NOB1,上游引物5′-GAAAGAACAACGCCCTGGAG-3′;下游引物5′-CAGCCTTGAGATGACCTAAGC-3′;β-actin,上游引物5′-GTGGACATCCGCAAAGAC-3′;下游引物5′-AAAGGGTGTAACGCAACTA-3′。
1.3 RNA提取及逆转录当细胞生长至95%融合度后,用无菌细胞刮分别刮取实验组和对照组的贴壁细胞;按照Trizol reagent试剂盒说明书提取RNA;应用QIAGEN RNeasy Mini Kit纯化质检合格的总RNA;按照标准流程应用Agilent表达谱芯片配套试剂盒对纯化后的总RNA中的mRNA进行放大和标记。
1.4 基因芯片的制备按Gene Expression Hybridization Kit说明书,采用如下比例配置混合液:Cy3标记的线性扩增的cRNA样品5 μg,10× Gene Expression Blocking Agent50 μL,25×Fragmentation Buffer10 μL, Nuclease-free water补齐体系总体积至250 μL;60℃水浴孵育混合液30 min,冰上淬冷1 min;加入250 μL 2× Hi-RPM Hybridization Buffer终止RNA裂解,吹打混匀。然后13 000×g离心1 min,将样品置于冰上并立刻上样;在载玻片的上样孔中滴加490 μL样品,盖上芯片并将芯片置于杂交盒中,杂交炉中65℃滚动杂交17 h。应用基因表达洗涤缓冲液试剂盒(Gene Expression Wash Buffer Kit)对芯片进行洗涤。
1.5 芯片扫描所用芯片为Agilent Human Gene Expression (8*60K,Design ID:039494)。洗涤后的芯片通过Agilent Scanner G2505C进行扫描。染色通道为Green,扫描分辨率3 μm, 20bit。采用配套软件Feature Extraction(version 10.7.1.1)读取原始数据,采用Genespring软件(version 12.5)以及Quantile算法进行归一化处理,得到每个基因在各样品中的相对含量。
1.6 数据分析和统计学方法应用GenBank(http://www.ncbi.nlm.nih.gov/genbank/)全库对基因芯片所得表达谱数据进行注释,对应蛋白产物注释应用Swissport/UniProt(http://web.expasy.org/docs/swiss-prot_guideline.html)全库。Lv-shNOB1-U2OS组和Lv-shCon-U2OS组两两配对做3个生物学重复。差异基因的筛选采用SPSS 10.0软件进行配对t检验和SAM运算。首先对差异基因进行生物信息学数据库肿瘤基因GO注释,然后将差异基因向Gene Ontology数据库(http://www.geneontology.org/)的GO条目进行映射,从而得到各GO条目的差异基因数。进一步利用SPSS10.0软件进行超几何检验,找出差异基因明显富集的GO条目。同样,采用与GO富集相似的方法对通路(pathway)进行富集,将差异基因向京都基因与基因组百科全书(KEGG)数据库(http://www.genome.jp/kegg/)中的通路进行映射,再采用超几何检验找出差异基因明显富集的通路条目。
2 结果 2.1 NOB1干扰效率检测采用q-PCR法检测慢病毒载体干扰效率。在骨肉瘤细胞系U2OS中,NOB1 shRNA均能有效抑制NOB1的表达。与Lv-shCon组和Con组比较,干扰效率均大于50%,而Lv-shCon组与Con组之间比较差异无统计学意义(P>0.05)。通过Lv-shNOB1慢病毒载体转染成功构建了Lv-shNOB1-U2OS细胞株及相应的对照Lv-shCon-U20s细胞株。
2.2 差异表达谱分析在筛选差异基因之前,先进行探针过滤,用于比较的每组样本中至少有一组100%标记为Detected的探针留下进行后续分析。利用差异倍数Fold change值进行筛选,标准为Fold change值≥2.0。本研究对归一化后的芯片数据进行了差异表达分析,单荧光芯片的原始数据经过标准化处理,转化为log以2为底的对数后,在一个二维直角坐标系平面中,绘制散点图。芯片数据的散点图常用于评估2组数据总体分布集中趋势。
从基因芯片代表性荧光杂交信号散点图(图 1)可看出,在2个实验组间存在大量基因mRNA表达差异高于2倍,显示出极大的表达谱改变。NOB1干扰后共有792个基因mRNA表达水平上调以及1059个基因mRNA表达水平下调,总共差异变化基因为1851个。
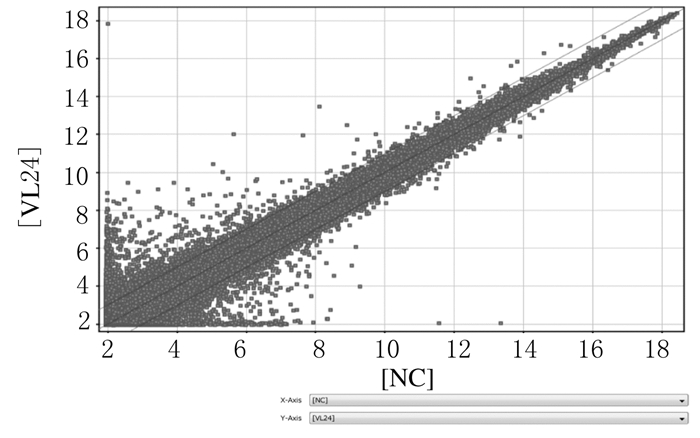
|
| 图 1 基因芯片数据散点图 Figure 1 Scatter plots of gene chip data |
|
|
本研究设定超几何检验所得P值小于0.05作为差异基因明显富集的筛选指标。分别从细胞位置(cellular component)、分子功能(molecular function)以及生化过程(biological_process)3个一级条目对所有差异基因进行富集分析。
从细胞位置条目的富集程度(图 2)来看,差异基因编码产物蛋白主要分布于细胞质膜,占56.9%;分布于细胞外区域的占39.4%;分布于细胞外空间占20%;其他所分布区域按比例由大到小依次为蛋白质的细胞外基质、细胞外间质、细胞质膜、基底膜、胶原蛋白、核小体、血小板和肌球蛋白。

|
| 图 2 差异基因的亚细胞位置分布 Figure 2 Subcellular location distribution of differential genes |
|
|
从分子功能条目的富集程度(图 3)来看,差异基因编码产物蛋白主要功能为钙离子结合相关功能,占总差异基因的22%;其次功能为转运子活性,占总差异基因的9.2%;第3位功能为肌动蛋白结合活性,占总差异基因的8.7%。

|
| 图 3 差异基因的分子功能分布 Figure 3 Distribution of molecular function of differential genes |
|
|
从生化过程条目的富集程度(图 4)来看,差异基因编码产物蛋白主要参与过程为信号转导,占总差异基因的18.7%;其次参与过程为多种细胞器生成,占总差异基因的15.6%;第3位过程为细胞黏附过程,占总差异基因的10.2%。

|
| 图 4 差异基因在生物学进程中的富集情况 Figure 4 Enrichment of differential genes in biological process |
|
|
利用KEGG数据库对差异基因进行Pathway分析,找到富集差异基因的Pathway条目,寻找不同样品的差异基因可能与哪些细胞通路的改变有关。对于KEGG数据库,满足条件的差异基因富集条目有上百个之多,这可能是由于KEGG数据库收录了大量正常代谢和生化途径相关通路,这些通路涵盖基因数过多造成的。在此本文作者选择性的列出富集程度前12的通路。由图 5可知,差异基因在系统性红斑狼疮相关通路(systemic lupus erythematosus)所占总差异基因的比例最大,为24.5%,其次主要分布的通路为细胞因子-细胞因子受体相互作用通路(cytokine-cytokine receptor interaction)和细胞的焦点黏连(focal Adhesion)信号通路,分别占总差异基因的19.4%和16.3%。

|
| 图 5 KEGG数据库中差异基因富集程度通路 Figure 5 Enrichment degree pathways of differential genes in KEGG database |
|
|
NOB1基因Nob1p(Nin one binding protein)蛋白最早通过利用酵母双杂交方法在酵母中鉴定为一种锌带蛋白,其在溶酶体的组装和成熟以及对于核糖体RNA (ribonsomal RNA, rRNA)的形成过程中发挥着重要的作用[11-12]。NOB1是一个与细胞周期及转录相关的基因,虽然关于NOBl在肿瘤方面的研究还很有限,其在肿瘤中的功能机制也并未明确,鉴于NOB1在UPP途径和核糖体合成过程中的重要作用,NOB1基因在肿瘤发生发展过程中的作用越来越受到重视[13-15]。
本研究利用mRNA表达谱芯片对Lv-shCon-U2OS组和Lv-shNOB1-U2OS组细胞分别行表达谱分析,结果显示:NOB1干扰后共有792个基因mRNA表达水平上调,1059个基因mRNA表达水平下调,总共差异变化基因为1 851个。上调基因数和下调基因数相仿,整体差异基因数较多,说明NOB1干扰后影响的表达谱范围较广,预示NOB1在骨肉瘤细胞中可能具有重要的功能。本研究分别从细胞位置、分子功能以及生化过程3个一级条目对所有差异基因进行GO分析。细胞质膜是指包围在细胞表面的一层极薄的膜,主要由膜脂和膜蛋白所组成。质膜的基本作用是维护细胞内微环境的相对稳定,并参与同外界环境进行物质交换、能量和信息传递。另外,细胞质膜在肿瘤细胞的生存、生长、分裂和分化中起重要作用。细胞受体能与细胞外专一信号分子(配体)结合引起细胞反应的蛋白质,可分为细胞表面受体和细胞内受体。受体与配体结合即发生分子构象变化,从而引起细胞反应,如介导细胞间信号转导、细胞间黏合和细胞胞吞等细胞过程[16]。细胞信号转导是指细胞外因子通过与受体(膜受体或核受体)结合,引发细胞内的一系列生物化学反应以及蛋白间相互作用,直至细胞生理反应所需基因开始表达、各种生物学效应形成的过程。细胞信号转导是一个十分复杂的网络系统,对肿瘤细胞的各个方面都有极其重要的影响[17]。
本研究利用KEGG数据库对差异基因进行Pathway分析,结果显示:差异基因在系统性红斑狼疮相关通路所占总差异基因的比例最大;其次主要分布的通路为细胞因子-细胞因子受体相互作用通路和细胞的焦点粘连信号通路。其中,细胞的许多重要功能均由细胞因子介导,细胞因子通过特定的细胞表面受体激活相关信号通路,并在细胞内发挥作用以协调生物反应,其中比较热门的相关通路有JAK/STAT通路(Janus kinase/signal transducer and activator of transcription)[18-19]、MAPK通路(mitogen-activated protein kinases)[20]和核因子κB(NF-κB)信号通路等。JAK/STAT通路、MAPK通路以及NF-κB信号通路作为应激反应通路,对胞内或胞外的多种刺激做出反应,调节细胞内增殖、分化、凋亡以及炎症反应等多种重要生物学过程的发生[21-22]。
综上所述,本研究阐明了NOB1在骨肉瘤中的重要作用,提示NOB1可能参与骨肉瘤的发生发展和转移过程。本研究为骨肉瘤的基因靶向治疗提供了新的具有潜力的治疗靶点。
| [1] | Marina N, Gebhardt M, Teot L, et al. Biology and therapeutic advances for pediatric osteosarcoma[J]. Oncologist, 2004, 9(4): 422–441. DOI:10.1634/theoncologist.9-4-422 |
| [2] | PosthumaDeBoer J, Witlox MA, Kaspers GJ, et al. Molecular alterations as target for therapy in metastatic osteosarcoma:a review of literature[J]. Clin Exp Metastasis, 2011, 28(5): 493–503. DOI:10.1007/s10585-011-9384-x |
| [3] | Kim C, Shin E, Hong S, et al. Clinical value of ezrin expression in primary osteosarcoma[J]. Cancer Res Treat, 2009, 41(3): 138–144. DOI:10.4143/crt.2009.41.3.138 |
| [4] | Zhao Q, Wang C, Zhu J, et al. RNAi-mediated knockdown of cyclooxygenase2 inhibits the growth, invasion and migration of SaOS2 human osteosarcoma cells:a case control study[J]. J Exp Clin Cancer Res, 2011, 30: 26. DOI:10.1186/1756-9966-30-26 |
| [5] | Hughes DP. How the NOTCH pathway contributes to the ability of osteosarcoma cells to metastasize[J]. Cancer Treat Res, 2009, 152: 479–496. DOI:10.1007/978-1-4419-0284-9 |
| [6] | Dai H, Hou K, Cai Z, et al. Low-level miR-646 in colorectal cancer inhibits cell proliferation and migration by targeting NOB1 expression[J]. Oncol Lett, 2017, 14(6): 6708–6714. |
| [7] | Lin Y, Xu T, Zhou S, et al. MicroRNA-363 inhibits ovarian cancer progression by inhibiting NOB1[J]. Oncotarget, 2017, 8(60): 101649–101658. |
| [8] | Kong R, Liu W, Guo Y, et al. Inhibition of NOB1 by microRNA-330-5p overexpression represses cell growth of non-small cell lung cancer[J]. Oncol Rep, 2017, 38(4): 2572–2580. DOI:10.3892/or.2017.5927 |
| [9] | Liu K, Chen H, You Q, et al. miR-145 inhibits human non-small-cell lung cancer growth by dual-targeting RIOK2 and NOB1[J]. Int J Oncol, 2018, 3. DOI:10.3892/ijo.2018.4393 |
| [10] | Lin Y, Xu T, Teng H, et al. Anticancer activity of NOB1-targeted shRNA combination with TRAIL in epithelial ovarian cancer cells[J]. Int J Clin Exp Pathol, 2015, 8(9): 10061–10071. |
| [11] | Gao X, Wang J, Bai W, et al. NOB1 silencing inhibits the growth and metastasis of laryngeal cancer cells through the regulation of JNK signaling pathway[J]. Oncol Rep, 2016, 35(6): 3313–3320. DOI:10.3892/or.2016.4707 |
| [12] | Niinaka Y, Harada K, Fujimuro M, et al. Silencing of autocrine motility factor induces mesenchymal-to-epithelial transition and suppression of osteosarcoma pulmonary metastasis[J]. Cancer Res, 2010, 70(22): 9483–9493. DOI:10.1158/0008-5472.CAN-09-3880 |
| [13] | Luo L, Wang Y, Yin Y, et al. Effects of NOB1 on the pathogenesis of osteosarcoma and its expression on the chemosensitivity to cisplatin[J]. Oncol Lett, 2018, 15(3): 3548–3551. |
| [14] | Huang P, Xi J, Liu S. MiR-139-3p induces cell apoptosis and inhibits metastasis of cervical cancer by targeting NOB1[J]. Biomed Pharmacother, 2016, 83: 850–856. DOI:10.1016/j.biopha.2016.07.050 |
| [15] | Ma Y, Zhou W, Hong L, et al. Establishment of a novel monoclonal antibody L6 specific to NOB1[J]. Monoclon Antib Immunodiagn Immunother, 2016, 35(2): 100–103. DOI:10.1089/mab.2015.0065 |
| [16] | Lingwood D, Binnington B, Róg T, et al. Cholesterol modulates glycolipid conformation and receptor activity[J]. Nat Chem Biol, 2011, 7(5): 260–262. DOI:10.1038/nchembio.551 |
| [17] | Teschendorff AE, Severini S. Increased entropy of signal transduction in the cancer metastasis phenotype[J]. BMC Syst Biol, 2010, 4: 104. DOI:10.1186/1752-0509-4-104 |
| [18] | Darnell JE Jr. The JAK-STAT pathway at 20[J]. JAKSTAT, 2012, 1(1): 2. DOI:10.4161/jkst.18726 |
| [19] | 张朝亚, 赵相轩, 卢再鸣, 等. 索拉非尼诱导肝癌细胞凋亡信号通路的研究进展[J]. 临床肝胆病杂志, 2016, 32(4): 816–820. DOI:10.3969/j.issn.1001-5256.2016.04.048 |
| [20] | Xie C, Kang J, Ferguson ME, et al. Blueberries reduce pro-inflammatory cytokine TNF-α and IL-6 production in mouse macrophages by inhibiting NF-κB activation and the MAPK pathway[J]. Mol Nutr Food Res, 2011, 55(10): 1587–1591. DOI:10.1002/mnfr.201100344 |
| [21] | Quintás-Cardama A, Verstovsek S. Molecular pathways:Jak/STAT pathway:mutations, inhibitors, and resistance[J]. Clin Cancer Res, 2013, 19(8): 1933–1940. DOI:10.1158/1078-0432.CCR-12-0284 |
| [22] | Ghosh G, Wang VY, Huang DB, et al. NF-κB regulation:lessons from structures[J]. Immunol Rev, 2012, 246(1): 36–58. DOI:10.1111/j.1600-065X.2012.01097.x |
 2018, Vol. 44
2018, Vol. 44


