扩展功能
文章信息
- 严彩芳, 赵红庆, 高源, 朱兵清, 徐丽, 高朝辉, 邵祝军
- Yan Caifang, Zhao Hongqing, Gao Yuan, Zhu Bingqing, Xu Li, Gao Chaohui, Shao Zhujun
- 中国侵袭性肺炎链球菌19A、19F血清型的基因分型分析
- Genotyping of invasive Streptococcus pneumoniae serotype 19A and 19F in China
- 疾病监测, 2018, 33(2): 168-171
- Disease Surveillance, 2018, 33(2): 168-171
- 10.3784/j.issn.1003-9961.2018.02.012
-
文章历史
- 收稿日期:2017-11-29
2. 中国疾病预防控制中心传染病预防控制所, 传染病预防控制国家重点实验室, 北京 102206
2. State Key Laboratory of Infectious Disease Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing, 102206, China
肺炎链球菌(Streptococcus pneumoniae,Spn)分为48个血清群,90多个血清型[1],可引起侵袭性肺炎球菌性疾病(invasive pneumococcal disease,IPD),包括菌血症、菌血症性肺炎和脑膜炎等,在<5岁儿童和老年人群中具有严重的疾病负担[2]。肺炎球菌疫苗的应用可有效预防IPD,但也会造成肺炎链球菌血清型的替换[3-7],导致疫苗免疫逃逸。19A和19F血清型的替换备受关注。有研究表明,通过多位点序列分型(multilocus sequence type,MLST)分析,CC320/271的ST型克隆群在侵袭性肺炎链球菌19A和19F血清型中,可发生荚膜的转换导致血清型的替换[8-9]。ST分型是针对肺炎链球菌7个管家基因进行的基因分型,当不同ST型的7个管家基因中有4个以上等位基因位点相同时,可将这些ST型聚类为一个克隆群(clonal complex,CC)。同一个CC中的菌株可以有不同的ST型,也可以有不同的血清型,等位基因的变化,可以作为研究菌株基因变异的根据。我国关于侵袭性肺炎链球菌19A和19F基因型研究的数据较少,本研究针对侵袭性肺炎链球菌19A和19F血清型菌株进行多位点序列分型和聚类分析,以掌握肺炎链球菌荚膜转换和血清型替换的趋势,有助于肺炎球菌疫苗应用效果的评价。
1 材料与方法 1.1 菌株信息本研究中57株侵袭性肺炎链球菌分离自肺炎、脑膜炎患者的脑脊液、血液等无菌部位,来自全国各疾病预防控制中心(CDC)细菌性疫苗可预防疾病监测网络实验室。
1.2 菌株鉴定所有菌株均经革兰染色(革兰染液试剂盒,法国生物梅里埃公司)、奥普托欣敏感性实验(DDI Optochin,英国OXOID公司)以及胆汁溶解实验(脱氧胆酸钠,美国Sigma公司)进行生化鉴定,采用肺炎链球菌血清荚膜肿胀实验(肺炎链球菌分型诊断血清,丹麦Statens Serum Institut公司)和血清型特异的PCR分型方法确定血清型。
1.3 MLST分析用Promega Wizard®基因组DNA纯化试剂盒提取肺炎链球菌基因组DNA,PCR扩增7个管家基因(aroE、gdh、gki、recP、spi、xpt、ddl),引物序列参照肺炎链球菌MLST网站(http://spneumoniae.mlst.net/)所提供序列,见表 1。PCR反应体系为30 μl,其中15 μl GoTaq® Green Master Mix(美国Promega公司),正向引物(10 μmol/L)1.5 μl,反向引物(10 μmol/L)1.5 μl,DNA模板2 μl,无菌双蒸水10 μl。PCR反应条件为94 ℃预变性3 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸30 s,30个循环;72 ℃再延伸5 min。扩增引物即为测序引物,引物合成及基因测序由北京天一辉远生物科技有限公司完成。使用DNAStar软件进行序列分析,序列提交肺炎链球菌MLST网站,获得管家基因的等位基因号及ST型。采用BioNumerics软件处理数据并进行ST型聚类分析。
| 基因 | 引物名称 | 引物序列(5’~3’) |
| aroE | aroE-F | GCC TTT GAG GCG ACA GC |
| aroE-R | TGC AGT TCA (G/A)AA ACA T(A/T)T TCT AA | |
| gdh | gdh-F | ATG GAC AAA CCA GC(G/A/T/C) AG(C/T) TT |
| gdh-F | GCT TGA GGT CCC AT(G/A) CT(G/A/T/C) CC | |
| gki | gki-F | GGC ATT GGA ATG GGA TCA CC |
| gki-R | TCT CCC GCA GCT GAC AC | |
| recP | recP-F | GCC AAC TCA GGT CAT CCA GG |
| recP-R | TGC AAC CGT AGC ATT GTA AC | |
| spi | spi-F | TTA TTC CTC CTG ATT CTG TC |
| spi-R | GTG ATT GGC CAG AAG CGG AA | |
| xpt | xpt-F | TTA TTA GAA GAG CGC ATC CT |
| xpt-R | AGA TCT GCC TCC TTA AAT AC | |
| ddl | ddl-F | TGC (C/T)CA AGT TCC TTA TGT GG |
| ddl-R | CAC TGG GT(G/A) AAA CC(A/T) GGC AT |
经生化鉴定以及血清分型,侵袭性肺炎链球菌19A血清型菌株有35株,19F血清型菌株22株。
2.2 侵袭性肺炎链球菌19A、19F血清型菌株的ST型分布35株侵袭性肺炎链球菌19A血清型菌株共有6种ST型,其中ST320(29株,82.9%),ST876(2株,5.7%),ST4560(1株,2.9%),ST9230(1株,2.9%),ST7759(1株,2.9%),ST6429(1株,2.9%)。22株侵袭性肺炎链球菌19F血清型菌株共有7种ST型,其中ST271(14株,63.6%),ST236(1株,4.5%),ST320(2株,9.1%),ST1937(2株,9.1%),ST6993(1株,4.5%),ST9236(1株,4.5%),ST9240(1株,4.5%)。结果表明在侵袭性肺炎链球菌19A血清型菌株中,ST320为主要的ST型;在19F血清型菌株中,ST271为主要的ST型。
2.3 侵袭性肺炎链球菌19A、19F血清型的ST型聚类分析利用BioNumerics软件对19A和19F血清型肺炎链球菌ST数据进行分析,见图 1。
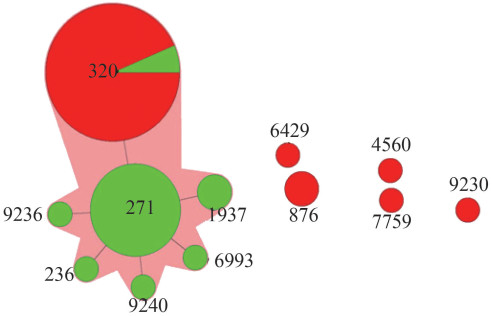
|
| 图 1 19A、19F血清型肺炎链球菌ST型的BioNumerics聚类分析 Figure 1 BioNumerics cluster analysis of STs of S. pneumoniae serotype 19A and 19F strains 注:红色部分代表 19A血清型肺炎链球菌菌株,绿色部分代表 19F血清型肺炎链球菌菌株,圆圈大小代表菌株数目多少 |
| |
聚类分析结果表明,ST320/271型作为优势ST型,两者的同源性较高。在MLST数据库中查询发现,ST320和ST271中6个管家基因(aroE、gdh、gki、recP、spi、xpt)的等位基因号相同,只有1个管家基因ddl的等位基因号不同,在ST320中ddl的等位基因号为1,而在ST271中ddl的等位基因号为26,提示ST320和ST271可能起源于同一个ST型。
3 讨论肺炎链球菌有众多血清型,但引起IPD的主要有20多个血清型。目前肺炎球菌疫苗主要包括两类:肺炎球菌多糖疫苗(PPV)和肺炎球菌多糖蛋白结合疫苗(PCV)[10-11],其中,PCV7疫苗已被PCV13疫苗所替代。PCV7疫苗中包含19F血清型,PCV13相比于PCV7增加了19A血清型,主要是基于美国及其他各国肺炎链球菌流行病学监测数据,发现19A导致的侵袭性肺炎球菌病例上升[9, 12-14]。
对本研究所收集的57株侵袭性肺炎链球菌19A和19F血清型菌株的BioNumerics聚类分析发现,19F血清型的肺炎链球菌不同ST型聚类集中,以ST271为中心,其余6种ST型均匀分布在ST271周围。在MLST数据库中查询发现,ST271与其他6种ST型均只有一个管家基因的等位基因号不同;相比较而言,19A血清型的肺炎链球菌不同ST型分布松散,与其主要基因型别ST320相比,ST4560、ST7759和ST9230的7个管家基因的等位基因位点均与其不同,表明肺炎链球菌19A血清型菌株的基因型更具有多变性和复杂性。
另外,本研究发现ST320/271为侵袭性肺炎链球菌19A和19F血清型菌株的主要基因型,ST271型全部为19F血清型菌株,19A血清型菌株中未检测到ST271。19A血清型肺炎链球菌主要型别为ST320,19F血清型菌株中也检测到2株ST320型别,表明如果有荚膜转换发生,ST320基因型的肺炎链球菌更易在19A和19F血清型中进行荚膜转换,可能与ST320的多药耐药(MDR)特点有关[15]。
有研究表明,CC320/271克隆群菌株最初出现于19F血清型肺炎链球菌中,后出现于19A血清型肺炎链球菌中,故认为19F和19A血清型肺炎链球菌之间发生了荚膜转换[8]。CC和ST是基因分型,而荚膜则与肺炎链球菌血清型相关,属于表型分型,根据荚膜多糖免疫化学特性的不同,将肺炎链球菌分为多个血清型。因此,研究认为CC320/271克隆群菌株的基因型并未发生改变,只是荚膜转换导致了19F血清型和19A血清型这一表型的变化。关于其转换机制,有研究认为PCV7应用产生的疫苗选择压力可能导致了这种转变[16],也有研究表明19A血清型菌株的增加可能与抗生素的选择压力有关[17]。
本研究发现19F菌株中的ST236,ST320,ST1937,ST6993,ST9236,ST9240均与ST271仅有1个等位基因位点的差异,除ST320型外,这些ST型都有可能在基因型不发生改变的情况下,发生荚膜转换而导致表型的改变,从而表现为19A血清型肺炎链球菌。
随着PCV13的应用,疫苗所覆盖血清型引起的疾病减少,某些ST型别会在选择压力下发生荚膜转换产生“疫苗逃避”。对此,我国在未来推广肺炎球菌疫苗的同时,对于19F与19A之间的荚膜转换和流行趋势应引起关注,为评价PCV13疫苗效果提供依据。
作者贡献:
严彩芳 ORCID:0000-0002-3844-3590
赵红庆 ORCID:0000-0002-4423-0612
严彩芳:负责文献收集,实验室检测工作,数据处理及论文撰写
赵红庆:参与文献收集,数据处理
高源、朱兵清、徐丽:数据处理
高朝辉、邵祝军:指导工作,参与论文修改
| [1] |
Marimón JM, Ercibengoa M, Santacatterina E, et al. Single-step multiplex PCR assay for determining 92 pneumococcal serotypes[J]. J Clin Microbiol, 2016, 54(8): 2197-2200. DOI:10.1128/JCM.01156-16 |
| [2] |
GBD 2015 Mortality and Causes of Death Collaborators. Global, regional, and national life expectancy, all-cause mortality, and cause-specific mortality for 249 causes of death, 1980-2015:a systematic analysis for the Global Burden of Disease Study 2015[J]. Lancet, 2016, 388(10053): 1459-1544. DOI:10.1016/S0140-6736(16)31012-1 |
| [3] |
Weinberger DM, Grant LR, Weatherholtz RC, et al. Relating pneumococcal carriage among children to disease rates among adults before and after the introduction of conjugate vaccines[J]. Am J Epidemiol, 2016, 183(11): 1055-1062. DOI:10.1093/aje/kwv283 |
| [4] |
Suga S, Chang B, Asada K, et al. Nationwide population-based surveillance of invasive pneumococcal disease in Japanese children:Effects of the seven-valent pneumococcal conjugate vaccine[J]. Vaccine, 2015, 33(45): 6054-6060. DOI:10.1016/j.vaccine.2015.07.069 |
| [5] |
Hicks LA, Harrison LH, Flannery B, et al. Incidence of pneumococcal disease due to non-pneumococcal conjugate vaccine (PCV7) serotypes in the United States during the era of widespread PCV7 vaccination, 1998-2004[J]. J Infect Dis, 2007, 196(9): 1346-1354. DOI:10.1086/521626 |
| [6] |
Gertz RE Jr., McEllistrem MC, Boxrud DJ, et al. Clonal distribution of invasive pneumococcal isolates from children and selected adults in the United States prior to 7-valent conjugate vaccine introduction[J]. J Clin Microbiol, 2003, 41(9): 4194-4216. DOI:10.1128/JCM.41.9.4194-4216.2003 |
| [7] |
Beall B, McEllistrem MC, Gertz RE Jr., et al. Pre-and postvaccination clonal compositions of invasive pneumococcal serotypes for isolates collected in the United States in 1999, 2001, and 2002[J]. J Clin Microbiol, 2006, 44(3): 999-1017. DOI:10.1128/JCM.44.3.999-1017.2006 |
| [8] |
Tyrrell GJ. The changing epidemiology of Streptococcus pneumoniae serotype 19A clonal complexes[J]. J Infect Dis, 2011, 203(10): 1345-1347. DOI:10.1093/infdis/jir056 |
| [9] |
Beall BW, Gertz RE, Hulkower RL, et al. Shifting genetic structure of invasive serotype 19A pneumococci in the United States[J]. J Infect Dis, 2011, 203(10): 1360-1368. DOI:10.1093/infdis/jir052 |
| [10] |
Murray D, Jackson C. A conjugate vaccine for the prevention of pediatric pneumococcal disease[J]. Mil Med, 2002, 167(8): 671-677. DOI:10.1093/milmed/167.8.671 |
| [11] |
Cillóniz C, Amaro R, Torres A. Pneumococcal vaccination[J]. Curr Opin Infect Dis, 2016, 29(2): 187-196. DOI:10.1097/QCO.0000000000000246 |
| [12] |
Marimón JM, Alonso M, Rolo D, et al. Molecular characterization of Streptococcus pneumoniae invasive serotype 19A isolates from adults in two Spanish regions(1994-2009)[J]. Eur J Clin Microbiol Infect Dis, 2012, 31(6): 1009-1013. DOI:10.1007/s10096-011-1399-3 |
| [13] |
Setchanova LP, Alexandrova A, Dacheva D, et al. Dominance of multidrug-resistant Denmark(14)-32(ST230) clone among Streptococcus pneumoniae serotype 19A isolates causing pneumococcal disease in Bulgaria from 1992 to 2013[J]. Microb Drug Resist, 2015, 21(1): 35-42. DOI:10.1089/mdr.2014.0076 |
| [14] |
Tarragó D, Aguilar L, García R, et al. Evolution of clonal and susceptibility profiles of serotype 19A Streptococcus pneumoniae among invasive isolates from children in Spain, 1990 to 2008[J]. Antimicrob Agents Chemother, 2011, 55(5): 2297-3302. DOI:10.1128/AAC.01494-10 |
| [15] |
Shin J, Baek JY, Kim SH, et al. Predominance of ST320 among Streptococcus pneumoniae serotype 19A isolates from 10 Asian countries[J]. J Antimicrob Chemother, 2011, 66(5): 1001-1004. DOI:10.1093/jac/dkr048 |
| [16] |
Pai R, Moore MR, Pilishvili T, et al. Postvaccine genetic structure of Streptococcus pneumoniae serotype 19A from children in the United States[J]. J Infect Dis, 2005, 192(11): 1988-1995. DOI:10.1086/498043 |
| [17] |
刘春林, 赵春江, 刘昱东, 等. 侵袭性肺炎链球菌148株血清型、耐药性及分子分型研究[J]. 中华医学杂志, 2010, 90(22): 1565-1570. Liu CL, Zhao CJ, Liu YD, et al. Study of serotype distridbution, antimicrobial resistance patterns and molecular epidemiology in 148 isolates of invasive Streptococcus pneumoniae[J]. Nat Med J China, 2010, 90(22): 1565-1570. DOI:10.3760/cma.j.issn.0376-2491.2010.22.014 |
 2018, Vol. 33
2018, Vol. 33


