扩展功能
文章信息
- 杨晶, 王红, 朱文涛, 张桂, 金东
- YANG Jing, WANG Hong, ZHU Wen-tao, ZHANG Gui, JIN Dong
- 四川省自贡市食品和环境携带屎肠球菌的耐药性和多位点序列分型研究
- Antibiotic susceptibility and multilocus sequence typing of Enterococcus faecium strains isolated from foods and environment in Zigong, Sichuan
- 疾病监测, 2017, 32(12): 925-930
- Disease Surveillance, 2017, 32(12): 925-930
- 10.3784/j.issn.1003-9961.2017.12.008
-
文章历史
- 收稿日期:2017-06-30
2. 自贡市疾病预防控制中心, 四川 自贡 643000;
3. 河南省疾病预防控制中心, 河南 郑州 450016
2. Zigong Center for Disease Control and Prevention, Zigong 643000, Sichuan, China;
3. Henan Provincial Center for Disease Control and Prevention, Zhengzhou 450016, Henan, China
肠球菌耐酸、耐盐、对多种抗生素天然耐药等特性而广泛存在于多种动物、媒介、食品和环境中[1-2]。肠球菌是人和动物肠道的常见细菌,同时也是院内感染的病原菌,可造成尿路感染、腹腔和盆腔感染、败血症以及心内膜炎等多种感染性疾病[3]。其作为饲料添加的菌种之一,在畜、禽养殖场所和畜、禽产品生产加工的各个环节中都有分布。随着养殖业中长期使用低治疗浓度的抗生素,动物中出现了大量耐药菌株,这些耐药菌株可以污染食物,进而使人类暴露于耐药菌株[4]。2015年中国细菌耐药监测网(CHINET)细菌耐药性监测结果显示,在革兰阳性临床分离株中,肠球菌是仅次于葡萄球菌的重要院内感染致病菌[5]。我国临床分离的肠球菌中,屎肠球菌(Enterococcus faecium)占50.14%,其耐药情况比粪肠球菌(Enterococcus faecalis)严重[5]。肠球菌的耐药现象日益严重,特别是万古霉素耐药肠球菌(Vancomycin Resistant Enterococcus,VRE)的出现,造成临床治疗的困难。同时,VRE还可以将万古霉素耐药基因水平转移给其他多重耐药的革兰阳性菌,如耐甲氧西林金黄色葡萄球菌(MRSA),给临床抗感染治疗带来了巨大的挑战[6]。
多位点序列分型(multilocus sequence typing, MLST)是基于多个管家基因的序列分型(ST)方法,因其操作简便,结果客观可靠且易于实验室间横向比较的特点而广泛用于多种病原菌的分子分型研究。近年来的基于MLST方法的分子流行病学研究显示,引起院内感染的屎肠球菌多属于17(clonal complex 17,CC17)克隆复合体,主要包括ST16、ST17、ST18和ST78等多个ST型别[7-8]。该群的菌株能够获得多种抗生素耐药性(如β-内酰胺类、喹诺酮类抗生素和万古霉素等)和毒力特性(肠球菌表面蛋白和/或透明质酸酶基因), 造成院内感染患者治疗难度加大[9-10]。
研究表明食源性途径是耐药菌株(包括其耐药基因)由动物到人的主要途径[11-12]。本研究对四川省自贡市多个农贸市场、餐馆的肉制品和环境标本携带的屎肠球菌进行分离,并对其进行耐药性检测和MLST分子分型研究,以评估食品和环境中携带屎肠球菌对人类健康的潜在影响。
1 材料与方法 1.1 材料 1.1.1 样品采集和菌株157份食品及环境标本采集于2013年4月17-21日,来自四川省自贡市6个辖区(县)的菜市场、农贸市场和餐馆。样品包括147份肉及其制品,包括生肉(生牛肉)、腊肉、熟肉制品和酱卤肉制品以及10份采样点冰箱拭子标本。药敏实验质控菌株为粪肠球菌ATCC 29212,为本实验室保藏。
1.1.2 主要仪器和试剂Senso Quest Labcycler PCR仪(德国Senso公司),全自动微生物鉴定仪为Micro Scan Walk Away 40 SI(美国贝克曼库尔特公司),Gel Doc XR+ System凝胶成像系统(美国Bio-Rad公司),BAGMIXER均质器(法国INTERSCIENCE公司)。Ex TaqTM Version 2.0 plus dye购自宝生物工程(大连)有限公司;Qiagen核酸提取试剂盒(QIAamp DNA Mini Kit),购自Qiagen公司;革兰阳性菌药敏板购自Pos MIC Panel Type 29(美国贝克曼库尔特公司); KF链球菌选择性培养基,购自北京陆桥技术有限公司,该培养基常用于食品中肠球菌的选择性分离及计数,其对食品样品中的革兰阴性菌有很好的抑制作用,而粪肠球菌在该培养基上生长良好;API 20 Strep生化鉴定系统购自法国生物梅里埃公司;引物由生工生物工程(上海)有限公司合成,PCR产物纯化及测序委托北京天一辉远生物科技有限公司完成。
1.2 方法 1.2.1 样品预处理在无菌条件下取25 g生肉及肉制品放入装有225 ml缓冲蛋白胨水的均质袋中,放入均质器中,以8 000 r/min均质1~2 min,制成1 : 10的样品匀液。冰箱拭子标本直接进行检测。
1.2.2 菌株分离和鉴定将处理后的样品匀液和冰箱拭子接种于KF链球菌选择性培养基上,37 ℃,5%CO2条件下培养24 h。KF链球菌选择性培养基能抑制革兰阴性菌生长,屎肠球菌在该培养基上生长良好。每份标本挑选3~5个可疑的单菌落(紫色、圆润、大菌落)接种于哥伦比亚血平板上进行纯化培养。采用API 20 Strep生化鉴定系统对纯培养物进行生化鉴定;同时使用Qiagen核酸提取试剂盒(QIAamp DNA Mini Kit)按照说明书提取纯培养物的基因组核酸,进行16S rDNA序列(27F:5′-AGA GTT TGA TCC TGG CTC AG-3′; 1492R:5′-ACG GCT ACC TTG TTA CGA CTT-3′)和rpoA基因序列鉴定[13], 16S rDNA序列初步比对能判定为肠球菌,而rpoA基因序列比对相似度高于97%即鉴定为屎肠球菌。每份阳性标本最终保留1株屎肠球菌分离株进行后续分析。
1.2.3 药敏试验使用美国贝克曼库尔特公司MicroScanWalkAway 40 SI全自动微生物鉴定仪及其配套的Pos MIC Panel Type 29革兰阳性菌药敏板,按照仪器和药敏板使用说明,使用快速接种法进行药敏试验。药敏结果判断参照2015年美国临床和实验室标准协会(CLSI)标准[14]。
1.2.4 MLST分析屎肠球菌实验方案参考E. faecuim MLST数据库(http://pubmlst.org/efaecium/),7个管家基因(atpA、ddl、gdh、purK、gyd、pstS和adk)引物序列和扩增产物长度见表 1。聚合酶链式反应(polymerase chain reaction, PCR)产物经纯化后,进行双向测序。序列分析采用SeqMan软件(DNAStar Inc., 美国)对等位基因序列进行拼接和校正,提交MLST数据库比对确定每个管家基因等位基因号,7个等位基因号组合查询即获得该菌株的ST型别。使用Mega 6.0软件,将7个管家基因的串联序列构建系统进化树,选用Neighbor-joining方法。参考序列来自屎肠球菌CC17克隆群的代表ST型别,包括ST16、ST17、ST18和ST78。
| 引物 | 检测基因编码产物 | 引物序列(5′~3′) | 退火温度(℃) | 产物大小(bp) |
| adk1n | 腺苷酸激酶 | GAA CCT CAT TTT AAT GGG G | 52 | 437 |
| adk2n | TGA TGT TGA TAG CCA GAC G | |||
| atpA1n | ATP合成酶,α亚基 | TTC AAA TGG CTC ATA CGG | 52 | 556 |
| atpA2n | AGT TCA CGA TAA GCA ACA GC | |||
| ddl1 | D-丙氨酸:D-丙氨酸连接酶 | GAG ACA TTG AAT ATG CCT TAT G | 52 | 465 |
| ddl2 | AAA AAG AAA TCG CAC CG | |||
| gdh1 | 6-磷酸葡萄糖脱氢酶 | GGC GCA CTA AAA GAT ATG GT | 52 | 530 |
| gdh2 | CCA AGA TTG GGC AAC TTC GTC CCA | |||
| gyd1 | 3-磷酸甘油醛脱氢酶 | CAA ACT GCT TAG CTC CAA TGG C | 52 | 395 |
| gyd2 | CAT TTC GTT GTC ATA CCA AGC | |||
| purK1n | 磷酸核糖酰氨咪唑羧化酶 | CAG ATT GGC ACA TTG AAA G | 52 | 492 |
| purK2n | TTC ATT CAC ATA TAG CCC G | |||
| pstS1n | 磷酸ABC转运蛋白 | TTG AGC CAA GTC GAA GC | 55 | 583 |
| pstS2n | CGT GAT CAC GTT CTA CTT CC | |||
| esp-F | 肠球菌表面蛋白 | AGA TTT CAT CTT TGA TTC TTG G | 52 | 510 |
| esp-R | AAT TGA TTC TTT AGC ATC TGG | |||
| hyl-F | 透明质酸酶 | ACA GAA GAG CTG CAG GAA ATG | 52 | 276 |
| hyl-R | GAC TGA CGT CCA AGT TTC CAA | |||
| 注:ATP:三磷酸腺苷;ABC转运蛋白: ATP结合盒式转运蛋白 | ||||
使用PCR方法对屎肠球菌2种常见的毒力基因:透明质酸酶(hyalronidase, hyl)编码基因hyl和肠球菌表面蛋白(enterococcal surface protein, esp)编码基因esp进行检测。引物序列及扩增产物长度见表 1。PCR反应体系:2×Ex Taq Mix 10 μl,10 μmol/L的上下游引物各1 μl,模板DNA 1.0 μl,加水至20 μl。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,52 ℃退火30 s,72 ℃延伸45 s,30个循环;72 ℃延伸10 min。阳性对照基因为屎肠球菌(ATCC BAA-472,hyl基因阳性)和粪肠球菌(ATCC 51299,esp基因阳性)。
2 结果 2.1 菌株分离和药敏情况从157份样本中共分离到44株屎肠球菌,其中43株分离自肉及其制品样品,1株分离自环境样品,阳性率为28.03% (44/157)。44株屎肠球菌对14种常见抗生素的耐药情况见表 2。44株粪肠球菌对红霉素耐药率最高,达到65.91% (29/44);对四环素和利福平的耐药率分别为31.82% (14/44)和27.27% (12/44),对环丙沙星、高浓度链霉素和高浓度庆大霉素以及氯霉素亦也有一定的耐药率,分别为9.09%(4/44)、11.36%(5/44)、9.09%(4/44)和6.82%(3/44)。44株屎肠球菌中,耐药菌株为38株,耐药率86.36%(38/44)。其中,对1种抗生素耐药的菌株22株,对2种抗生素耐药的菌株8株,对≥3种抗生素耐药的菌株为7株。仅有1株菌株(E365)对14种检测的抗生素全部敏感,另有5株菌对1~3种抗生素中介。分离到1株喹奴普丁-达福普丁耐药的菌株。本研究未分离到对青霉素类、糖肽类、脂肽类、喹诺酮类以及恶唑烷酮类抗生素耐药的粪肠球菌。
| 抗菌药物 | 敏感(S)菌株数量(%) | 中度敏感(I)菌株数量(%) | 耐药(R)菌株数量(%) |
| 红霉素 | 9(20.45) | 6(13.64) | 29(65.91) |
| 四环素 | 30(68.18) | 0(0.00) | 14(31.82) |
| 利福平 | 28(63.64) | 4(9.09) | 12(27.27) |
| 环丙沙星 | 36(81.82) | 4(9.09) | 4(9.09) |
| 高浓度链霉素 | 39(88.64) | 0(0.00) | 5(11.36) |
| 高浓度庆大霉素 | 40(90.91) | 0(0.00) | 4(9.09) |
| 氯霉素 | 39(88.64) | 2(4.55) | 3(6.82) |
| 喹奴普丁-达福普丁 | 9(20.45) | 34(77.27) | 1(2.27) |
| 利奈唑胺 | 43(97.73) | 1(2.27) | 0(0.00) |
| 左旋氧氟沙星 | 40(90.91) | 4(9.09) | 0(0.00) |
| 氨苄西林 | 44(100.00) | 0(0.00) | 0(0.00) |
| 达托霉素 | 44(100.00) | 0(0.00) | 0(0.00) |
| 青霉素 | 44(100.00) | 0(0.00) | 0(0.00) |
| 万古霉素 | 44(100.00) | 0(0.00) | 0(0.00) |
| 注:括号内为菌株数/总菌株数×100% | |||
44株屎肠球菌分为38个ST型别,包括ST94(4株)、ST301(3株),以及ST69、ST361和ST957各1株,其余菌株均属于不同的未知ST型别。44株屎肠球菌分离自22个不同的农贸市场或餐馆来源的样本,主要为熟肉制品(C,cooked meat),其次为生肉制品(R,raw meat)和冰箱拭子(RS, refrigerator swab)。通过样本溯源分析菌株来源可以看出,本研究涉及的13株ST94克隆群的菌株分离自11个不同的农贸市场或餐馆,表明该克隆群是自贡地区的熟肉制品中主要的ST型别,见表 3。
| 分离株(来源) | ST型别 | 红霉素 | 环丙沙星 | 喹奴普丁-达福普丁 | 利福平 | 利奈唑胺 | 高浓度链霉素 | 氯霉素 | 高浓度庆大霉素 | 四环素 | 左旋氧氟沙星 |
| E074_2(C7) | ST-94 | R | S | I | S | S | S | S | S | S | S |
| E096_2(C10) | ST-94 | R | S | I | S | S | S | S | S | S | S |
| E142_2(C17) | ST-94 | R | S | I | S | S | S | S | S | S | S |
| E141_1a(RS16) | ST-94c | R | S | I | S | S | S | S | S | S | S |
| E078_1(C8) | ST-94c | R | S | I | I | S | S | S | S | S | S |
| E134b(C15) | ST-94c | R | S | I | R | S | S | S | S | S | S |
| E363_2c(C21) | ST-94c | S | S | I | S | S | S | S | S | S | S |
| E133_1(C13) | ST-94 | R | S | I | S | S | R | I | R | R | S |
| E109(C11) | ST957 | R | R | R | R | I | R | R | R | R | I |
| 注:菌株来源:C为熟肉制品,RS为冰箱拭子,数字代表不同的采样农贸市场和饭店;aE141_1(RS16), E064-1(C6), E362_2(C21)和E082_2(C8), representIsolates(Samples) E141_1(RS16), E064-1(C6), E362_2(C21) and E082_2(C8); bE134(C15)和E344_2(C19), representIsolates(Samples) E134(C15) and E344_2(C19); cE363_2(C21)和E355_2(C20), representIsolates(Samples) E363_2 (C21) and E355_2(C20) | |||||||||||
利用Mega 6.0软件中的Neighbor-joining方法构建系统进化树,并与屎肠球菌CC17克隆群中的代表ST型别ST16、ST17、ST18和ST78进行亲缘关系分析,结果见图 1。处于同一分支上的ST94克隆群(2个等位基因的差异)是主要的克隆群,包括13株菌,而这13株菌具有相似的耐药谱(表 3)。在13株ST94克隆群的菌株中,有7株菌(E074_2等)耐药情况一致, 为红霉素耐药,喹奴普丁-达福普丁中敏;1株菌(E078_1)红霉素耐药,喹奴普丁-达福普丁, 利福平中敏;2株菌(E134和E344_2)红霉素和利福平耐药,喹奴普丁-达福普丁中敏;2株菌(E363_2和E355_2)对喹奴普丁-达福普丁中敏,对其他13种抗生素敏感;1株(E133_1)为红霉素-链霉素-庆大霉素-四环素耐药,喹奴普丁-达福普丁中敏。需注意的是有1株屎肠球菌E139_2属于CC17克隆群,该菌株对环丙沙星-利福平-四环素耐药,对红霉素,喹奴普丁-达福普丁,氯霉素和左旋氧氟沙星中敏。另外属于ST957的菌株(E109)对8种抗生素耐药(表 3)。
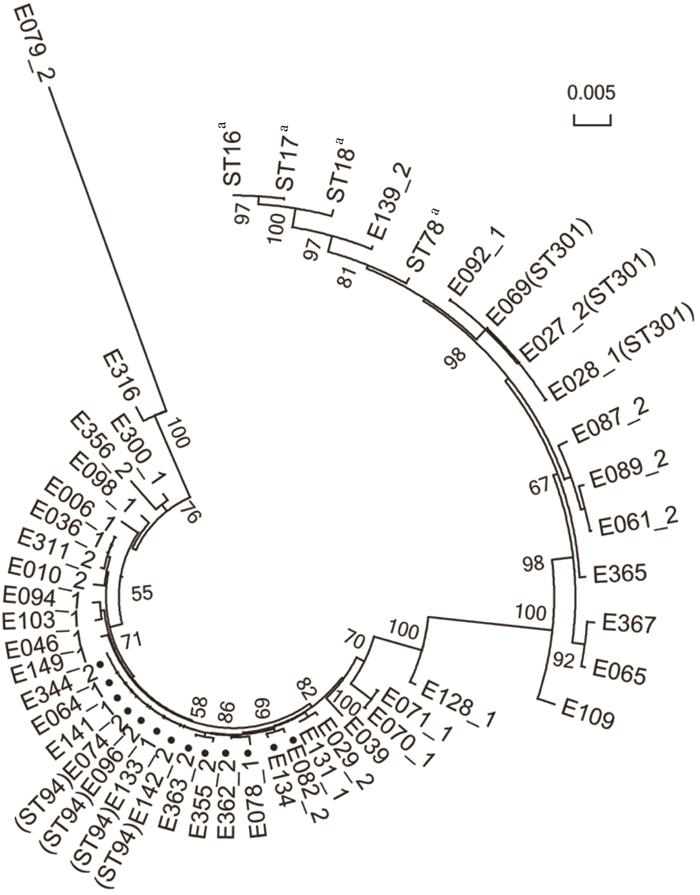
|
| 图 1 44株屎肠球菌分离株及CC17代表菌株MLST序列的Neighbour-joining系统发生树 Figure 1 Neighbor-joining phylogenetic tree inferred from comparison of MLST sequences of 44 E. faecium strains from food and environment samples and representative CC17 strains 注:aCC17代表序列; ●ST94克隆群菌株 |
| |
对44株屎肠球菌进行毒力基因检测后,在2株菌(E029_2和E355_2)中检测到esp基因,携带率为4.55%(2/44), 这2个分离株均对喹奴普丁-达福普丁中敏,对其他抗生素敏感。未检测到hyl基因阳性的屎肠球菌。
3 讨论肠球菌作为动物和人类肠道的正常菌群,因为获得耐药性/抗生素抗性基因成为机会致病菌[15]。耐药菌的出现增加了感染概率,造成抗感染治疗的失败;耐药菌感染也增加了感染严重性,导致病程延长和死亡率增高,对人类健康和公共卫生造成严重的影响。抗生素由于能够治疗和预防动物疾病、促进动物生长和提高经济效益,被广泛用于畜牧和水产养殖业[16]。抗生素的大量使用,造成动物体内携带大量的耐药菌,后者通过直接接触、摄食被耐药菌污染的食品和水等方式从动物传递给人类。据报道,欧洲联盟每年有1 500多起因对第三代头孢药物耐药的大肠埃希菌感染死亡案例,这都直接与在禽类养殖中过度使用头孢和其他种类的抗生素有关[17],而MRSA可以通过密切接触牲畜的农民、兽医和其他人群传播,导致严重的感染[18]。研究表明耐药菌的来源主要是临床使用、自我药疗以及摄取了受抗生素污染的水或食物,其中食源性途径可能是主要的传播方式[19]。
本研究表明,自贡地区食品中屎肠球菌污染率较高。耐药性分析表明,多数食品来源的屎肠球菌分离株(38/44)对1种及其以上的抗生素耐药,红霉素耐药率最高(65.91%), 四环素、利福平和氨基糖苷类抗生素耐药率也较高,应重视食品中屎肠球菌耐药菌株对人类健康的威胁。除了治疗难度大的万古霉素耐药菌株外,目前临床来源的屎肠球菌对青霉素、红霉素、环丙沙星和左氧氟沙星的耐药率依次为92.6%、90.9%、90.0%和86.9%[5]。本研究食品中污染的屎肠球菌,其耐药谱与临床菌株间存在差异,提示食品和环境中的屎肠球菌与医院来源的菌株所面临的抗生素选择压力不同。相比于粪肠球菌,屎肠球菌更易获得和转移耐药基因,除少数抗生素外,屎肠球菌对常见抗生素的耐药率明显高于粪肠球菌[20-21]。
44株食品来源的屎肠球菌ST型别组成多样,ST94克隆群是优势ST型,包含13个分离株,且这些菌株的耐药谱相似。有文献报道,ST94的屎肠球菌在新鲜蔬菜等食品中是较常出现的ST型别[22]。河南省睢县3岁以下儿童肠道中ST94克隆群的屎肠球菌是仅次于CC17的主要克隆群菌株,该群菌株的耐药性和毒力基因携带情况与本研究类似[23]。虽然标本采集的区域不同,但仍提示该复合群的屎肠球菌有通过食物链传播到人类,且在人类肠道长期存留的可能。
世界范围内的分子流行病学研究显示,医院内感染VRE菌株主要为CC17克隆复合群,该复合群的屎肠球菌对多种抗生素耐药,同时携带多种毒力基因,且通过获得外源基因片段而产生更强的院内生存的能力[7-8, 24]。本研究从食品中也分离到1株CC17克隆群的多重耐药菌(E139_2),同时还分离到1株ST957(E109)菌株对8种抗生素耐药,包括红霉素、环丙沙星、利福平等多种常见的抗生素,这2株菌株均分离自熟食肉制品,应对该地区食品中耐药屎肠球菌污染状况引起重视。
作者贡献:
杨晶 ORCID:0000-0002-5865-3165
杨晶:MLST,数据整理,文章撰写
王红:样本采集
朱文涛:毒力基因检测
张桂:药敏实验
金东:实验设计,MLST数据分析,文章修改指导
| [1] |
Aarestrup FM, Butaye P, Witte W. Nonhuman reservoirs of enterococci[M]//Gilmore MS, Clewell DB, Courvalin P, et al. The Enterococci:Pathogenesis, Molecular Biology, and Antibiotic Resistance. Washington, DC:ASM Press, 2002:55-100.
|
| [2] |
Byappanahalli MN, Nevers MB, Korajkic A, et al. Enterococci in the environment[J]. Microbiol MolBiol Rev, 2012, 76(4): 685-706. DOI:10.1128/MMBR.00023-12 |
| [3] |
Higuita NIA, Huycke MM. Enterococcal disease, epidemiology, and implications for treatment[M]//Gilmore MS, Clewell DB, Ike Y, et al. Enterococci:From commensals to leading causes of drug resistant infection. Boston:Massachusetts Eye and Ear Infirmary, 2014:45-86.
|
| [4] |
Kruse H, Johansen BK, Rørvik LM, et al. The use of avoparcin as a growth promoter and the occurrence of vancomycin-resistant Enterococcus species in Norwegian poultry and swine production[J]. Microb Drug Resist, 1999, 5(2): 135-139. DOI:10.1089/mdr.1999.5.135 |
| [5] |
Hu FP, Zhu DM, Wang F, et al. Report of CHINET antimicrobial resistance surveillance program in 2015[J]. Chinese Journal of Infection and Chemotherapy, 2016, 16(6): 685-694. (in Chinese) 胡付品, 朱德妹, 汪复, 等. 2015年CHINET细菌耐药性监测[J]. 中国感染与化疗杂志, 2016, 16(6): 685-694. |
| [6] |
Weigel LM, Clewell DB, Gill SR, et al. Genetic analysis of a high-level vancomycin-resistant isolate of Staphylococcus aureus[J]. Science, 2003, 302(5650): 1569-1571. DOI:10.1126/science.1090956 |
| [7] |
Leavis HL, Bonten MJM, Willems RJL. Identification of high-risk enterococcal clonal complexes:global dispersion and antibiotic resistance[J]. Curr Opin Microbiol, 2006, 9(5): 454-460. DOI:10.1016/j.mib.2006.07.001 |
| [8] |
Top J, Willems R, van der Velden S, et al. Emergence of clonal complex 17Enterococcus faecium in the Netherlands[J]. J Clin Microbiol, 2008, 46(1): 214-219. DOI:10.1128/JCM.01351-07 |
| [9] |
Willems RJL, Top J, van Santen M, et al. Global spread of vancomycin-resistant Enterococcus faecium from distinct nosocomial genetic complex[J]. Emerg Infect Dis, 2005, 11(6): 821-828. DOI:10.3201/1106.041204 |
| [10] |
Hallgren A, Claesson C, Saeedi B, et al. Molecular detection of aggregation substance, enterococcal surface protein, and cytolysin genes and in vitro adhesion to urinary catheters of Enterococcus faecalis and E. faecium of clinical origin[J]. Int J Med Microbiol, 2009, 299(5): 323-332. DOI:10.1016/j.ijmm.2008.10.001 |
| [11] |
Giraffa G. Enterococci from foods[J]. FEMS Microbiol Rev, 2002, 26(2): 163-171. DOI:10.1111/j.1574-6976.2002.tb00608.x |
| [12] |
Hammerum AM, Lester CH, Heuer OE. Antimicrobial-resistant enterococci in animals and meat:a human health hazard?[J]. Foodborne Pathog Dis, 2010, 7(10): 1137-1146. DOI:10.1089/fpd.2010.0552 |
| [13] |
Naser SM, Thompson FL, Hoste B, et al. Application of multilocus sequence analysis (MLSA) for rapid identification of Enterococcus species based on rpoA and pheS genes[J]. Microbiology, 2005, 151(7): 2141-2150. DOI:10.1099/mic.0.27840-0 |
| [14] |
Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing:twenty-fifth informational supplement:M100-S25[S]. Wayne:CLSI, 2015.
|
| [15] |
Gilmore MS, Lebreton F, van Schaik W. Genomic transition of enterococci from gut commensals to leading causes of multidrug-resistant hospital infection in the antibiotic era[J]. Curr Opin Microbiol, 2013, 16(1): 10-16. DOI:10.1016/j.mib.2013.01.006 |
| [16] |
Dibner JJ, Richards JD. Antibiotic growth promoters in agriculture:history and mode of action[J]. Poult Sci, 2005, 84(4): 634-643. DOI:10.1093/ps/84.4.634 |
| [17] |
Collignon P, Aarestrup FM, Irwin R, et al. Human deaths and third-generation cephalosporin use in Poultry, Europe[J]. Emerg Infect Dis, 2013, 19(8): 1339-1340. DOI:10.3201/eid1908.120681 |
| [18] |
Petinaki E, Spiliopoulou I. Methicillin-resistant Staphylococcus aureus among companion and food-chain animals:impact of human contacts[J]. Clin Microbiol Infect, 2012, 18(7): 626-634. DOI:10.1111/j.1469-0691.2012.03881.x |
| [19] |
Wang B, Wang HX, Zhou W, et al. Urinary excretion of phthalate metabolites in school children of China:implication for cumulative risk assessment of phthalate exposure[J]. Environ Sci Technol, 2015, 49(2): 1120-1129. DOI:10.1021/es504455a |
| [20] |
Mundy LM, Sahm DF, Gilmore M. Relationships between enterococcal virulence and antimicrobial resistance[J]. Clin Microbiol Rev, 2000, 13(4): 513-522. DOI:10.1128/CMR.13.4.513-522.2000 |
| [21] |
Palmer KL, Kos VN, Gilmore MS. Horizontal gene transfer and the genomics of enterococcal antibiotic resistance[J]. Curr Opin Microbiol, 2010, 13(5): 632-639. DOI:10.1016/j.mib.2010.08.004 |
| [22] |
Burgos MJG, Aguayo MCL, Pulido RP, et al. Multilocus sequence typing and antimicrobial resistance in Enterococcus faecium isolates from fresh produce[J]. Antonie Leeuwenhoek, 2014, 105(2): 413-421. DOI:10.1007/s10482-013-0073-4 |
| [23] |
Hu SK, Zhou J, Hu YY, et al. Antibiotic resistance and multilocus sequence typing of Enterococcus faecium strains isolated from children under 3 years old in Sui county, Henan province[J]. Chinese Journal of Microbiology and Immunology, 2016, 36(8): 588-592. (in Chinese) 胡守奎, 周娟, 胡园园, 等. 河南睢县3岁以下儿童携带屎肠球菌耐药性及多位点序列分型研究[J]. 中华微生物学和免疫学杂志, 2016, 36(8): 588-592. |
| [24] |
Ruiz-Garbajosa P, de Regt M, Bonten M, et al. High-density fecal Enterococcus faecium colonization in hospitalized patients is associated with the presence of the polyclonal subcluster CC17[J]. Eur J Clin Microbiol Infect Dis, 2012, 31(4): 519-522. DOI:10.1007/s10096-011-1342-7 |
 2017, Vol. 32
2017, Vol. 32


