扩展功能
文章信息
- 刘笑舒, 廖凤, 李文革, 古文鹏, 张文竹, 陈小萍, 吴媛, 卢金星
- LIU Xiao-shu, LIAO Feng, LI Wen-ge, GU Wen-peng, ZHANG Wen-zhu, CHEN Xiao-ping, WU Yuan, LU Jin-xing
- 艰难梭菌五种实验室检测方法的评价
- Evaluation of five Clostridium difficile detection methods
- 疾病监测, 2017, 32(4): 351-354
- Disease Surveillance, 2017, 32(4): 351-354
- 10.3784/j.issn.1003-9961.2017.04.022
-
文章历史
- 收稿日期:2016-10-12
2. 云南省第一人民医院, 云南 昆明 650034;
3. 云南省疾病预防控制中心, 云南 昆明 650022
2. The First People's Hospital of Yunnan Province, Kunming 650034, Yunnan, China;
3. Yunnan Provincial Center for Disease Control and Prevention, Kunming 650022, Yunnan, China
艰难梭菌 (Clostridium difficile,C. difficile, C.diff) 是一种严格厌氧的革兰阳性芽孢杆菌,是造成抗生素相关腹泻与伪膜性肠炎的最主要病原体[1]。艰难梭菌主要通过产生毒素A和/或毒素B以及少数菌株产生的二元毒素来致病。近年来,艰难梭菌已经成为医院感染中疾病负担最高的病原体,每年造成巨大的经济损失。艰难梭菌感染 (Clostridium difficile infection,CDI) 逐年升高,成为全球公共卫生面临巨大挑战[2-8]。而我国国内对于艰难梭菌的检测,大部分实验室及医院缺乏厌氧培养条件,检测手段相对局限。
本实验对包括tcdB基因普通PCR检测、tcdB基因Real-time PCR检测、艰难梭菌毒素A/B检测试剂盒 (酶联免疫法)、艰难梭菌谷氨酸脱氢酶抗原及毒素检测试剂盒 (酶联免疫层析法) 以及环丝氨酸-头孢西丁-果糖琼脂 (cycloserin-cefoxitin-fructose agar,CCFA) 常规培养法共5种方法进行评价,以CCFA常规培养联合16S rDNA测序鉴定的结果作为参考,对2种艰难梭菌检测试剂盒检测和2种PCR检测方法进行评价比较,以期为实验室诊断艰难梭菌提供依据和参考。
1 材料与方法 1.1 样本及来源收集2016年来自云南省腹泻患者的粪便标本共125份。
1.2 主要仪器及试剂酶标仪 (BioTek,Elx808),洗板机 (BioTek,Elx50),PCR仪 (德国SENSO,labcycler),实时荧光定量PCR仪 (新加坡ABI,7500FAST),恒温培养箱 (德国Memmert),温控高速离心机 (德国Eppendorf),凝胶成像仪 (Bio-Rad),电泳仪 (百晶通,BG-Submzd1);粪便基因组提取试剂盒,细菌基因组提取试剂盒 (TIANGEN),试剂盒C.difficile Tox A/BⅡ[美国Alere,艰难梭菌毒素A/B检测 (酶联免疫法)]、试剂盒C.diff Quik Chek Complete[美国Alere Techlab,艰难梭菌谷氨酸脱氢酶抗原及毒素检测 (酶联免疫层析法)],CCFA培养基及其添加剂 (英国OXOID),Premix ExTaqTM(日本TaKaRa),Premix Ex TaqTM(Perfect Real Time)(日本TaKaRa),引物合成,16S rDNA测序由生工生物工程 (上海) 有限公司完成。
1.3 方法 1.3.1 tcdB基因普通PCR检测使用粪便基因组提取试剂盒,按照说明书操作提取粪便样本中的DNA,并对tcdB进行PCR扩增。所使用引物及其序列为:NK104:5′-GTG TAG CAA TGA AAG TCC AAG TTT ACG C-3′和NK105:5′-CAC TTA GCT CTT TGA TTG CTG CAC CT-3′[9]。反应体系 (50 μl):Premix Ex TaqTM(2×)25 μl,NK104 (20 pmol/μl)1 μl,NK105(20 pmol/μl)1 μl,DNA模板 (20~80 ng/μl) 3 μl,去离子水20 μl。反应条件:95 ℃ 10 min;95 ℃ 20 s,55 ℃ 30 s,72 ℃ 60 s,35个循环;延伸72 ℃ 5 min。扩增产物大小为203 bp,产物经1.5%琼脂糖凝胶电泳进行鉴定。
1.3.2 tcdB基因Real-time PCR检测使用粪便基因组提取试剂盒 (TIANGEN),提取粪便样本中的DNA,并对毒素B基因tcdB进行PCR扩增。所使用引物、探针及其序列为:stcdb-f:5′-ATA TCA GAG ACT GAT GAG-3′,stcdb-r:5′-TAG CAT ATT CAG AGA ATA TTG T-3′,stcdb-p:5′FAM-CTG GAG AAT CTA TAT TTG TAG AAA CTG-BHQ3′[10]。反应体系 (25 μl):Premix Ex TaqTM(Perfect Real Time)12.5 μl,stcdb-f (10 pmol/μl)2.5 μl,stcdb-r (10 pmol/μl)5 μl,stcdb-p (10 pmol/μl)2.5 μl,ROXⅡ0.5 μl,DNA模板2 μl,去离子水2.5 μl。反应条件采用两步法PCR扩增标准程序:第一步,预变性95 ℃ 30 s,1个循环;第二步,PCR反应95 ℃ 3 s,60 ℃ 30 s,40个循环。扩增产物大小为103 bp。
1.3.3 艰难梭菌毒素A/B检测 (酶联免疫法)将试剂盒放置于室温,按照说明书操作,对粪便样本进行处理,使用酶标仪通过酶联免疫法检测艰难梭菌的A/B毒素,从而鉴定粪便样本中有无艰难梭菌。在取到样品的72 h内检测。
1.3.4 艰难梭菌谷氨酸脱氢酶抗原及毒素检测 (酶联免疫层析法)将试剂盒放置于室温,按照说明书操作,通过胶体金方法检测艰难梭菌谷氨酸脱氢酶抗原及毒素A/B,从而鉴定粪便样本中有无艰难梭菌。在取到样品的72 h内检测。
1.3.5 CCFA常规培养法取粪便样本20 μl涂布于CCFA培养皿,37 ℃厌氧培养48 h。典型的艰难梭菌菌落呈灰白色、扁平、干燥、边缘粗糙、有特殊臭味。对判断为艰难梭菌的菌落进行传代培养48 h,使用细菌基因组提取试剂盒提取其DNA,tcdBPCR检测结果阳性,同时进行16S rDNA扩增测序,BLAST比对鉴定,从而得出最后鉴定结果。
1.4 数据分析本实验采用了5种方法对临床粪便样本中的艰难梭菌进行同步检测。其中,采用CCFA常规培养法的结果作为评价的参考标准。该方法通过培养得到病原体,并联合tcdB基因普通PCR检测,16S rDNA测序鉴定,确定样本为艰难梭菌阳性。使用SAS 9.3软件对检测方法与标准方法进行Kappa一致性检验,并分别计算检测方法的灵敏度、特异度、阳性预测值以及阴性预测值,评估方法的正确率。
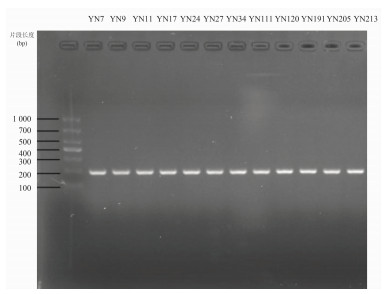
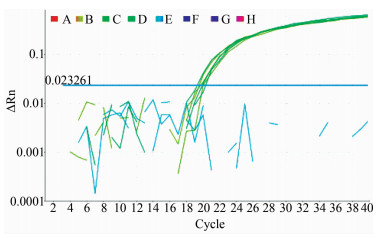
2 结果在125份样本中,参考CCFA培养法检出阳性11份,tcdB基因普通PCR检测见图 1、tcdB基因Real-time PCR检测见图 2、艰难梭菌毒素A/B检测 (酶联免疫法)、艰难梭菌谷氨酸脱氢酶抗原及毒素检测 (酶联免疫层析法) 分别检出阳性12、12、50、25份,不同方法间检出阳性率差异有统计学意义 (χ2=61.452,P < 0.000 1),见表 1。其中tcdB基因普通PCR和Real-time PCR检测结果完全一致。
|
| 图 1 tcdB基因普通PCR检测结果 Figure 1 PCR results of tcdB detection |
| |
|
| 图 2 tcdB基因Real-time PCR检测 Figure 2 Real-time PCR results of tcdB detection |
| |
| 方法 | 阳性标本数 | 阳性率 (%) |
| 普通PCR | 12 | 9.60 |
| Real-time PCR | 12 | 9.60 |
| 酶联免疫法 | 50 | 40.00 |
| 酶联免疫层析法 | 25 | 20.00 |
| CCFA培养 | 11 | 8.80 |
经一致性检验得到4种方法与参考方法的Kappa值分别为0.569、0.569、0.100和0.241,普通PCR与Real-time PCR检测方法与参考方法的一致性最高。4种方法与参考方法之间的比较,以及其敏感度、特异度、阳性预测值和阴性预测值见表 2。4种方法之间敏感度差异无统计学意义 (χ2=0.288, P=0.962),特异度差异有统计学意义 (χ2=63.597,P < 0.000 1)。
| 方法 | CCFA培养 | 敏感度 | 特异度 | 阳性预测值 | 阴性预测值 | Kappa值 | ||
| 阳性 | 阴性 | 合计 | ||||||
| PCR | ||||||||
| 阳性 | 7 | 5 | 12 | 63.64% | 95.61% | 58.33% | 96.46% | 0.569 |
| 阴性 | 4 | 109 | 113 | |||||
| 合计 | 11 | 114 | 125 | |||||
| Real-time PCR | ||||||||
| 阳性 | 7 | 5 | 12 | 63.64% | 95.61% | 58.33% | 96.46% | 0.569 |
| 阴性 | 4 | 109 | 113 | |||||
| 合计 | 11 | 114 | 125 | |||||
| 酶联免疫法 | ||||||||
| 阳性 | 7 | 43 | 50 | 63.64% | 62.28% | 14.00% | 94.67% | 0. 100 |
| 阴性 | 4 | 71 | 75 | |||||
| 合计 | 11 | 114 | 125 | |||||
| 酶联免疫层析法 | ||||||||
| 阳性 | 6 | 19 | 25 | 54.55% | 83.33% | 24.00% | 95.00% | 0.241 |
| 阴性 | 5 | 95 | 100 | |||||
| 合计 | 11 | 114 | 125 | |||||
自1978年首次报道艰难梭菌是伪膜性肠炎的病原体以来,全球各地的艰难梭菌感染逐年升高,并在欧美地区引起了几次暴发流行。目前,艰难梭菌已经成为医院感染的重要病原体[1-8]。我国国内未报道过艰难梭菌的流行暴发,流行情况并不清楚。2010年美国医疗保健流行病学学会和感染性疾病学会提出对CDI的诊断应结合临床表现和实验室检测结果进行。随着艰难梭菌感染的不断增加,对其进行临床检测和流行病学调查刻不容缓。
本研究5种检测方法中,标准参考方法需要对细菌培养,所耗时间较长,需要厌氧设备及相关操作,在国内实验室和医院中普及率不高。同时,该方法以培养出活菌为基本标准,而艰难梭菌作为一种严格厌氧的细菌,在取样、转移至实验室以及后续的实验过程中都有可能部分或全部死亡,采用本方法可保证诊断的准确性,但存在漏检的可能。
2种分子生物学检验手段在灵敏度、特异度上并无差别。操作快速、实验相对简单,能够在较短时间内给出检测结果,在本次试验中与参考方法的一致性最高。在无厌氧培养条件或是需快速检测的情况下,这2种方法综合实用性较高。其中普通PCR方法仪器、试剂、操作条件等更为简单、便捷、节约,而Real-time PCR仪器试剂较贵,操作要求高。在进行大范围、多样本流行病学调查中,PCR方法在实验条件以及准确性方面更符合要求。
2种使用免疫学方法的检测试剂盒,酶联免疫层析方法操作简单快捷,该方法可以同时检测艰难梭菌谷氨酸脱氢酶抗原及毒素A/B,由于艰难梭菌有毒株和无毒株都产生大量的谷氨酸脱氢酶[12-14],此方法能在更大程度上保证检出艰难梭菌。而酶联免疫法使用特异抗体检测毒素A/B,从而进行艰难梭菌的体外诊断,该方法灵敏、快速,在艰难梭菌体外诊断中实用性好。但是这2种方法特异度、阳性预测值和阴性预测值均较PCR方法低;容易产生假阳性和假阴性。不正确的样本采集、处理和/或存储、毒素含量低于试剂盒检测限等因素,均有可能对检测结果产生影响,同时,样本冻融,尤其是反复冻融可能会导致毒素降解从而失活。建议用于先期筛查用,应结合患者的病史考虑检测结果,可作为临床诊断的辅助手段。
综上所述,2种免疫学方法虽有一定局限性,但可用于样本的快速检测和先期筛查。PCR方法灵敏度和特异度较高,在无培养条件以及需快速检测的情况下,建议使用PCR方法进行艰难梭菌的快速检测,其中普通PCR的实用性更高,成本较小。
目前,对于艰难梭菌的检测方法的研究方向依然是检测样品中毒素的有无。免疫学方法的试剂盒的假阳性以及假阴性过高是需要克服的关键问题。艰难梭菌作为一种严格厌氧的细菌,暴露在空气中一段时间后死亡,所检测毒素等蛋白的性状也随之变化,如何保证快速准确鉴定是一大难题。
作者贡献:
刘笑舒 ORCID:0000-0001-9233-1576
刘笑舒:实验操作、论文撰写
廖凤:样本采集,实验操作
李文革:实验操作
古文鹏:实验操作,样本初筛
张文竹、陈小萍:实验操作
吴媛、卢金星:论文修改、指导
| [1] | Carroll KC, Carroll JG. Biology of Clostridium difficile:implications for epidemiology and diagnosis[J]. Annu Rev Microbiol, 2011, 65: 501–521. DOI:10.1146/annurev-micro-090110-102824 |
| [2] | Gerding DN. Global epidemiology of Clostridium difficile infection in 2010[J]. Infect Control Hosp Epidemiol, 2010, 31(Suppl 1): S32–34. |
| [3] | Rupnik M, Wilcox MH, Gerding DN. Clostridium difficile infection:new developments in epidemiology and pathogenesis[J]. Nat Rev Microbiol, 2009, 7(7): 526–536. DOI:10.1038/nrmicro2164 |
| [4] | Bauer MP, Notermans DW, van Benthem BHB, et al. Clostridium difficile infection in Europe:a hospital-based survey[J]. Lancet, 2011, 377(9759): 63–73. DOI:10.1016/S0140-6736(10)61266-4 |
| [5] | Cornely OA, Crook DW, Esposito R. Fidaxomicin versus vancomycin for infection with Clostridium difficile in Europe, Canada, and the USA:a double-blind, non-inferiority, randomised controlled trial[J]. Lancet Infect Dis, 2012, 12(4): 281–289. DOI:10.1016/S1473-3099(11)70374-7 |
| [6] | He M, Miyajima F, Roberts P, et al. Emergence and global spread of epidemic healthcare-associated Clostridium difficile[J]. Nat Genet, 2013, 45(1): 109–113. |
| [7] | Lessa FC, Mu Y, Bamberg WM, et al. Burden of Clostridium difficile infection in the United States[J]. N Engl J Med, 2015, 372(9): 825–834. DOI:10.1056/NEJMoa1408913 |
| [8] | Wiegand PN, Nathwani D, Wilcox MH, et al. Clinical and economic burden of Clostridium difficile infection in Europe:a systematic review of healthcare-facility-acquired infection[J]. J Hosp Infect, 2012, 81(1): 1–14. DOI:10.1016/j.jhin.2012.02.004 |
| [9] | Kato H, Kato N, Watanabe K, et al. Identification of Toxin A-negative, Toxin B-positive Clostridium difficile by PCR[J]. J Clin Microbiol, 1998, 36(8): 2178–2182. |
| [10] | Bélanger SD, Boissinot M, Clairoux N, et al. Rapid detection of Clostridium difficile in feces by real-time PCR[J]. J Clin Microbiol, 2003, 41(2): 730–734. DOI:10.1128/JCM.41.2.730-734.2003 |
| [11] | Cohen SH, Gerding DN, Johnson S, et al. Clinical practice guidelines for Clostridium difficile infection in adults:2010 update by the society for healthcare epidemiology of america (SHEA) and the infectious diseases society of America (IDSA)[J]. Infect Control Hosp Epidemiol, 2010, 31(5): 431–455. DOI:10.1086/651677 |
| [12] | Zheng L, Keller SF, Lyerly DM, et al. Multicenter evaluation of a new screening test that detects Clostridium difficile in fecal specimens[J]. J Clin Microbiol, 2004, 42(8): 3837–3840. DOI:10.1128/JCM.42.8.3837-3840.2004 |
| [13] | Miles BL, Siders JA, Allen SD. .Evaluation of a commercial latex test for Clostridium difficile for reactivity with C.difficile and cross-reactions with other bacteria[J]. J Clin Microbiol, 1988, 26(11): 2452–2455. |
| [14] | Lyerly DM, Wilkins TD. Commercial latex test for Clostridium difficile Toxin A does not detect Toxin A[J]. J Clin Microbiol, 1986, 23(3): 622–623. |
 2017, Vol. 32
2017, Vol. 32


