2 自然资源部第一海洋研究所, 海洋沉积与环境地质国 家海洋局重点实验室, 山东 青岛 266061;
3 中国科学院大学, 北京 100049;
4 中国科学院海洋大科学研究中心, 山东 青岛 266071)
深海海底是地球上最庞大的生态系统,拥有极其多样的底栖生物群落,生物多样性以及生物资源非常丰富[1~2]。底栖有孔虫是深海底栖动物群落的主要组成部分,在许多深海区域,它们在真核生物中的占比能达到50 %甚至更多[3~5]。它们分布广泛,能够栖息在几乎所有的深海海洋环境中[6~7],在能量流动与物质循环中起着重要作用,并且能通过多种方式影响深海生物群落的结构和动态,例如改变沉积物结构、影响海底颗粒沉积、为后生动物提供庇护所及竞争后生动物的稀缺资源等[3, 8]。
深海海底不仅拥有丰富的生物资源,还蕴藏着大量矿产资源和油气资源,是目前世界各国争相探索和开发的战略要地。早在第一次世界大战前后,美国科学家就利用底栖有孔虫来鉴定地层年代并依此开展石油勘探,大获成功[9]。底栖有孔虫生长速度快,生命周期短,对环境变化高度敏感,其壳体形态、群落组成和分布特征(如不同底栖有孔虫的组合、特定属种的丰度、群落多样性参数)与其栖息环境的温度、盐度、氧含量、有机质输入、表层沉积物的粒度等密切相关[10~13]。在底栖有孔虫死后,它们的壳体(以钙质为主)能够在深海沉积物中形成化石并被大量保存下来[14]。底栖有孔虫的这些特点使其成为反演古海洋和古气候变化、重建古环境的重要指标[15~16]。
底栖有孔虫的上述应用主要基于对其物种多样性和群落组成的研究。传统的研究方法是在显微镜下对它们的壳体进行观察、鉴定和计数。这种方法不仅要求研究人员具有良好的有孔虫分类学知识,而且极其耗费时间和人力[17~18]。为了便于观察,在镜检前通常会对沉积物进行过筛处理,筛子的尺寸严重影响单个物种的丰度[19]。大多数形态学研究是针对粒径大于125 μm或150 μm的沉积物,忽略了个体微小的有孔虫类群的丰度,从而影响了对有孔虫多样性和群落组成的研究结果[20~21]。与浅水区相比,深海底栖有孔虫大多个体微小、形态特征不明显,在某些海区尤以单房室软壳类有孔虫为优势种,这些有孔虫类群主要集中在粒径较小(63~125 μm)的细组分中,忽略它们将会丢失许多有价值的信息[22]。目前,DNA高通量测序技术的应用为有孔虫生态调查提供了一种全新的替代方法。它能一次并行对数百万个DNA分子进行测序,效率高、成本低。与镜检法相比,它避免了繁琐的样品前期处理过程,降低了对有孔虫特别是软壳类的破坏。它能通过使用特定的基因标记(如有孔虫特异性DNA条形码)将沉积物中的有孔虫(包括低丰度和个体微小的有孔虫类群)全部检测出来,因此能更真实、更全面地反映环境中有孔虫的多样性[23]。
由于深海海底广阔且遥远,使深海采样变得异常困难,采样不足限制了人们对深海底栖有孔虫多样性的全面评估[24~25]。与大西洋、印度洋及太平洋周边浅海区域相比,关于太平洋深海底栖有孔虫的研究资料十分有限[7, 26~27]。海洋科学家[19, 24, 26~29]对太平洋深海底栖有孔虫进行了一些调查,但主要集中在太平洋东部海区。目前,国际上对西太平洋深海底栖有孔虫的研究仍相对薄弱。此次,我们在西北太平洋深海采集了11个站位(平均水深超5000 m)的表层沉积物,提取了样品中的总基因组DNA,利用有孔虫特异性引物扩增其中的有孔虫DNA,应用高通量测序技术对获得的基因片段测序,评估了西北太平洋深海沉积物中有孔虫的物种多样性、群落组成和分布特点,以期为探索有孔虫全球地理分布模式以及该海区丰富的自然资源提供基础生物信息支撑。
1 材料与方法 1.1 样品采集和处理本文的研究样品来自于近年来“科学”号考察船在西北太平洋开展的航次,利用0.25 m2箱式采泥器对11个目标站位(32.2°~36.0°N,145.5°~156.5°E) (图 1和表 1)的沉积物进行定量采样,使用采样勺刮取未受扰动的表层2 cm沉积物,将其放入封口袋中,充分混合后放置-80 ℃冰箱中冷冻保存直至回到实验室进行下一步处理。

|
图 1 采样站位图 Fig. 1 Location map of the stations |
| 表 1 各个站位的经度、纬度和水深 Table 1 Longitude, latitude and water depth of each station |
将沉积物样品带回实验室后,对样品中的总基因组DNA进行提取。每次从样品中称取大约0.25 g沉积物,使用Power Soil DNA提取试剂盒(MoBio Laboratories,美国)提取总基因组DNA,除细胞裂解外,所有步骤均按照试剂盒说明书提供的实验方案进行。细胞裂解过程则按照Lecroq等[25]所述,将裂解时间延长至40 min。每个样品重复提取3份DNA,每3份DNA对应一个空白对照。
1.3 PCR扩增为了鉴定DNA提取是否成功,首先进行了一系列聚合酶链式反应(Polymerase Chain Reaction,简称PCR),在此过程中,每3份DNA对应一个阴性对照(即每个沉积物样品对应一个空白对照)。利用有孔虫特异性引物s14F3和s17 (它们分别是5′-ACGCAMGTGTGAGAACTTG-3′和5′-CGGTCACGTTCGTTGC-3′)来扩增核糖体小亚基DNA(Small Subunit Ribosomal DNA,简称SSU rDNA)片段[5, 30~31],目标区域长度约为330 bp。每个PCR扩增体系为25 μL,包括12.5 μL的2×High-Fidelity PCR Master Mix,0.5 μL的10 μm引物,2 μL DNA模板和9.5 μL双蒸水(distilled deionized water, 简称ddH2O)。PCR反应程序为:在94 ℃下预变性90 s,然后在94 ℃下变性60 s,在50 ℃下退火60 s,在72 ℃下延伸45 s,此过程进行25个循环周期;之后紧接着进行另外10个循环周期,变性和退火时间缩短至30 s,最后在72 ℃下保持2 min。将得到的PCR产物与1×loading buffer等体积混合,并在浓度为1 %的琼脂糖凝胶上进行电泳检测。将凝胶放置凝胶成像系统下,标记出长度约为400 bp的单一明亮条带所对应的DNA,即为提取成功的样品。对提取失败的样品按照上述DNA提取步骤重新进行提取,直至从每份沉积物样品中获得3份总DNA。
1.4 文库构建和上机测序在每个站位均成功提取到3份总DNA后,利用带有barcode标记的正、反向引物重新进行PCR扩增反应。此处使用的barcode是一段包含6个核苷酸的序列,它们被添加在引物5′末端以标记样品,一组barcode标记一个目标站位。将同一站位的3份PCR产物混合,并在浓度为2 %的琼脂糖凝胶上进行电泳(100 V;35 min)以检测是否成功获得目的条带。在凝胶成像系统下将目的条带切下,利用E. Z.N. ATM凝胶提取试剂盒(Omega Bio-tek,Inc)回收PCR产物,并用Qubit @ 2.0荧光计(Thermo Scientific)检测纯化后的PCR产物的浓度。使用TruSeqⓇDNA PCR-free样品制备试剂盒(Illumina,美国)进行建库,具体操作按照说明书进行。文库质量通过Qubit @ 2.0荧光计(Thermo Scientific)和Agilent Bioanalyzer 2100系统进行评估[32]。使用Illumina HiSeq 2500平台对文库进行双末端测序,测序工作由北京诺禾致源生物信息技术有限公司完成,原始下机数据是长度为250 bp的双端配对reads。
1.5 数据质控从Illumina HiSeq测序平台获得的原始下机数据(Raw paired-end reads),存在一定数量的干扰数据,为使分析结果更加准确、可靠,对得到的测序数据进行了一系列质量控制。首先根据barcode序列和引物序列将下机数据拆分到各个样品,之后切除barcode和引物序列,使用FLASH(V1.2.7)[33]将每个样品的Raw paired-end reads拼接起来,得到的拼接序列我们称之为Raw reads。根据QIIME(V1.9.1)[34]质量控制流程对Raw reads进行质量过滤,去除嵌合体和低质量的序列,得到高质量的有效数据(effective reads)。使用unoise3非聚类直接去噪的方法生成操作分类单元(operational taxonomic units,简称OTUs),丰度阈值设为10,即丢弃丰度小于10的reads。去噪后,利用Usearch软件(v11.0.667)[35]的usearch_global命令以97 %的相似性阈值计算OTU丰度以生成OTU表。使用BLAST(V2.7.1)将OTU代表序列与PR2数据库(V4.11.1)[36]中的序列进行比对以获得该OTU的分类信息。在注释之前,我们根据对Rotaliida的最新分类[37]更新和完善了PR2数据库[36]中底栖有孔虫的分类。
1.6 统计分析利用iNEXT软件包[38]和ggplot2软件包[39]绘制11个样品的稀释曲线rarefaction curve(图 2)。根据注释到不同有孔虫目的reads丰度和OTU丰度,利用ggplot2软件包[39]和reshape2软件包[40]绘制有孔虫群落组成柱状图bar chart(图 3)。基于获得的所有有孔虫属的丰度,利用pheatmap软件包[41]绘制热图heatmap(图 4)。在进行alpha多样性和beta多样性分析之前,以11个样品中最少的数据量(47823)为标准对数据进行均一化处理。基于均一化数据,利用QIIME(V1.9.1)[34]计算3个alpha多样性指数并用ggplot2软件包[39]绘制成柱状图bar chart(图 5)。根据11个站位的共有OTUs和特有OTUs,利用UpSetR软件包[42]绘制韦恩图venn diagram(图 6)。同样基于均一化数据,利用vegan软件包[43]计算11个样品之间的bray-curtis相似性系数和spearman's相关系数。基于bray-curtis相似性系数通过R(Version 2.15.3)绘制UPGMA聚类树clustering tree(图 7a),基于spearman's相关系数利用pheatmap软件包[41]绘制样品间群落结构相似性热图heatmap(图 7b)。
|
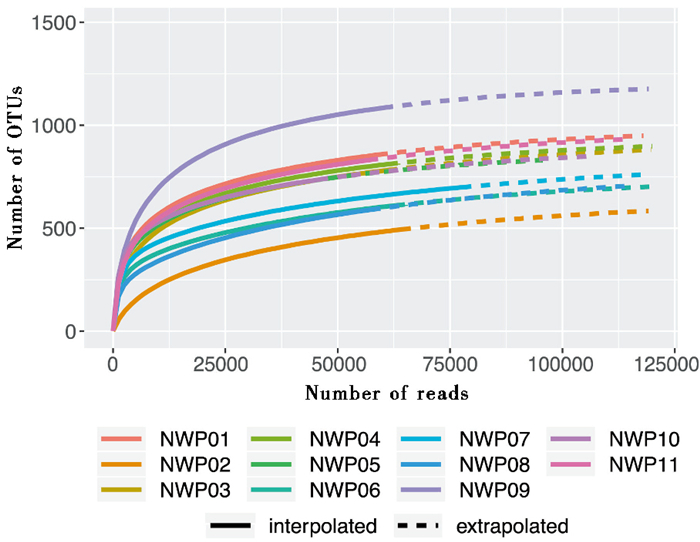
图 2 基于reads和OTUs数量绘制的稀释曲线 不同颜色的曲线表示不同的样品,实线是基于实际获得的测序数据所作,虚线是根据外推的数据所作 Fig. 2 Rarefaction curve based on the number of reads and OTUs. Different colored curves represent different samples. The solid lines were drawn based on the abundance of obtained data, and the dotted lines were drawn based on the abundance of extrapolated data |
|
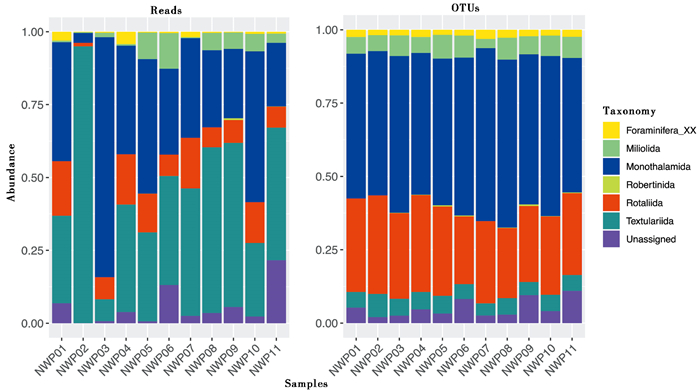
图 3 基于reads和OTUs绘制的目级水平物种相对丰度柱形图 Fig. 3 Proportions of reads and OTUs assigned to different foraminiferal order |

|
图 4 基于注释到各个有孔虫属的reads数绘制的有孔虫属级水平物种分布热图 横轴为站位,纵轴为不同的有孔虫属,左侧为聚类树;红色表示高丰度,蓝色表示低丰度;右侧图例表示变异范围为±1 Fig. 4 Heatmap based on the number of reads assigned to different foraminiferal genera. The horizontal axis represents the station, the vertical axis represents different genus. The left part is a clustering tree. Red color indicates high abundance and blue color indicates low abundance. The legend on the right indicates that the variation range is ±1 |
|
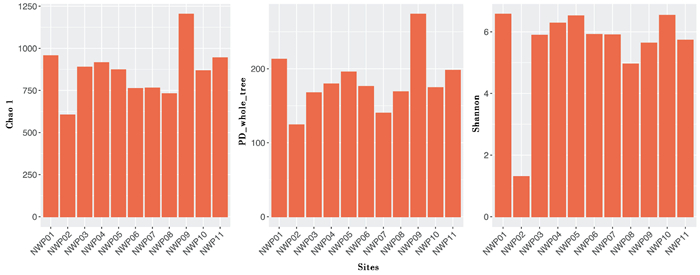
图 5 基于均一化处理后的数据计算所得的3个alpha多样性指数 横轴为站位,纵轴为指数大小 Fig. 5 The three alpha diversity indices calculated based on the homogenized data. The horizontal axis is the station, and the vertical axis is the index |

|
图 6 基于11个站位的共有OTUs和特有OTUs绘制的韦恩图 左侧绿色部分为各样本OTUs数据集大小;右侧红色点阵区域,为样本间交叉数据集,样本间若存在共有OTUs,则使用连线将样本相连;右侧蓝色柱状图表示样本间交叉数据集中OTUs的数量 Fig. 6 The Venn diagram drawn based on the common OTUs and unique OTUs of 11 stations. The green part on the left is the number of OTU in each sample. The red dot matrix area on the right is the cross dataset between samples. If there are common OTUs between the samples, the samples are connected with a line. The blue bar graph on the right indicates the number of OTUs between samples |

|
图 7 基于bray-curtis相似性系数绘制的UPGMA聚类树(7a)和基于spearman's相关系数绘制的样品间群落结构相似性热图(7b) 图中红色表示相似性高,蓝色表示相似性低;右侧图例表示变异范围为±1 Fig. 7 UPGMA clustering tree based on bray-curtis similarity coefficient(7a), and heatmap for showing the similarity of community structure between samples based on spearman's correlation coefficient(7b). Red indicates high similarity and blue indicates low similarity; the legend on the right indicates that the variation range is ±1 |
利用Illumina HiSeq测序平台对西北太平洋11个沉积物样品中的有孔虫DNA进行测序,共获得988949条Raw paired-end reads,拼接后得到762941条Raw reads,在对获得的Raw reads进行质量过滤后,得到702620条Effective reads,其中Effective reads数最多的是NWP07 (79542),最少的是NWP05 (50782)。利用unoise3降噪法生成OTU,共得到1548个OTUs。之后,利用usearch_global命令以97 %的相似性阈值计算OTU丰度并生成OTU表,最终OTU表中共保留了1536个OTUs和661388条reads。OTUs数量最多的站位是NWP09 (1085),最少的是NWP02 (494)(表 2)。
| 表 2 测序数据统计及质控结果 Table 2 Results of sequencing data and quality control |
稀释曲线能直接反映测序深度,检验样本的测序数据量是否合理。根据最终保留的reads数和OTUs数绘制了西北太平洋11个样品的稀释曲线(图 2)。此次11个样品的测序数据量均高于47000。由图 2可知,当随机抽取的reads数小于25000时,11个样品的代表曲线急剧升高,当reads数超过25000时,曲线变为缓慢升高,说明测序数据量渐进合理,更多的数据量只会产生少量新的物种(OTUs),之后随着reads数的不断增多,曲线逐渐趋向平坦,最终达到饱和状态,说明此次测序数据量足以覆盖样品中绝大部分物种(OTUs)。
2.2 分类组成和相对丰度利用BLAST对保留的1536个OTUs进行注释,共有1330个OTUs(624967条reads)被注释到有孔虫并获得了详细的分类信息,剩下的206个OTUs(36421条reads)未能在PR2数据库[36]中找到符合条件的注释结果,被归入Unassigned组。在获得注释结果的1330个OTUs中,Identity值(OTU代表序列与PR2数据库[36]中物种参考序列的相似度)的平均值为92.9 %,Identity值大于或等于97 %的OTUs仅274个,Identity值小于95 %的OTUs有839个,Identity值小于90 %的OTUs有358个。在有孔虫OTUs中,属于单房室软壳类有孔虫纲Monothalamea的OTUs最多,共721个,包含235873条reads;归入多房室硬壳类有孔虫纲Globothalamea的OTUs次之,共493个,其代表的reads数目最多,共有355586条;还有84个OTUs(25821条reads)被注释到Tubothalamea,32个OTUs(7687条reads)被注释到Foraminifera X。
分别基于reads数和OTUs数绘制了有孔虫在目级水平上的物种相对丰度柱形图(图 3)。总体来看,多房室胶结质有孔虫目Textulariida所占reads数最多,平均占比高达42 %,但归入该类群的OTUs数极少,平均占比仅为5 %;单房室软壳有孔虫目Monothalamida所占reads数略低于Textulariida,平均占比为36 %,但属于该类群的OTUs数最多,平均占比高达52 %;在传统研究中占主导地位的多房室钙质有孔虫目Rotaliida所占reads比仅为11 %,但归入该类群的OTUs平均占比达30 %。样品间注释到各个有孔虫目的reads数差异极大:在NWP01、NWP03、NWP04、NWP05和NWP10这5个站位,Monothalamida所占reads比最高,其中在NWP03,Monothalamida所占reads比接近82 %;在其余6个站位,Textulariida所占reads比最高,其中在NWP02,Textulariida所占reads比高达95 %。样品间的OTUs组成和相对丰度相差无几,均以Monothalamida为主。Textulariida在reads中占比高,在OTUs中占比极小,很可能是该类群在西北太平洋深海中数量多,但种类稀少;而Monothalamida在reads和OTUs中占比都极高,说明该古老类群的多样性和相对丰度在该海区有孔虫群落中均占绝对优势。
2.3 群落变化与地理分布由热图(图 4)可知,在获得的所有有孔虫属中,有1/3在11个深海站位中均具有极高的丰度,如多房室胶结质类Trochammina,单房室软壳类Saccamminidae_X、Psammophaga、Nemogullmia、Vanhoeffenella、Ovammina等,说明这些有孔虫类群能很好地适应西北太平洋深海环境,是该海区的广布属。有1/3有孔虫属以极低的丰度分布在所有站位,如单房室软壳类Psammosphaera、Cribrothalammina、Rhabdammina,这表明西北太平洋深海环境不利于这些类群的生存和繁殖。还有1/3有孔虫属在少数几个站位丰度极高,而在其他站位丰度极低,例如Arnoldiellina、Crithionina和Eggerelloides等,这些类群在西北太平洋深海呈明显的斑块性分布。
基于均一化数据,计算了11个站位的3个alpha多样性指数:Chao 1、PD_whole_tree和Shannon指数(图 5)。结果显示,11个站位之间有孔虫群落的alpha多样性存在明显差异,表明有孔虫群落分布具有显著的空间异质性。NWP09站位的3个指数都较大,说明该站位有孔虫群落的物种多样性较高;NWP02站位的3个指数均最小,说明该站位有孔虫群落不仅物种(OTU)丰度低,且物种(OTU)均匀度也低。
根据共有OTUs和特有OTUs绘制的韦恩图(图 6)显示:11个站位的共有OTUs有125个,特有OTUs数最多的站位是NWP09,有33个,其次是NWP07,有28个,NWP01仅有9个特有OTUs,NWP02、NWP03、NWP04和NWP08这4个站位各自的特有OTUs少于8个;NWP01、NWP02、NWP03、NWP04、NWP05、NWP09和NWP11这7个站位共有且特有的(在其余4个站位未出现)的OTUs有54个;仅在NWP06、NWP07、NWP08、NWP09和NWP10这5个站位出现,在其余6个站位未出现的OTUs有46个;只在NWP01、NWP02、NWP03、NWP04、NWP05和NWP11这6个站位出现,在其余5个站位未出现的OTUs有41个。由UPGMA聚类树(图 7a)和样品间群落结构相似性热图(图 7b)可知,11个样品可以分为两组:一组包括NWP01、NWP02、NWP03、NWP04、NWP05和NWP11;另一组包括NWP06、NWP07、NWP08、NWP09和NWP10。前一组样品(除NWP11)采自纬度位于34°N至36°N之间、水深在5100 m至5800 m的深水区;后一组样品(除NWP7)采自纬度在32.5°N附近、水深4400 m至5000 m的相对较浅水区。这表明西北太平洋深海底栖有孔虫的群落结构随纬度和水深变化而产生了明显差异。
3 讨论 3.1 深海底栖有孔虫的DNA分子多样性关于深海真核微生物多样性的研究显示,深海中真核微生物的多样性极为丰富,且存在大量未知种类[1, 44~45]。Zhao等[46]利用高通量测序技术研究了西太平洋海山及邻近深海平原的沉积物中纤毛虫的多样性,结果显示有73 %的OTUs与数据库中参考序列的相似性小于97 %,有25 %的OTUs与参考序列的相似性小于90 %,说明西太平洋海山及邻近深海平原的纤毛虫具有极高的遗传新颖性。Cordier等[47]在西北太平洋千岛-堪察加海沟的5个站位(水深5741 m至9444 m)采集了18个沉积物样品,利用有孔虫特异性引物s14F1和s17扩增了有孔虫的DNA片段,通过高通量测序技术检测了该海区底栖有孔虫的分子多样性,结果显示在丰度最高的100个OTUs中,只有5个OTUs与数据库中参考序列的相似度达到100 %,有51个OTUs未匹配到相似度大于90 %的参考序列,这说明即便是在深海底栖有孔虫的优势类群中也存在大量至今未知的种类。
本研究采用有孔虫特异性引物s14F3和s17[5, 30~31]扩增了有孔虫特有可变区的DNA片段,并且在数据处理过程中,对得到的序列进行了严格的质量控制[34],过滤掉了嵌合体和低质量的干扰数据,尽可能保证最终保留的reads和OTUs属于有孔虫。在使用BLAST将OTUs的代表序列与PR2数据库[36]中的参考序列进行比对后,仅有20.6 %的有孔虫OTUs的Identity值大于或等于97 %,有63.1 %的有孔虫OTUs的Identity值小于95 %,有26.9 %的有孔虫OTUs的Identity值小于90 %,这说明西北太平洋深海底栖有孔虫与目前检获到的有孔虫之间存在较明显的遗传差异性。在基于DNA的真核微生物遗传分类中,一般将序列相似度大于95 %的个体定义为同一物种[48]。未能在PR2数据库[36]中找到高度相似序列(Identity值>95 %)的有孔虫OTUs(约63.1 %)极有可能代表的是至今未被发现的“新物种”或新的高级分类阶元[46~47]。上述研究均表明目前人类所掌握的深海底栖有孔虫的遗传资料仅仅是其遗传基因库的冰山一角,深海有孔虫的分子系统分类学研究任重而道远。
3.2 深海底栖有孔虫群落组成的影响因素Lecroq等[25]利用Illumina测序技术评估了采自5个深海区域的31个未筛分的沉积物样品中的有孔虫多样性和群落组成,他们发现在传统有孔虫研究中经常被忽视的单房室软壳类有孔虫Monothalamids具有超乎想像的丰富度,有约40 %的OTUs被注释到该类群。之后,研究人员利用高通量测序技术对全球不同深海生境中的有孔虫群落进行了调查,结果均显示单房室软壳类有孔虫在群落中占据绝对优势[47, 49~50]。本文共调查了11个西北太平洋站位,平均水深超5000 m,最浅4080 m,最深5830 m,结果显示有54 %的有孔虫OTUs注释到单房室软壳类。
在水深超过4000 m的深海,碳酸盐溶解是影响深海底栖生物生存的一个重要因素[26]。随着水深的增加,碳酸盐的溶解度逐渐增加,在碳酸盐补偿深度以下(CCD:通常>4000~5500 m,在南极洲的某些地区较浅),钙质类群难以形成外壳,因此底栖动物群落几乎完全由具有胶结质或有机质壳体的类群组成[14]。深海除高压、低温和恒定的盐度外,最显著的特征是缺乏光照,无法进行光合作用,因此深海沉积物是食物十分受限制的环境[51]。深海初级生产仅限于热液和冷泉附近的化能合成作用,除此之外,绝大多数深海生物直接或间接地依靠从海表面沉降下来的浮游植物初级生产产物来维持生命活动[14, 52]。东北太平洋M站的投喂实验证实:单房室有孔虫占较高比例可能与它们对源自海表面浮游植物生产的新鲜有机物的依赖性低有关,与多房室特别是钙质类群相比,单房室软壳类有孔虫“saccamminids”对所提供的13 C标记的硅藻未表现出明显的反应[53]。上述因素都导致钙质类群在深海有孔虫群落中的比例低,单房室软壳类有孔虫占据优势。
3.3 深海底栖有孔虫的空间分布特点我们的结果还显示站位间有孔虫的DNA分子多样性、群落组成和分布特点存在显著差异:在1330个有孔虫OTUs中,11个站位的共有OTUs仅125个(图 6);站位间OTUs数目差异巨大,最多的有1085个,最少的仅494个(表 2);有1/3有孔虫属仅集中分布在少数站位,表现出明显的地理限定性;各个站位的物种组成(图 3)和alpha多样性(图 5)也大不相同。上述结果均表明西北太平洋深海底栖有孔虫的分布呈明显的空间异质性。
空间异质性是深海底栖生物群落的一个典型分布特征,这种特征在小到1 m或大到几千米的空间尺度上均存在,研究人员已证实无论是大型底栖动物,如多毛类、双壳类等[54]、线虫[55],还是有孔虫[56],它们在深海的分布都表现出这种复杂性和不均匀性。Lejzerowicz等[49]利用高通量测序技术调查了南大洋深海底栖有孔虫的分子多样性和空间分布特征,结果表明:即使是取自同一站位、同一采样管的3个子样品之间OTUs和reads的相对丰度和组成也存在显著差异。深海有孔虫的这种分布特征通常与沉积物特性、生物扰动、食物碎屑的斑块性沉降有关[12, 56]。深海底栖生物分布的这种局部高度差异性增加了对其多样性调查的难度[49]。就深海底栖有孔虫调查而言,无论是传统的镜检法还是新兴的高通量测序技术,研究人员都是取小范围、一定量的沉积物作为样本,有孔虫的斑块性分布很可能导致某些重要的物种未能被样本所包含,这样就导致了调查结果与真实情况之间存在偏差[17]。例如,底栖有孔虫Epistominella exigua是典型的深海种,在各大洋均有分布,但是以往的研究表明并非每克深海沉积物样品中都能检测到该物种的基因[57]。虽然无法完全消除空间异质性导致的偏差,但可以通过扩大采样范围、采集多个重复样本、反复多次PCR等措施来降低它的影响,更确切地反映物种的真实多样性和空间分布情况[49]。
3.4 高通量测序技术的不足尽管高通量测序技术一直在不断发展和改进,但是目前依然存在许多不可忽视的缺陷和有待完善的地方[17]。核糖体基因是研究真核生物分子多样性最常用的基因,SSU rDNA被证实能够鉴别有孔虫绝大多数种类,但是不同可变区域对不同有孔虫种类的识别能力不同[58]。较短的片段PCR扩增较容易,成功率更高,能够尽可能多地将环境样品中包含的有孔虫种类鉴别出来,但是片段越短其包含的序列信息也就越少,很难进行其他分析,比如区分有孔虫基因型、确定分化时间等[59]。高通量测序数据分析过程中,OTU聚类时选择的序列相似性阈值会严重影响有孔虫多样性的评估结果[17]。不同的研究采用的序列相似性阈值往往不同,如95 %或98 % [60]、96 % [25]和97 % [61]都被采用过,序列相似性阈值的不一致性导致不同研究之间无法进行OTUs相对丰度的比较。OTU的分类学解释取决于现有参考数据库的完整性和所选条形码的分类学解决方案。分类信息不全是目前数据库普遍存在的一个问题[62]。PR2数据库[36]和PFR2数据库[63]是有孔虫序列注释常用的2个数据库,它们针对性强、分类信息准确性高,但仍未能涵盖所有有孔虫种类的参考序列。高通量测序技术在有孔虫分子生物学研究中存在的这些不足极大地阻碍了对有孔虫在全球范围内的分子多样性和地理分布模式的探索,而解决这些问题将是一项艰巨而长久的任务。
4 结论本工作利用高通量测序技术测定了西北太平洋深海11个站位的表层沉积物中的有孔虫DNA,分析了该海区有孔虫的多样性和群落组成特征,得到以下结论:
(1) 共获得1536个OTUs,有1330个OTUs成功注释到有孔虫。其中,注释到Monothalamids的OTUs最多,有721个,这说明该海区的优势类群不是传统形态学中研究较多的多房室钙质类有孔虫,而是起源最早但研究较少的单房室软壳类有孔虫。
(2) 在1330个有孔虫OTUs中,Identity值大于或等于97 %的OTUs只有274个,Identity值小于95 %的OTUs有839个,这表明西北太平洋深海底栖有孔虫的DNA与现存数据库中已有的有孔虫DNA之间存在高度的遗传差异性,该海区极可能蕴藏着大量至今未被发现的“新物种”甚至新的高级分类阶元。
(3) 在获得的有孔虫属中,广布属和稀有属各占1/3,还有1/3有孔虫属呈典型的斑块性分布,且站位间的有孔虫群落结构也表现出明显的差异性。
致谢: 感谢中国科学院海洋研究所“科学”号考察船的全体队员在航次中给予的无私帮助,感谢实验员王雪皎、郑萌萌、曹丽娜协助工作。
| [1] |
赵峰, 徐奎栋. 深海真核微生物多样性研究进展[J]. 地球科学进展, 2014, 29(5): 551-558. Zhao Feng, Xu Kuidong. Advances in the diversity of microbial eukaryotes in deep sea[J]. Advances in Earth Science, 2014, 29(5): 551-558. |
| [2] |
Sinniger F, Pawlowski J, Harii S, et al. Worldwide analysis of sedimentary DNA reveals major gaps in taxonomic knowledge of deep-sea benthos[J]. Frontiers in Marine Science, 2016, 3: 92. DOI:10.3389/fmars.2016.00092 |
| [3] |
Gooday A J, Levin L A, Linke P, et al. The role of benthic foraminifera in deep-sea food webs and carbon cycling[M]//Rowe G T, Pariente V eds. Deep-Sea Food Chains and the Global Carbon Cycle. Kluwer, Dordrecht: NATO ASI Ser., C360, 1992: 63-91. doi: 10.1007/978-94-011-2452-2_5.
|
| [4] |
Nomaki H, Heinz P, Nakatsuka T, et al. Species-specific ingestion of organic carbon by deep-sea benthic foraminifera and meiobenthos:In situ tracer experiments[J]. Limnology and Oceanography, 2005, 50(1): 134-146. DOI:10.4319/lo.2005.50.1.0134 |
| [5] |
Shi J, Lei Y, Li Q, et al. Molecular diversity and spatial distribution of benthic foraminifera of the seamounts and adjacent abyssal plains in the tropical Western Pacific Ocean[J]. Marine Micropaleontology, 2020, 156: 101850. https://doi.org/10.1016/j.marmicro.2020.101850.
|
| [6] |
Goldstein S T. Foraminifera: A biological overview[M]//Sen Gupta B K ed. Modern Foraminifera. Dordrecht: Springer, 1999: 37-55. doi: 10.1007/0-306-48104-9_3.
|
| [7] |
Murray J W. Ecology and Palaeoecology of Benthic Foraminifera[M]. New York: Longman Scientific & Technical, 1991: 397. DOI:10.4324/9781315846101
|
| [8] |
Levin L A. Interactions between metazoans and large, agglutinated protozoans:Implications for the community structure of deep-sea benthos[J]. American Zoologist, 1991, 31(6): 886-900. DOI:10.1093/icb/31.6.886 |
| [9] |
张江勇, 汪品先. 深海研究中的底栖有孔虫:回顾与展望[J]. 地球科学进展, 2004, 19(4): 545-551. Zhang Jiangyong, Wang Pinxian. Benthic foraminifera in deep-sea research:Retrospect and prospect[J]. Advances in Earth Science, 2004, 19(4): 545-551. DOI:10.3321/j.issn:1001-8166.2004.04.009 |
| [10] |
李铁刚, 李绍全, 苍树溪, 等. YSDP102钻孔有孔虫动物群与南黄海东南部古水文重建[J]. 海洋与湖沼, 2000, 31(6): 588-595. Li Tiegang, Li Shaoquan, Cang Shuxi, et al. Paleo-hydrological reconstruction of the Southern Yellow Sea inferred from foraminiferal fauna in core YSDP102[J]. Oceanologia et Limnologia Sinica, 2000, 31(6): 588-595. DOI:10.3321/j.issn:0029-814X.2000.06.002 |
| [11] |
翦知盡, 陈荣华, 李保华. 冲绳海槽南部20 ka来深水底栖有孔虫的古海洋学记录[J]. 中国科学(D辑), 1996, 26(5): 467-473. Jian Zhimin, Chen Ronghua, Li Baohua. Paleoceanography record of deep-sea benthic foraminifera in the Southern Okinawa Trough over the last 20000 years[J]. Science in China(Series D), 1996, 26(5): 467-473. DOI:10.3321/j.issn:1006-9267.1996.05.008 |
| [12] |
Gooday A J. The biology of deep-sea foraminifera:A review of some advances and their applications in paleoceanography[J]. Palaios, 1994, 9(1): 14-31. DOI:10.2307/3515075 |
| [13] |
Lei Y, Li T, Nigam R, et al. Environmental significance of morphological variations in the foraminifer Ammonia aomoriensis (Asano, 1951) and its molecular identification:A study from the Yellow Sea and East China Sea, PR China[J]. Palaeogeography, Palaeoclimatology, Palaeoecology, 2017, 483: 49-57. DOI:10.1016/j.palaeo.2016.05.010 |
| [14] |
Gooday A J. Benthic foraminifera(Protista) as tools in deep-water palaeoceanography:Environmental influences on faunal characteristics[J]. Advances in Marine Biology, 2003, 46: 1-90. DOI:10.1016/S0065-2881(03)46002-1 |
| [15] |
Loeblich A R, Tappan H. Foraminiferal genera and their classification[M]. New York: Van Nostrand Reinhold, 1988: 970. DOI:10.1007/978-1-4899-5760-3
|
| [16] |
Pawlowski J, Fahrni J, Lecroq B, et al. Bipolar gene flow in deep-sea benthic foraminifera[J]. Molecular Ecology, 2007, 16(19): 4089-4096. DOI:10.1111/j.1365-294X.2007.03465.x |
| [17] |
Pawlowski J, Lejzerowicz F, Esling P. Next-generation environmental diversity surveys of foraminifera:Preparing the future[J]. The Biological Bulletin, 2014, 227(2): 93-106. DOI:10.1086/BBLv227n2p93 |
| [18] |
Sen A, Bhadury P. Molecular diversity of benthic foraminifera from tropical and subtropical coastal settings-A concise review[J]. Indian Journal of Geo-Marine Sciences, 2015, 44(7): 951-960. |
| [19] |
Gooday A J, Goineau A. The contribution of fine sieve fractions (63-150μm) to foraminiferal abundance and diversity in an area of the eastern Pacific Ocean licensed for polymetallic nodule exploration[J]. Frontiers in Marine Science, 2019, 6: 114. DOI:10.3389/fmars.2019.00114 |
| [20] |
Kurbjeweit F, Schmiedl G, Schiebel R, et al. Distribution, biomass and diversity of benthic foraminifera in relation to sediment geochemistry in the Arabian Sea[J]. Deep-Sea Research Part Ⅱ:Topical Studies in Oceanography, 2000, 47(14): 2913-2955. DOI:10.1016/S0967-0645(00)00053-9 |
| [21] |
Rathburn A E, Corliss B H. The ecology of living(stained) deep-sea benthic foraminifera from the Sulu Sea[J]. Paleoceanography, 1994, 9(1): 87-150. DOI:10.1029/93PA02327 |
| [22] |
Schrøder C J, Scott D B, Medioli F S. Can smaller benthic foraminifera be ignored in paleoenvironmental analyses?[J]. Journal of Foraminiferal Research, 1987, 17(2): 101-105. DOI:10.2113/gsjfr.17.2.101 |
| [23] |
Shokralla S, Spall J L, Gibson J F, et al. Next-generation sequencing technologies for environmental DNA research[J]. Molecular Ecology, 2012, 21(8): 1794-1805. DOI:10.1111/j.1365-294X.2012.05538.x |
| [24] |
Goineau A, Gooday A J. Diversity and spatial patterns of foraminiferal assemblages in the eastern Clarion-Clipperton zone (abyssal eastern equatorial Pacific)[J]. Deep-Sea Research Part Ⅰ:Oceanographic Research Research Papers, 2019, 149: 103036. DOI:10.1016/j.dsr.2019.04.014 |
| [25] |
Lecroq B, Lejzerowicz F, Bachar D, et al. Ultra-deep sequencing of foraminiferal microbarcodes unveils hidden richness of early monothalamous lineages in deep-sea sediments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(32): 13177-13182. DOI:10.1073/pnas.1018426108 |
| [26] |
Goineau A, Gooday A J. Novel benthic foraminifera are abundant and diverse in an area of the abyssal equatorial Pacific licensed for polymetallic nodule exploration[J]. Scientific Reports, 2017, 7: 45288. DOI:10.1038/srep45288 |
| [27] |
Nozawa F, Kitazato H, Tsuchiya M, et al. 'Live' benthic foraminifera at an abyssal site in the equatorial Pacific nodule province:Abundance, diversity and taxonomic composition[J]. Deep-Sea Research Part Ⅰ:Oceanographic Research Research Papers, 2006, 53(8): 1406-1422. DOI:10.1016/j.dsr.2006.06.001 |
| [28] |
Gooday A J, Holzmann M, Goineau A, et al. Five new species and two new genera of xenophyophores (Foraminifera:Rhizaria) from part of the abyssal equatorial Pacific licensed for polymetallic nodule exploration[J]. Zoological Journal of the Linnean Society, 2018, 183(4): 723-748. DOI:10.1093/zoolinnean/zlx093 |
| [29] |
Ohkawara N, Kitazato H, Uematsu K, et al. A minute new species of Saccammina (monothalamous Foraminifera; Protista) from the abyssal Pacific[J]. Journal of Micropalaeontology, 2009, 28(2): 143-151. DOI:10.1144/jm.28.2.143 |
| [30] |
Esling P, Lejzerowicz F, Pawlowski J. Accurate multiplexing and filtering for high-throughput amplicon-sequencing[J]. Nucleic Acids Research, 2015, 43(5): 2513-2524. DOI:10.1093/nar/gkv107 |
| [31] |
Holzmann M, Habura A, Giles H, et al. Freshwater foraminiferans revealed by analysis of environmental DNA samples[J]. Journal of Eukaryotic Microbiology, 2003, 50(2): 135-139. DOI:10.1111/j.1550-7408.2003.tb00248.x |
| [32] |
Zhao F, Xu K. Molecular diversity and distribution pattern of ciliates in sediments from deep-sea hydrothermal vents in the Okinawa Trough and adjacent sea areas[J]. Deep-Sea Research Part Ⅰ:Oceanographic Research Research Papers, 2016, 116: 22-23. DOI:10.1016/j.dsr.2016.07.007 |
| [33] |
Magoč T, Salzberg S L. FLASH:Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [34] |
Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [35] |
Edgar R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [36] |
Guillou L, Bachar D, Audic S, et al. The Protist Ribosomal Reference database(PR2): A catalog of unicellular eukaryote small sub-unit rRNA sequences with curated taxonomy[J]. Nucleic Acids Research, 2013, 41(Database issue): D597-D604. doi: 10.1093/nar/gks1160.
|
| [37] |
Holzmann M, Pawlowski J. An updated classification of rotaliid foraminifera based on ribosomal DNA phylogeny[J]. Marine Micropaleontology, 2017, 132: 18-34. DOI:10.1016/j.marmicro.2017.04.002 |
| [38] |
Hsieh T C, Ma K H, Chao A. iNEXT:An R package for rarefaction and extrapolation of species diversity (Hill numbers)[J]. Methods in Ecology and Evolution, 2016, 7: 1451-1456. DOI:10.1111/2041-210X.12613 |
| [39] |
Wickham H. Ggplot 2:Elegant Graphics for Data Analysis[M]. New York: Springer Verlag, 2016: 1-260. DOI:10.1007/978-0-387-98141-3
|
| [40] |
Wickham H. Package 'reshape2'. CRAN, 2019: 1-14[DB]. Retrieved fr om: https://cran.r-project.org/web/packages/reshape2/reshape2.pdf.
|
| [41] |
Kolde R. Package 'pheatmap'. CRAN, 2019: 1-8[DB]. Retrieved from: https://cran.r-project.org/web/packages/pheatmap/pheatmap.pdf.
|
| [42] |
Conway J R, Lex A, Gehlenborg N. UpSetR:An R package for the visualization of intersecting sets and their properties[J]. Bioinformatics, 2017, 33(18): 2938-2940. DOI:10.1093/bioinformatics/btx364 |
| [43] |
Oksanen J, Blanchet F G, Friendly M, et al. Package 'vegan'. CRAN, 2019: 1-296[DB]. Retrieved from: https://cran.r-project.org/web/packages/vegan/vegan.pdf.
|
| [44] |
Rothe N, Gooday A J, Cedhagen T, et al. Biodiversity and distribution of the genus Gromia (Protista, Rhizaria) in the deep Weddell Sea(Southern Ocean)[J]. Polar Biology, 2011, 34(1): 69-81. |
| [45] |
Scheckenbach F, Hausmann K, Wylezich C, et al. Large-scale patterns in biodiversity of microbial eukaryotes from the abyssal sea floor[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(1): 115-120. DOI:10.1073/pnas.0908816106 |
| [46] |
Zhao F, Filker S, Stoeck T, et al. Ciliate diversity and distribution patterns in the sediments of a seamount and adjacent abyssal plains in the tropical Western Pacific Ocean[J]. BMC Microbiology, 2017, 17: 192. DOI:10.1186/s12866-017-1103-6 |
| [47] |
Cordier T, Barrenechea I, Lejzerowicz F, et al. Benthic foraminiferal DNA metabarcodes significantly vary along a gradient from abyssal to hadal depths and between each side of the Kuril-Kamchatka trench[J]. Progress in Oceanography, 2019, 178: 102175. DOI:10.1016/j.pocean.2019.102175 |
| [48] |
Caron D A, Countway P D, Savai P, et al. Defining DNA-based operational taxonomic units for microbial-eukaryote ecology[J]. Applied and Environmental Microbiology, 2009, 75(18): 5797-5808. |
| [49] |
Lejzerowicz F, Esling P, Pawlowski J. Patchiness of deep-sea benthic Foraminifera across the Southern Ocean:Insights from high-throughput DNA sequencing[J]. Deep-Sea Research Part Ⅱ:Topical Studies in Oceanography, 2014, 108: 17-26. DOI:10.1016/j.dsr2.2014.07.018 |
| [50] |
Lejzerowicz F, Esling P, Majewski W, et al. Ancient DNA complements microfossil record in deep-sea subsurface sediments[J]. Biology Letters, 2013, 9(4): 20130283. |
| [51] |
Tyler P A. Conditions for the existence of life at the deep-sea floor:An update[J]. Oceanography and Marine Biology, An Annual Review, 1995, 33: 221-244. DOI:10.1016/0029-8018(95)80003-4 |
| [52] |
Herguera J C, Berger W H. Paleoproductivity from benthic foraminifera abundance:Glacial to postglacial change in the west-equatorial Pacific[J]. Geology, 1991, 19(12): 1173-1176. DOI:10.1130/0091-7613(1991)019<1173:PFBFAG>2.3.CO;2 |
| [53] |
Enge A J, Nomaki H, Ogawa N O, et al. Response of the benthic foraminiferal community to a simulated short-term phytodetritus pulse in the abyssal North Pacific[J]. Marine Ecology Progress Series, 2011, 438: 129-142. DOI:10.3354/meps09298 |
| [54] |
McClain C R, Nekola J C, Kuhnz L, et al. Local-scale faunal turnover on the deep Pacific seafloor[J]. Marine Ecology Progress Series, 2011, 422: 193-200. DOI:10.3354/meps08924 |
| [55] |
Danovaro R, Carugati L, Corinaldesi C, et al. Multiple spatial scale analyses provide new clues on patterns and drivers of deep-sea nematode diversity[J]. Deep-Sea Research Part Ⅱ:Topical Studies in Oceanography, 2013, 92: 97-106. DOI:10.1016/j.dsr2.2013.03.035 |
| [56] |
Griveaud C, Jorissen F, Anschutz P. Spatial variability of live benthic foraminiferal faunas on the Portuguese margin[J]. Micropaleontology, 2010, 56(3-4): 297-322. |
| [57] |
Lecroq B, Gooday A J, Pawlowski J. Global genetic homogeneity in deep-sea foraminiferan Epistominella exigua (Rotaliida:Pseudoparrellidae)[J]. Zootaxa, 2009, 2096(1): 23-32. DOI:10.11646/zootaxa.2096.1.4 |
| [58] |
Pawlowski J, Lecroq B. Short rDNA barcodes for species identification in foraminifera[J]. Journal of Eukaryotic Microbiology, 2010, 57(2): 197-205. |
| [59] |
Ujiié Y, Ishitani Y. Evolution of a planktonic foraminifer during environmental changes in the tropical oceans[J]. PLoS One, 2016, 11(2): e0148847. |
| [60] |
Lejzerowicz F, Pawlowski J, Fraissinet-Tachet L, et al. Molecular evidence for widespread occurrence of Foraminifera in soils[J]. Environmental Microbiology, 2010, 12(9): 2518-2526. DOI:10.1111/j.1462-2920.2010.02225.x |
| [61] |
Bernhard J M, Edgcomb V P, Visscher P T, et al. Insights into foraminiferal influences on microfabrics of microbialites at Highborne Cay, Bahamas[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(24): 9830-9834. DOI:10.1073/pnas.1221721110 |
| [62] |
Nowrousian M. Next-generation sequencing techniques for eukaryotic microorganisms:Sequencing-based solutions to biological problems[J]. Eukaryotic Cell, 2010, 9(9): 1300-1310. DOI:10.1128/EC.00123-10 |
| [63] |
Morard R, Darling K F, Mahé S, et al. PFR2:A curated database of planktonic foraminifera 18S ribosomal DNA as a resource for studies of plankton ecology, biogeography and evolution[J]. Molecular Ecology Resources, 2015, 15(6): 1472-1485. DOI:10.1111/1755-0998.12410 |
2 Key Laboratory of Marine Sedimentology and Environment Geology, First Institute of Oceanography, Ministry of Natural Resources, Qingdao 266061, Shandong;
3 University of Chinese Academy of Sciences, Beijing 100049;
4 Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, Shandong)
Abstract
The deep sea has become a strategic area for countries around the world to develop natural resources, but the diversity and distribution of abyssal organisms are still poorly known. By using high-throughput sequencing technology, we evaluated the molecular diversity of benthic foraminifera, a major component of abyssal communities and a useful proxy in paleoceanography, in the northwest Pacific Ocean(32.2°~36.0°N, 145.5°~156.5°E). We collected eleven sediment samples from the northwest Pacific Ocean with water depths between 4080 and 5830 m and extracted the total genomic DNA from each sample. Foraminiferal DNA fragments were amplified with foraminiferal-specific primers and were sequenced by high-throughput sequencing technology. Finally, we obtained a total of 1536 operational taxonomic units(OTUs). 1330 OTUs were successfully assigned to Foraminifera, of which 721 OTUs(about 54%)were placed to early(ancient)lineages of soft-walled, single-chambered(monothalamous)foraminifera. The mean identity value of all detected foraminiferal OTUs was 92.9%. Only 20.6% of OTUs had an identity value greater than or equal to 97% and 63.1% of OTUs had an identity value less than 95%. This indicated that the benthic foraminifera in our survey regions had great genetic divergence with those species obtained in previous studies and the northwest Pacific Ocean might contain a large number of "new species" or even high-level taxonomic groups. A third of foraminiferal genera obtained in our study had spatial heterogeneity, and the foraminiferal community structure in different sites showed obvious variation. This work is the first attempt to investigate deep-sea benthic foraminifera in the northwest Pacific Ocean using high-throughput sequencing technology in China and aims to provide bioecological and genetic data for studying the global geographic distribution of foraminifera and exploring deep-sea resources. 2020, Vol.40
2020, Vol.40

