形状是人类认知物体的一个重要线索。某些情况下,物体可能并不具备亮度、纹理或颜色等信息,只能通过其形状来表达。仅通过形状信息,人类仍然可以轻松识别不同的物体及其类别。而且,形状对于物体的颜色、光照和纹理的变化是具有稳定性的[1],也可以作为其他物体表示的一种辅助,以提高物体表示及识别的准确性。由于形状信息具备上述优点,基于形状的物体高效表示及识别一直是研究的热点。
计算机视觉中研究的形状一般分为二维(平面)形状和三维形状两种数据类型,两类形状有多种应用领域。其中,二维形状被广泛地应用于诸如商标检索、指纹表示及识别、物体定位、图像检索等领域中[2]。这些应用的一个基本问题是如何形成一种丰富、简洁和高效的形状特征表示[3]。典型的二维形状表示方法可以大体上分为三类:基于轮廓的形状表示、基于区域的形状表示和基于骨架的形状表示。本文主要关注的是基于轮廓和基于骨架的二维形状表示及识别方法。
形状的轮廓提供了形状大部分的整体及细节信息,是形状表示中最为频繁采用的对象。基于轮廓的形状表示[1-2]多数把轮廓进行离散化采样,通过统计轮廓序列上采样点的空间位置分布关系,来提取形状轮廓上一些稳定的特征量,一般称为形状特征描述符。这些特征量可以是全局的[3]或者是局部的[1],其中最为典型的特征子应该是形状上下文(Shape Context, SC)[1]。形状上下文在每个采样点上,利用二维直方图统计其余采样点相对于该点的距离和角度变化信息。两个形状之间的相似性通过各自采样点集的最优匹配计算,采样点之间的匹配误差由其形状上下文描述向量衡量。实验表明,基于轮廓的形状表示方法,在形状简单变化时具备较好的稳定性,可以有效应对形状中的噪声和轻度的变形。
不过在形状发生一些等距变换(如关节变换)时,基于轮廓的表示方法往往稳定性不好,而此时形状骨架的拓扑结构会保持不变。可以借助骨架提出形状更简洁且稳定的描述方法。一般来说,骨架是形状内所有最大内切圆圆心的集合,提供了形状的厚度沿骨架的变化信息,对非脊变形和关节具有不变性,且对于轮廓上的噪声比较鲁棒。这些优点使得基于骨架的形状表示独具优势。
然而现实中的形状往往存在较多变形,如放缩、遮挡、大尺度噪声、同一形状的多样性等,给形状的准确表示和识别带来很大挑战。近期,二维形状方面的研究进展包括基于深度神经网络的方法[4]和基于生物信息学的方法[5]等。其中,深度神经网络一般需要大规模的标记样本,而二维形状公开数据集的样本规模往往较小,制约了深度神经网络在二维形状研究领域中的推广。与传统研究方法不同,Bicego和Lovato[5]提出了利用生物信息学模型解决二维形状的识别问题,其基本思想是把形状轮廓按照其空间分布编码为生物信息序列,即DNA分子序列,然后借助标准的生物信息序列分析工具来进行序列之间的相似性分析,以及在此基础上的形状识别或分类。
而目前两个形状之间的相似性分析,通用的方法还是借助于其特征描述点集之间的最优匹配。匹配过程由传统的动态规划算法实现,存在算法复杂度较高且匹配精确度不够等问题。利用生物信息序列分析工具替代传统基于动态规划的匹配方法,无疑将有助于丰富轮廓之间相似性的计算方法,同时,生物序列相似性匹配具有几十年的研究历史,涌现出了多种针对不同场景的匹配算法。所以,利用生物信息序列分析方法,进行二维形状分析显然颇具新意,是一个有不错前景且值得进一步深入研究的方向。
基于上述思路,引入生物信息序列分析方法到二维形状分析及识别中。研究中发现,轮廓中的细长分支会使得生物信息编码序列中出现大量方向相反且长度相近的编码,这些编码对于形状相似性匹配贡献较少,且有时会制约匹配的精度。区别于Bicego和Lovato[5]的工作,提出了结合形状轮廓和骨架的协同编码方案。
本文的主要贡献:①提出了一种新型的二维形状表示及识别方法,该方法基于生物信息学,把二维形状转化为生物信息序列,可以借助生物信息学领域的序列匹配方法提高二维形状的识别精度。②提出了一种二维形状的轮廓及骨架协同编码的方案。该方案利用骨架表示形状中的细长分支,并分别对轮廓和骨架进行不同类型的编码,具备编码简洁、后续匹配准确性高等优点。同时在基于草图图像检索(SBIR)也有一定的应用前景[6]。③在三个公开形状数据集上做了大量的形状识别实验,并采用不同的准确性评价指标,与多种通用形状识别方法进行了详细的比较,证明了本文方法的优势和有效性。
1 形状描述及匹配首先简单介绍一些典型的基于轮廓和基于骨架的二维形状表示及识别方法,然后给出在形状匹配中所采用的生物信息序列分析工具的相关概述。
1.1 基于轮廓的形状描述基于轮廓的形状表示方法是二维形状描述中最为通用的一类方法,在二维形状识别中有着广泛的应用。其描述方法的共同特点是通过统计轮廓序列上点的空间位置分布关系来提取形状轮廓某种不变特征量。
Belongie等[1]根据轮廓点空间位置关系提出了一种形状上下文的描述方法。该描述方法对轮廓序列上的每个点采用一个向量来表示,通过构造一组特征向量对形状目标进行描述。该方法着重考察轮廓序列上的一个点与该轮廓序列上的其他所有采样点的空间位置分布关系,具有包含信息量大、描述能力强和鲁棒性良好等特点。
Ling和Jacobs[2]基于形状上下文的思想,提出了利用轮廓点间的内部距离(Inner-Distance Shape Context,IDSC)来代替Belongie等[1]计算形状上下文中采用的欧氏距离,把轮廓序列上两个采样点经形状内部的最短路径长度定义为内部距离。该方法适用于产生柔性变化目标的识别,实验中取得了较好效果。
Biswas等[7]基于内部距离的形状上下文描述方法,提出了一个新的形状索引和检索框架。通过索引,高效计算待检索形状与数据库中形状的相似度,改进了传统基于动态规划的形状匹配算法的效率。总体上说,基于轮廓形状描述方法的优点主要表现在以下三个方面:
1) 可以把二维形状整体信息与局部信息有机地结合在一起,较准确地描述形状结构特征。
2) 可以与多种形状匹配算法组合,灵活地采用基于动态规划的形状匹配或基于词典的形状索引及匹配。
3) 可以通过图像分割及物体边界提取,方便地应用于自然图像的形状识别,具有较好的实用性。
这类方法主要存在以下两个方面的不足:
1) 仅考虑了目标形状的轮廓边界信息,而轮廓容易受到形状变形的影响,导致提取的特征量有时无法准确反映物体信息。
2) 对于复杂场景中的多个物体,基于轮廓的表示方法往往存在轮廓线参数化困难等问题,大多只能处理简单场景下的物体图像。
1.2 基于骨架的形状描述骨架是形状数据一种重要的几何特征描述符,由Blum在1967年首次提出[8],并将其应用到形状识别中。使用一种基于最大内切圆模型的骨架表示方法,具体指圆心在形状内部、且至少内切形状轮廓上两个点的圆形。形状的骨架点就是这些内切圆圆心的集合。
陈展展等[9]提出利用树形结构表示形状骨架,首先根据欧氏距离变换定义一个骨架点的中心点,然后构造以骨架中心点为根节点的骨架树,使用骨架中心点到骨架端点路径上经过的骨架点构造特征向量,并利用改进的最优子序列双射算法来进行骨架点之间的匹配,以用于二维形状的分类和识别。
Bai和Latecki[10]提出了一种基于骨架路径相似性的形状识别,属于整体对局部的形状描述方法。通过提取当前骨架端点到其他所有骨架端点间骨架路径上的特征量,对骨架端点进行描述,结合最优匹配算法对形状目标进行识别。
基于骨架形状描述方法的优点主要表现在以下三个方面:
1) 骨架表示了目标形状的整体结构,也能反映目标形状的边界。
2) 骨架简化了目标形状的描述难度,也能方便目标形状的匹配。
3) 骨架保持了形状的重要拓扑结构和几何特征,也能降低因噪声而引起的轮廓失真。
这类方法存在以下两个方面的不足:
1) 骨架不能有效抵抗大尺度噪声的影响,会产生较多的冗余骨架枝,易出现主次不分、结构混乱的现象,从而影响对物体真实形状和连接关系的判断。
2) 轮廓边缘的突起或遮挡会引起形状骨架的变化,有时会较大改变骨架的拓扑连接,造成目标形状骨架的信息多余,从而影响骨架的度量和匹配。
1.3 生物信息序列分析工具生物信息学中,基因序列在区分生物种类上有决定性的作用。经过过去40年生物信息学的研究,在基因序列分析方面已经有了大量的工具和解决方案。在基因序列分析工具中,主要分为双序列比较工具和多序列比较工具。
双序列比较工具分为全局比较和局部比较:全局比较尝试将两个完整的基因序列进行对准,而局部比较则是将两个基因序列的高相似性短区域进行对准。最有名的双序列比对算法是Needleman-Wunsch (全局比较)[11]和Smith-Waterman (局部比较)[12]。近些年,比较通用的基因序列比较方法是BLAST工具(基本局部对比较工具)[13],在比对的准确性和效率上都有着较好的表现。
多序列比较工具是对三个以上基因序列比较,首先对所有序列对准,然后进行多序列的比较。其最广泛使用的方法是渐进技术,该技术通过从最相似的一对基因序列进行对齐到最不相似的基因序列,近些年主要使用多序列比较工具包括Clustal[14]、MUSCLE等。
基于生物信息序列进行形状分析的优点主要表现在以下两个方面:
1) 把二维形状编码为生物信息序列,是一种新型的描述二维形状几何分布的方法,拓展了传统的二维形状表示技术。
2) 可以利用多种生物信息序列比对工具,进行形状编码的相似性匹配,从而改进了传统基于动态规划的匹配算法,提高了算法效率。
这类方法存在以下两个方面的不足:
1) 本质上,生物信息序列表示属于一种形状签名技术,相比于形状上下文,其表示准确性需要进一步提高。
2) 目前二维形状的生物信息编码技术,尚存在编码冗余问题,同时也未充分考虑如何使形状编码序列具有更多基因层面的信息。
2 基于轮廓及骨架序列的编码方法形状编码及匹配流程如图 1所示。本节具体给出形状轮廓和骨架的协同表示及编码方案。首先,假定二维形状数据F的轮廓已经得到,C(F)={c1, c2, …, cn}为轮廓上的n个等距采样点,并按顺时针排列,其中ci=(xi, yi)。其次,利用二维形状骨架提取方法[15]得到形状F的骨架S(F)。
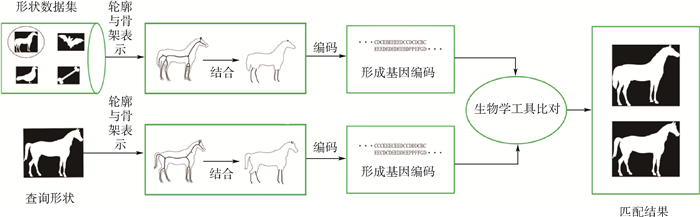
|
| 图 1 二维形状匹配流程图 Fig. 1 Flowchart of 2D shape matching |
在形状编码过程中,形状的方向对编码有着很大的影响。两个相同的形状,如果编码方向不同,得到的形状编码也会不同,从而影响编码之间的距离。为了解决这个问题,需要对形状进行归一化。首先,旋转形状使其长轴与x轴平行,消除不同旋转角度对形状分类结果的影响。
形状的长轴可以由形状数据的主成分分析得到,也可以通过形状中距离最长的两个点简单确定。实验中,发现两种选择的形状识别结果相近。简单起见,本文选择了后者。旋转对齐后,还可能会存在形状与目标匹配形状180°翻转的情况。本文采用的解决方案是:以形状长轴作为分隔线,把形状分为两个部分,使得面积较大的那一部分处于长轴的上方。
此时,形状与目标匹配形状可能还存在大小比例不一致的问题,本文编码方法本质上可以有效应对形状之间的比例尺度不一致的问题,不过,为了与其他形状匹配方法进行比较,实验中,还是通过标准的尺度归一公式(式(1))对形状进行尺度归一化。其中,D为目标长轴大小,D0为当前形状长轴大小。

|
(1) |
给定形状F,利用上述标准化方法得到其轮廓C(F),同时提取骨架S(F),如图 2所示。图中左边是获取了轮廓点与骨架点之后的形状,右边蓝色代表骨架点,绿色代表轮廓点。假定在每个轮廓点或骨架点周围至多有两个轮廓点或骨架点位于其8个连通方向上。这里的骨架点不包括骨架的关节点。
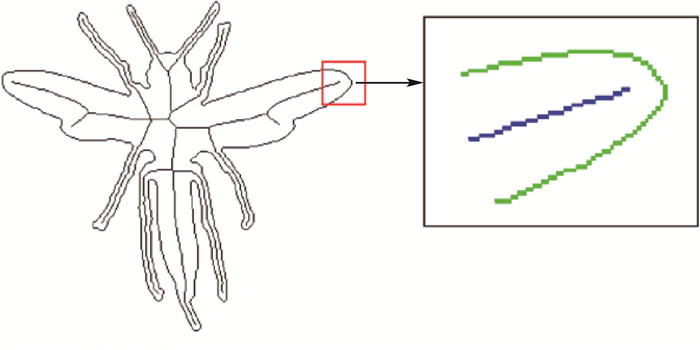
|
| 图 2 形状的轮廓与骨架 Fig. 2 Contour and skeleton of a shape |
Bicego和Lovato[5]提出可以把形状轮廓转化为生物信息序列即DNA分子序列,本质上是对每个轮廓点的8个方向上的轮廓点,利用字母A~H依次编码。不过发现,这种编码方案在形状细支处两边的编码会产生过多的冗余,使大量重复的编码被考虑进形状分类,如图 3所示。图 3中细支上,a到b点的相对位置关系可以近似另一边c到b点相对位置关系。也即轮廓点从a到b的编码和从b到c的编码虽然不一样,但是存在一个变换,在该变换下,两个编码是相似的。所以,a到b的编码和b到c的编码存在冗余。而发现,如果利用蓝色的骨架表示这个细支, 也即通过e到d的编码代替a到c的编码可以有效减少编码的冗余。同时,这种编码方案也可以继承骨架表示的优点。
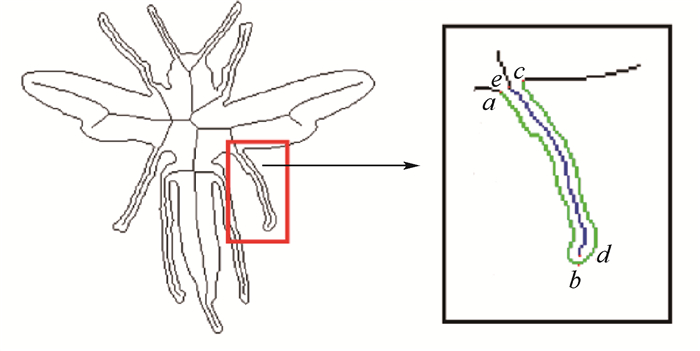
|
| 图 3 形状细支处的轮廓与骨架 Fig. 3 Contour and skeleton of branch of a shape |
另外,如果使用整个轮廓点进行编码,如编码图 3所示的昆虫。昆虫在细支处发生变形,细支处两边轮廓上一般会产生大体相似的变形,从而形状的一处变形会产生编码两处较大的改变。而在非细支处,轮廓的一侧变形,很难使这段轮廓在骨架上对应的另一端轮廓发生变形。所以,仅依靠轮廓信息进行编码无法表示形状的这种特性,从而降低了编码的准确性。显然,如果在细支处采用骨架点编码,在主体轮廓处采用轮廓点编码,可以兼顾编码的简洁性和准确性,有效解决上述问题。
同时,形状中往往会存在形变的情况,如动物形状和日常物体形状等。常见的形变包括轻度变形、遮挡和关节变换等。而骨架处理这些变形问题独具优势[10],由于本文引入了针对骨架的编码,所以也具备了对于形变物体一定的鲁棒性。
当然,在形状中准确定位其细支位置,本身也是一个比较有挑战性的问题。不过,由于本文编码方案可以单独在轮廓上编码,所以并不要求对于形状中分支的准确定位。实验中,认为骨架特征尺寸(内切圆半径)低于一个给定阈值的骨架点对应的轮廓点处于一个细支中,这些轮廓点不参与编码过程,由其骨架点的编码代替。图 3的形状经上述方法可以确定形状中所有的细支和主体,从而得到一个联合部分骨架和部分轮廓所形成的形状表示,如图 4黑色点所示。

|
| 图 4 轮廓与骨架的联合表示及默认的编码方向 Fig. 4 A combined representation of contour and skeleton and default encoding direction |
在得到形状的轮廓和骨架的联合表示之后,可以对形状进行编码。区别于Bicego和Lovato[5]单一编码形式,对形状轮廓和骨架分别采用不同的编码形式,即把二维形状转化为字母和数字联合表示的字符编码。
同时,Bicego和Lovato[5]形成编码序列的方法是对每个轮廓点的8个方向上的轮廓点依次使用字母A~H进行编码。如果轮廓存在一些局部变形,这种编码方式的结果是不太稳定的。为了改善这一方案,提出以下编码方法。
在轮廓上,每隔两个轮廓点取一个轮廓点,这样可以在一定程度上解决轮廓发生局部变形时稳定性不足的问题,也可以精简轮廓部分编码的长度,最大限度地保留轮廓上的大部分细节信息。在实验部分,给出了轮廓点的选取方式对准确率影响的比较,在实验结果看来,目前的方案是最优的。由于骨架部分往往仅占全部形状编码的一小部分,所以取所有骨架点进行编码,这样可以使骨架部分的编码长度最长,所占全部编码的比重也最大,使骨架编码的贡献度最高。为了能在字符编码中分别区分轮廓信息给予的贡献和骨架信息给予的贡献,分别用字母和数字来编码轮廓和骨架。
按照上述编码方式,在每个轮廓点周围会存在24个方向可能出现下一个轮廓点,对每个可能出现的下一个轮廓点的位置赋予从A~X的字母表示。起始点设为图像最左上方的轮廓点,即通过下一个轮廓点与此轮廓点的相对位置关系,来表示此轮廓点的编码。每个骨架点周围会有8个方向出现下一个骨架点,对每个可能出现的下一个骨架点的位置赋予从1~8的数字表示。起始点为离轮廓点最近的骨架点,结尾的骨架点用0表示,即通过下一个骨架点与此骨架点的相对位置关系,来表示此骨架点的编码。通过把骨架和轮廓进行编码,会得到图像的编码,如图 5所示。
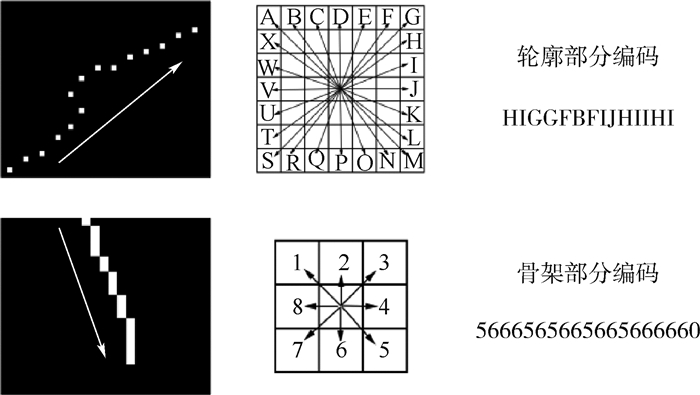
|
| 图 5 编码构造规则 Fig. 5 Encoding construction rule |
关于编码的方向如图 4红色箭头所示。编码开始的起点为轮廓部分最靠近左上方的点,沿逆时针方向对形状上所有点按照上述规则进行编码,最后回到起点。
2.4 使用生物学工具形状分类在通过2.3节转换后,可以得到不同形状唯一的字符编码表示。通过生物信息学的映射规则,把字符编码映射为基因序列,在分类时,就可以使用大量生物信息学领域的对齐工具。区别于Bicego和Lovato[5]使用的NW (全局比较)和SW(局部比较)对齐工具,使用更为高效和准确的MUSCLE对齐工具。因为NW和SW对齐工具只能比较2个基因序列,效率不高且对齐效果太过依赖生物基因信息的氨基酸占比。在此使用的是支持多序列同时比较、对生物多样性更为适合的MUSCLE对齐工具。之后实验会给出使用不同对齐工具对结果的影响。
通过对齐工具的比较,可以得到形状之间的相似度的评分,由于该评分是通过生物学的基因对齐工具获得,为了使该评分能表现形状上的基本信息,引入惩罚项,使评分更为合理。
在惩罚项的选择中,使用简单形状描述符。文献[16]已证实简单形状描述符能达到这一目的。这里选用最小边界矩形面积及离心率作为惩罚项。惩罚项如式(2)。最小边界矩形是通过在原形状上利用周长最小原则做外界矩形,计算此矩形的面积为最小边界矩形面积,离心率是最小外界矩形的宽度与长度之比。
加入惩罚项之后,通过计算式(2),最终得到2个形状之间的相似度评分:
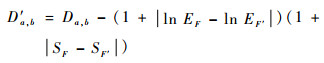
|
(2) |
式中:SF、SF′表示2个形状最小边界矩形面积;EF、EF′表示2个形状所形成的最小边界离心率。
3 实验及分析使用不同的数据集对本文方法进行性能评价。思路来源于文献[5]。首先把二维形状通过轮廓信息转换为生物信息编码,然后使用现有的编码分析工具:全局对齐工具(NW)和局部对齐工具(SW)计算形状之间的相似性。提出了一种结合轮廓和骨架的混合编码方案,并采用MUSCLE对齐工具,以进一步提高形状相似性度量的准确性。在形状的识别实验中,论文采用文献[5]中方法,利用K近邻分类器进行形状的分类和识别。
3.1 数据集和分类方法 3.1.1 MPEG-7数据集MPEG-7数据集是形状分析研究领域中最流行的数据集之一。有70个不同的类别组成,每个类别中有20个形状,因此数据集中总计有1400个形状。比较方法使用牛眼比较法(bulls-eye),即对每个查询形状,计算按相似度排序的前40个结果中,属于同一类的形状的百分比。同时,本文也采用留一法(leave one out)和留半法(half training)进行识别准确性的评价。留一法,即每次在同一类形状中抽取一个作为测试样本,其余作为训练集,检测样本集被准确分类的百分比。留半法,即每次在一类中选取一半作为测试集,其余全部作为训练集,检测样本集被成功分类的百分比。
3.1.2 ETH-80数据集ETH-80数据集包含8类形状,每个类别中有10个对象,每个对象具有41幅不同角度拍摄的彩色图片。由于本文主要是对形状进行分类,不考虑图片中的颜色问题。在识别准确率评价中,采用基于K最近邻的留一法。即对每个形状找与其相似度最高的K个形状(不包括本身),记该形状的类别为K个形状中类别出现频次最高的那一类。最后统计所有形状识别(分类)成功的个数,从而得到总体的识别准确率。
3.1.3 Swedish leaf数据集Swedish leaf数据集是全部由树叶图片组成的数据集,由15个类别组成,每个类别包含75个样本。Swedish leaf数据集由高分辨率的彩色图像组成,虽然提供了丰富的信息,但是与形状分类无关。所以,在原分辨率不变的情况下,本文对图像取二值处理,即只保留图像中叶子的形状信息,这样既可以保留叶子的细节,也可以去除图像中的多余信息。识别准确率的评价采用基于K最近邻的留一法进行。
3.2 MPEG-7数据集的识别结果MPEG-7数据集每一类形状的代表如图 6所示。

|
| 图 6 MPEG-7数据集 Fig. 6 MPEG-7 dataset |
MPEG-7数据集利用牛眼比较法,和现有的方法的对比结果如表 1所示。可以看出,在牛眼比较的准确率上,本文方法并不是最好的。离性能表现最好的方法尚有一定的差距,但是对于文献[5]得到的77.24%的准确率,本文提出的编码方案还是有较大提升。主要得益于形状中细支处使用骨架和主体上使用轮廓的协同编码方式。
性能表现最好的2种方法是IDSC+LP和IDSC+SSP,准确率均在90%以上。不过,这2种方法性能的提升均建立在对于形状相似度矩阵的距离学习上,属于形状识别领域中的后处理方法。该类方法必须提前得到一个质量较好的形状相似度矩阵,且并不能提供形状的表示。这与本文方法及形状上下文(SC和IDSC)等方法有本质上的区别。
进一步,在MPEG-7数据集上,本文分别使用了留一法和留半法的评价原则和现有的方法进行了比较,结果如表 2所示。
由表 2可见,本文编码方法在使用留一法和留半法时都取得了一个较高的准确率,和文献[5]的方法结果相当,不过本文的编码较为简短,冗余较小。由于一些形状存在比较明显的细支,另一些形状不存在。下面分别给出含有细支处形状和没有含有细支处形状对于形状编码的可视化查询,如图 7所示。
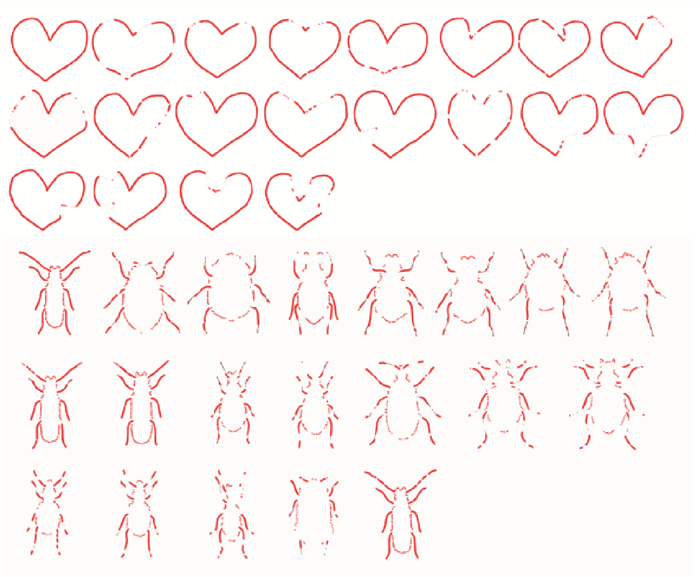
|
| 图 7 MPGE-7数据集可视化查询 Fig. 7 Visual query of MPEG-7 dataset |
图 7中,形状上点的颜色表示点与点匹配相似度的高低。通过可视化查询的片段中可以看出,在未含有细支的形状中,如上半部分所示,主体轮廓所形成的编码可以准确地描述形状。在含有细支的形状中,如下半部分所示,本文的方法由于在细支处使用骨架来进行编码,可以更有效地表示出细支处的形状信息,对该类形状的贡献度比较大。所以,在细支处使用骨架、在主体处使用轮廓的编码方案可以有效地表示形状信息。
3.3 ETH-80数据集的识别结果在ETH-80数据集,每组中随机选取一个作为测试形状,然后对所有形状进行编码,计算测试形状和训练形状之间的相似度,然后通过相似度排序选取前79个形状,统计它们的类别,得到测试形状的分类,并最终计算识别准确率。ETH-80数据集中8类物体的代表性形状如图 8所示。

|
| 图 8 ETH-80数据集 Fig. 8 ETH-80 dataset |
本文方法和现有方法的识别准确率如表 3所示。可以看出,本文方法取得了较高的准确率,相对于传统的描述子如基于内部距离的形状上下文(IDSC + DP),准确率提高了将近5%。
3.4 Swedish leaf数据集的识别结果
由于不同叶子类之间的高度相似性以及叶子类中往往存在较大变形,Swedish leaf数据集具有较大挑战性。识别准确率的计算方式和ETH-80数据集方式相同,在Swedish leaf数据集每类中选取一个作为测试形状,然后对所有形状进行编码,通过相似度降序排序选取前14个形状的相似度,判定该形状的类别为14个形状中比重最大的类别,最终得到识别准确率。Swedish leaf数据集15类叶子的代表性形状如图 9所示。

|
| 图 9 Swedish leaf数据集 Fig. 9 Swedish leaf dataset |
本文方法和现有方法的识别准确率如表 4所示。可以看出本文在Swedish leaf数据集上取得了较高的准确率。
当然,本文方法的识别准确率,与BCF和CNN方法的准确率相比还有一些差距。BCF方法利用多个轮廓片段及其池化表示,借助支持向量机(SVM)进行分类及识别。而CNN方法把形状表示和识别融合于卷积神经网络的训练和学习,其准确性依靠于样本的规模和有效标记。和这2种方法不同,本文的方法基于生物信息序列分析的理论。把形状数据表示为基因序列,可以直接用于形状数据的表示与匹配,也是一个颇具前景的方向。
3.5 深入分析在结合轮廓与骨架的编码方案中,除了本文提出的策略外,还有其他多种选择,不同的编码策略会产生不同的编码形式和编码长度。本节通过实验说明本文选择策略的有效性。
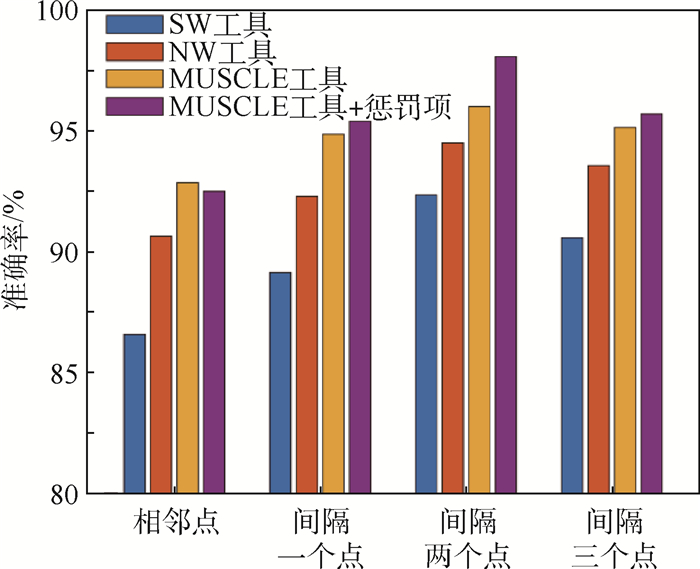
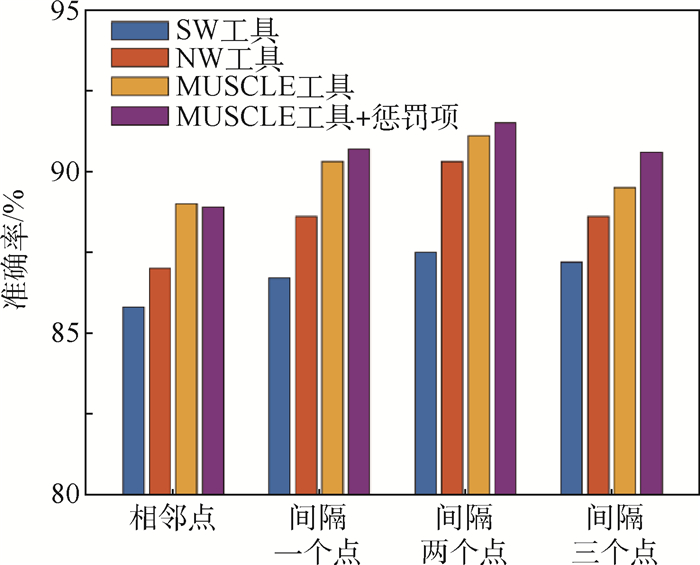
在主体轮廓上中,选取不同间隔的轮廓点:相邻点、间隔一个点、间隔两个点和间隔三个点,产生了不同的生物信息序列;并结合不同的序列对齐工具:局部对齐(SW)、全局对齐(NW)、MUSCLE对齐以及MUSCLE对齐加惩罚项,进行不同组合之间的比较。实验中,选取MPEG-7数据集和ETH-80数据集,使用上述的留一法进行比较,实验结果如图 10和图 11所示。
|
| 图 10 MPEG-7数据集上不同编码策略分类准确率的比较 Fig. 10 Comparison of classification accuracy rate on MPEG-7 dataset by different encoding strategies |
|
| 图 11 ETH-80数据集上不同编码策略分类准确率的比较 Fig. 11 Comparison of classification accuracy rate on ETH-80 dataset by different encoding strategies |
可以看出,使用本文的编码选择策略,即在主体轮廓上间隔两个点选取一个轮廓点进行编码,同时采用MUSCLE对齐工具和惩罚项,得到了最高的识别准确率。
4 结论本文提出了一种新型的二维形状识别方法。该方法首先得到形状主体轮廓和细支骨架的组合表示,其次通过一个轮廓及骨架的协同编码方案,把形状编码为生物信息序列,最后由成熟的生物序列对齐工具进行形状的分类及识别。在3个公开形状数据集的实验表明,本文方法相较于现有方法,有一定准确度的提升。
本文方法的优势在于可以有效降低原始编码方案的信息冗余。同时,骨架和轮廓的组合表示及编码可以增强形状编码的鲁棒性,提高后续编码匹配的效率和准确性。最后,把形状识别转化为生物信息序列匹配,可以采用广泛的生物序列对齐工具。
在对形状转化为基因编码的过程中,如何使形状编码序列具有更多基因层面的信息、几何信息与基因信息有更深层次的映射关系[29]仍是一个颇具前景的研究方向。
| [1] |
BELONGIE S, MALIK J, PUZICHA J. Shape matching and object recognition using shape contexts[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2002, 24(4): 509-522. DOI:10.1109/34.993558 |
| [2] |
LING H, JACOBS W D. Shape classification using the inner-distance[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2007, 29(2): 286-299. DOI:10.1109/TPAMI.2007.41 |
| [3] |
LING H, YANG X, LATECKI J L.Balancing deformability and discriminability for shape matching[C]//11th European Conference on Computer Vision.Berlin: Springer-Verlag, 2010, 6313(3): 411-424.
|
| [4] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E.ImageNet classification with deep convolutional neural networks[C]//NIPS'12 Proceedings of the 25th International Conference on Neural Information Processing Systems, 2012: 1097-1105.
|
| [5] |
BICEGO M, LOVATO P. A bioinformatics approach to 2D shape classification[J]. Computer Vision and Image Understanding, 2016, 145: 59-69. DOI:10.1016/j.cviu.2015.11.011 |
| [6] |
XU D, ALAMEDA-PINEDA X, SONG J, et al. Cross-paced representation learning with partial curricula for sketch-based image retrieval[J]. IEEE Transactions on Image Processing, 2018, 27(9): 4410-4421. DOI:10.1109/TIP.2018.2837381 |
| [7] |
BISWAS S, AGGARWAL G, CHELLAPPA R.Efficient indexing for articulation invariant shape matching and retrieval[C]//2007 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2007: 1-8.
|
| [8] |
BLUM H.A transformation for extracting new descriptors of shape[M]//WATHEN DUNN W.Models for the perception of speech and visual form model.Cavnbridge: MIT Press, 1967: 362-380.
|
| [9] |
陈展展, 汤进, 罗斌, 等. 基于最优子序列双射的骨架树匹配[J]. 计算机工程与应用, 2011, 41(7): 162-165. CHEN Z Z, TANG J, LUO B, et al. Skeleton tree matching based on optimal subsequence bijection[J]. Computer Engineering and Applications, 2011, 41(7): 162-165. DOI:10.3778/j.issn.1002-8331.2011.07.047 (in Chinese) |
| [10] |
BAI X, LATECKI J L. Path similarity skeleton graph matching[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2008, 30(7): 1282-1292. DOI:10.1109/TPAMI.2007.70769 |
| [11] |
NEEDLEMAN S, WUNSCH C. A general method applicable to the search for similarities in the amino acid sequence of two proteins[J]. Journal of Molecular Biology, 1970, 48(31): 443-453. |
| [12] |
SMITH T, WATERMAN S M M. Identification of common molecular subsequences[J]. Journal of Molecular Biology, 1981, 147(1): 195-197. DOI:10.1016/0022-2836(81)90087-5 |
| [13] |
ALTSCHUL S, GISH W, MILLER W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 2145(3): 403-410. |
| [14] |
LARKIN M, BLACKSHIELDS G, BROWN N, et al. Clustal w and clustal x version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [15] |
MARIE R, LABBANI-IGBIDA O, MOUADDIB M E. The delta medial axis:A fast and robust algorithm for filtered skeleton extraction[J]. Pattern Recognition, 2016, 56: 26-39. DOI:10.1016/j.patcog.2016.02.011 |
| [16] |
GOS'CIEWSKA K, FREJLICHOWSKI D.Silhouette-based action recognition using simple shape descriptors[C]//Lecture Notes in Computer Science.Berlin: Springer-Verlag, 2018, 11114: 413-424.
|
| [17] |
BAI X, YANG X, LATECKI J L, et al. Learning context-sensitive shape similarity by graph transduction[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2009, 32(5): 861-874. |
| [18] |
WANG J, LI Y, BAI X, et al. Learning context-sensitive similarity by shortest path propagation[J]. Pattern Recognition, 2011, 44(10): 2367-2374. |
| [19] |
LIN L, ZENG K, LIU X, et al.Layered graph matching by composite cluster sampling with collaborative and competitive interactions[C]//2009 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2009: 1351-1358.
|
| [20] |
MCNEILL G, VIJAYAKUMAR S.Hierarchical procrustes matching for shape retrieval[C]//2006 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2006: 885-894.
|
| [21] |
DALIRI R M, TORRE V. Robust symbolic representation for shape recognition and retrieval[J]. Pattern Recognition, 2008, 41(5): 1782-1798. DOI:10.1016/j.patcog.2007.10.020 |
| [22] |
BAI X, LIU W, TU Z.Integrating contour and skeleton for shape classification[C]//2009 IEEE 12th International Conference on Computer Vision Workshops.Piscataway, NJ: IEEE Press, 2009: 360-367.
|
| [23] |
SUN B K, SUPER J B.Classification of contour shapes using class segment sets[C]//2005 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2005: 727-733.
|
| [24] |
WANG X, FENG B, BAI X, et al. Bag of contour fragments for robust shape classification[J]. Pattern Recognition, 2014, 47(5): 2116-2125. |
| [25] |
LEIBE B, SCHIELE B.Analyzing appearance and contour based methods for object categorization[C]//2003 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2003: 409-415.
|
| [26] |
HU X R, JIA W, ZHAO Y, et al. Perceptually motivated morphological strategies for shape retrieval[J]. Pattern Recognition, 2012, 45(9): 3222-3230. DOI:10.1016/j.patcog.2012.02.020 |
| [27] |
DALIRI R M, TORRE V. Shape recognition based on Kernel-edit distance[J]. Computer Vision and Image Understanding, 2010, 114(10): 1097-1103. DOI:10.1016/j.cviu.2010.07.002 |
| [28] |
ATABAY A H. A convolutional neural network with a new architecture applied on leaf classification[J]. ⅡOAB Journal, 2016, 7(5): 326-331. |
| [29] |
GAO L, SONG J, NIE F, et al.Optimal graph learning with partial tags and multiple features for image and video annotation[C]//2015 IEEE Conference on Computer Vision and Pattern Recognition.Piscataway, NJ: IEEE Press, 2015: 4371-4379.
|



